莱茵衣藻CrPGP3和CrFAT1基因克隆及其序列分析与原核表达
2016-10-08费小雯李亚军邓晓东余丽芸黑龙江八一农垦大学生命科学技术学院黑龙江大庆69中国热带农业科学院热带生物技术研究所海南海口570海南医学院理学院海南海口570
高 寒,夏 斌,费小雯,李亚军,邓晓东,余丽芸(.黑龙江八一农垦大学生命科学技术学院,黑龙江 大庆 69;.中国热带农业科学院热带生物技术研究所,海南 海口 570;.海南医学院理学院,海南 海口 570)
莱茵衣藻CrPGP3和CrFAT1基因克隆及其序列分析与原核表达
高 寒1,2,夏 斌2,费小雯3,李亚军2,邓晓东2,余丽芸1
(1.黑龙江八一农垦大学生命科学技术学院,黑龙江 大庆 163319;2.中国热带农业科学院热带生物技术研究所,海南 海口 571101;3.海南医学院理学院,海南 海口 571101)
克隆莱茵衣藻(Chlamydomonas reinhardtii)的CrPGP3和CrFAT1基因,并对其序列进行生物信息学分析和原核表达。选取莱茵衣藻CC124提取总RNA后,反转录cDNA,PCR获得CrPGP3和CrFAT1的全长编码区,构建原核表达载体CrPGP3-PGEX-6p-1和CrFAT1-PGEX-6p-1,转入大肠杆菌DL21 (DE3)中,在37℃、1.0mmol/L IPTG诱导整合蛋白表达。结果表明, CrPGP3和CrFAT1的全长编码区分别为786 bp和1 188 bp,分别编码261和395个氨基酸;CrPGP3是与脂类合成相关磷脂酰甘油磷酸合成酶(Phosphatidylglycerophosphate synthase)属于CDP-alcohol phosphatidyltransferase家族,CrFAT1是酰基载体蛋白硫脂酶(Acyl-ACP thioesterase)属于hot dog家族;SDS-PAGE结果表明所表达蛋白与预期蛋白大小一致。本文成功克隆了CrPGP3和CrFAT1基因的全长编码区,并在原核生物中表达得到了预期蛋白。
CrPGP3;CrFAT1;莱茵衣藻;基因克隆;原核表达
高寒,夏斌,费小雯,等. 莱茵衣藻CrPGP3和CrFAT1基因克隆及其序列分析与原核表达[J].广东农业科学,2016,43 (8):163-168.
莱茵衣藻是一类利用光合作用的自养生物,具有陆地、海洋分布广泛,营养丰富、生长周期短、生物产量高、环境适应能力强等优点,是开发新生物质能源的理想材料[1-2]。光合作用对于藻的生长至关重要,也决定着藻类代谢产物的积累,磷脂酰甘油(Phosphatidylglycerol,PG)是植物在叶绿体中合成的磷脂,其在植物的光合作用中扮演着重要角色,而酰甘油磷酸合成酶(Phosphatidyl glycerophos phate synthase,PGPS)参与了PG合成的最后一步,对植物的生长和光合作用的活性有一定的影响[3-5]。通过将相关PGPS基因的敲除发现,PGPS基因的缺失对光合作用产生了严重的影响[6-8]。也有研究表明PGP基因对于拟南芥叶绿体分化也起到重要作用[7]。酰基载体蛋白硫脂酶(Acyl-ACP thioesterase)是游离脂肪酸合成的关键酶,它的参与直接决定了脂肪酸的产量和成分,在脂肪酸合成中具有重要作用[9-10]。不同酰基载体蛋白硫脂酶的数量和成分会对脂肪酸积累造成影响[11]。如今生物柴油等生物燃料已经是全世界的热门话题,而脂肪酸的积累则对于生物质油的产量至关重要[12-13]。本研究通过对两基因的生物信息分析及其所编码蛋白的结构、性质与功能进行预测,为今后其功能研究提供了理论基础。
1 材料与方法
1.1试验材料
供试材料为莱茵衣藻CC124购自中国科学院水生生物研究所。大肠杆菌(Escherichia coli)菌株DH5α和BL21两种菌株购置于上海生工生物工程有限公司。克隆载体PMD18-T购自Takara公司,原核表达载体、过量表达载体和亚细胞定位载体为作者实验室保存。
试剂:Trizol试剂、PCR产物纯化试剂盒、琼脂粮凝胶回收试剂盒和质粒抽提试剂盒均购自上海生工生物工程有限公司;反转录酶PrimeScript® Reverse Transcriptase、RNase 抑制剂、T4DNA连接酶、DL2000 Marker、DL15000 Marker、限制性内切酶EcoR Ⅰ和Sal Ⅰ、SYBR ® Premix Ex Taq™(TakaRa)购自宝生物工程(大连)有限公司;蛋白分子量标准proteinRule ®IV购自北京全式金生物技术有限公司。氯仿、异戊醇等为分析纯试剂。
仪器:恒温恒湿培养箱(赛福PRX-250B,宁波);冷冻调整离心机(eppendorf 5804R,德国);PCR仪(Biometra Tgradient T1 德国);干式恒温器(奥盛 MK-20,杭州);电泳仪(PowerPac™ HC,新加坡)
培养基:培养莱茵衣藻所用的培养基是TAP培养基。大肠杆菌用LB培养基培养。LB培养基成分为胰蛋白胨10 g/L、酵母提取物5 g/L和氯化钠10 g/L。固体培养基则添加1.5%的琼脂。
1.2试验方法
1.2.1CrPGP3和CrFAT1基因的PCR扩增 总RNA的提取采用Trizol法[14]抽提,并用超微量分光光度计、1%琼脂糖凝胶电泳参考反转录酶PrimeScript® Reverse Transcriptaser的使用说明以Oligo(dT)18为引物,以1 μg总RNA为模板合成cDNA。根据Phytozome 11 (https://phytozome.jgi.doe.gov/pz/portal.html)Chlamydomonas reinhardtii v5.5数据库中的序列信息(CrPGP3:Cre02.g095106.t1.1 CrFAT1:Cre06.g256750.t1.2),分别设计两对RT-PCR扩增引物(表1),并以cDNA为模板进行PCR扩增。PCR反应体系如下:灭菌 ddH2O 水稀释10×的 cDNA 1 μL,10 mmol/L dNTPs 0.4μL,10× LA Taq buffer 2.5μL,LA Taq 酶0.3 μL,5 mol/L 甜菜碱2.5 μL,DMSO 1μL,灭菌 ddH2O 15.3μL。PCR 反应条件为:95℃预变性 5 min,然后按95℃变性45s,58℃退火1 min,72℃延伸2 min,3个反应循环30次;最后72℃延伸 10 min;4℃结束。用1%琼脂糖凝胶电泳检测PCR扩增产物后,用凝胶回收试剂盒回收目的条带,连接到pMD18-T载体上,然后转化大肠杆菌DH5α,筛选阳性克隆,菌液PCR鉴定,送至上海生工生物工程有限公司测序。

表1 CrPGP3 和 CrFAT1基因PCR扩增引物信息
1.2.2序列分析方法 在phytozome网站找到基因的DNA序列和cDNA序列,然后分别输入NCBI的Spidey分析工具(http://www.ncbi. nlm.nih.gov/IEB/Research/Ostell/Spidey/),分析外显子和内含子数目,并用DNAMAN软件绘制基因结构图;利用MEGA5构建系统进化树;利用ExPASy-ProtParam tool (http://web.expasy. org/protparam/)进行蛋白质基本理化性质预测;利用NetPhos 2.0 Server(http://www.cbs.dtu. dk/services/NetPhos/)进行蛋白质磷酸化位点预测。
1.2.3CrPGP3和CrFAT1原核表达载体的构建 设计两对引物:GEX CrPGP3正反向引物、GEX CrFAT1正反向引物,序列信息见表1,分别在CrPGP3和CrFAT1编码区序列的两端引入EcoRⅠ和SalⅠ酶切位点,用LA Taq酶进行PCR扩增,纯化后的PCR产物和载体质粒pGEX-6p-1分别利用EcoRⅠ和SalⅠ进行双酶切。目的条带用DNA凝胶回收试剂盒回收,并用T4 DNA连接酶在16℃连接8h,随后将连接产物转化大肠杆菌DH5α。阳性克隆经菌液PCR和双酶切鉴定,送到上海生工生物工程有限公司测序。
1.2.4CrPGP3和CrFAT1融合蛋白的诱导表达 吸取50 μL含重组质粒的DH5α菌液至50 mL含100 μg/mL Amp的LB液体培养基中,在37℃,200 r/min下振荡过夜培养,吸取过夜培养物1 mL加入到10 mL含100 μg/mL Amp的LB液体培养基中,在37℃,200 r/min下振荡培养2 h,加入IPTG至终浓度为1 mmol/L诱导表达,在37℃,200 r/min下诱导培养,于培养2、4、6 h分别收集处理组和对照组诱导物1 mL,12 000 r/min,4℃离心1 min,弃上清,用100 μL的1×SDS凝胶加样缓冲液重悬菌体沉淀,100℃加热3 min,12 000 r/min,4℃离心1 min,每种悬液取10 μL进行15%SDS聚丙稀酰胺凝胶电泳,电泳结束后用考马斯亮蓝对凝胶进行染色,再对凝胶条带进行扫描。
2 结果与分析
2.1莱茵衣藻CrPGP3和CrFAT1基因的生物信息学分析
2.1.1莱茵衣藻CrPGP3和CrFAT1基因全长cDNA分析 PCR获得的基因中间片段经过测序对比发现与Chlamydomonas reinhardtii v5.5数据库中的序列信息完全匹配,将中间片段与3′端和5′端进行拼接,获得CrPGP3和CrFAT1基因的全长。CrPGP3的cDNA序列全长为1 853 bp,开放阅读框(ORF)长786 bp,编码261个氨基酸,其基因区的DNA序列长4 423 bp,包含7个内含子,8个外显子(图1A黑色方形)。CrFAT1的cDNA序列全长为2 362 bp,ORF长1 188 bp,编码395个氨基酸,其基因区的DNA序列长4 692 bp,包含6个内含子,7个外显子(图1B黑色方形)。
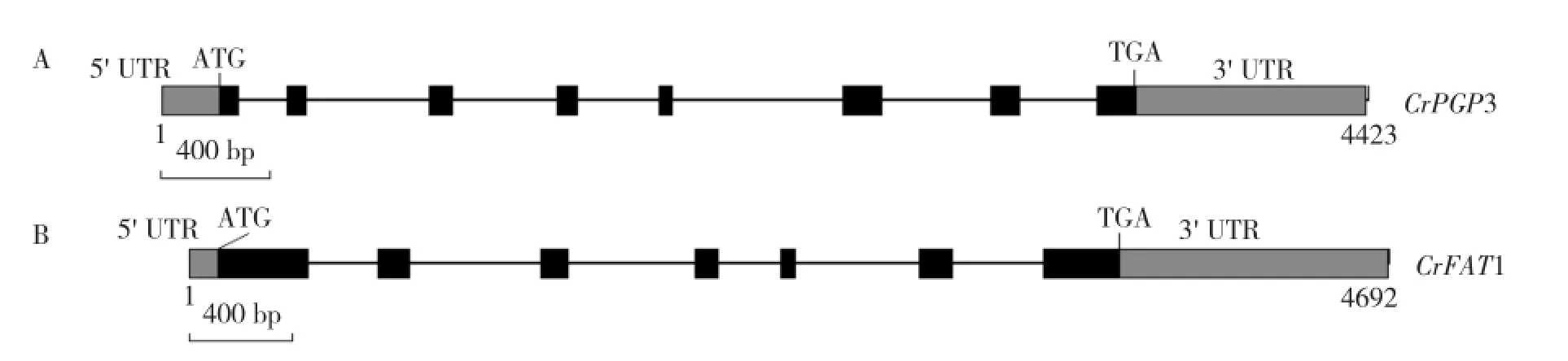
图1 CrPGP3和CrFAT1基因的结构
将两基因的氨基酸序列在GenBank数据库中进行BLAST对比,分析其氨基酸在其他物种中的同源性,结果发现,CrPGP3氨基酸序列为CDP-alcohol phosphatidyltransferase家族,CrFAT1氨基酸序列为hot_dog 家族,两者与团藻(Volvox carteri f. nagariensis)的相关氨基酸序列相似度均为98%以上,与小球藻(Chlorella variabilis)、绿藻(Coccomyxa subellipsoidea C-169)都具有很高的同源性。利用MEGA5对两者氨基酸进行NJ系统进化树分析。从图2可以看出,不同科属类植物之间相关基因有明显差异。CrPGP3和CrFAT1都与团藻的进化关系最为亲近,然后与蓝藻(Monoraphidium neglectum)、绿藻、小球藻等在同一分枝。高等植物如烟草(Nicotiana tomentosiformis)、番茄(Solanum lycopersicum)、油菜(Brassica napus)、向日葵(Helianthus annuus)等都在另一个分枝,表明莱茵衣藻CrPGP3和CrFAT1与高等植物同源基因之间的亲缘关系较远。

图2 CrPGP3和CrFAT1氨基酸的系统进化树
2.1.2蛋白基本理化性质,磷酸化位点的预测与分析 用ExPASy-ProtParam在线分析软件预测CrPGP3和CrFAT1基因所编码的蛋白质基本理化性质,结果表明CrPGP3所编码的蛋白包含261个氨基酸,分子量为28.7 kDa,理论等电点pI为9.06,预测分子式为C1351H2107N331O343S8,不稳定系数为49.44,总平均亲水系数(GRAVY)为0.561,推测CrPGP3蛋白为不稳定的亲水蛋白。CrFAT1蛋白包含395个氨基酸,分子量约为44.4kDa,理论等电点pI为8.42,预测分子式为C1894H3014N556O575S19,不稳定系数为58.71,总平均亲水系数(GRAVY)为-0.351,推测CrFAT1所编码的蛋白为不稳定疏水蛋白。
通过NetPhos 2.0 Server在线分析两基因所编码蛋白的磷酸化位点结果显示:CrPGP3基因所编码的蛋白可发生磷酸化位点有7个:包括7、26、29、95位点的丝氨酸(Ser)和11、85位点的苏氨酸(Thr),以及99位点的酪氨酸(Tyr)。CrFAT1基因所编码的蛋白可能发生磷酸化的位点有16个,包括10个丝氨酸(Ser)位点、6个苏氨酸(Thr)位点,各自分别在肽链第16、19、20、23、36、49、141、213、290位,第6、17、109、240、372、373位。
2.2原核表达载体构建和重组质粒鉴定
利用限制性内切酶EcoRⅠ和SalⅠ分别对重组质粒CrPGP3-Pgex-6p-1和CrFAT1-pGEX-6p-1进行双酶切,结果见图3,重组质粒CrPGP3-PGEX-6p-1在5 000 bp左右和750 bp左右的位置上各出现一条亮带,同样重组质粒CrFAT1-pGEX-6p-1也分别在5 000 bp左右和1 000 bp以上的位置上出现一条亮带,重组质粒中目的基因的测序结果与目的片段大小一致,表明莱茵衣藻CrPGP3和CrFAT1原核表达载体构建成功。

图3 重组质粒酶切验证
2.3CrPGP3 和CrFAT1蛋白原核表达检测
将构建好的原核表达载体转入BL21宿主菌中,筛选阳性单克隆,在37℃条件下利用IPTG诱导时间分别为2、4、6 h表达外源蛋白,通过SDS-PAGE检测蛋白表达情况,其电泳结果如图4。插有外源片段的重组质粒CrPGP3-PGEX-6p-1经过IPTG诱导后,在预期的蛋白分子量50 kD左右有1条蛋白条带(图4A),空载体转化子在26 kD左右有1条蛋白条带;重组质粒CrFAT1-pGEX-6p-1在70 kD左右出现1条蛋白条带(图4B),空载体转化子同样在26 kD左右出现1条蛋白条带,经过计算重组质粒CrPGP3-pGEX-6p-1和CrFAT1-pGEX-6p-1所表达的融合蛋白大小约与预期蛋白大小相符,表明重组质粒在大肠杆菌BL21中成功诱导表达了其相应所编码的蛋白。不同的诱导时间会影响蛋白的表达量,两个重组质粒约在IPTG终浓度为1 mmol/L、诱导时间为6 h时,蛋白表达量最多。
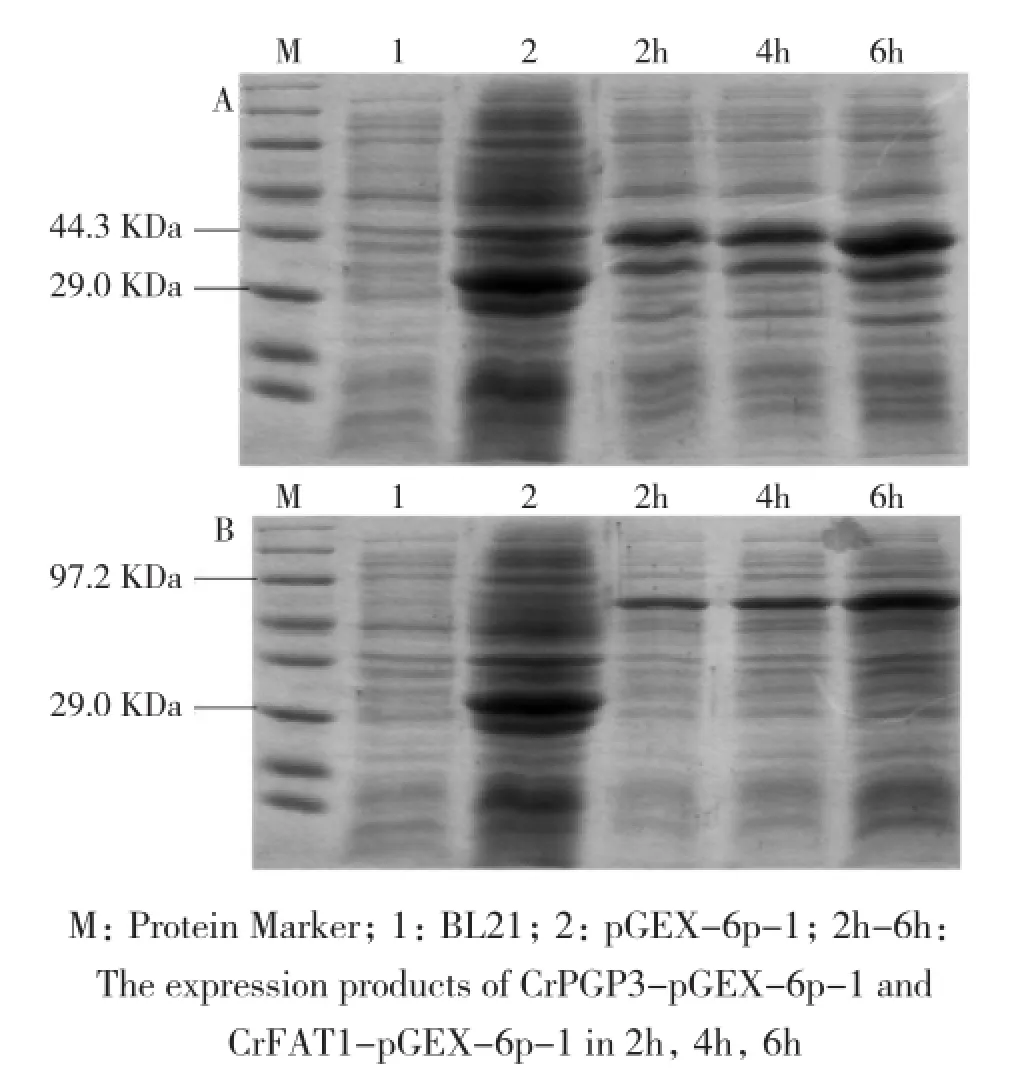
图4 CrPGP3(A) 和CrFAT1(B)在大肠杆菌中表达 SDS-PAGE 分析
3 结论与讨论
莱茵衣藻是单细胞真核生物,作为真核模式生物,其基因组测序已经基本完成,是一种重要的油料作物,莱茵衣藻体形小、繁殖快,光合效率高,培养条件简单,被称为“光合酵母”[15],是生产生物质能源的热门材料。通过对莱茵衣藻与光合作用相关和脂肪酸代谢相关基因的研究,对于生物质能源的产物具有重要研究意义。
通过对莱茵衣藻CrPGP3、CrFAT1氨基酸序列同源性和系统进化树的分析显示,莱茵衣藻与团藻具有很高的同源性。如与团藻序列的相似度达到了98%,在系统进化树分析中,团藻与莱茵衣藻以100匹配度同在一个分支中,与小球藻、绿藻在一个大分支中,但是与高等生物如烟草,番茄亲缘关系较远。
本试验成功将莱茵衣藻CrPGP3和CrFAT1全长编码区克隆到pGEX-6p-1表达载体上并在大肠杆菌BL21中成功表达,表达出蛋白大小分别约为50、70 kDa的融合蛋白,pGEX-6p-1作为融合蛋白的一部分,可表达大小26 kDa蛋白。通过计算,目的蛋白大小与预测蛋白大小相符。对于下一步纯化蛋白以及进行生物功能分析奠定基础。CrPGP3编码酰甘油磷酸合成酶,是参与光合作用的重要酶,对于莱茵衣藻的生长,代谢产物都具有深远影响。CrFAT1是编码酰基载体蛋白硫脂酶的基因,通过对此类酶的研究,阐明与脂肪酸代谢的关系,进而对藻类产油新类型能源奠定了基础,为解决能源危机做出了贡献。
[1] Milne T A,Evans R J,Nagle N. Catalytic conversion of microalgae and vegetable oils to premium gasoline,with shape-selective zeolites [J]. Biomass,1990,21(3):219-232.
[2] Minowa T,Yokoyama S,Kishimoto M,et al. Oil production from algal cells of Dunaliella tertiolecta by direct thermochemical liquefaction [J]. Fuel,1995,74(12):1735-1738.
[3] Hagio M,Gombos Z,Varkonyi Z,et al. Direct evidence for requirement of phosphatidylglycerol in photosystem Ⅱ of photosynthesis[J]. Plant Physiology,2000,124(2):795-804.
[4] Sato N,Hagio M,Wada H,et al. Requirement of phosphatidylglycerol for photosynthetic function in thylakoid membranes[J]. Proceedings of the National Academy of Sciences,2000,97(19):10655-10660.
[5] Gombos Z,Varkonyi Z,Hagio M,et al. Phosphatidylglycerol requirement for the function of electron acceptor plastoquinone Q (B)in the photosystem Ⅱ reaction center[J]. Biochemistry,2002,41(11):3796-3802.
[6] Xu C,Hartel H,Wada H,et al. The pgp1 mutant locus of Arabidopsis encodes a phosphatidylglyce rolphosphate synthase with impaired activity[J]. Plant Physiology,2002,129(2):594-604.
[7] Babiychuk E,Müller F,Eubel H,et al. Arabidopsis phosphatidylglycerophosphate synthase 1 is essential for chloroplast differentiation,but is dispensable for mitochondrial function[J]. Plant Journal for Cell and Molecular Biology,2003,33 (5):899-909.
[8] Kobayashi K,Fujii S,Sato M,et al. Specific role of phosphatidylglycerol and functional overlaps with other thylakoid lipids in Arabidopsis chloroplast biogenesis[J]. Plant Cell Reports,2015,34(4):631-642.
[9] Cao Y,Yang J,Xian M,et al. Increasing unsaturated fatty acid contents in Escherichia coli by co-expression of three different genes[J]. Applied Microbiology and Biotechnology,2010,87(1):271-280.
[10] Steen E J,Kang Y S,Bokinsky G,et al. Microbial production of fatty-acid-derived fuels and chemicals from plant biomass[J]. Nature,2010,463(463):559-563.
[11] Zhang X,Li A,Agrawal A,K Y San. Efficient free acid production in Escherichia coli using plant acyl-ACP thioesterases[J]. Metabolic Engineering,2011,13(6):713-722.
[12] Dong T,Wang J,Miao C,Zheng Y,et al. Twostep in situ biodiesel production from microalgae with high free fatty acid content[J]. Bioresource Technology,2013,136C(3):8-15.
[13] Lennen R M,Kruziki M A,Kumar K,et al. Membrane stress induced by overproduction of free fatty acids in Escherichia coli[J]. Applied and Environmental Microbiology,2011,77 (22):8114-8128.
[14] 李亚军,费小雯,邓晓东. 2种微藻总RNA提取方法的比较[J]. 热带农业科学,2011,31 (5):24-27.
[15] Rochaix J D. Chlamydomonas reinhardtii as the photosynthetic yeas[J]. Annual Review of Genetics,1995,29(4):209-230.
(责任编辑 白雪娜)
Cloning,sequence analysis,and prokaryotic expression of Chlamydomonas reinhardtii gene CrPGP3 and CrFAT1
GAO Han1,2,XIA Bin2,FEI Xiao-wen3,LI Ya-jun2,DENG Xiao-dong2,YU Li-yun1
(1.College of Life Science,Heilongjiang Bayi Agricultural University,Daqing 163319,China;2.Institute of Tropical Bioscience and Biotechnology,Chinese Academy of Tropical
Agicultural Sciences,Haikou 571101,China;3.Department of Basic Medicine,Hainan Medical College,Haikou 571101,China)
In order to express the CrPGP3 and CrFAT1 genes of Chlamydomonas reinhardtii in prokaryotic cell,we cloned and analyzed these two sequences. After the total RNA in C. reinhardtii CC124 was extracted,the revers transcription were performed. The full length cDNA obtained by PCR was inserted into the prokaryotic expression vector PGEX-6p-1 and transformed into Escherichia coli BL21(DE3). These positive clones were called CrPGP3-PGEX-6p-1 and CrFAT1-PGEX-6p-1. The target proteins were expressed after adding 1.0 mmol/L IPTG. The results showed that the CrPGP3 and CrFAT1 gene had 786 bp and 1 188 bp coding 261 and 395 amino acids,respectively. Sequence analysis indicated that CrPGP3 was a member of super family “CDP-alcohol phosphatidyltransferase”,and the CrFAT1 belonged to a member of “hot dog” super family. The SDS-PAGEdisplayed that the expressed proteins were consistent with the anticipated size. The recombinant plasmids expressed the target proteins in E. coli BL21.
CrPGP3;CrFAT1;Chlamydomonas reinhardtii;gene clone;prokaryotic expression
Q945
A
1004-874X(2016)08-0163-06
2016-02-28
国家自然科学基金(31360051,31160050);海南省重大科技专项(ZDZX2013023);海南省工程技术研究中心专项(GCZX2012004,GCZX2013004);中央级公益性科研院所基本科研业务费(ITBB)
高寒(1989-),男,在读硕士生,E-mail:hanksg@foxmail.com
余丽芸(1966-),女,博士,教授,E-mail:yuliyun1227@126.com
