基于ITS序列位点特异性PCR的酸枣仁及其混伪品的鉴别△
2016-09-25李桂林宋雅迪吕振晖刘春晖李佳张夏楠
李桂林,宋雅迪,吕振晖,刘春晖,李佳,张夏楠
(首都医科大学 中医药学院,北京 100069)
基于ITS序列位点特异性PCR的酸枣仁及其混伪品的鉴别△
李桂林,宋雅迪,吕振晖,刘春晖,李佳,张夏楠*
(首都医科大学 中医药学院,北京 100069)
目的:基于ITS序列建立酸枣仁及其市场混伪品的分子鉴定方法。方法:收集酸枣仁及其常见混伪品理枣仁、枳椇子、紫荆子、兵豆,提取总DNA,基于ITS序列设计引物并进行PCR扩增,对扩增产物进行测序,用BioEdit分析软件进行序列比对,针对变异位点用Primer Premier 5.0设计特异性鉴别引物,进行PCR反应并考察反应条件。结果:ITS序列可以用于酸枣仁及其易混淆品种的鉴别研究,在位点特异性PCR反应条件考察中,DNA模板用量范围应为0.7~2.1 ng·μL-1,退火温度在59 ℃时特异性最佳,实验建立的方法对不同Taq酶和PCR仪具有普遍适应性。结论:研究设计的位点特异性引物在一定条件下的PCR反应中,酸枣仁能扩增出66 bp的条带,而其他混伪品不能扩增出条带,从而实现了酸枣仁与其混伪品的快速、准确鉴别。
酸枣仁;ITS;特异性PCR;分子鉴定
酸枣仁为鼠李科植物酸枣ZiziphusjujubaMill.var.spinosa(Bunge)Hu ex H.F.Chou 的干燥成熟种子。产自河北、陕西、辽宁、山东、河南、安徽等省。酸枣仁性平,味甘、酸,具有养心补肝、宁心安神、敛汗、生津的功能,用于虚烦不眠、惊悸多梦、体虚多汗、津伤口渴[1]。现代药理学研究表明,本品具有镇静催眠、镇痛抗惊厥、降压、降血脂、增强免疫功能、抗缺氧、抗心律失常、抗衰老、抗辐射及抗血小板聚集等作用[2]。由于酸枣仁野生资源减少,市场货源供不应求,药材市场上出现以同属植物滇刺枣ZizyphusmauritianaLam.的种子理枣仁混作酸枣仁出售的情况,此外还有枳椇子Hoveniadulcis(又名拐枣仁)、兵豆Lensculinaris、紫荆子Cercischinensis等混伪品出现。目前对于酸枣仁的鉴别研究多采用药材性状[3]、显微特征[4]、薄层色谱[5]、电泳[6-7]、紫外光谱[8-9]、扫描电镜[10]等传统鉴别方法,但由于该5个品种外观性状相近,显微组织特征不明显,化学成分复杂,以上这些方法专属性不强,且容易受到外加因素的干扰[11]。本实验即以ITS序列作为分子标记,应用位点特异性PCR技术筛选特异性鉴别引物,建立一种快速、稳定的分子鉴别方法以区分酸枣仁及其混伪品药材。
1 仪器与试药
1.1 仪器
DYY-6D电泳系统(北京市六一仪器厂),PCR仪(Life Technologies 公司,型号 Veriti;德国Eppendorf AG 22331 Hamburg;德国Eppendorf Mastercycler pro),1-14台式离心机(德国SiGMR公司),Vilber Lourmat凝胶成像仪(法国),Plant Genomic DNA kit 50 Preps植物基因组DNA提取试剂盒(TianGen)。
1.2 试剂
2x Easy Taq®PCR SuperMix,2x Phusion High-Fidelity PCR MM W/HF Buffer,Trans FastTaq DNA Polymerase(5U·μL-1),TaKaRa dNTP Mixture 2.5 mm each,Trans 10x FastTaq Buffer(Mg2+),Trans2K®DNA Marker(北京全式金生物技术有限公司),琼脂糖Biowest®Regular Agarose G-10,Tiangen 10 000x GeneGreen Nucleic Acid Dye,10x Mops 电泳缓冲液[生工生物工程(上海)股份有限公司]。
1.3 药材
收集安国、亳州药材市场的20份材料,包括11份酸枣仁、5份理枣仁、2份枳椇子、1份紫荆子、1份兵豆,鉴定人为首都医科大学中医药学院李佳副教授。
2 方法
2.1 提取样品总DNA
取适量样品研磨成细粉,采用植物基因组DNA提取试剂盒提取样品总DNA,于-20 ℃保存备用。
2.2 ITS片段扩增与测序
各样品采用ITS通用引物进行扩增[12],ITS1:5′-AGAAGTCGTAACAAGGTTTCCGTAGG-3′;ITS 4:5′-TCCTCCGCTTATTGATATGC-3′。PCR反应体系总体积为50 μL,包括25 μL 2x Phusion High-Fidelity PCR MM W/HF Buffer,正反引物各2.5 μL,DNA 5 μL,无菌水15 μL。反应条件为98 ℃ 30 s,35个循环(98 ℃ 7 s,55 ℃ 20 s,72 ℃ 15 s),72 ℃ 7 min。产物由北京睿博兴科生物技术有限公司测序,各样品均采用正反双向测序,以保证结果的准确性。
2.3 构建系统发育树
根据测序结果利用CLUSTAL X软件进行序列排序,使用MEGA6进行遗传分析构建系统发育树。
2.4 设计位点特异性鉴别引物
根据2.2测序结果将酸枣仁、理枣仁、兵豆、枳椇子、紫荆子的ITS序列用CONTIG软件同源对齐并辅以人工校对,多重序列比对进行差异变异位点分析,根据变异位点采用Primer Premier 5.0软件设计特异性鉴别引物。引物序列由北京睿博兴科生物技术有限公司合成。引物序列为ZmITS3F:5′-CAACCAGCGAACCCGTGAA-3′;ZmITS3R:5′-CCAAGGAAGGCTCCGTGACA3′。
2.5 PCR扩增条件的确定
用设计的特异性鉴别引物进行PCR反应,反应体系为25 μL,包括12.5 μL 2x Easy Taq®PCR SuperMix缓冲液,正反引物各0.5 μL,DNA 35 ng·μL-1共1.5 μL,无菌水10 μL。PCR反应条件为94 ℃ 5 min,35个循环(94 ℃ 30 s,55 ℃ 15 s,72 ℃ 30 s),72 ℃ 7 min。反应结束后取反应产物5 μL,用1%琼脂糖凝胶电泳检测,SYNGENE凝胶成像系统观察、成像。为使PCR鉴别反应准确高效,考察了DNA模板量(0.4~2.6 ng·μL-1)、退火温度(53、55、56、57、59、61、63 ℃)、Taq酶种类(2x EasyTaq®PCR SuperMix、Trans FastTaq DNA Polymerase)以及不同PCR仪(Life Technologies 公司,型号 Veriti;德国Eppendorf AG 22331 Hamburg;德国Eppendorf Mastercycler pro)对PCR鉴别反应的影响。
3 结果与分析
3.1 酸枣仁及其混伪品的ITS分析及系统发育树的构建
采用Mega 6分析软件进行酸枣仁及其混伪品系统发育树的构建,采集NJ法构建系统发育树,见图1,采用Kimura2-parameter 距离,系统树各分支的置信度用bootstrap method检验,共进行1000次循环。从图1可知,酸枣仁与理枣仁的亲缘关系最近,其次为枳椇子,与紫荆子和兵豆的关系较远,都可以区分开,可信度在98%以上,符合植物分类结果。证明ITS序列可以用于鉴别酸枣仁及其混伪品。
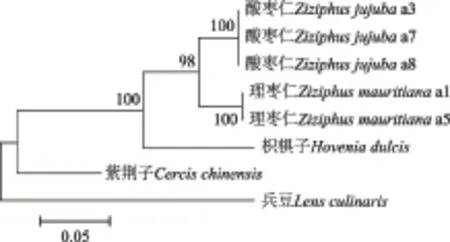
图1 基于ITS序列构建酸枣仁及其混伪品的系统发育树
3.2 PCR反应条件考察
为建立准确、简便的酸枣仁及其混伪品分子鉴别方法,本文对设计的鉴别引物在PCR反应中的应用进行了全面考察。
3.2.1 DNA模板量的考察 对25 μL PCR 反应体系中的模板DNA用量进行了考察,分别设置为0.42、0.70、0.98、1.26、1.54、1.82、2.10、2.38、2.66 ng·μL-1,反应体系为:12.5 μL 2x Easy Taq®PCR SuperMix 缓冲液,正反向引物各0.5 μL,无菌水补齐。图2结果显示,模板浓度为0.42 ng·μL-1时正伪品均未出现条带,而提高到2.10 ng·μL-1及以上则全部出现条带,其余模板量均能区分开酸枣仁及其混伪品,因此确定本方法DNA模板浓度范围应为0.70~2.10 ng·μL-1。实验采用0.70 ng·μL-1DNA模板浓度继续进行其他条件的考察。
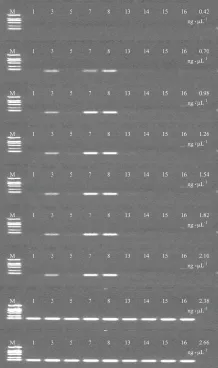
注:(1)M.2 000 bp DNA Marker,3、7、8.正品酸枣仁,1、5.理枣仁,13、14.枳椇子,15.紫荆子,16.兵豆;(2)从上至下DNA模板浓度分别为0.42、0.70、0.98、1.26、1.54、1.82、2.10、2.38、2.66 ng·μL-1。图2 不同DNA模板浓度PCR反应结果
3.2.2 退火温度的考察 设置退火温度分别为53、55、56、57、59、61、63 ℃进行考察,结果见图3。退火温度为53、55、56、57 ℃时伪品依稀可见假阳性条带,退火温度提高至59 ℃时伪品无条带,而正品酸枣仁有清晰的特异性扩增条带,退火温度为61、63 ℃时,伪品无条带,而正品条带也不易扩增出来。因此,为保证PCR反应具有良好的可重复性及区分度,采用59 ℃作为此方法的退火温度。
3.2.3 两种DNA聚合酶的考察 除上述方法中使用的聚合酶2x EasyTaq®PCR SuperMix,本研究还考察了另一种常用聚合酶Trans FastTaq DNA Polymerase(5 U·μL-1)的反应效果。PCR反应体系为25 μL,其中Trans FastTaq DNA Polymerase(5 U·μL-1)0.2 μL,TaKaRa dNTP Mixture 2.5 mm each 2 μL,Trans 10x FastTaq Buffer(Mg2+)2.5 μL,正反引物各 1 μL,DNA模板 1.40 ng·μL-1(1 μL),无菌水17.3 μL。PCR反应条件为94 ℃ 5 min,35个循环(94 ℃ 30 s,59 ℃ 15 s,72 ℃ 30 s),72 ℃ 7 min,结果见图4。采用两种聚合酶的PCR反应条件下,伪品无条带,而正品均有66 bp特异性扩增条带,说明特异性引物可用多种酶进行实验,无单一局限性。
3.2.4 PCR仪对实验结果重复性的影响 选用3台不同PCR仪器及同一仪器多次实验,结果均能区分开酸枣仁及其混伪品。

注:(1)M.2 000 bp DNA Marker;(2)从上到下依次为退火温度53、55、56、57、59、61、63 ℃;(3)3、4、7、8、9、10、11、12、17、18、19.正品酸枣仁,1、2、5、6、20.理枣仁,13、14.枳椇子,15.紫荆子,16.兵豆。图3 不同退火温度下PCR反应结果图

注:(1)M.2000 bp DNA Marker,3、4、7、8、9、10、11、12、17、18、19.正品酸枣仁;1、2、5、6、20.理枣仁;13、14.枳椇子;15.紫荆子;16为兵豆;(2)上为酶Trans FastTaq DNA Polymerase(5U·μL-1),下为酶2x Easy Taq®PCR SuperMix。图4 两种酶进行PCR实验的结果
4 讨论
ITS序列是位于高等植物核糖体rRNA基因上高度重复的串联序列单位,被广泛用于被子植物系统学的研究,尤其是近源属间和属内系统学的研究。目前ITS序列作为植物分子鉴定的特异性片段在泽泻[13]、红景天[14]、肉苁蓉[15]等多种药用植物中得到探讨和应用,陈士林等[16]以ITS2序列为主体推出了植物类中药材DNA条形码鉴定的通用引物,并已得到了广泛应用。本实验即选用DNA条形码中的ITS序列作为分子标记,使用现有文献记录的ITS序列通用引物对收集的样品进行测序,构建系统发育树的结果也证实了ITS序列可以作为分子标记来区分酸枣仁及其混伪品,这与任莉等的研究结果相一致[17]。实验收集的样品是当前药材市场较为常见的混伪品,其中理枣仁与酸枣仁为同属来源,形态相近最不容易区分。本实验基于ITS序列设计筛选鉴别酸枣仁及其混伪品的位点特异性引物,建立了位点特异性PCR方法,并对其反应条件进行了一系列考察,力求得到稳定、可重复的鉴别体系,可以准确鉴别出酸枣仁及其混伪品。
位点特异性引物PCR具有操作简单、成本低、重复性好、鉴定条带单一、样品用量少等优点,此外可以极大降低PCR的假阳性率,保证鉴定结果的准确性[18]。本实验建立的分子鉴别方法,将为应用DNA条形码鉴别中药材提供理论依据,同时也为开发酸枣仁快速鉴别试剂盒打下理论基础。
[1] 国家药典委员会.中华人民共和国药典:一部[S].北京:中国医药科技出版社,2015:366-367.
[2] 郑晔,钱苏瑜,游自立.酸枣仁药理作用研究进展[J].四川生理科学杂志,2006,28(1):35-37.
[3] 孙柳源.酸枣仁的两种混伪品[J].浙江中医学院学报,2000,24(3):73.
[4] 梅华,毕飞霞,张荣.酸枣仁、理枣仁、枳椇子三者之间性状、显微的区别[J].时珍国医国药,1996,(3):51.
[5] 兴宇清,秦伟华,李学彬,等.酸枣仁及其混淆品滇刺枣仁的鉴别[J].时珍国医国药,2001,12(12):1100.
[6] 帅智翠,岳林峰,张惠珍,等.酸枣仁滇枣仁和枳椇子光谱鉴别[J].时珍国医国药,1999,10(8):594-595.
[7] 刘文泉,谭亚非,赵庆忠,等.酸枣仁及其混淆品滇刺枣仁的鉴别[J].中医药学刊,2005,23(12):2281.
[8] 陈振德,许重远,谢立.酸枣仁及其混淆品蛋白质高效毛细管电泳法鉴别[J].中国药学杂志,2001,36(9):625-626.
[9] 吴建方.酸枣仁及其混淆品理枣仁的鉴别[J].中国实用医药,2008,3(31):155-156.
[10] 张淑华,李连环,刘振通,等.几种易混种子类重要的扫描电镜鉴别[J].中草药,1995,26(8):427.
[11] 刘萍,古今,周文斌,等.酸枣仁与其混淆品及伪品的RAPD分析[J].解放军药学学报,2006,22(1):67-70.
[12] CPB Group,LI D Z,GAO L M,et al.Comparative analysis of a large dataset indicates that internal transcribed spacer(ITS)should be incorporated into the core barcode for seed plants[J].PNAS,2011,108(49):19641-19646.
[13] 马晓冲,姚辉,辛天怡,等.基于DNA条形码SNP鉴别东方泽泻、泽泻及市售泽泻药材[J].中国药学杂志,2015,50(17):1474-1478.
[14] XIN T Y,LI X J,YAO H,et al.Survey of commercial Rhodiola products revealed species diversity and potential safety issues[J].Scientific Reports,2015,5:8337.
[15] 李振华,龙平,邹德志,等.基于ITS序列位点特异性PCR的肉苁蓉与其混伪品的鉴别研究[J].中国中药杂志,2014,39(19):3684-3688.
[16] 陈士林.中药DNA条形码分子鉴定[M].北京:人民卫生出版社,2012.
[17] 任莉,陈新连,石林春,等.应用DNA条形码技术鉴定中药材酸枣仁[J].世界科学技术—中医药现代化,2016,18(1):25-29.
[18] 黄璐琦.中药分子鉴定操作指南[M].上海:上海科学技术出版,2014:25.
IdentificationofZizyphusjujubawiththeCounterfeitProductsbyAllele-specificPCRBasedonITSGenesSequence
LI Guilin,SONGYadi,LYUZhenhui,LIUChunhui,LIJia,ZHANGXianan*
(SchoolofTraditionalChineseMedicine,CapitalMedicalUniversity,Beijing100069,China)
Objective:To establish molecular identification method forZizyphusjujubaand its counterfeit species based on ITS genes sequence.Methods:Total DNAs ofZ.jujuba,Z.mauritiana,Hoveniadulcis,LensculinarisandCercischinensiswere extracted,and the internal transcribed spacer(ITS)regions were amplified by PCR and the products were sequenced.BioEdit analysis software was used for sequence comparison.Primer Premier 5.0 was applied to design specific primers according to the mutation sites,the PCR reactions were performed and the conditions were examined.Results:ITS sequences were used in identification research ofZ.jujubaand its counterfeit species.To investigate the specific PCR reaction conditions,the scope of DNA template should variate from 0.7-2.1 ng·μL-1,the annealing temperature was proper at 59 ℃,different Taq enzymes and PCR instruments were available for this PCR process.Conclusion:In the same PCR reaction,66 bp DNA band could be amplified fromZ.jujubawhile nothing from the counterfeit species.It is a fast and accurate method to identifyZ.jujubafrom the counterfeit products using specific PCR primer.
Zizyphusjujuba;ITS;specific PCR;molecular identification
2016-01-18)
国家自然科学基金(81303166);国家中医药管理局中医药行业专项(201407003)
*
张夏楠,讲师,研究方向:分子生药学;Tel:(010)83911633,E-mail:xnzhang@ccmu.edu.cn
10.13313/j.issn.1673-4890.2016.12.007
