基于知识组织系统的生物医学文本挖掘研究
2016-09-23中国医学科学院医学信息研究所北京100020
钱 庆(中国医学科学院医学信息研究所,北京 100020)
基于知识组织系统的生物医学文本挖掘研究
钱庆
(中国医学科学院医学信息研究所,北京 100020)
随着生物医学信息技术的飞速发展,生物医学文献呈“指数型”增长,单纯依靠人工阅读获取和理解所需知识变得异常困难,如何从海量生物医学文献中整合已有知识、挖掘新知识成为当前研究热点。生物医学领域的知识组织系统建设相比其他领域更加规范和完整,为生物医学文本挖掘奠定了基础,大量基于知识组织系统的文本挖掘方法、系统得到快速发展。本文主要梳理现有医学知识组织系统,归纳生物医学文本挖掘的主要流程,按照挖掘任务探讨当前的主要研究和进展情况,并进一步分析基于知识组织系统的生物医学文本挖掘的特点,对知识组织系统在生物医学文本挖掘中发挥的主要作用和当前研究面临的挑战进行总结,以期为生物医学工作者提供借鉴。
知识组织系统;文本挖掘;信息检索;信息抽取;知识发现
1 引言
随着生物医学信息技术的飞速发展,生物医学信息资源增长快速,特别是文献资源呈“指数型”增长。PubMed是生物医学文献的主要仓储,包括MEDLINE、生命科学期刊和在线图书等,现有数据2 300多万条,以每年100多万篇的速度增长,并且这个数字在未来还会不断增加。在科学领域的开放获取期刊中,生物医学资源也是数量最多、增长最快的。急剧增长的生物医学文献为生物医学研究提供了丰富的资源,但是也造成信息获取的困难。因为大多数信息都隐含在无结构或者半结构的文本中,采用自然语言描述。自然语言虽然有助于人们直接理解和交流,但缺乏规范性,计算机难以理解。文本挖掘能够帮助人们从大量非结构化、半结构化生物医学文本中挖掘提取隐含的、事先不知道的但又具有潜在价值的信息和知识,现在被广泛应用于生物医学研究中,如生物医学实体识别、药物发现、靶标选择、药物副作用识别、蛋白质交互作用预测等方面。大量国际会议如BioNLP、SIGIR、BioCreative、TREC Genomics Track等,提出生物医学文本挖掘的任务,通过不同方法进行探索和实践,推动本领域研究的发展。在生物医学文本挖掘过程中,不可避免地需要应用大量特定领域知识,利用知识组织系统,特别是医学知识组织系统可以对概念进行规范、知识组织序化、关系发现和推理等,能够有助于提高人们获取新知识及其关联的能力。
2 医学知识组织系统
医学知识组织系统(Medical Knowledge Organization Systems,MedKOS)涵盖医学领域中的各种词汇列表、概念及概念间关系、分类体系及相应代码标识等,其对医学知识内容、概念及其相互关系进行描述和组织,具有词义消歧、同义词和近义词的控制、揭示概念之间的语义关系-等级关系、揭示概念之间的语义关系-非等级(相关)关系、揭示事物的类型及关系类型、描述事物的属性特征等功能[1]。医学知识组织系统形式多样,包括一体化语言系统、本体、叙词表、语义网络、分类表、权威规范术语表等。典型代表有医学主题词表(Medical Subject Headings,MeSH)、一体化医学语言系统(Unified Medical Language System,UMLS)以及各种医学本体等。MeSH词表是由美国国立医学图书馆(National Library of Medicine,NLM)编制的权威主题词表,在医学领域被广泛使用。1954年MeSH正式对外发布,1979年授权中国医学科学院医学信息研究所开始中文翻译,2007年推出网络版在线查询系统MeSH Browser。UMLS由NLM 于1986年主持启动,是生物医学领域、跨语言多表集成的知识组织系统,2015AB版集成了来自超过190万个来源词表的320多万个概念和128万个唯一概念名称,在医疗信息系统、病案系统、文本自动标注、智能检索等领域广泛应用。医学本体是对生物医学领域共享概念的明确形式化、规范化说明,也是生物医学文本挖掘中非常重要的知识组织系统之一,生物医学领域已建立大量本体,如基因本体(Gene Ontology,GO)、解剖学本体(The Foundational Model of Anatomy,FMA)、通用解剖参考本体(Common Anatomy Reference Ontology,CARO)、解剖实体本体(Anatomical Entity Ontology,AEO)、转化医学本体(Translational Medicine Ontology,TMO)、序列本体(Sequence Ontology,SO)、蛋白质本体(Protein Ontology,PRO)以及语言、百科和命名的通用架构(Generalized Architecture for Languages,Encyclopedias and Nomenclatures,GALEN)等。最常用的GO,其最初收录的基因信息来源于3个模式生物数据库:果蝇、酵母和小鼠,随后相继收录了更多数据,包括国际上主要的植物、动物和微生物基因组数据库。GO通过控制注释词汇的层次结构,使研究人员能够从不同层面查询和使用基因注释信息。从整体上来看,GO注释系统是一个有向无环图(Directed Acyclic Graphs,DAG),包含三个分支,即生物学过程(Biological process)、分子功能(Molecular function)和细胞组分(Cellular component)。注释系统中每一个结点(node)都是基因或蛋白质的一种描述,结点之间保持严格的关系,即“is a”或“part of”。开放生物医学本体(Open Biomedical Ontologies,OBO)是一系列关于生物和医学本体的集合。其中有些本体是通用的,应用于所有的生物体;有些本体是特殊的,只局限于某个领域。除此之外,还有ConceptWiki、Wikigenes、Wikipedia等。李丹亚等对医学知识组织系统进行了系统性总结,对203部医学知识组织系统的特征、构建模式等进行了分析和归纳[2],如表1所示。
Bodenreider也总结了生物学文本挖掘中常用的词典、术语集和本体,介绍了它们在实体识别和关系抽取中的应用[3]。
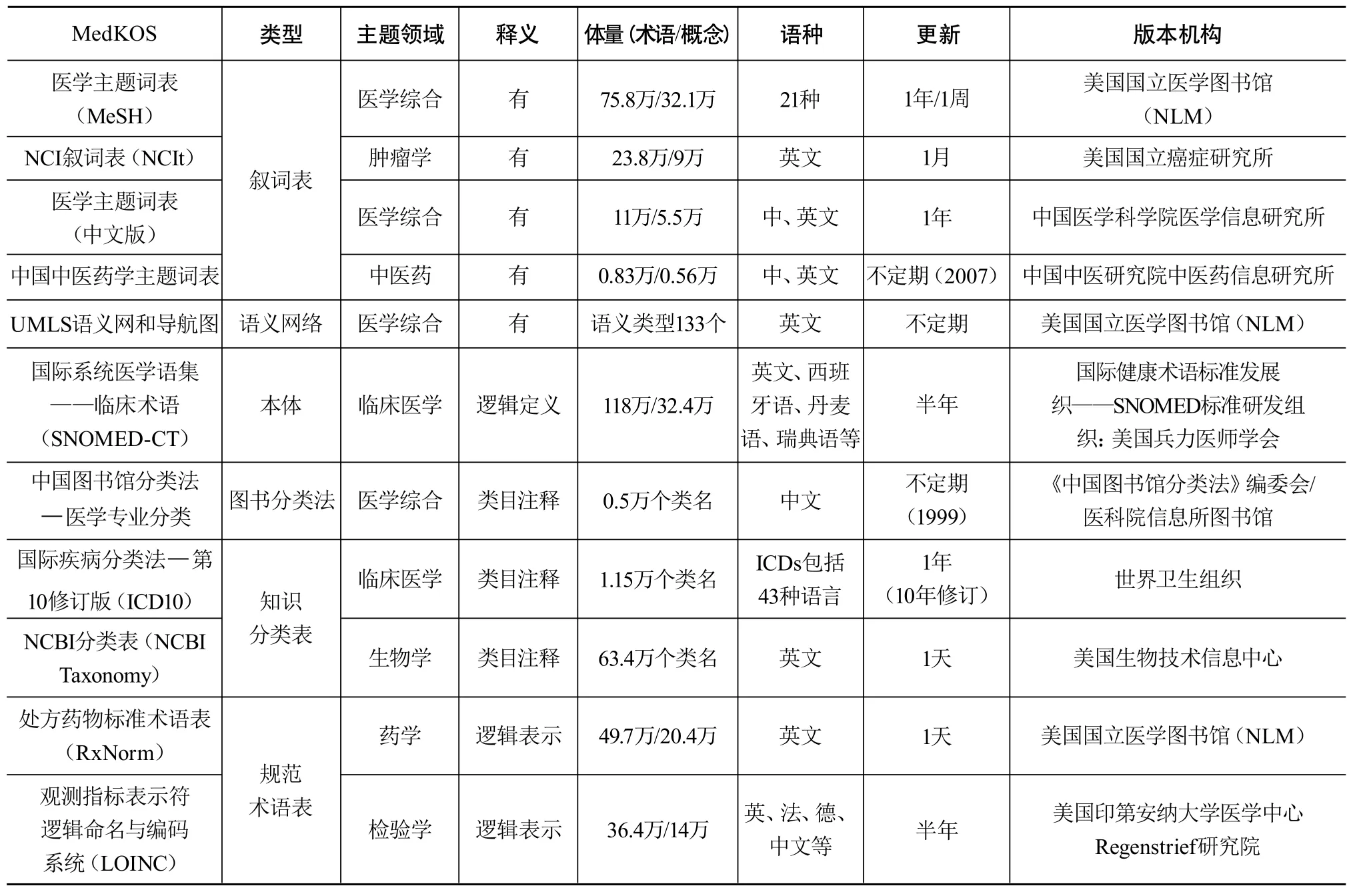
表1 医学知识组织系统[2]
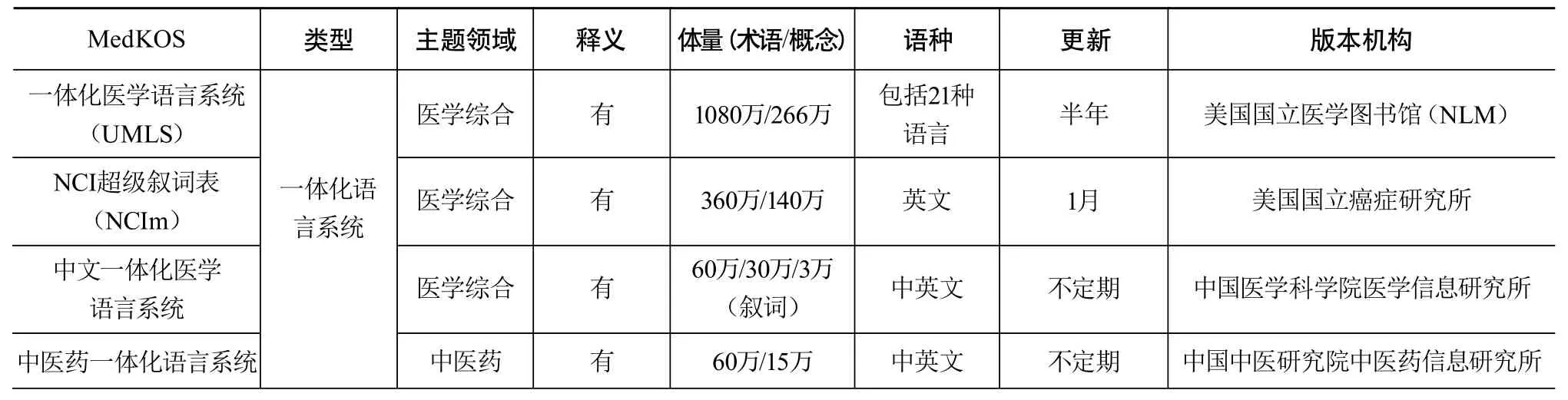
续表
3 生物医学文本挖掘的主要流程与典型应用
如图1所示,生物医学文本挖掘的主要目标是通过计算机辅助将非结构化或半结构化数据转换为结构化数据,将隐性知识转变为显性知识,帮助研究者进行知识发现。它的主要流程包括信息检索、信息抽取和知识发现三个步骤,这三个步骤也是生物医学文本挖掘的主要任务。信息检索的目标是获取关于某一主题的相关文本;信息抽取是抽取已定义类型的信息,如概念、实体或关系;知识发现是帮助从文本中抽取出潜在知识或基于文本推理获取未知的新知识。这三个步骤相互支撑,信息检索的结果可以缩小后两个步骤处理的文献数据范围,而信息抽取及知识发现的结果可以用于进一步优化信息检索结果,如提供深入文本内容的高级信息检索,提供相关类型实体、概念、实体间的隐含关系等。相比其他领域,生物医学领域的语义资源建设更加规范和完整,大量知识组织系统为文本挖掘奠定了基础。在生物医学文本挖掘过程中,知识组织系统被作为资源、工具、标准规范或专家知识等,发挥了重要作用。其中包含的大量术语,以及以树状或网状结构记录的术语间的关联,可用于支持生物医学文本挖掘应用。同时,文本挖掘的结果所生成的结构化知识也可以用于构建知识组织系统,用于丰富词表或本体的实体及语义关系。下面主要按照这三个关键任务,组织、论述基于知识组织系统的生物医学文本挖掘的最新研究情况,并分析和归纳知识组织系统在其中的具体作用。
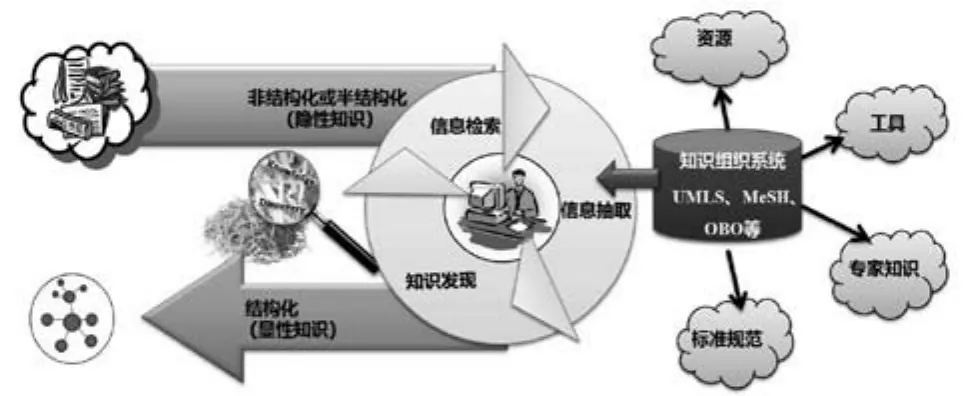
图1 生物医学文本挖掘流程
3.1 信息检索
传统信息检索方法如关键词检索或布尔逻辑检索等具有一定缺陷,如用户输入的检索词可能不能充分代表其真实需求;检索系统对文本的标引不能完全表达文献的内容,特别是缺乏考虑信息资源之间的语义关系,不能提供深层次的信息关联;检索结果使用线性排序,导致用户不能从多维度探测检索结果。针对现有信息检索系统难以满足用户知识获取需求的问题,大量具有标准化可控词汇并具有层次结构(树状或网状)的知识组织系统被引入检索系统中,用于对生物医学文献进行深度标引、拓展用户查询、对信息资源进行深度语义关系提取和分析、对检索结果进行多层次或多维揭示等,实现基于语义的知识检索和智能检索。PubMed是基于WEB的生物医学信息检索系统,它能自动地为输入的检索词寻找相应的MeSH词,用户利用MeSH词能找出所有有关该主题的文献,提高了检索的准确性和专指性。GoPubMed使用GO和MeSH标引检索结果,将来自GO、MeSH及UniProt的术语映射到PubMed数据库的文献中,生成基于本体的检索结果浏览,并对检索结果进行组织、分类,提供与检索词相关的来自GO等本体的相关术语[4]。美国孟菲斯大学的CVPIA实验室开发了SEGoPubMed检索系统,该系统以PubMed为数据源,利用GO本体,在PubMed检索时使用潜在语义分析技术和语义相关度排序大大提高了查准率和查全率[5]。为了解决研究者的问题,如“哪些疾病和一个特定基因相关”或“哪些化学物质和一种特定疾病相关”,现有研究也构建了能够揭示这些关联的检索系统。FACTA是一个基于MEDLINE数据库摘要的文本搜索引擎,用于查找关联的生物医学概念,不仅标引了文本中的词,而且标引了概念,能够让用户进行灵活查询并且用户可以看到来自MEDLINE的文献片段,包括检索词或概念的关联证据[6],如图2所示。FACTA覆盖六大类生物医学概念,包括人类基因/蛋白质、疾病、症状、药物、酶和化学化合物,通过词典匹配判断这些概念是否出现在文本中。一共标引了80 260个唯一概念,使用UniProt访问号作为基因/蛋白质的概念ID,收集了来自多个知识组织系统的基因/蛋白质的名称和同义词,疾病和症状主要来自UMLS,药物、酶和化学化合物的概念ID和名称则来自HMDB、KEGG和DrugBank等数据库。
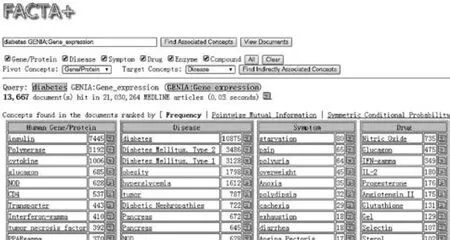
图2 FACTA检索结果[6]
PubOnto也是基于本体的MEDLINE文献浏览检索系统,使用来自OBO的多种本体,包括GO、Foundational Model of Anatomy(FMA)、Mammalian Phenotype Ontology、Environment Ontology等,帮助研究者从不同角度浏览文献,并快速定位最相关的MEDLINE记录用于进一步研究[7]。PubOnto如图3所示,基于AdobeFlex3.0平台,将本体术语自动映射到MEDLINE摘要,提供交互式探索和检索结果过滤,交互的本体过滤模式有助于找到不同本体间的交叉文献。PubOnto还提供定制检索、客户端过滤、定制本体检索、引文链接到PubMed、概念链接到Wikipedia、可视化统计分析、对检索文献的MeSH进行词频统计和打分等功能。
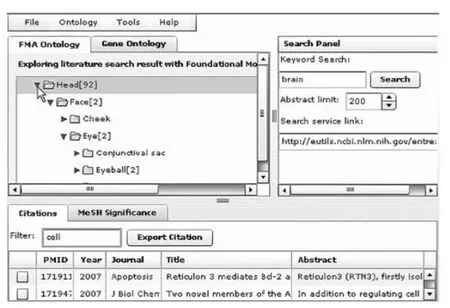
图3 PubOnto的检索结果[7]
3.2 信息抽取
信息抽取包括对生物医学文本中的概念、实体(如疾病、症状、药物、基因、蛋白质、器官、化学物质等)及各种关系(基因间的关系、蛋白质间的关系、基因和疾病间的关系、疾病和药物间的关系、药物和治疗间的关系等)的抽取。特别是随着生物医学领域对生物数据保存、编审的日益关注,计算机实体抽取技术得到进一步促进和发展,用以辅助人工编审。
3.2.1 概念及实体识别
典型的概念识别系统是NLM开发的初步标引系统MetaMap,用于图书馆半自动和全自动的生物医学文献标引。其基于UMLS叙词表通过切分、产生变形体、检索候选词、候选词的评价、建立匹配等一系列流程,将生物医学文本与UMLS超级词表中的概念进行匹配和筛选排序,能够有效识别文本中来自UMLS的概念。UMLS每一次改版,MetaMap也需要更新其数据库文件,包括预先计算变形词表、语义类型和MeSH树状结构号的信息,以及按照超级词表中含有单词的字串索引[8]。
实体识别是对词或短语的识别,并将分类对应到预先定义的分类上,如疾病、症状、药物或基因。现有实体识别方法可归纳为三类,分别为基于词典的实体识别、基于规则的实体识别和基于机器学习的实体识别。基于词典的实体识别方法是最基础的识别方法,识别来自词表等资源中的实体名称,如使用ICD识别疾病名称、使用GO识别基因名称等,能够保证识别的准确率,但是也存在局限性,因为很多实体不一定会在已有词典中出现,因此,一般会与基于规则的方法结合使用。Fang等开发的一个癌症命名实体识别器MeinfoText系统,采用结合癌症词典和基于正则表达的方法挖掘基因甲基化和癌症关联信息[9]。UEMUC3M是一个基于本体的生物医学文本的实体识别系统,能够用于识别药学领域中的化学物质[10]。该系统通过应用生物医学本体和外在资源,决定是否将识别的术语作为一个药品名称。从文本中找到概念的过程被称为Megrep,又分为两个步骤:首先扫描、识别实体;其次,通过规则对实体分类。识别过程是利用UMLS和药物领域本体、主药物数据库本体(Master Drug Data Base,MDDB)、国家药物数据文件(National Drug Data File,NDDF)、药物发现调查本体(Ontology for Drug Discovery Investigetions,ODDI)等进行药物名称识别。鉴于基于规则和基于词典的实体识别存在不足,大量基于机器学习的生物医学实体识别方法如基于HMM的方法、基于SVM的方法、基于CRF的方法等被提出。机器学习方法需要使用训练集进行模型训练。训练集是经人工或机器已经标注实体特征的文本集。实体特征可归纳为5类:语言特征、拼写特征、形态学特征、上下文特征和词典特征[11]。其中,词典特征使用来自特定领域的术语或实体名称和文本中的术语进行匹配和识别,用于进一步优化实体识别功能。
PubTator是基于网络用于帮助人工生物编审(biocuration)和文本注释的工具[12]。它支持对PubMed检索结果的标注,识别的生物医学实体包括基因、化学物质、疾病、变异、物种,标注结果如图4所示。它由多种实体识别工具组成,包括跨物种基因标注工具GenNorm[13]、基于成对学习排序的疾病实体识别工具Dnorm[14]、化学命名实体识别工具tmChem[15]、基因标准化的物种识别工具SR4GN[16]、抽取序列变异的工具tmVar[17]。这些工具使用了MeSH、MEDIC和来自NLM的词典用于实体特征训练和词典查找。PubTator提供在线使用和调用URL的使用方式。

图4 PubTator文本标注结果[12]
NCBO Annotator是基于本体的网络服务,用于对公共数据集文本进行标注[18]。其使用来自BioPortal和UMLS的本体概念,便于数据集成和转化发现。如图5所示,其工作流程包括两个关键步骤:(1)直接注释:通过使用一个由来自UMLS和NCBO本体的术语(概念名称和同义词)构成的词典进行语法概念识别;(2)语义拓展注释:组件使用本体语义拓展直接生成注释的集合,其中用到的组件包括is_a传递闭包、本体间映射、相似度算法等。
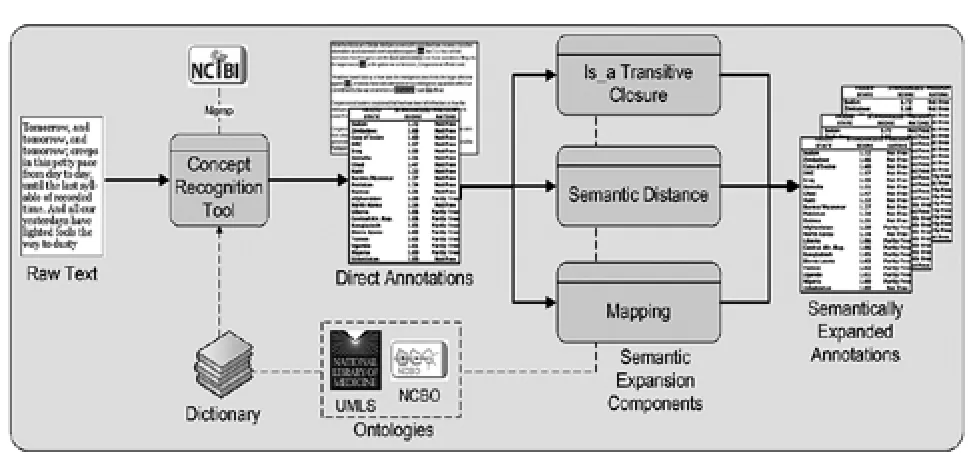
图5 NCBO Annotator的工作流程[18]
3.2.2 关系抽取
关系抽取是信息抽取的关键技术之一,比实体抽取更为复杂。通过关系抽取可以建立实体之间的信息关联,用于构建领域本体、支持文本聚类、构建生物医学知识网络、构建自动问答系统等。关系抽取的主要方法包括基于共现的抽取、基于自然语言处理的抽取、基于词典的抽取、基于模式匹配的抽取、基于机器学习的方法等。其中,基于词典的关系抽取主要利用生物医学词表、本体、语义网络等中的同义关系、层级关系、具体类型关系等进行关系的抽取。基于模式匹配的方法,通过定义规则进行关系抽取,依赖于规则的数量,难以涵盖全部关系。医学信息检索平台CoremineMedical(见图6)利用本体语言技术支持MEDLINE数据库的相关数据、文献、信息、知识资源的检索、分析和获取[19],通过构建术语关联共现网络和术语类型组织来发现相关的概念,这些概念来自MeSH、GO等知识组织系统。
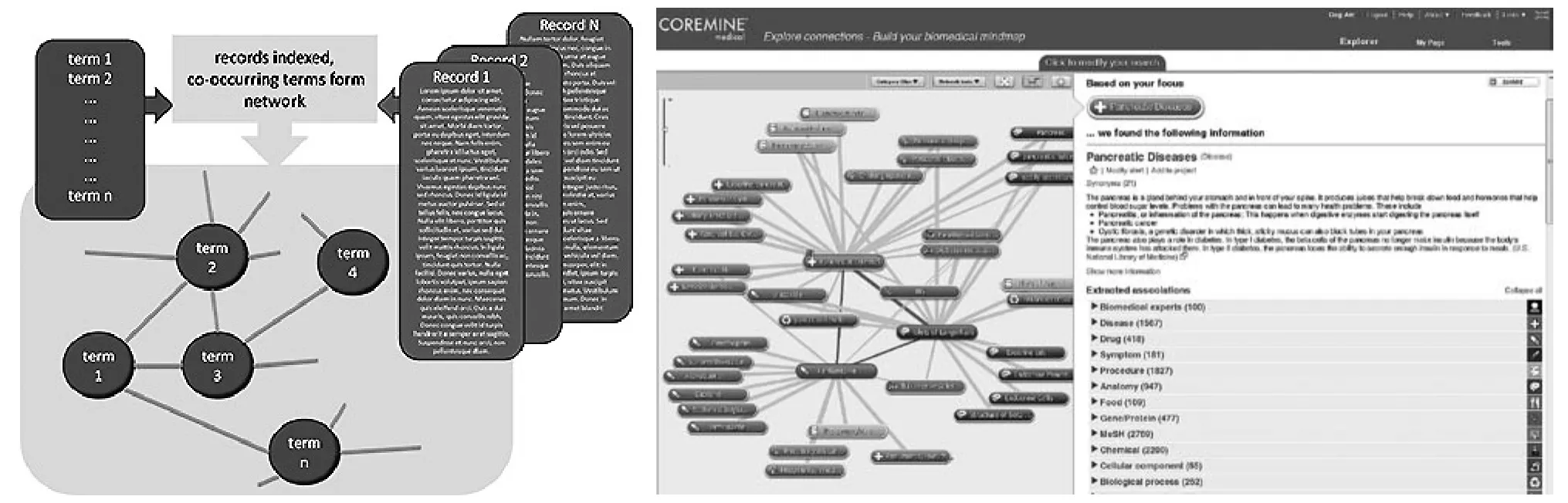
图6 CoreMine medical的主要流程[19]
SemRep是基于UMLS语义关系的工具,首先利用MetaMap识别文本中的概念及其语义类型,而后对文本进行浅层语义分析,根据UMLS语义网络定义的54种关系,判断两个概念之间的关系[20]。Embarek和Ferret提出的MeTAE(Medical Texts Annotation and Exploration)系统从文本中抽取实体和关系[21],其对MetaMap进行了改进,用于抽取实体并提出一种基于语言模式的关系抽取方法,并基于UMLS语义网络中的语义类型进行过滤和识别,关系结构存储为RDF三元组格式。RINDFLESCH基于UMLS,利用领域知识和语法解析构建了ARBITER,使用两阶段法识别分子作用关联(Molecular Binding):首先利用MetaMap、语法解析器等识别作用关联术语集;其次,识别作用关联术语作为关系的论元(agruments)[22]。Sharma等提出构建以动词为中心的关系抽取系统,利用UMLS语义网络、WordNet和VerbNet从生物医学文献中识别包含关系的句子,而后利用深层解析器和语义角色分析器抽取关系的描述短语,并识别及抽取涉及的生物医学实体,最后输出抽取的关系[23]。他将其应用于MEDLINE文摘构成的三个数据集进行测评,其算法达到0.86~0.95的准确率和0.88~0.92的召回率。Pustejovsky等使用UMLS和Brill词法解析器,通过浅层语法解析,从文献摘要中抽取蛋白质抑制关系信息[24]。
3.3 知识发现
基于文献的知识发现,包括开放发现和闭合发现模式,可以通过开放发现模式生成新的假设,或通过闭合发现模式检验一个假设,从而发现新的知识。基于文献的知识发现理论是1986年由美国芝加哥大学的医学教授D. R.Swanson最早提出的,指出非相关的生物文献中可能隐含大量不为人知的科学知识[25]。Swanson将基于文献的知识发现定义为:如果有两类文献集A和C,其中,A讨论了概念M和概念集B之间的关系,而C则讨论了概念N和概念集B之间的关系,但是没有任何文献直接讨论过M和N的关系,那么M与N之间通过共同的桥梁B,隐含地存在某种关系,这就可能是一个新的科学发现。这时的A和C被称为非相关互补的文献,而概念集B则被称为中间集。他将该理论应用于发现镁缺乏与神经系统疾病、消炎痛与阿尔兹海默病、雌激素与阿尔兹海默病、游离钙磷脂酶A2与精神分裂症、镁缺乏与偏头痛以及可作为生物武器的潜在病毒间的关系。Swanson教授与Neil Smalheise构建了Arrowsmith系统,用于处理从PubMed数据库检索出的A和C文献集,而后对中间集B进行过滤和排序,按照相对频次排列的列表提供给用户[26]。Smalheise将UMLS引入Arrowsmith系统的处理过程中,基于UMLS对中间集B进行语义归类、筛除低频共现词、基于共现的统计学模型对中间集B聚类、去低频特征词等[27]。BITOLA(见图7)也是一个基于文献的交互式生物医学发现支持系统,系统采用闭合式和开放式两种发现模式,目标是帮助生物医学研究者发现生物医学概念间潜在的新关系[28]。系统采用来自MeSH中的主题词表达概念和来自HUGO的人类基因名称。
Hu提出一种新的基于语义分析的知识发现系统(Biomedical Semantic-based Association Rule System,Bio-SARS),该系统使用MeSH词表示文献内容,通过UMLS语义类型和基于语义的关联规则减少候选术语的数量和过滤无关联的关系[29]。Litlinker系统使用基于文献的开放知识发现系统,利用MetaMap获取MeSH术语[30]。Literby系统使用两阶段方法,利用MetaMap获取MeSH术语,通过UMLS过滤数据类型[31]。Srinivasan等开发了文本挖掘系统Manjal,该系统使用MeSH词和关键词来代表文献的内容,根据语义类型来过滤词汇并利用词的权重确定词间的关系[32]。
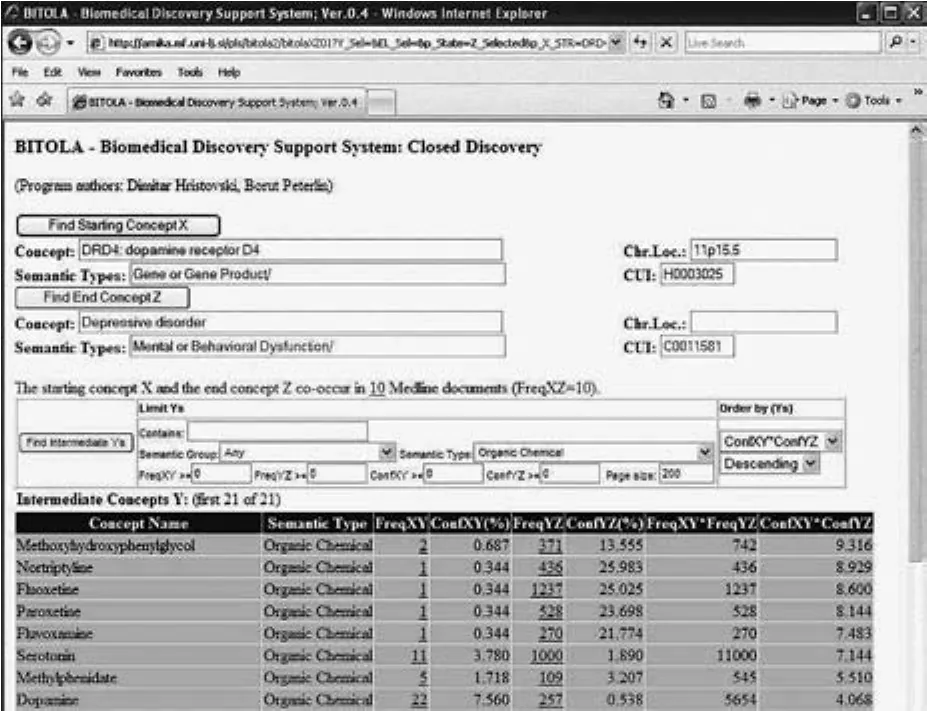
图7 BITOLA挖掘结果界面
4 特点分析
综上可知,生物医学文本挖掘得到快速发展,基于知识组织系统的生物医学文本挖掘体现出如下特点:
(1)知识组织系统在文本挖掘各阶段中发挥了重要作用。其可归纳为:①在信息检索中,被用于文献内容的标引、用户检索词的扩展、对检索结果的组织浏览、作为外部注释资源解释和理解文本内容、检索结果的可视化;②在信息抽取中,可用于对术语进行匹配映射、消歧去重、规范表达,用于术语或实体分类及进行训练集的标注,用于抽取结果后处理优化;③在知识发现中,被用于抽取实体和关系类型的过滤。此外,通过知识组织系统中包含的可控词汇对生物医学文献进行语义标注,搭建起生物医学文献与生物医学数据之间的桥梁。
(2)生物医学文献挖掘结果也可用于构建知识组织系统。知识组织系统和生物医学文本挖掘过程相互支撑,从本体中获得的实体或关系可以支持文本挖掘过程;反之,通过生物医学文本挖掘识别的概念、术语、关系,也可以用于构建本体和词表,或对现有本体词表中的术语或语义关系的语义。
(3)面向特定文本挖掘任务选择特定知识组织系统。在现有生物医学文本挖掘中,需根据特定目标选择相应的本体或词表。此外,现有生物医学文本挖掘研究中使用单一本体或词表难以满足应用需求,而需使用集成词表(如UMLS)、集成本体(OBO)或将多词表或多本体联合使用以满足挖掘应用。
(4)多方法融合的生物医学文本挖掘。无论是实体识别还是关系抽取,单一识别或抽取方法往往不能取得较好的效果。通过现有研究可以发现,研究者趋向于多方法融合的挖掘方法,用于弥补单一方法的不足,提高实体识别及关系抽取的准确率和召回率。
5 结语
基于知识组织系统的生物医学文本挖掘取得了一定的进展,而仍然面临诸多挑战。虽然大量医学知识组织系统被用于生物医学挖掘系统中,但是当前医学知识组织系统对生物医学术语的覆盖有限,不能覆盖所有文献中出现的术语,如UMLS叙词表中记录了超过1 600万个关系,而这些关系也不能全面反映文献中术语间或实体间的关系;并且,当前文本挖掘研究逐渐趋向面向开放资源的抽取任务。因此,如何优化现有的基于知识组织系统的生物医学文本挖掘方法,成为未来研究需要进一步思考的问题。
[1] 曾蕾.在浏览和检索界面设计中利用知识组织系统(KOS)[EB/OL].[2015-12-01]. http://www.libnet.sh.cn/upload/htmleditor/File/071213121516.pdf.
[2] 李丹亚,李军莲,李晓瑛,等.医学知识组织体系发展现状及研究重点[J].数字图书馆坛, 2012(12):12-20.
[3] Bodenreider O. Lexical, Terminological, and Ontological Resources for Biological Text Mining[EB/OL].[2015-12-01].http://www.artechhouse. com/uploads/public/documents/chapters/ananiadou_984_samplech03. pdf.
[4] Delfs R ,Doms A, Kozlenkov A,et al.GoPubMed:ontology-based literature search applied to Gene Ontology and PubMed[EB/OL].[2015-12-01]. http://www.biotec.tu-dresden.de/fileadmin/groups/schroeder/group/ papers/gopubmedGCB.pdf.
[5] Yeasin M,Vanteru B,Shaik J,et al. i-SEGOPubmed: a web interface for semantic enabled browsing of PubMed using Gene Ontology[EB/ OL].[2015-12-01]. http://www.biomedcentral.com/content/pdf/1471-2105-9-S7-P20.pdf.
[6] Tsuruoka Y, Tsujii J, Ananiadou S. FACTA: a text search engine for finding associated biomedical concepts[EB/OL].[2015-12-01]. http:// bioinformatics.oxfordjournals.org/content/24/21/2559.long.
[7] PubOnto provides multiple ontologies from the Open Biomedical Ontology [EB/OL].[2015-12-01].http://brainarray.mbni.med.umich.edu/ brainarray/prototype/PubOnto/.
[8] 张云秋,冷伏海.MetaMap的文本映射原理及其对信息检索效果的影响[J].情报学报, 2007, 26(3):344-349.
[9] Yu C F, Po T L, Hong JD,et al. MeInfo Text2.0:gene methylation and cancer relation extraction from biomedical literature[J].BMC BIOINFOR MATICS,2011,12(1):471.
[10] Fernando.UEM-UC3M: An Ontology-based named entity recognition system for biomedical texts[EB/OL].[2015-12-01].http://aclweb.org/ anthology/S/S13/S13-2104.pdf.
[11] Prikshit S. A survey on Name Entity Extraction in the Biomedical Domain [EB/OL].[2015-12-1]. http://sifaka.cs.uiuc.edu/~sondhi1/survey1. pdf.
[12] Wei C H, Kao H Y, Lu Z Y. PubTator:A PubMed-like interactive curation system for document triage and literature curation[EB/OL].[2015-12-01]. http://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/PubTator/tutorial/ PubTator.pdf.
[13] GenNorm[EB/OL].[2015-12-01].http://ikmbio.csie.ncku.edu.tw/GN/.
[14] Leaman R, Dogan R I, Lu Z Y. DNorm:Disease Named Entity Recognition and Normalization with Pairwise Learning to Rank[EB/ OL].[2015-12-01]. http://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/ DNorm/.
[15] Leaman R,Wei C H, Lu Z Y. tmChem: a high performance approach for chemical named entity recognition and normalization [EB/ OL].[2015-12-01]. http://www.ncbi.nlm.nih.gov/CBBresearch/Lu/Demo/ tmChem/.
[16] Wei C H, Kao H Y, Lu Z Y. SR4GN:a species recognition software tool for gene normalization [EB/OL].[2015-12-01].http://www.ncbi.nlm.nih. gov/CBBresearch/Lu/downloads/SR4GN/.
[17] Wei C H,Harris B R, Kao H Y,et al.tmVar:A text mining approach for extracting sequence variants in biomedical literature [EB/ OL].[2015-12-01]. http://www.ncbi.nlm.nih.gov/CBBresearch/Lu/pub/ tmVar/.
[18] Jonquet C, Shah N H, Musen M A,et al. NCBO Annotator: Semantic Annotation of Biomedical Data[EB/OL].[2015-12-01]. http://www.lirmm. fr/~jonquet/publications/documents/Demo-ISWC09-Jonquet.pdf.
[19] Coremine medical [EB/OL].[2015-12-01]. http://www.coremine.com/ medical/#search?ids=519944&tt=8191&org=hs&i=5199441.
[20] Semantic Knowledge Representation[EB/OL].[2015-12-01].http://semrep. nlm.nih.gov/.
[21] Abacha A B, Zweigenbaum P. Automatic extraction of semantic relations between medical entities: a rule based approach[J].JOURNAL OF BIOMEDICAL SEMANTICS,2011,2(5): 1-11.
[22] Rindflesch T C, Rajan J V, Hunter L. Extracting Molecular Binding Relationships from Biomedical Text[EB/OL].[2015-12-01]. http://165.112.8.46/files/archive/pub2000016.pdf.
[23] Sharma A, Swaminathan R,Yang H. A Verb-centric Approach for Relationship Extraction in Biomedical Text[EB/OL].[2015-12-01]. http:// cs.sfsu.edu/~huiyang/publications/ICSC10-rel-ex.pdf.
[24] Verhagen M, Pustejovsky M. Medstract - The NextGeneration[EB/ OL].[2015-12-01]. http://www.aclweb.org/anthology/W11-0224.
[25] Swanson D R, Smalheiser N R, Bookstein A. Information discovery from complementary literatures:categorizing viruses as potential weapons[J]. JOURNAL OF AMERICAN SOCIETY FOR INFORMATION SCIENCE AND TECHNOLOGY,2001,52(10):797-812.
[26] Swanson D, Smalhesier N. An interactive system for finding complementary literatures: a stimulus to scientific discovery[J]. ARTIFICIAL INTELLIGENCE,1997,91(2):183-203.
[27] Smalheiser N R. The Arrowsmith project:2005 status report[EB/ OL].[2015-12-01] . http://pdf.aminer.org/000/039/534/the_arrowsmith_pr oject_status_report.pdf.
[28] Hristovski D, Peterlin B. BITOLA-Biomedical Dicovery Support System [EB/OL].[2015-12-01]. http://ibmi.mf.uni-lj.si/bitola/.
[29] Hu X,Yoo I, Rumm P, et al.Mining Candidate Viruses as Potential Bioterrorism Weapons from Biomedical Literature[EB/OL].[2015-12-01]. http://link.springer.com/content/pdf/10.1007/11427995_6.pdf.
[30] Pratt W,Yildiz M. LitLinker: Capturing Connections across the Biomedical Literature[EB/OL]. [2015-12-01].http://staff.washington.edu/ melihay/publications/KCAP2003.pdf.
[31] Weeber M. Drug Discovery as an Example of Literature-Based Discovery[EB/OL]. [2015-12-01]. http://link.springer.com/content/pdf/10. 1007/978-3-540-73920-3_14.pdf.
[32] Sehgal A K, Srinivasan P. Manjal:A Text Mining System for MEDLINE[EB/OL]. [2015-12-01]. http://dl.acm.org/ft_gateway. cfm?id=1076192&type=pdf.
Research on Biomedical Text Mining Based on Knowledge Organization System
QIAN Qing
(Chinese Academy of Medical Sciences, Institute of Medical Informatics, Beijing 100020, China)
With the rapid development of biomedical information technology, biological medical literatures grow exponentially. It's hard to read and understand the required knowledge by manual, how to integrate knowledge from huge amounts of biomedical literatures, mining new knowledge has been becoming the current hot spot. Knowledge organization system construction in the field of biological medicine is more normative and complete than other fields, which is the foundation for biomedical text mining. A large number of text mining methodsand systems based on knowledge organization system have fast development. This paper investigates the existing medical knowledge organization systems and summarizes the process of biomedical text mining. It also summaries the researches andrecentprogressand analyzes the characteristics of biomedical text mining based on knowledge organization system. The knowledge organization systems play an important role in biomedical text mining and the challenge for the current study are summarized, so as to provide references for biomedical workers.
Knowledge Organization System; Text Mining; Information Retrieval; Information Extraction; Knowledge Discovery
G254
10.3772/j.issn.1673-2286.2016.4.001
钱庆,男,1970年生,中国医学科学院医学信息研究所副所长,研究员,研究方向:数据挖掘,E-mail:qian.qing@imicams.ac.cn。
2016-01-19)
