尼罗罗非鱼干扰素调节因子1的表达与细胞定位
2016-09-23解文杰朱叶飞赵金良陈晓武
王 丹,解文杰,朱叶飞,赵金良,陈晓武
(上海海洋大学 水产种质资源发掘与利用教育部重点实验室,上海 201306)
尼罗罗非鱼干扰素调节因子1的表达与细胞定位
王丹,解文杰,朱叶飞,赵金良,陈晓武
(上海海洋大学 水产种质资源发掘与利用教育部重点实验室,上海201306)
为了分析尼罗罗非鱼干扰素调节因子1(IRF1)的表达规律及其功能,克隆了尼罗罗非鱼IRF1基因cDNA全长,与其他脊椎动物IRF1基因进行比对,构建IRF家族系统进化树。然后使用PolyI∶C对尼罗罗非鱼进行体内注射诱导抗病毒免疫反应,利用荧光定量PCR技术检测处理组和对照组中尼罗罗非鱼各组织IRF1基因的表达差异。最后构建尼罗罗非鱼IRF1基因真核表达载体,对其进行细胞定位。结果表明,尼罗罗非鱼IRF1有888 bp的开放读码框,编码295个氨基酸的蛋白序列。该蛋白N端高度保守,并含有6个保守的色氨酸,具有DNA结合结构域。对蛋白相似率的计算发现尼罗罗非鱼与斑马鱼IRF1相似度为52.1%。而与大鼠IRF1的相似率最低,为36.2%。IRF家族成员蛋白序列构建的系统进化树显示,IRF家族共分为4个亚群。将pCMV3-FLAG-IRF1真核表达载体转染到Hela细胞中后,对IRF1进行细胞定位发现,尼罗罗非鱼IRF1主要分布在胞质中,胞核中有少量分布。对尼罗罗非鱼使用PolyI∶C进行体内注射诱导抗病毒应答反应,利用实时荧光定量PCR检测到各个组织中IRF1在不同时间下的表达水平有显著差异。本研究完成了尼罗罗非鱼IRF1的表达和细胞定位,对各种脊椎动物IRF家族成员进行了系统进化分析,结果有助于深入了解干扰素系统在机体免疫系统中的调控机制。
干扰素调节因子;尼罗罗非鱼;系统进化;细胞定位
干扰素调节因子(Interferon regulation factor,IRF)能够调节干扰素基因(Interferon,IFN)的转录。至今为止,在哺乳动物中发现9个IRF成员(IRF1~9),鸟类中共有10个IRF家族成员被鉴定出(IRF1~10),而在鱼类,IRF家族第11号成员IRF11只在斑马鱼等硬骨鱼类中[1-2]。IRF除了可以诱导IFN的产生及抗病毒功能之外,还在细胞因子信号转导、细胞生长及凋亡等方面发挥着广泛作用[3-4]。所有的IRF家族成员在结构上均有较高的保守性,IRF基因N端由大约120个高度同源的氨基酸组成,为DNA结合结构域(DNA-binding domain,DBD)。在DBD结构域内有5个高度保守的色氨酸,形成螺旋-转角-螺旋结构。通过DBD结构域,IRF基因能够结合DNA以发挥各种生物学功能。IRF1由于能够结合在Ⅰ型IFN的病毒诱导类增强子元件上诱导IFN的表达而最早被发现,随后家族其他成员被陆续报道。除了IRF1外,其他IRF成员的羧基端均有一个能够介导与其他转录因子互作的干扰素调节因子相关域(Interferon associate domain,IAD) 1区域[5]。而在IRF1亚家族羧基端为IAD2,能够与IRF8发生互作。IRF1最先作为Ⅰ型干扰素基因病毒诱导增强子样元件的调节子被发现,也是目前研究最多的IRF家族成员。IRF1能够结合IFN启动子调控域,诱导I型IFN的表达,参与机体抗病毒过程[6]。对哺乳动物IRF1的研究发现,IRF1能够在各种细胞中组成性表达,不仅存在于细胞核中,也存在细胞质中。在斑马鱼中,IRF1与其他IRF成员相比,在免疫器官中表达量最高,并且病毒感染后的IRF1表达变化最为显著[7]。通过利用IRF1基因构建质粒,并用荧光素酶分析发现草鱼体内IFN的转录水平提高,从而证明IRF1是草鱼IFN转录的正调节因子[8]。在鱼类中,牙鲆的IRF1是较早被鉴定出来的。当被感染牙鲆弹状病毒后,IRF1基因在牙鲆各组织中的表达量都出现增高[9]。IRF家族成员众多,功能复杂,有的相互协同,也有相互拮抗的关系[7]。在细胞定位研究中也有不同之处。目前,在少数动物中有报道。鱼类作为种类最多的脊椎动物,是研究IRF系统最好的选择。
尼罗罗非鱼(Oreochromisniloticus)隶属于鲈形目(Percifomes)、丽鱼科(Cichlidae),其适应性强、食性广、生长快、繁殖力高[10]。我国是尼罗罗非鱼养殖的主要国家,现已成为主要淡水养殖品种。近年来,由于养殖密度高、养殖环境变化等原因导致养殖尼罗罗非鱼发病率逐渐升高,威胁着尼罗罗非鱼养殖业的健康发展[11-12]。对尼罗罗非鱼抗病毒研究,特别是IRF家族在病毒性疾病中的功能研究还很少。尼罗罗非鱼基因组已经比较完整,多数IRF家族成员已经被注释,但尚需试验验证。IRF1表达水平相对其他IRF家族成员较高,也是重要的代表性成员。因此,本试验以尼罗罗非鱼为研究对象,对IRF1基因结构、系统进化、抗病毒效应、以及细胞定位方面进行分析探讨。
1 材料和方法
1.1尼罗罗非鱼养殖和取材
试验用尼罗罗非鱼是由上海海洋大学尼罗罗非鱼种质资源试验站提供的新吉富品系,选择体重200 g左右的试验鱼,暂养于室内循环水养殖中心,试验期间水温25 ℃,每天投喂饲料2次,14 d后挑选摄食正常,体表完好无伤,健康状况良好的尼罗罗非鱼用于试验。暂养后的尼罗罗非鱼随机分为2组。使用聚肌胞苷酸(PolyI∶C)对其中一组尼罗罗非鱼进行腹腔注射以模拟病毒刺激。所使用的PolyI∶C购自Sigma公司,溶于灭菌后的磷酸缓冲液中,注射量为每克鱼0.5 μg。另一组使用不含PolyI∶C的PBS腹腔注射作为对照。
1.2组织总RNA的提取
PolyI∶C处理12,24 h后,将处理组及对照组尼罗罗非鱼用M-222麻醉,取脑、鳃、肝、脾和肾脏组织迅速冻存于液氮中,约30 mg组织在1 mL TRIzol进行裂解,裂解使用AP公司的裂解柱。RNA提取按照TRIzol说明书中的操作步骤进行。提取完毕后,使用1% 琼脂糖凝胶电泳检测 RNA 的完整性,并用分光光度仪检测 RNA 的产量和纯度。合格的RNA 样品存放于-80 ℃备用。
1.3IRF1基因的克隆
第一链cDNA合成使用逆转录PrimeScriptTMRT regent Kit with gDNA Eraser试剂盒(TaKaRa公司),具体操作按试剂盒说明书操作。接着对尼罗罗非鱼IRF1 基因全长进行克隆。首先,利用GenBank数据库中尼罗罗非鱼IRF1(XM_003451380.3)预测序列设计引物OnIRF1F:TCCTGGAATCAAAGCAAATT,OnIRF1R:TGAGGGTAGCTCTGCAAACT。以对照组尼罗罗非鱼头肾为模板,对IRF1基因进行PCR扩增。PCR 反应条件为:94 ℃预变性 3 min;94 ℃变性 30 s,57 ℃退火30 s,72 ℃延伸 1 min 30 s,35个循环;72 ℃延伸 7 min。扩增产物经 1.0%的琼脂糖凝胶电泳并使用 DNA 凝胶回收试剂盒(Qiagen 公司)割胶回收,回收后的目的片段使用pGEM T-easy Vector (Promega公司)为载体进行连接,连接后转染至DH5α大肠杆菌中培养扩增,后经菌液PCR鉴定,挑选阳性克隆提取质粒并使用EcoR Ⅰ进行酶切,电泳鉴定后,将样品送至上海生工生物工程技术有限公司测序。
1.4序列比对和系统发育分析
获得的IRF1序列使用DNAStar等软件分析序列信息,确定其开放阅读框进而预测氨基酸序列,并且在NCBI网站进行BlastP分析。从 GenBank 下载脊椎动物模式物种的IRF家族蛋白序列,使用ClustalX及MEGA6软件对IRF家族成员蛋白序列进行比对和系统发育分析。用邻接法(Neighbor-Joining)构建系统进化树。MEGA软件中邻接法采用Poission correction替换模型和配对删除法,系统树分支的可信度测试采用100次重复抽样检验。
1.5IRF1在不同组织中的表达和比较
根据尼罗罗非鱼IRF1基因cDNA序列,设计1对基因特异引物qOnIRF1F:AACAGTCGAGATTG GCCCTG,qOnIRF1R:AGAGTTGTGTACTGTGGCCG。以β-actin作为内参基因,引物为qOnactbF:GAATC CTGCGGAATCCACGA,qOnactbR:ATTTACGCTCAG GTGGGGC。20 μL PCR 反应液中包括 2 μL cDNA 模板,10 μL 2 ×Roche SYBR Master Mix,正反向混合引物共0.8 μL (10 μmol/L)和 7.2 μL双蒸水。反应程序为95 ℃预变性5 min;再运行 95 ℃ 5 s,60 ℃ 34 s退火共40 个循环;接着72 ℃延伸5 min,最后绘制熔解曲线,生物学重复每组3个(n=3)。每个样品重复 3管以保证试验的准确性,利用公式 2-ΔΔCt计算出每一个样本IRF1 基因的相对表达量,使用GraphPad Prism 5软件进行显著差异分析和作图。
1.6荧光表达载体构建和细胞定位
使用OnIRF1F和OnIRF1R扩增产物为模板,使用两端包含限制性内切酶位点的引物IRF1-CMVs:GACAAGCTTATGCCAGTGTCAAGA (Hind Ⅲ)和IRF1-CMVa:GGCCTAGGTAAAGCAAAAGTGG (BamH Ⅰ),进行第2轮PCR,再经过克隆验证。提取质粒进行双酶切,和经过同样酶切的p3XFLAG-CMV-10表达载体进行连接。构建重组质粒pCMV3-FLAG-IRF1,进行测序验证。将构建好的载体转染到培养的Hela细胞中,培养后进行免疫荧光染色,主要方法如下:转染48 h后,使用3%的多聚甲醛固定细胞10 min;将固定好的细胞置于含0.2%TritonX-100的PBS溶液通透15 min;之后置于5%的山羊血清37 ℃封闭1 h,接着加入免抗FLAP抗体(1∶200稀释浓度)37 ℃下孵育1 h;再使用FITC标记的抗山羊IgG二抗孵育1 h;使用PBS漂洗3次后,使用4′、6-二脒基-2-苯基吲哚(DAPI)染核1 min,荧光显微镜下观察和拍照。

起始密码子(ATG)、终止密码子(TAA)加粗显示。下划线为DBD结构域区。
2 结果与分析
2.1尼罗罗非鱼IRF1基因序列特征
尼罗罗非鱼IRF1基因克隆测序的结果显示其读码框长度为888 bp,编码295个氨基酸的蛋白序列(图1)。蛋白质分子量是34.15 kDa。氨基酸含量最高的是丝氨酸(Ser,S),占12.0%,最低的是酪氨酸,占2.0%。蛋白序列不含信号肽。
比较斑马鱼、河鲀及尼罗罗非鱼IRF1基因结构图中可以看出,尼罗罗非鱼IRF共有9个外显子和8个内含子,尼罗罗非鱼与河鲀比较接近。而斑马鱼只有8个外显子和7个内含子,内含子区较长。可见,尼罗罗非鱼IRF1基因与河鲀IRF1相比,基因排列基本一致,存在高度保守的基因组共线性关系,上下游包括了ACS16、FNIP1和SLC22A4等基因(图2)。
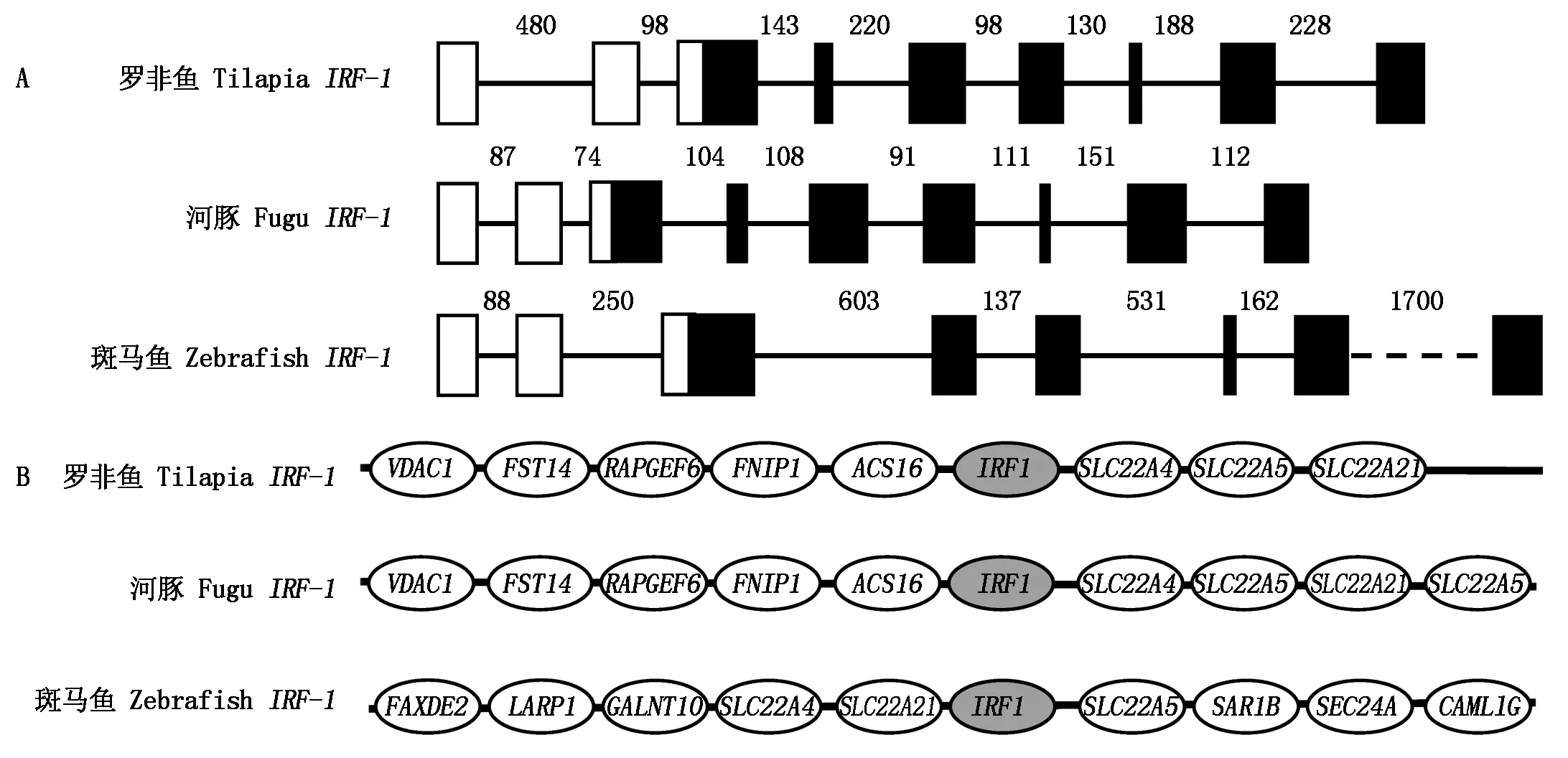
A.罗非鱼、河豚和斑马鱼IRF1基因结构。方框代表外显子,水平线代表内含子,不保守外显子和保守外显子分别用黑色框和白色框表示,数字分别代表核苷酸数目。B.罗非鱼、河豚和斑马鱼IRF1基因在基因组中的共线性分析。
A.Schematic diagram of gene structure ofIRF1 in tilapia,fugu and zebrafish.Exons were expressed as boxes,and introns as lines,black and white boxes indicated conserved and unconserved regions,and the figure means the number of nucleotides;B.Diagram to show gene synteny atIRF1 loci in nile tilapia,fugu and zebrafish.
图2IRF1基因结构和共线性分析
Fig.2Analysis of gene structure and synteny ofIRF1
尼罗罗非鱼和斑马鱼(Daniorerio)、虹鳟(Onchorynchusmykiss)等硬骨鱼类及其他脊椎动物如鸡(Gallusgallus)、大鼠(Rattusnorvegicus)、小鼠(Musmusculus)及人(Homosapiens)IRF1比对结果显示,IRF是一类结构特征上保守的转录因子家族。所有物种IRF1蛋白的N 端约120个氨基酸的同源性较高,越靠近C端,保守性越低。氨基酸的N端包含DBD结构域,而在C端含有IAD2结构域。在尼罗罗非鱼IRF1氨基端DBD结构域内含有 IRF家族特有的色氨酸(Try,W)重复残基,分别为第11,26,38,46,58及77位,共6个(图3)。蛋白同源性分析结果显示,尼罗罗非鱼与斑马鱼的IRF1蛋白序列相似度最高,为52.1%,其次是鲑鱼(44.3%)及虹鳟(42.6%),相似度最小的为大鼠(36.2%)。
选取脊椎动物亚门中的代表物种IRF家族各成员蛋白序列构建系统进化树(图4)。从图4可以看出,13个IRF1蛋白聚集在一起,其中尼罗罗非鱼IRF1 与黄鳝和青鳉IRF1相似性最高,而与亲缘关系较远的鱼和其他脊椎动物IRF1相似性较低。其他IRF也基本聚集成单独的家族,最后又分别聚为4大亚群:一支包含所有IRF1和IRF2;一支由IRF4、IRF8和IRF9组成;一支由IRF5和IRF6组成;一支由IRF3和IRF7组成。
2.2尼罗罗非鱼IRF1 mRNA的组织特异性表达及刺激后的表达变化
尼罗罗非鱼IRF1在脑、鳃、肝、脾和头肾中均有表达。脾脏及鳃中IRF1表达量较高,而在脑中的表达量比其他组织低。尼罗罗非鱼腹腔注射PolyI∶C 12 h后,除脑无显著差异外,其他各组织的对照组和处理组均有显著差异。差异最大的为肝脏,表达量约增加12倍。在处理24 h后,头肾及脾脏中IRF1表达水平仍有显著上调变化。在处理24 h后的各个组织中,IRF1表达水平均出现显著上调(图5)。
实线方框显示DBD结构域,其中包括高度保守的6个色氨酸;虚线方框代表IAD2结构域。
2.3IRF1表达载体的构建及免疫荧光染色定位
构建真核表达重组质粒pCMV3-FLAG-IRF1,转染Hela细胞并进行荧光染色,转染重组质粒pCMV3-FLAG-IRF1的细胞能够检测到绿色荧光,说明IRF1重组蛋白能够在细胞中正常表达,荧光主要定位在细胞质中,细胞核中也能检测到少量绿色荧光蛋白。这表明尼罗罗非鱼IRF1主要在细胞质中表达(图6)。
3 讨论
尼罗罗非鱼作为重要的经济养殖品种,近10年在我国的产量以平均每年9%左右的速度递增,稳居世界首位。其养殖产量约占世界总产量50%。但随着尼罗罗非鱼养殖规模扩大,各种传染性疾病的发生呈现不断增长的趋势,尤其是近年来链球菌病和一些病毒性疾病的大范围流行,给尼罗罗非鱼养殖产业造成了严重的经济损失[11]。因此,对于尼罗罗非鱼免疫学的研究越来越受到重视。
随着尼罗罗非鱼基因组信息的逐步完善,通过生物信息学方法能获得大量注释的编码基因,包括IRF家族的成员。但是相关结果仍然需要进一步的试验验证,但尼罗罗非鱼IRF1基因的功能研究也未见报道。通过将尼罗罗非鱼与其他脊椎动物的IRF1氨基酸序列进行多重比对后发现,IRF1家族蛋白序列比较保守。特别是氨基端有高度保守的DBD结构域,并含有6个色氨酸残基,说明尼罗罗非鱼IRF1蛋白序列拥有保守的二级结构特征。在羧基端,IRF1和IRF2具有与其他成员不同的IAD2结构域。尽管IRF1和IRF2序列比较接近,但是在干扰素转录调节及促细胞生长方面具有不同的效应[13-14]。IRF1具有激活干扰素转录功能,而IRF2通过与IRF1竞争相同的顺式调控元件而发挥相反的抑制作用[15]。通过来自脊椎动物的IRF家族基因蛋白序列系统进化树显示,IRF1~IRF 9都可以单独聚为1支。而IRF家族又可分为四大支:IRF1 亚家族;IRF4亚家族;IRF5亚家族和IRF3亚家族。结果表明IRF在分化中的时间上先后不同。
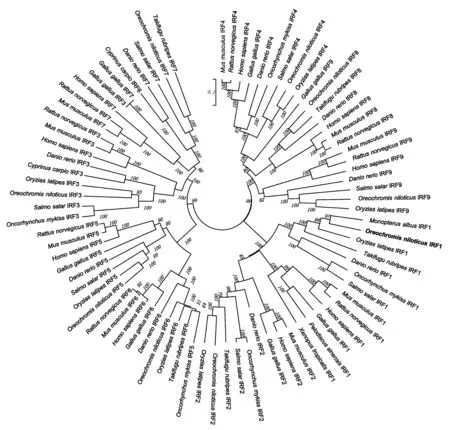
系统树构建使用MEGA 6.0软件,根据Neighbor-joining法进行计算,自举检测100次重复检验,蛋白序列来自GenBank数据库。
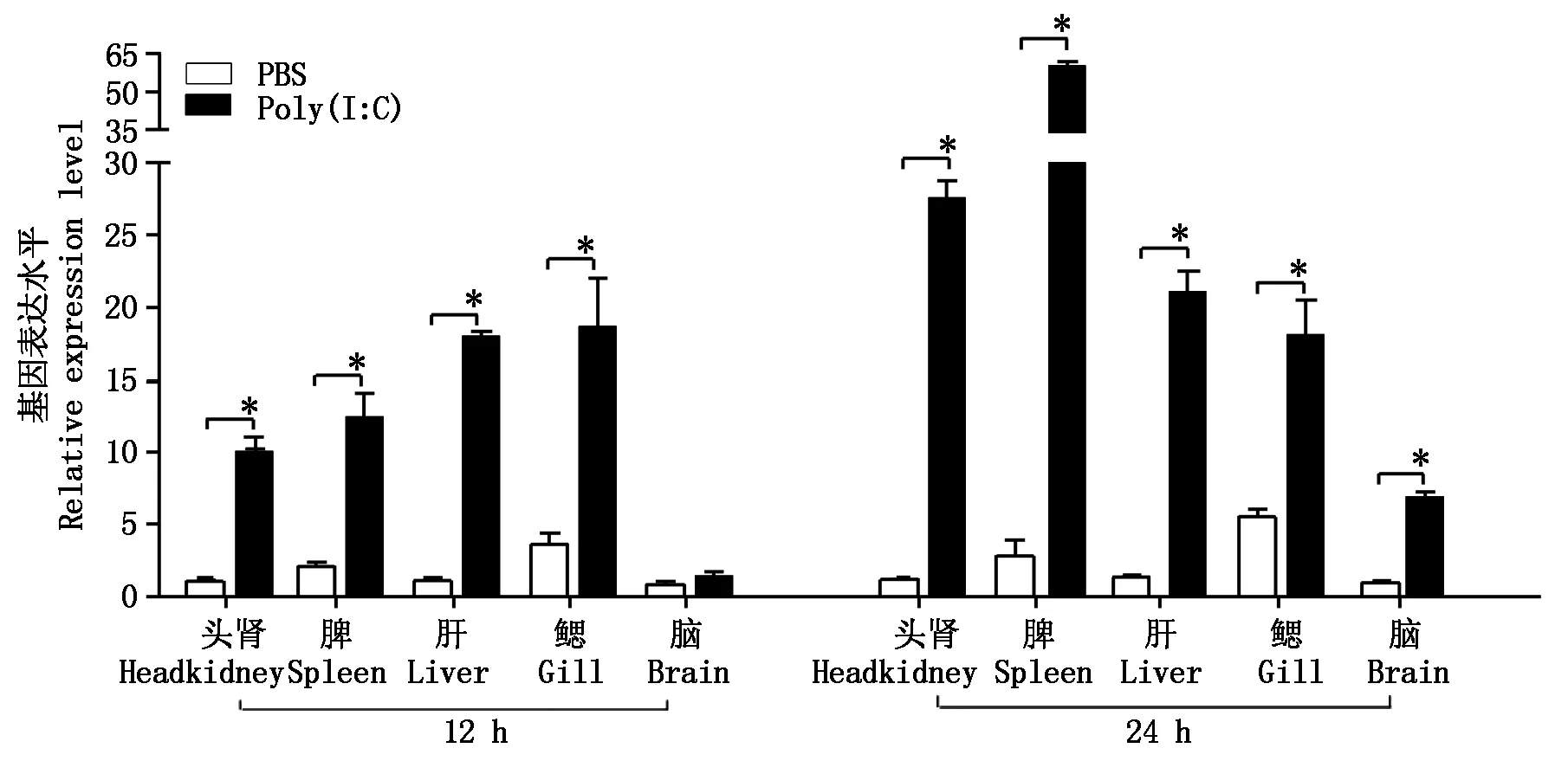
IRF1基因表达量计算以β-actin作为内标,每组至少3条鱼,每组计算mean±SEM;*.P<0.05。
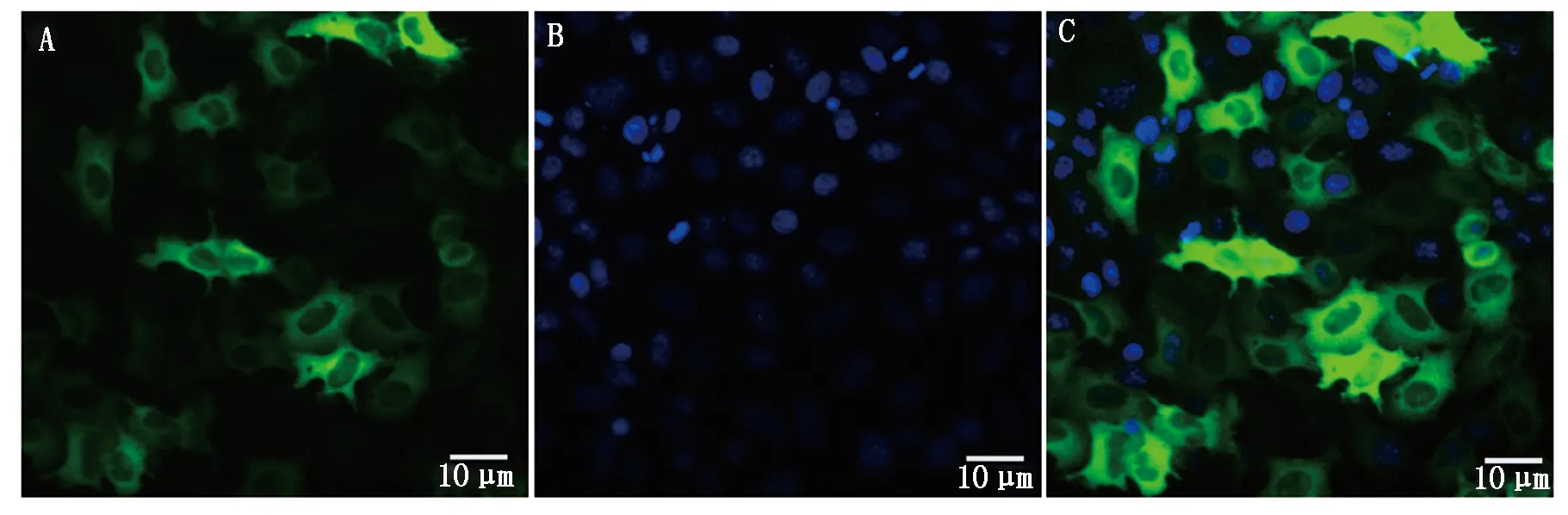
A.IRF1在细胞中表达的荧光照片;B.细胞核DAPI染色;C.IRF1荧光染色和DAPI染色图像重合。
作为低等脊椎动物,鱼类具有固有免疫和适应性免疫[16]系统。由于鱼类适应性免疫系统的不完善,固有免疫系统在鱼类整个免疫系统中占十分重要的地位。固有免疫利用模式识别受体(Pattern recognition receptors,PRRs)对细菌、病毒等病原体中保守结构、病原体相关分子模式(Pathogen associated molecular patterns,PAMPs)进行识别,从而引起免疫反应[17]。在识别PAMPs的过程中,Toll样受体(Toll-like receptors,TLRs)是研究最多的模式识别受体之一,能够诱导I型干扰素的表达,从而使机体形成抗病毒状态。I型IFN系统在固有免疫及适应性免疫系统中扮演重要的角色,能参与抗病毒免疫反应,抑制肿瘤细胞调节,巨噬细胞活性及T、B淋巴细胞增殖[18-20]。而这些过程离不开IRF家族成员的参与。IRF家族为IFN重要的转录调控因子。关于哺乳动物IRF家族功能的研究表明,IRF1、IRF3、IRF5和IRF7能够参与I型干扰素的正反馈调节,以此发挥抗病毒作用[21]。如在哺乳动物TLR信号通路中,当机体受到病毒感染时,细胞质中的IRF3受到磷酸化并向细胞核转移,以激活IFN-beta的低量表达。而IFN-beta又能促进IRF7的大量表达,从而IRF3和IRF7的大量表达激活I型干扰素及抗病毒基因的表达。在鱼类中已有十几种鱼类IRF1的研究,包括虹鳟、河鲀、斑马鱼及草鱼等硬骨鱼类。经聚肌胞苷酸刺激的鱼类,其各个组织中IRF1的表达量会出现显著上调[22-24]。在草鱼抗病毒感染的过程中,IRF3在受到刺激后能大量表达,从而诱导IFN和ISG的转录[25]。对斑马鱼的研究显示,IRF1与其他IRF家族成员相比,在免疫器官中的表达量最高,并且病毒感染后的IRF1表达变化最为显著[7]。对牙鲆IRF1的研究发现,IRF1能够使机体形成抗病毒状态[15]。因此,在牙鲆的养殖过程中,IRF1可以作为一种DNA疫苗辅剂而增强鱼体抗病能力。本试验结果显示了尼罗罗非鱼各种组织能表达IRF1,其表达量也会随着组织种类的不同而不同。试验利用荧光定量PCR技术检测尼罗罗非鱼各组织中IRF1表达量,发现IRF1基因在脾脏和鳃中表达量较高,脑中的表达水平较低。同时利用聚肌胞苷酸PolyI∶C模拟病毒刺激,促使尼罗罗非鱼产生抗病毒免疫反应。结果显示,各个组织中IRF1的表达量会出现显著上调趋势。可见,IRF1对病毒刺激有相同的反应模式,其功能具有保守性。
对哺乳动物IRF1细胞定位的研究发现,IRF1主要存在于细胞核中,少量在细胞质中[26-27]。而斑马鱼IRF1在正常状态下主要存在于细胞质中,经过刺激后会聚集在细胞核中[7]。为了对尼罗罗非鱼IRF1进行细胞定位,本试验构建IRFl融合蛋白的真核表达载体pCMV3-FLAG-IRF1。转染到Hela细胞进行细胞定位,这种方法可以方便地使用抗flag的抗体来识别目的蛋白,有利于目的蛋白检测和分离纯化[28]。免疫荧光染色结果显示,尼罗罗非鱼IRF1蛋白能在Hela细胞中表达,而且在细胞质中表达更多,在细胞核中仅存在少量表达。结果和斑马鱼IRF1的研究结果比较接近,和哺乳动物结果有一定差异,推测与表达的蛋白种类和选择的细胞类型有关系。本试验对尼罗罗非鱼IRF1基因进行克隆,系统分析和细胞定位,初步探讨了其在抗病毒中的组织表达和反应模式。这些结果将有助于更深入了解脊椎动物干扰素表达调控的特点,为尼罗罗非鱼抗病研究提供基础理论依据。
[1]Strausberg R L,Feingold E A,Grouse L H,et al.Generation and initial analysis of more than 15 000 full-length human and mouse cDNA sequences[J].Proceedings of the National Academy of Sciences of the United States of America,2002,99(26):16899-16903.
[2]Shu C,Sun Y,Xu T.Molecular characterization of threeIRF1 subfamily members reveals evolutionary significance ofIRF11 in miiuy croaker[J].Developmental and Comparative Immunology,2015,53(2):385-391.
[3]沈金花,吕印,刘庆华.干扰素调节因子的研究进展[J].中南民族大学学报:自然科学版,2014,33(2):41-44.
[4]Tamura T,Yanai H,Savitsky D,et al.The IRF family transcription factors in immunity and oncogenesis[J].Annual Review of Immunology,2008,26(1):535-584.
[5]Inkpen S M,Hori T S,Gamperl A K,et al.Characterization and expression analyses of five interferon regulatory factor transcripts (IRF4a,IRF4b,IRF7,IRF8,IRF10) in Atlantic cod (Gadusmorhua)[J].Fish & Shellfish Immunology,2015,44(1):365-381.
[6]Foss G S,Prydz H.Interferon regulatory factor 1 mediates the interferon-gamma induction of the human immunoproteasome subunit multicatalytic endopeptidase complex-like 1[J].The Journal of Biological Chemistry,1999,274(49):35196-35202.
[7]Feng H,Zhang Y B,Zhang Q M,et al.ZebrafishIRF1 regulates IFN antiviral response through binding to IFNø1 and IFNø3 promoters downstream of MyD88 signaling[J].Journal of Immunology,2015,194(3):1225-1238.
[8]Lai Q,Lin G,Ma M,et al.IRF-1 acts as a positive regulator in the transcription of grass carp (Ctenopharyngodonidella)IFNgene[J].Fish & Shellfish Immunology,2013,34(6):1432-1438.
[9]Yabu T,Hirose H,Hirono I,et al.Molecular cloning of a novel interferon regulatory factor in Japanese flounder,Paralichthysolivaceus[J].Molecular Marine Biology and Biotechnology,1998,7(2):138-144.
[10]林勇,卢其西,杨慧赞,等.八种品系罗非鱼及其尼奥罗非鱼耐寒性能的比较试验[J].华北农学报,2011,26(S1):278-282.
[11]郭玉娟,张德锋,樊海平,等.中国南方地区罗非鱼无乳链球菌的分子流行病学研究[J].水产学报,2012,36(3):399-406.
[12]朱佳杰,周宇,沈夏霜,等.吉富品系尼罗罗非鱼抗病性状分子标记筛选及遗传多样性分析[J].华北农学报,2013,28(S1):16-21.
[13]Choo A,Palladinetti P,Passioura T,et al.The role ofIRF1 andIRF2 transcription factors in leukaemogenesis[J].Current Gene Therapy,2006,6(5):543-550.
[14]Ren G,Cui K,Zhang Z,et al.Division of labor betweenIRF1 andIRF2 in regulating different stages of transcriptional activation in cellular antiviral activities[J].Cell & Bioscience,2015,5(1):17.
[15]Christopher M C,Ikuo H,Takashi A.Induction of antiviral state in fish cells by Japanese flounder,Paralichthysolivaceus,interferon regulatory factor-1[J].Fish & Shellfish Immunology,2005,19(1):79-91.
[16]Plouffe D A,Hanington P C,Walsh J G,et al.Comparison of select innate immune mechanisms of fish and mammals[J].Xenotransplantation,2005,12(4):266-277.
[17]Netea M G,Van Der Meer J W.Immunodeficiency and genetic defects of pattern-recognition receptors[J].The New England Journal of Medicine,2011,364(1):60-70.
[18]Bouker K B,Skaar T C,Riggins R B,et al.Interferon regulatory factor-1 (IRF-1) exhibits tumor suppressor activities in breast cancer associated with caspase activation and induction of apoptosis[J].Carcinogenesis,2005,26(9):1527-1535.
[19]Schoggins J W,Wilson S J,Panis M,et al.A diverse range of gene products are effectors of the type I interferon antiviral response[J].Nature,2011,472(7344):481-485.
[20]Xu H C,Grusdat M,Pandyra A A,et al.Type I interferon protects antiviral CD8+ T cells from NK cell cytotoxicity[J].Immunity,2014,40(6):949-960.
[21]Honda K,Taniguchi T.IRFs:master regulators of signalling by Toll-like receptors and cytosolic pattern-recognition receptors[J].Nature Reviews Immunology,2006,6(9):644-658.
[22]Collet B,Hovens G C,Mazzoni D,et al.Cloning and expression analysis of rainbow troutOncorhynchusmykissinterferon regulatory factor 1 and 2 (IRF-1 andIRF-2)[J].Developmental and Comparative Immunology,2003,27(2):111-126.
[23]Ordás M C,Abollo E,Costa M M,et al.Molecular cloning and expression analysis of interferon regulatory factor-1 (IRF-1) of turbot and sea bream[J].Molecular Immunology,2006,43(7):882-890.
[24]Sun B J,Chang M X,Song Y,et al.Gene structure and transcription ofIRF-1 andIRF-7 in the Mandarin fishSinipercachuatsi[J].Veterinary Immunology and Immunopathology,2007,116(1/2):26-36.
[25]Sun F,Zhang Y B,Liu T K,et al.Characterization of fishIRF3 as an IFN-inducible protein reveals evolving regulation of IFN response in vertebrates[J].Journal of Immunology,2010,185(12):7573-7582.
[26]Andersen P,Pedersen M W,Woetmann A,et al.EGFR induces expression ofIRF-1 via STAT1 and STAT3 activation leading to growth arrest of human cancer cells[J].International Journal of Cancer,2008,122(2):342-349.
[27]Moschonas A,Kouraki M,Knox P G,et al.CD40 induces antigen transporter and immunoproteasome gene expression in carcinomas via the coordinated action of NF-kappaB and of NF-kappaB-mediated de novo synthesis ofIRF-1[J].Molecular and Cellular Biology,2008,28(20):6208-6222.
[28]梁芸,魏凤菊.拟南芥 CPK10双元 TAP 载体的构建及原生质体表达[J].华北农学报,2015,30(4):43-46.
Expression Characterization of Interferon Regulatory Factor 1 in Nile Tilapia
WANG Dan,XIE Wenjie,ZHU Yefei,ZHAO Jinliang,CHEN Xiaowu
(Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources,Shanghai Ocean University,Ministry of Education,Shanghai201306,China)
In order to study the expression and function of interferon Regulatory Factor (IRF1) in nile tilapia,the full length cDNA ofIRF1 was cloned and applied for phylogenetic analysis,and the expression variation was checked by qRT-PCR after PolyI∶C stimulation.Cell transfection by eukaryotic expression vector was used to analyzing for subcellular localization.The opening reading frame of nile tilapiaIRF1 contains 888 bp with deduced protein of 295 amino acids.Structurally,nile tilapia IRF1 protein typically shares the conserved characterizations with other species′ IRF1 homologues,displaying conserved DNA-binding domain,IRF association domain,serine-rich C terminal domain,and 6 tryptophan residues in the N terminal.The results of phylogenetic analysis showed that the IRF family was divided into four subfamily:IRF1,IRF3,IRF4 and IRF5.The nile tilapia IRF1 was found to be merged into the teleost subgroup,amphibian and mammalian homologues were clustered into their corresponding subgroups.Real-time PCR revealed a broad expression pattern of nile tilapiaIRF1 in various tissues.The expression ofIRF1 was mainly in the head kidney,spleen,liver and gill,furthermore,which increased obviously after PolyI∶C stimulation,especially for the spleen and head kidney.Subcellular localization analysis showed that nile tilapia IRF1 mainly resides in the cytoplasm.These data supported the view that nile tilapia IRF1 was a potential molecule in IFN immune defense system against viral infection.
Interferon regulatory factor;Nile tilapia;Phyletic evolution;Subcellular localization
2016-02-25
现代农业产业技术体系专项(CARS-49);水产动物遗传育种中心上海市协同创新中心 (ZF1206)
王丹(1991-),女,安徽淮南人,在读硕士,主要从事鱼类免疫学研究。
陈晓武(1976-),男,安徽六安人,副教授,博士,主要从事水生生物分子遗传学研究。
Q785;S917
A
1000-7091(2016)04-0031-08
10.7668/hbnxb.2016.04.006
