干酪中肠球菌种群的遗传差异及万古霉素抗性基因分析
2016-09-19倪永清
于 洋,倪永清*
(石河子大学食品学院,新疆石河子832000)
干酪中肠球菌种群的遗传差异及万古霉素抗性基因分析
于洋,倪永清*
(石河子大学食品学院,新疆石河子832000)
通过种、属引物特异性扩增、重复序列PCR(rep-PCR)技术和万古霉素抗性基因检测对新疆北疆地区干酪样品中球菌的遗传结构差异进行分析。结果表明,15份样品共分离52株肠球菌,包括31株耐久肠球菌(Enterococcusdurans)、18株粪肠球菌(Enterococcusfaecalis)和3株屎肠球菌(Enterococcusfaecium)。依据rep-PCR遗传指纹带谱分析,52株菌可以聚类成7个群,其中4个由E.durans构成。24株肠球菌检测到了万古霉素抗性基因,17株E.durans为VanC2/C3型,4株E.faecalis为VanC1型,2株E.faecium为VanB型,只有1株E.faecium为VanA型。新疆北疆地区干酪中肠球菌种群分布较为广泛,地域之间优势种群和基因型不同,同种肠球菌菌株之间存在广泛的遗传差异。
干酪;肠球菌;重复序列PCR;万古霉素
肠球菌(enterococci)属于乳酸菌(lactic acid bacteria,LAB),是一类共生于人和其他哺乳动物肠道的革兰氏阳性菌,也是人和哺乳动物肠道菌群的重要组成。最早分离的菌株大多来自人和哺乳动物肠道,陆续研究发现肠球菌广泛分布于自然界的其他生态环境中,如健康人体的上呼吸道、口腔,甚至昆虫体内[1]。除此之外,肠球菌更广泛的存在于各种食品中,如干酪、牛奶、肉制品和蔬菜等[2]。
我国新疆北疆地区气候季节变化明显,有辽阔的天然牧场,生活在该地区的哈萨克族和维吾尔族等少数民族世代以原奶制作各种传统发酵乳制品,其中干酪(维语“库鲁特”)是生活在牧区的牧民最常见的食物。到目前为止,虽然关于新疆地区乳制品中乳酸菌多样性的研究报道较多[3-6],但针对专一属的同一类乳酸菌深入开展群体表型和遗传差异的研究很少。此外,由于人类对抗生素的滥用,人们非常关注栖息于人体的肠球菌耐药性及其致病性,对其遗传结构差异的研究非常深入,其中万古霉素抗性肠球菌(vancomycin-resistant enterococci,VRE)可携带相关抗性基因通过食物链进入人体,大大增强了致病风险而更加备受关注[7]。与此相比较,对于栖息于乳制品中的肠球菌,作为非主流的乳酸菌,对它们的遗传多样性和代谢特征、耐药性研究报道相对较少。
本研究对新疆北疆3个地区5个县的15份干酪样品中的肠球菌进行了分离筛选,在种属特异引物鉴定的基础上,采用基于聚合酶链反应(polymerase chain reaction,PCR)的两种指纹分型技术和万古霉素抗性基因检测,对不同地域来源干酪中肠球菌的种群分布和遗传差异进行了分析,为开发具有特殊益生功能的、安全的肠球菌菌种资源,加工制作不同风味、不同感官品质的肉、乳制品的生产实践提供参考依据。
1 材料与方法
1.1材料与试剂
1.1.1样品来源及模式菌株
样品采集自新疆北疆地区5个县,即伊宁县、昭苏县、尼勒克县、哈巴河县及和丰县,每个采样区各3份样品,共15份。贴上标签,置于4℃车载冰箱内,运回实验室,保存于-20℃。模式菌株粪肠球菌(Enterococcus faecalis)CGMCC 1.2135T、屎肠球菌(Enterococcus faecium)CGMCC 1.2136T、耐久肠球菌(Enterococcus durans)CGMCC 1.2489T、海氏肠球菌(Enterococcus hirae)CGMCC1.2490T和鸟肠球菌(Enterococcus avium)CGMCC 1.2505T购于中国普通微生物菌种保藏管理中心(ChinaGeneralM icrobiologicalCulture Collection Center,CGMCC);万古霉素抗性菌株粪肠球菌(E.faecalis)ATCC 700802、鹑鸡肠球菌(Enterococcus gallinarum)ATCC 49573T和铅黄肠球菌(Enterococcus cas seliflavu)ATCC 25788T购于美国典型培养物保藏中心(American for Type Culture Collection,ATCC)。
1.1.2培养基
MRS培养基、胆汁七叶灵苷叠氮钠(bile esculin azide,BEA)培养基:青岛高科技园海博生物技术有限公司。
1.1.3主要试剂
PCR反应试剂:生工生物工程(上海)有限公司;扩增引物:上海捷瑞生物工程有限公司;脱氧核糖核酸(deoxyribonucleic acid,DNA)、不同分子质量Marker:北京全式金生物技术(TransGen Biotech)有限公司。
1.2仪器与设备
5810R高速冷冻离心机:德国Eppendorf公司;TC-512 PCR扩增仪:英国Techne公司;PowerPacUniversal水平电泳仪、SUBCELLGT(20 cm×25 cm)电泳槽:美国Bio-Rad公司;QUANTUM-ST5凝胶成像系统:法国Vilber Lourmat公司。
1.3方法
1.3.1菌株的分离与纯化
每份样品取10 g加入190 m L无菌水于37℃摇床振荡1 h制成样品菌悬液。采用梯度稀释法稀释至10-5,取0.2 m L 10-3、10-4、10-5的稀释液分别涂布于MRS和BEA选择性琼脂培养基上,置于37℃恒温培养箱中,培养48 h。根据菌落颜色、形态、大小进行初步筛选,对疑似菌落继续进行纯化培养,纯化2~3次,直至为单一菌落。然后对单菌落进行革兰氏染色以及接触酶试验[8]进一步筛选疑似菌株。将筛选出的菌株液体纯培养物补充20%的灭菌甘油,冷冻保存于-80℃冰箱。
1.3.2菌株DNA的提取
参考ATUL K S等[9]提取菌株DNA的方法,略有改动。1 m L菌株纯培养物5 000 r/m in离心3 m in,弃上清液,用磷酸缓冲液(pH 7.4)洗涤3次,无菌水洗涤1次;加入0.5 m L 6mol/L尿素和0.1m L 10%十二烷基硫酸钠(sodium dodecyl sulfate,SDS)重悬;37℃水浴20min,沸水浴5min;8000 r/min离心10 m in,弃上清液,加入0.1 m L 0.2 mol/L NaOH溶液37℃水浴10 min;3 000 r/m in离心3 min,吸取上清液,加入2.5倍体积的无水乙醇-20℃条件下静置2 h;4℃条件下12 000 r/min离心15min,体积分数为70%预冷酒精洗涤2次,最后溶解于0.1 m L TE缓冲溶液中。
1.3.3肠球菌种属特异引物鉴定
先采用肠球菌属特异引物(Ent1/Ent2)[7]扩增疑似菌株确定其为肠球菌属,模式菌株作为阳性对照,再采用种特异引物(Fk1/Fk2、Fae1/Fae2、Dur1/Dur2、Hi1/Hi2和Av1/Av2)[7]进行多重PCR扩增,确定其具体种。属扩增PCR反应程序为:变性96℃、3 min;变性95℃、1 min,退火55℃、30 s,延伸72℃、1 min,35个循环,终延伸7 min。种扩增PCR反应程序为:变性96℃、3 m in;变性95℃、1 m in,退火60℃、30s,延伸72℃、1 min,35个循环,终延伸7 min。PCR反应体系详见参考文献[7]。
1.3.4rep-PCR分型
指纹图谱所采用的引物及PCR反应条件如表1所示,取10 μL扩增产物用1.5%琼脂糖凝胶、0.5×Tris硼酸电泳缓冲液(TBE),在160 V条件下电泳2 h 15 min,凝胶用溴化乙锭(ethidium bromide,EB)染色30 min,于凝胶成像系统观察。对所得基因组重复序列聚合酶链式反应(repetitive extragenic palindromic polymerase chain reaction,rep-PCR)指纹图谱采用GelCompar II 5.10软件进行分析,相似性分析采用Pearson系数,使用UPGMA输出树状图。

表1 rep-PCR引物和PCR反应条件Table 1 Primers of rep-PCR and PCR reaction conditions
1.3.5万古霉素抗性基因扩增
采用多重PCR来鉴定万古霉素抗性肠球菌(VRE),引物和反应体系详见文献[7],菌株E.faecium BM 4147(van A)、E.faecalis ATCC 700802(van B)、E.gallinarum ATCC 49573T(van C1)、E.casseliflavu ATCC 25788T(van C2/C3)作为阳性对照。扩增PCR反应程序为:变性94℃、3min;变性94℃、1 min,退火56℃、1 min,延伸72℃、1 min,30个循环,终延伸5 min。
2 结果与分析
2.1菌株分离鉴定及种群分布
利用MRS和BEA选择性琼脂培养基对干酪中肠球菌进行初筛,依据菌落大小、形态和颜色,油镜观察以及革兰氏染色和接触酶试验对菌株进行筛选,呈现球状,革兰氏阳性,接触酶阴性的疑似菌株87株。图1显示了部分菌株扩增结果,经属特异性引物鉴定,有52株菌株鉴定结果显示为阳性(图1a)属特异引物扩增产物大小为112 bp),再利用种特异性引物鉴定,52株肠球菌分别隶属于3个种,耐久肠球菌(E.durans)31株、粪肠球菌(E.faecalis)18株和3株屎肠球菌(E.faecium)(图1b)耐久肠球菌(E.durans)特异引物扩增产物大小为295 bp,粪肠球菌(E.faecalis)特异引物扩增产物大小为941 bp,屎肠球菌(E.faecium)特异引物扩增产物大小为550 bp)。其中伊宁县样品9株(9株E.durans),昭苏县样品13株(3株耐久肠球菌(E. durans),9株粪肠球菌(E.faecalis),1株屎肠球菌(E.faecium),尼勒克县样品12株(2株耐久肠球菌(E.durans),9株粪肠球菌(E.faecalis),1株屎肠球菌(E.faecium)),哈巴河县10株(10株均为耐久肠球菌(E.durans)),和丰县8株(7株耐久肠球菌(E.durans),1株屎肠球菌(E.faecium)。

图1 部分菌株属特异引物(a)及种特异引物(b)扩增产物电泳图Fig.1 Am plification products electrophoregram of genus specific primers(a)and species specific primers(b)of partial strains
不同取样点的干酪中肠球菌种群的分布情况见图2。由图2可知,伊宁县、哈巴河县及和丰县样品中的优势种为耐久肠球菌(E.durans),而昭苏县和尼勒克县样品的优势种为粪肠球菌(E.faecalis)。IRIGOYEN A等[7,11-12]研究发现干酪中粪肠球菌(E.faecalis)和屎肠球菌(E.faecium)是常见的优势种,也有研究表明屎肠球菌(E.faecium)和耐久肠球菌(E.durans)是干酪中的优势种[13]。结果表明,在完全成熟的干酪中粪肠球菌(E.faecalis)和屎肠球菌(E.faecium)占据优势,而耐久肠球菌(E.durans)则在未完全成熟的干酪中大量存在[14-15]。

图2 各采样点肠球菌种群的分布Fig.2 Species d istribution o f enterococci at various sam p ling points
2.2rep-PCR分型结果
为了得到准确的分析,对所得52株肠球菌的rep-PCR指纹图谱采用GelCompar II 5.10凝胶分析软件进行分析,输出树状图,结果如图3所示。由图3可知,引物BOXAIR扩增产物的大小在300~3 000 bp之间,条带数为2到16不等,大多数产物的条带数都多于4条。引物(GTG)5扩增产物的大小在100~5 000 bp之间,条带数为3到20不等,大多数产物的条带数在10条以上。
由图3可知,52株肠球菌大约在40%的相似水平上被聚类为7群(Cluster),ClusterⅠ为5株耐久肠球菌(E.durans),带型相近;ClusterⅡ仅仅包括带型极为相近的2株屎肠球菌(E.faecium);ClusterⅢ包括4株耐久肠球菌(E.durans)和1株屎肠球菌(E.faecium),4株耐久肠球菌(E.durans)中,菌株HF3-B-1、HF3-R-6和HF3-R-3的带型更相似,菌株ZS2-R-4的带型与它们相差较大,尤其是BOXAIR引物扩增图谱中比较明显,而另1株屎肠球菌(E.faecium)HF3-R-2也被划分在这一组中,与另外2株屎肠球菌(E.faecium)不同,预示了该菌株独特的遗传结构;ClusterⅣ由15株耐久肠球菌(E.durans)组成,在45%的相似性水平上可进一步分为三小组,其中菌株NLK3-R-2与其它菌株的带型相差很大,独自成1组,菌株YN3-B-4是第二小组中差异最大的菌株;ClusterⅤ由16株带型相近粪肠球菌(E.faecalis)组成,在45%的相似性水平上仍然可分为3小组,其中菌株NLK 3-B-4两种指纹带谱与其他菌株明显不同,遗传差异较大;ClusterⅥ仅有2株E.faecalis组成,与划分在ClusterⅤ的所有其他粪肠球菌(E.faecalis)菌株相比,BOXAIR指纹带谱差异更显著,显示了明显的种内遗传差异;耐久肠球菌(E.durans)显示了最大种内遗传差异,ClusterⅦ也由7株耐久肠球菌(E.durans)构成,但与隶属于前四组的耐久肠球菌(E.durans)相比,带谱差异极其明显,其中HBH3-R-2是本组遗传差异最大的菌株。带型统计显示,31株耐久肠球菌(E.durans)有10种带型,18株粪肠球菌(E.faecalis)有3种带型,3株屎肠球菌(E.faecium)有2种带型。

图3 52株肠球菌基于rep-PCR扩增的聚类图及万古霉素抗性肠球菌鉴定结果Fig.3 Dendrogram of 52 strains of enterococci based on rep-PCR am plification and identification results of enterococci w ith vancomycin-resistance
rep-PCR分型结果显示3个肠球菌种间具有明显的差异,种内带型多样,存在极高的遗传多态性,与DALBB等[16]的研究结果一致。屎肠球菌(E.faecium)和耐久肠球菌(E.durans)两个种间的系统发育关系较粪肠球菌(E.faecalis)更为接近,但作为优势种群的耐久肠球菌(E.durans)表现出更大的遗传多态性。指纹图谱以及综合实验结果发现,引物(GTG)5产生的指纹更清晰、条带数有较高的多样性,扩增效果明显优于引物BOXAIR的扩增效果。因此,(GTG)5更适合于Enterococcus的遗传差异研究,与VANCANNEYT M等[17]的研究结论基本一致。
2.3万古霉素抗性基因肠球菌的鉴定
本研究分离的52株肠球菌,采用4种万古霉素抗性基因特异引物检测到其中24株肠球菌携带有万古霉素抗性基因,包括4种基因型,其中VanC2/C3型出现在17株耐久肠球菌(E.durans)中,这17株菌均来自耐久肠球菌(E.durans)构成的ClusterⅠ、Ⅲ和Ⅳ,而同样由耐久肠球菌(E.durans)菌株组成的ClusterⅦ中没有检测到(见图3)。在4株粪肠球菌(E.faecalis)中检测到VanC1型,而隶属于ClusterⅡ的2株屎肠球菌(E.faecium)为VanB型,仅有屎肠球菌(E.faecium)HF3-R-2检测到vanA基因(见图3)。部分菌株万古霉素抗性基因扩增产物电泳图见图4。由图4可知,van A基因扩增产物大小为732 bp;van B基因扩增产物大小为635 bp,van C基因扩增产物大小为822 bp,van C2/C3基因扩增产物大小为438 bp。
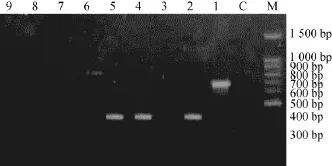
图4 部分菌株万古霉素抗性基因扩增产物电泳图Fig.4 Am plification products electrophoregram of vancomycinresistance genes of partia l strains
2株屎肠球菌(E.faecium)含有基因van B,1株屎肠球菌(E.faecium)含有基因van A,而在干酪中分布广泛的耐久肠球菌(E.durans)和粪肠球菌(E.faecalis)种群均没有检测到这两种抗性基因。迄今为止,中国的临床环境中万古霉素抗性肠球菌普遍存在[18],但是养殖动物中很少报道万古霉素抗性肠球菌的存在。虽然可以认为新疆地区干酪中的肠球菌有可能来自养殖动物,但是干酪大部分是家庭作坊式手工加工,来自加工者自身的肠球菌也有可能进入干酪,而且可能携带万古霉素抗性肠球菌的概率更高。本研究从干酪中筛选到的屎肠球菌(E.faecium)菌株较少,但都携带有万古霉素抗性基因,因此实验室后期对于屎肠球菌(E.faecium)将会进一步关注,分离到更多的菌株,以便更全面地评价干酪、人体和动物原奶中肠球菌的安全性,为科学评价近年来我国畜牧养殖业中滥用抗生素引起的耐药基因扩散问题提供理论参考依据。
3 结论
与食品相关的乳酸菌中,肠球菌是最受争议的一类,最近越来越受到大家的关注,成为国内外研究的热点。本研究从新疆北疆地区5个县的干酪样品中分离培养肠球菌,采用属、种特异引物扩增鉴定最终确定52株肠球菌,分别属于耐久肠球菌(E.durans)、粪肠球菌(E.faecalis)和屎肠球菌(E.faecium)这三个种。根据rep-PCR分型结果,52株菌可以聚类成7个群,其中4个由E.durans构成。万古霉素耐药基因检测结果显示,24株肠球菌含有万古霉素耐药基因,分属4种基因型。
新疆北疆地区干酪中肠球菌种群分布较为广泛,地域之间优势种群和基因型不同,同种内肠球菌菌株之间存在比较广泛的遗传差异。在24株肠球菌中检测到万古霉素抗性基因,且在两株E.faecium中检测到含有van B,1株E.faecium含有van A,这是国内首次报道干酪中肠球菌检测到有van A和van B抗性基因,本实验室后续会做进一步研究。
[1]DEVRIESE L A,COLLINS M D,WIRTH R.The Genus Enterococcus. In:BALLOWS A,TRQPER HG,DWORKIN M,et al(eds)The Prokaryotes[M].2nd edn.New York:Springer-Verlag,1992:1465-1478.
[2]BEN ON,CASTRO A,LUCAS R,et al.Functional and safety aspects of enterococci isolated from different Spanish foods[J].Syst App ld M icrobiol,2004,27(5):118-130.
[3]赵蕊,霍贵成.新疆酸奶子中乳酸菌多样性分析[J].山东大学学报,2008,43(7):27-31.
[4]蓝晓雪,胡晓晴,胡敏,等.新疆酸乳中益生菌的筛选与鉴定[J].中国乳品工业,2012,40(11):16-20.
[5]杨洁,张文亮,邹建军,等.新疆传统酸奶中乳酸菌的筛选鉴定及菌相分析[J].食品工业科技,2015,36(1):324-333.
[6]袁雪林,杨洁,胡敏,等.新疆喀什地区传统发酵酸乳中乳酸菌多样性的初步分析[J].食品工业科技,2015,36(10):202-204.
[7]IRIGOYEN A,URDIN M,GARCíA S,et al.Sources of enterococci in Idiazábal-type cheese[J].Int J Food M icrobiol,2008,125(3):146-152.
[8]东秀珠,蔡妙英.常见细菌系统鉴定手册[M].北京:科学出版社,2001:364-398.
[9]ATUL K S,AIYAGARI R.Evaluation of a facile method of template DNA preparation for PCR-based detection and typing of lactic acid bacteria[J].Food M icrobiol,2009,26(5):504-513.
[10]VERSALOVIC J,SCHNEIDER M,BRUIJN F J.Genomic fingerprinting of bacteria using repetitive sequence-based polymerase chain reaction[J].Meth M ol Cell Biol,1994,17(5):25-40.
[11]SUZZI G,CARUSO M,GARDINI F,et al.A survey of the enterococci isolated from an artisanal Italian goat's cheese(Sem icotto caprino)[J].Jf Appl M icrobiol,2000,89(1):267-274.
[12]SERIO A,PAPARELLA A,CHAVES-LóPEZ C,et al.Enterococcus populations in Pecorino Abruzzese cheese:biodiversity and safety aspects[J].J Food Protection,2007,70(7):1561-1568.
[13]SARANTINOPOULOS P,KALANTZOPOULOS G,TSAKALIDOU E. Effect of Enterococcus faecium on m icrobiological,physicochem ical and sensory characteristics of Greek Feta cheese[J].Int J Food M icrobiol,2002,76(1-2):93-105.
[14]GIRAFFA G.Functionality of enterococci in dairy products[J].Int J Food M icrobiol,2003,88(2-3):215-222.
[15]ANTONIO S V,NABIL B O,HIKMATE A,et al.Virulence factors,antibiotic resistance,and bacteriocins in enterococci from artisan foods of animal origin[J].Food Control,2009,20(5):381-385.
[16]DAL B B,RANTSIOU K,BELLIO A,et al.Microbial ecology of artisanal products from North West of Italy and antimicrobial activity of the autochthonous populations[J].LWT-Food Sci Tech,2010,43(7):1151-1159.
[17]VANCANNEYT M,SEMAN M,SNAUWAERT C,et al.Evaluation of(GTG)5-PCR for identification of Enterococcus spp.[J].FEMS M icrobiol Lett,2005,247(2):59-63.
[18]HO P L,LAI E,CHAN P Y,et al.Rare occurrence of vancomycin-resistant Enterococcus faecium among livestock animals in China[J].J Antim icrob Chemoth,2013,68(9):2948-2963.
Genetic divergence and vancomycin-resistance genes analysis ofenterococci isolated from cheese samples
YU Yang,NI Yongqing*
(School of Food Science,Shihezi University,Shihezi 832000)
The genetic structure differences of coccus from cheese samples in the north of Xinjiang were analyzed by specific amplification of species and genus primers,rep-PCR technology and the detection of vancomycin-resistance gene.The results showed that 52 strains of enterococci were obtained from 15 samples,including 31 strains of Enterococcus durans,18 strains of Enterococcus faecalis and 3 strains of Enterococcus faecium.According to the analysis of rep-PCR genetic fingerprint spectrum,the 52 strains could be clustered into seven groups,in which four groups were constituted by E.durans.The vancomycin-resistance gene was detected from 24 strains of enterococci,in which 17 strains of E.durans were identified as VanC2/C3,4 strains of E.durans were identified as VanC1,2 strains of E.durans were identified as VanB,only one of E.durans was VanA.The enterococci strains were w idely distributed in cheese from the north of Xinjiang.The dom inant population and genotype were different between different regions.There were high genetic differences among enterococci intraspecies.
cheese;enterococci;rep-PCR;vancomycin
Q566
0254-5071(2016)05-0149-05
10.11882/j.issn.0254-5071.2016.05.031
2016-03-25
国家自然科学基金项目(31360001);新疆兵团现代农业科技攻关与成果转化项目(2015AC003)
于洋(1989-),女,硕士研究生,研究方向为食品生物技术。
倪永清(1969-),男,教授,博士,研究方向为食品生物技术。
