DNA甲基转移酶抑制剂5-Aza-CdR对AID基因修饰的牛胎儿成纤维细胞的作用
2016-09-14奥旭东萨如拉王杰王会敏于海泉
奥旭东 萨如拉 王杰 王会敏 于海泉
(内蒙古大学实验动物研究中心,呼和浩特 010021)
DNA甲基转移酶抑制剂5-Aza-CdR对AID基因修饰的牛胎儿成纤维细胞的作用
奥旭东 萨如拉 王杰 王会敏 于海泉
(内蒙古大学实验动物研究中心,呼和浩特 010021)
以转AID基因细胞和AID基因敲减细胞为研究对象,旨在探讨5-氮-2'-脱氧胞苷(5-Aza-2'-deoxycytidine,5-Aza-CdR)对其细胞形态、细胞周期、相关基因表达及其基因启动子区甲基化状态变化的影响。此外,还分析了整体基因组去甲基化和位点特异性去甲基化之间存在的差别,探讨提高体细胞重编程效率的方法及其作用机制。通过Real-time PCR、BSP(Bisulfite Sequencing PCR)、Western blotting和流式细胞术等技术分析了5-Aza-CdR对转AID基因细胞和AID基因敲减细胞的影响。结果表明,5-Aza-CdR对转AID基因细胞的影响存在明显的剂量效应,浓度为4 μmol/L时,具有明显的细胞毒性,大量细胞死亡(P<0.05)。当使用处理浓度为1-3 μmol/L时,细胞形态发生改变,细胞增殖被抑制。核型分析显示,3 μmol/L浓度组处理就可以导致转AID基因细胞出现异常核型。使用1 μmol/L浓度处理细胞,明显增加了表达报告基因细胞的数量,细胞AID和SOX2的表达量都有所提高,并且SOX2基因启动子区的DNA甲基化水平也有所下降。5-Aza-CdR处理AID基因敲减细胞后,OCT4和SOX2的表达量较对照组明显升高,而NANOG却没有发生变化。结果显示,5-Aza-CdR处理转AID基因细胞可改变细胞形态、细胞周期和报告基因的表达,5-Aza-CdR处理后基因组整体去甲基化和AID基因的位点特异性去甲基化协同作用于体细胞重编程。
5-Aza-CdR;AID基因;DNA去甲基化;体细胞核移植
胚胎发育过程是细胞全能性逐渐丧失的过程[1],有研究显示,2-细胞期到4-细胞期的小鼠胚胎各个卵裂球已表现出不同的发育倾向[2],随着胚胎的发育,各个细胞不断地向既定的方向发生分化。在发育过程中,基因组经历多种表观遗传修饰,导致基因的表达状况也随之发生了改变,由此产生了不同的分化表型。已分化的细胞可以经过去分化被逆转到未分化状态,从而获得发育的全能性,这一过程被称为体细胞重编程[3]。诱导体细胞重编程的方式主要包括体细胞核移植(SCNT)、体细胞与多潜能细胞融合、胚胎干细胞提取物处理以及转录因子介导的诱导多能干细胞(iPS)。
核移植和iPS技术被认为是到目前为止诱导体细胞重编程最为完全、最具有再生医学临床应用潜能的技术。目前人们已经获得了多种核移植动物和iPS细胞[4-9],然而这两项技术也存在很大的局限性,如iPS细胞的效率低下,体细胞克隆胚胎发育率低,流产率高及异常个体等,这些都是制约这两项技术广泛应用的主要因素[10,11]。其中,造成体细胞克隆胚胎发育异常以及iPS细胞重编程的效率极低的主要原因是体细胞基因组不能完全的去分化、不能正常启动重编程过程。
基因组DNA甲基化是表观遗传调控的重要方式[12]。有研究表明,克隆胚胎基因组的异常甲基化是引起克隆胚发育率和出生率低的主要原因之一[13]。高甲基化的癌细胞DNA去甲基化后发生肌源性重组,可以提高其iPS的重编程效率[14]。目前研究表明,去甲基化药物可以使抑癌基因去甲基化,从而恢复其功能治疗肿瘤[15]。5-氮胞苷(5-azacytidine,5-AzaC)是一种原型的DNMT抑制剂,而5-氮-2'-脱氧胞苷(5-aza-2'-deoxy-cytidine,5-Aza-dC)是5-AzaC的脱氧核糖类似物。它们具有较强的DNMT抑制作用,其通过抑制了DNMT的作用来降低基因组的DNA甲基化水平,从而调节基因表达和介导细胞分化[16-18]。
活化诱导的胞嘧啶核苷脱氨酶蛋白(Activationinduced cytidine deaminase,AID)主要因其在B淋巴细胞中产生抗体多样性的作用被人们广泛认识[19-21]。随后,研究人员发现AID蛋白可以将5mC脱氨基成为T,从而导致在T-G不匹配[18]。而AID基因表达于具有多能性的组织中[22],这都暗示其具有DNA去甲基化作用。研究证实在小鼠[23,24]和斑马鱼[25]中AID基因具有DNA主动去甲基化作用。小鼠中的研究虽然表明AID基因具有DNA主动去甲基化作用,但是其作用并不是针对全基因组的而是具有一定的位点特异性[23]。目前,还未见用DNA去甲基化药物5-Aza-CdR与AID基因联合作用对重编程影响的报道。因此,5-Aza-CdR在体细胞重编程中与AID基因作用的关联还不是很清楚,还需进一步探讨。本研究以转AID基因细胞和AID基因敲减细胞为研究对象,探讨5-Aza-CdR对其细胞形态、细胞周期、相关基因表达及其基因启动子区甲基化状态变化的影响;并分析整体基因组去甲基化和位点特异性去甲基化之间存在的差别,以期为体细胞克隆效率的提高奠定理论基础。
1 材料与方法
1.1材料
5-Aza-CdR购自Sigma公司,MTT购自Promega公司,An-nexin-V购自Invitrogen公司。除特别提到的化学试剂,其余均购自Sigma公司。
1.2方法
1.2.1转AID基因细胞及AID敲减细胞的获得 转AID基因细胞的获得使用脂质体LipofectamineTM2000(Invitrogen)转染本实验室构建的过表达载体pCDsRed-PbAID。转染48 h后消化细胞,重新接种于10 cm培养皿中,加入G418,每3 d换液1次。待培养皿中形成表达红色荧光蛋白的抗药克隆时,用克隆杯消化后接种于24孔板后,扩大培养。
用于敲减AID基因的siRNA没有筛选标记,不能长期干扰AID基因的表达。为了保证AID-siRNA能够高效的转染牛胎儿成纤维细胞并起到干扰作用,本研究使用本实验室转染效率比较高的电转染方法来转染AID-siRNA。该方法转染pDsRed-C质粒转染效率能够达到80%左右。AID基因敲减细胞的获得参照以下步骤:第3代牛胎儿成纤维细胞培养2-3 d后,细胞单层汇合度达到70%-80%,胰酶消化细胞,1 200 r/min室温离心5 min。弃上清,根据细胞数量,加入适量的电转缓冲液重悬细胞并吹打均匀。加入终浓度为100 nmol/L siRNA,按照预定条件设置参数250 V,5 ms,2次,进行电击。将电转杯置于37℃培养箱中孵育8-12 min,然后将细胞悬液接种于培养皿中,正常培养4 h,待细胞贴壁后,换新鲜的完全培养液,以去除上层的死细胞。培养24 h后,进行相关的实验处理。
1.2.25-Aza-CdR处理体细胞及细胞毒性、凋亡与周期的分析 5-Aza-CdR用培养液DMEM/F12溶解,配制成1 mol/L的浓储液,-20℃储存。当细胞生长达到对数生长期,在培养孔里分别加入终浓度为1、2、3和4和5 μmol/L的5-Aza-CdR处理24 h或48 h。
收集对数生长期细胞,调整细胞浓度为2 000 cells /孔置于96孔板。37℃,5% CO2培养2 d后,加入梯度浓度的5-Aza-CdR。然后37℃,5% CO2孵育24 h或48 h,PBS洗2遍,加入100 μL细胞培养液。每孔加入20 μL 0.5% MTT溶液,继续培养4 h。每孔加入100 μL DMSO混匀后酶联免疫检测仪检测OD490 nm。生存细胞的百分比计算方法如下:细胞生存能力抑制率(%)=[1-(OD treatment-OD blank)/(OD control-OD blank)]×100%。
调整待检测的细胞浓度为2×106-5×106/mL。加入5 μL的Annexin V,室温孵育3 min,20 μg/mL 的PI染核。避光室温孵育10 min后,经流式细胞仪(Beckman Coulter,SC)检测。
5-Aza-CdR处理转AID基因细胞,20 μg/mL的PI染核后使用Beckman流式细胞仪检测细胞周期,使用Cell Cycle软件(Beckman Coulter)分析数据。处理后体细胞核型的检测方法使用本实验室常规制作检测方法。
1.2.35-Aza-CdR处理体细胞基因的表达及其甲基化变化分析 挑选生长状态良好的转AID基因细胞克隆提取RNA后,经反转录后进行Real-time PCR反应。PCR特异性引物以牛AID、OCT4、NANOG 和SOX2基因为模板进行设计。引物序列见表1。在ABI 7300实时荧光定量PCR仪反应后,使用2-ΔΔCT方法与GAPDH相比较后得到AID基因及多能基因的表达。
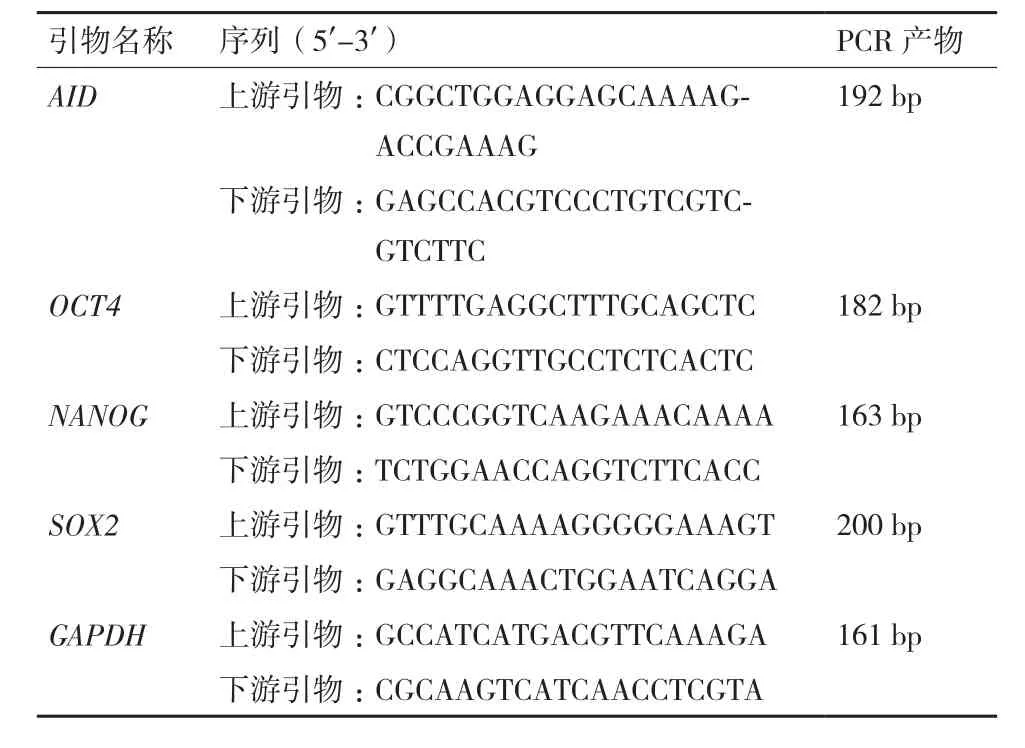
表1 牛Real-time PCR引物序列和反应产物
挑选生长状态良好的转AID基因细胞克隆提取DNA。基因组DNA经过Xho I(TaKaRa)酶切后,用亚硫酸氢盐处理,然后用乙醇沉淀的方法回收DNA用于PCR。引物序列见表2。
1.2.4统计分析 所有试验数据使用SPSS 14.0软件进行统计分析。Real-time PCR试验结果使用单因素分析,而DNA甲基化状态使用卡方检验。P<0.05确定为差异的显著性。
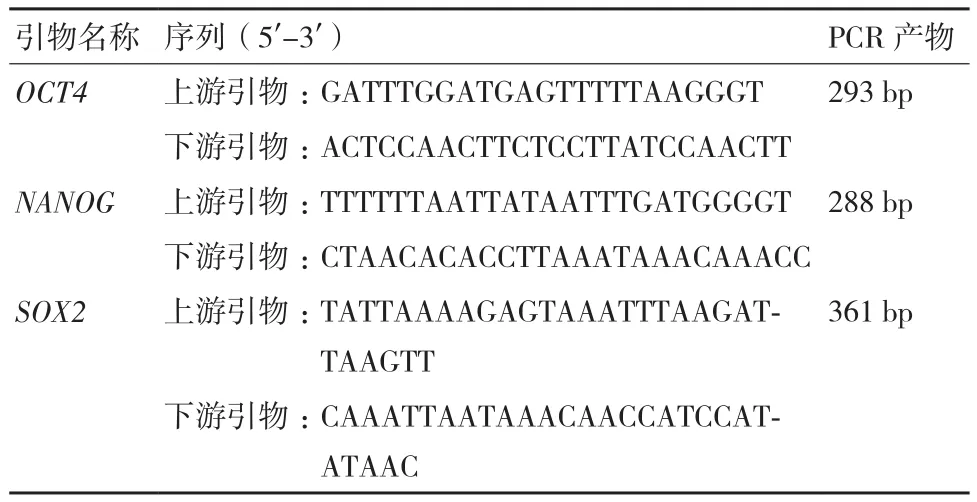
表2 牛BSP-PCR引物序列和反应产物
2 结果

图1 5-Aza-CdR对细胞增殖、细胞周期和细胞凋亡的影响
2.15-Aza-CdR对转AID基因细胞生长的影响
本实验使用梯度浓度的5-Aza-CdR处理转AID基因细胞,并分析了5-Aza-CdR处理后的细胞周期,细胞形态和其细胞毒性作用。结果(图1-A)显示,不同浓度的5-Aza-CdR处理24 h后,1、2 和3 μmol/L与对照组相比细胞活性没有明显差异(P>0.05),而且相邻浓度之间也不存在显著性差异。但随着5-Aza-CdR浓度的继续升高,细胞活性明显降低(P<0.01)。1 μmol/L 5-Aza-CdR处理48 h后,所有处理组都与对照组差异极显著,而不同浓度之间只有2 μmol/L 和3 μmol/L 之间没有差异(P>0.05)。由此可见,随着5-Aza-CdR作用浓度的增加,死细胞数明显增多,细胞耐受性减弱,活性明显下降;相同浓度作用下,随着处理时间的延长,细胞活性呈显著下降趋势(图1-B)。细胞凋亡实验(图1-D)表明,随着5-Aza-CdR处理浓度的增大,早期凋亡率并没有明显的升高趋势,晚期凋亡率在个别浓度处理下有所下降,但是死亡细胞的比例却显著增加(P<0.01),细胞发生凋亡的比例也与5-Aza-CdR处理浓度呈正相关。通过流式细胞仪,对5-Aza-CdR处理24 h组进行了细胞周期分析,结果(图1-C)显示经过1、2和3 μmol/L 5-Aza-CdR处理24 h后,G0/G1期和G2/M期细胞增多,S期细胞减少。而且发现在2 μmol/L和3 μmol/L处理组中出现了少量比例的非整倍体细胞,这说明高浓度的5-Aza-CdR会增加非整倍体的几率,对细胞有毒性作用。
此外,5-Aza-CdR处理24 h后,处理组细胞形态与对照组没有明显的变化(图2-A),但是48 h处理组与24 h相比,细胞形态更加扁平,细胞间隙变大(图2-B)。使用1、2和3 μmol/L浓度的5-Aza-CdR分别处理转AID基因细胞24 h后,检测细胞染色体倍性的变化(图3)。结果(表3)表明,1、2和3 μmol/L处理组都造成了体细胞整倍性率的降低,分别为78%、71%和60%,其中3 μmol/L处理组与对照组具有显著性差异。由此可见高浓度(3 μmol/L)的5-Aza-CdR处理会导致染色体数目的异常。

图2 不同浓度5-Aza-CdR处理转AID基因细胞24 h(A)及48 h(B)对其细胞形态的影响

表3 5-Aza-CdR处理转AID基因细胞染色体倍性分析
2.25-Aza-CdR对转AID基因细胞AID等基因表达的影响
使用1 μmol/L 5-Aza-CdR处理转AID基因细胞后,表达红色荧光蛋白(DsRed)的细胞数量及其荧光的表达强度都明显增加(图4-A)。说明5-Aza-CdR能够诱导失活的DsRed启动子CMV重新激活。
使用Real-time PCR的方法检测到稳定转染AID基因的牛胎儿成纤维细胞AID基因和多能性基因OCT4和NANOG表达明显上升,但是SOX2基因的表达却没有明显的改变;经5-Aza-CdR处理后,SOX2的表达量都明显提高(图4-B)。为了进一步分析5-Aza-CdR处理后SOX2的表达变化的原因,使用亚硫酸氢盐PCR测序的方法分析了1 μmol/L 5-Aza-CdR处理的AID转基因细胞中OCT4、NANOG 和SOX2启动子区DNA甲基化水平。结果(图4-C)显示,5-Aza-CdR的处理降低了这些基因的甲基化水平,其中SOX2的变化具有显著性差异。
2.35-Aza-CdR对AID基因敲减细胞的影响

图3 5-Aza-CdR处理转AID基因细胞对其染色体倍性的影响

图4 5-Aza-CdR处理转AID基因细胞对其基因表达的影响
为了进一步确认干扰片段能够有效地在牛胎儿成纤维细胞中起作用,对转染24 h后细胞中AID基因的表达情况进行了检测。结果(图5-A)显示,干扰片段能将AID基因的表达量降低至原水平的5.25%,可以高效的降解目标基因mRNA,为后续研究提供良好的基础。Western bloting结果(图5-B)也证实转染AID-siRNA干扰了正常AID基因的表达。
为了更好的分析AID基因对多能性基因的作用,本研究检测了电转染AID-siRNA 24 h后,细胞中多能性基因OCT4、NANOG和SOX2的表达变化。结果(图5-C)显示,干扰AID的表达后,OCT4和NANOG的表达没有变化,但是SOX2的表达量发生了明显的降低,提示AID对SOX2的表达起着至关重要的作用。1 μmol/L 5-Aza-CdR处理AID基因敲减细胞24 h后能够显著提高OCT4和SOX2基因的表达(图5-D),但是NANOG基因和AID基因的表达量没有明显的变化(图5-D)。
3 讨论

图5 5-Aza-CdR处理AID基因敲减细胞对其基因表达的影响
目前认为,供体细胞在体细胞克隆过程中必然要经历重编程的过程,而体细胞克隆效率低下和克隆动物异常的重要原因就是供体细胞错误的重编程或重编程不彻底[26]。而在克隆胚胎的重编程过程中DNA甲基化是很重要的事件,DNA甲基化水平的降低可以激活或促进多能基因的表达,进而改善体细胞重编程的效率[13]。目前,研究人员主要通过使用药物改变体细胞或重构胚的表观遗传特性来改善体细胞重编程。5-Aza-CdR作为一种DNMT抑制剂,能够抑制DNA的甲基化,从而降低基因组的整体甲基化水平,增强基因的表达量[16-18]。2002年,5-Aza-CdR成为美国 FDA 首批批准上市的去甲基化药物,其主要用途就是治疗MDS[27]。研究表明,使用5-Aza-CdR治疗41例白血病后有一半的患者临床症状减轻,通过检测发现患者的总体基因组DNA甲基化水平明显降低[17]。本研究检测了5-Aza-CdR处理的转AID基因细胞报告基因的表达、细胞周期的分布、细胞形态转变、多能性基因的表达及其启动子区DNA甲基化的变化,进而研究了5-Aza-CdR的整体去甲基化作用与AID的位点特异性去甲基化作用之间的关系。
本研究发现高浓度5-Aza-CdR或者是长时间的处理会明显增加细胞的毒性,而使细胞增殖受到抑制,并阻止细胞进入DNA复制期,细胞形态扁平化类似于生长过老的细胞的状态,此外,染色体整倍性也会发生改变。而在低浓度和短时间内处理对细胞的形态、周期和染色体倍性没有影响。以上研究说明5-Aza具有一定的细胞毒性作用,为了能够保证5-Aza不影响细胞的正常生长,本研究选择用1 μmol/L 5-Aza-CdR处理细胞24 h。这样既能保证药物的作用效果又可以避免其对细胞的毒性损伤。
吴侠等[28]研究显示,转基因体细胞中报告基因的表达会随着细胞的生长及传代次数的增加而明显下降,本研究也观察到了类似的现象。有研究显示,高浓度的5-Aza-CdR处理细胞能够降低其基因组DNA甲基化,进而增强外源报告基因的表达[29,30]。本研究所用过表达载体pCDsRed-PbAID的报告基因(DsRed)的表达是由CMV调控的,研究显示CMV启动子能够在哺乳动物体内广泛表达。但是,进一步的观察发现报告基因的表达随着细胞的传代培养被沉默[31]。通过分析CMV启动子在转基因细胞中其启动子的甲基化状态发现,其启动子区的8个CpG位点发生甲基化[32]。本研究证实,使用5-Aza-CdR处理后转基因体细胞基因组甲基化水平下降,报告基因的表达能够被恢复。因而推断,报告基因表达的下降,与CMV启动子在传代过程中的甲基化修饰有关。
供体细胞在受体卵母细胞胞质中不完全的重编程或错误重编程是导致克隆胚胎发育率低的重要原因[26]。使供体细胞的表观遗传特性更接近具有多能性的配子或卵裂球可能更有助于提高重编程的效率[33,34]。在猪的研究中,用5-Aza处理的细胞作为核供体,可以促进重构胚的发育[35]。在牛的研究中也有类似的发现。这些研究表明使用5-Aza处理能够改善供体核的表观遗传特性从而最终提高克隆胚胎的发育率。稳定转染AID基因的牛胎儿成纤维细胞其AID、OCT4和NANOG基因表达明显上升。这就提示在牛胎儿成纤维细胞中AID基因可以一定程度上改善其表观遗传特性。由于SOX2基因没有明显变化,暗示AID基因的DNA去甲基化作用具有位点特异性。5-Aza-CdR处理转AID基因细胞后检测发现OCT4和NANOG的表达量都没有明显的变化,但是SOX2的表达量明显提高。此外,通过BSP的方法研究了5-Aza-CdR处理转AID基因细胞中的OCT4、NANOG、SOX2的甲基化状态,发现5-Aza-CdR可以降低这几个基因的甲基化水平,尤其是SOX2 (P<0.05)。这说明5-Aza-CdR虽然具有广泛的DNA去甲基化作用,但是对于一些特定位点的去甲基化作用是不明显的。我们之前的研究表明,AID基因转入体细胞中,更有利于核移植胚胎的重编程,因而提高了体细胞克隆胚胎的卵裂率和发育率。综合本研究5-Aza-CdR处理细胞的实验结论,可以推测AID基因的导入和5-Aza-CdR的联合作用,可能更有利于体细胞克隆效率的提高。
此外,为了分析5-Aza-CdR的基因组整体去甲基化与AID基因的位点特异性去甲基化之间的关系,本研究使用电转的方法转染AID siRNA来干扰AID基因的表达,同时使用1 μmol/L 5-Aza-CdR对转染细胞进行处理。结果显示,干扰AID基因后OCT4 和NANOG的表达没有变化,但是SOX2的表达量发生了明显的降低,而使用5-Aza-CdR能够补偿SOX2的表达。这就再一次证实了AID在哺乳动物细胞中具有DNA去甲基化作用,并且AID的DNA去甲基作用具有位点特异性。同时也说明了5-Aza-CdR的DNA去甲基化作用与AID基因的导入联合作用,更有利于体细胞克隆效率的提高。目前研究证实AID基因可以对5mC/5hmC脱氨基,然后在如MBD4或胸腺嘧啶DNA糖基化酶(Thymine DNA glycosylase,TDG)这些糖基化酶的作用下通过碱基切除修复(Base excision repair,BER)和错配修复(Mismatch repair,MMR)的作用下修复T-G错配从而最终实现DNA去甲基化[22,36]。在所有可能的AID基因去甲基化作用过程中,AID基因只是通过脱氨基作用来确定一个修饰后的碱基位点,而这一位点可能被修复复合体所锚定并修复,同时这一位点也可能不被修复复合体所识别[37]。此外,虽然现在不清楚AID基因如何选择其靶位点,但是不可否认其选择具有一定的特殊性[36]。这两个原因就造成了AID基因的DNA去甲基化作用具有位点特异性。
4 结论
基因表达的调控是一个多种调节方式协同的结果,5-Aza-CdR的基因组整体去甲基化与AID基因的位点特异性去甲基化对于体细胞重编程都具有重要的作用,在特定基因位点这两种作用可以相互补充,二者联合处理供体细胞可能更有利于体细胞重编程效率的提高。
[1]Spivakov M, Fisher AG. Epigenetic signatures of stem-cell identity[J]. Nat Rev Genet, 2007, 8:263-271.
[2]Fujimori T, Kurotaki Y, Miyazaki J, et al. Analysis of cell lineage in two- and four-cell mouse embryos[J]. Development, 2003, 130:5113-5122.
[3]Hochedlinger K, Jaenisch R. Nuclear reprogramming and pluripotency[J]. Nature, 2006, 441:1061-1067.
[4]Takahashi K, Tanabe K, Ohnuki M, et al. Induction of pluripotent stem cells from adult human fibroblasts by defined factors[J]. Cell, 2007, 131:861-872.
[5]Chang MY, Kim D, Kim CH, et al. Direct reprogramming of rat neural precursor cells and fibroblasts into pluripotent stem cells[J]. PLoS One, 2010, 5(3):e9838.
[6]Liu H, Zhu F, Yong J, et al. Generation of induced pluripotent stem cells from adult rhesus monkey fibroblasts[J]. Cell Stem Cell,2008, 3:587-590.
[7]West FD, Terlouw SL, Kwon DJ, et al. Porcine induced pluripotent stem cells produce chimeric offspring[J]. Stem Cells Dev, 2010, 19:1211-1220.
[8]Honda A, Hirose M, Hatori M, et al. Generation of induced pluripotent stem cells in rabbits:potential experimental models for human regenerative medicine[J]. J Biol Chem, 2010, 285:31362-31369.
[9]Li Y, Cang M, Lee AS, et al. Reprogramming of sheep fibroblasts into pluripotency under a drug-inducible expression of mouse-derived defined factors[J]. PLoS One, 2011, 6:0015947.
[10]Li E. Chromatin modification and epigenetic reprogramming in mammalian development[J]. Nature Reviews Genetics, 2002, 3:662-673.
[11]Reik W. Stability and flexibility of epigenetic gene regulation in mammalian development[J]. Nature, 2007, 447:425-432.
[12]Duan B, Cheng L, Gao Y, et al. Silencing of fat-1 transgene expression in sheep may result from hypermethylation of its driven cytomegalovirus(CMV)promoter[J]. Theriogenology, 2012,78:793-802.
[13]Cedar H, Bergman Y. Linking DNA methylation and histone modification:patterns and paradigms[J]. Nature Reviews Genetics, 2009, 10:295-304.
[14]Xue L, Cheng L, Su G, et al. Nuclear transfer procedures in the ovine can induce early embryo fragmentation and compromise cloned embryo development[J]. Anim Reprod Sci, 2011, 126:179-186.
[15]Egger G, Liang G, Aparicio A, et al. Epigenetics in human disease and prospects for epigenetic therapy[J]. Nature, 2004, 429:457-463.
[16]Wu SC, Zhang Y. Active DNA demethylation:many roads lead to Rome[J]. Nature Reviews Molecular Cell Biology, 2010, 11:607-620.
[17]Kantarjian H, Oki Y, Garcia-Manero G, et al. Results of a randomized study of 3 schedules of low-dose decitabine in higherrisk myelodysplastic syndrome and chronic myelomonocytic leukemia[J]. Blood, 2007, 109:52-57.
[18]Yang AS, Doshi KD, Choi S-W, et al. DNA methylation changes after 5-aza-2'-deoxycytidine therapy in patients with leukemia[J]. Cancer Res, 2006, 66:5495-5503.
[19]Mayer W, Niveleau A, Walter J, et al. Demethylation of the zygotic paternal genome[J]. Nature, 2000, 403:501-502.
[20]Oswald J, Engemann S, Lane N, et al. Active demethylation of thepaternal genome in the mouse zygote[J]. Current Biology, 2000,10:475-478.
[21]Nakazawa Y, Shimada A, Noguchi J, et al. Replacement of nuclear protein by histone in pig sperm nuclei during in vitro fertilization[J]. Reproduction, 2002, 124:565-572.
[22]Morgan H, Dean W, Coker H, et al. Activation-induced cytidine deaminase deaminates 5-methylcytosine in DNA and is expressed in pluripotent tissues[J]. Biological Chemsitry, 2004, 279:52353-52360.
[23]Bhutani N, Brady JJ, Damian M, et al. Reprogramming towards pluripotency requires AID-dependent DNA demethylation[J]. Nature, 2010, 463:1042-1047.
[24]Popp C, Dean W, Feng S, et al. Genome-wide erasure of DNA methylation in mouse primordial germ cells is affected by AID deficiency[J]. Nature, 2010, 463:1101-1105.
[25]Rai K, Huggins IJ, James SR, et al. DNA demethylation in zebrafish involves the coupling of a deaminase, a glycosylase, and Gadd45[J]. Cell, 2008, 135:1201-1212.
[26]Wang Q, Yin S, Ai JS, et al. Histone deacetylation is required for orderly meiosis[J]. Cell Cycle, 2006, 5:766-774.
[27]Issa JPJ, Kantarjian HM, Kirkpatrick P. Azacitidine[J]. Nature Reviews Drug Discovery, 2005, 4:275-276.
[28]Wu X, Li Y, Li GP, et al. Trichostatin A improved epigenetic modifications of transfected cells but did not improve subsequent cloned embryo development[J]. Anim Biotechnol, 2008, 19:211-224.
[29]Krishnan M, Park JM, Cao F, et al. Effects of epigenetic modulation on reporter gene expression:implications for stem cell imaging[J]. FASEB J, 2006, 20:106-108.
[30]Mengxi D, Qian W, Nan W, et al. Effect of DNA methylation inhibitor on RASSF1A genes expression in non-small cell lung cancer cell line A549 and A549DDP[J]. Cancer Cell Int, 2013,13:1475-2867.
[31]Verma IM, Somia N. Gene therapy-promises, problems and prospects[J]. Nature, 1997, 389:239-242.
[32] Mutskov V, Felsenfeld G. Silencing of transgene transcription precedes methylation of promoter DNA and histone H3 lysine 9 [J]. Embo J, 2004, 23:138-149.
[33]Dean W, Santos F, Reik W. Epigenetic reprogramming in early mammalian development and following somatic nuclear transfer[J]. Seminars in Cell & Developmental Biology, 2003,14:93-100.
[34]Campbell KH, Loi P, Otaegui PJ, et al. Cell cycle co-ordination in embryo cloning by nuclear transfer[J]. Rev Reprod, 1996, 1:40-46.
[35]Diao YF, Naruse KJ, Han RX, et al. Treatment of fetal fibroblasts with DNA methylation inhibitors and/or histone deacetylase inhibitors improves the development of porcine nuclear transferderived embryos[J]. Anim Reprod Sci, 2013, 141:164-171.
[36]Dominguez PM, Shaknovich R. Epigenetic function of activationinduced cytidine deaminase and its link to lymphomagenesis[J]. Front Immunol, 2014, 5:642.
[37]Sadakierska-Chudy A, Kostrzewa RM, Filip M. A comprehensive view of the epigenetic landscape part I:DNA methylation, passive and active DNA demethylation pathways and histone variants[J]. Neurotox Res, 2015, 27:84-97.
(责任编辑 马鑫)
The Effect of DNA Methyltransferase Inhibitor 5-Aza-CdR on AID Gene-modified Bovine Fetal Fibroblasts
AO Xu-dong SA Ru-la WANG Jie WANG Hui-min YU Hai-quan
(Research Center for Laboratory Animal Science Inner Mongolia University,Hohhot 010021)
In order to improve the efficiency of somatic cell nuclear transfer,the researchers used drugs which can alter DNA methylation or histone modifications to benefit somatic cell nuclear transfer. Among them,5-aza-2'-deoxycytidine(5-Aza-CdR)blocked DNA methylation by inhibiting methyl group transfer to adenine or cytosine,and ultimately reduced genomic methylation. In this study,the AID-overexpressing and AID-knocked-down cells were used to study the effects of 5-Aza-CdR(1,2,3,4,and 5 μmol/L)on cell morphology,cell cycle,related gene expression and the changes of methylation status in the gene promoter region. Additionally,the differences between genomic DNA demethylation and site-specific DNA demethylation was analyzed,the methods and its action mechanism of increasing the reprogramming efficiency of somatic cells were discussed. Real-time PCR,BSP(Bisulfite Sequencing PCR),Western blotting,and flow cytometry were employed to analyze the effects of 5-Aza-CdR on the AID-transgenic and AID-knocked-down cells. The results showed that 5-Aza-CdR presented dose-dependent effect on AID-transgenic cells,while the 5-Aza-CdR concentration was 4 μmol/L,obvious toxicity was observed as a lot of cells were dead(P<0.05). When treated by 1-3 μmol/L,the cell's morphology changed,and proliferation of cells was inhibited. Karyotype analysis showed that 3 μmol/L treatments resulted in the emergence of a small proportion of aneuploidy cells,indicatingthat high concentrations of 5-Aza-CdR increased the rate of aneuploidy. The number of cells which expressed red fluorescent protein(DsRed)significantly increased after 1 μmol/L 5-Aza-CdR treatment,compared to the control group,the expression of AID and SOX2 were also increased,and the methylation levels in SOX2 promoter region were somehow decreased. After treated by 5-Aza-CdR,the expression of the OCT4 and SOX2 genes increased in AID-knocked-down cells,but the expression of the NANOG did not change. The above results revealed that 5-Aza-CdR treatment affected the cell morphology,cell cycle and reporter gene expression in bovine transgenic cells. In conclusion,while treated by 5-Aza-CdR,the genomic demethylation and loci-specific demethylation of AID act synergistically in somatic cell reprogramming.
5-Aza-CdR;AID;DNA demethylation;somatic cell nuclear transfer
10.13560/j.cnki.biotech.bull.1985.2016.08.016
2015-10-13
国家自然科学基金项目(31460311),内蒙古自然科学基金项目(2012ZD04)
奥旭东,男,博士,研究方向:哺乳动物生殖生物学;E-mail:aoxudong_123@126.com
于海泉,男,博士,研究员,研究方向:哺乳动物生殖生物学;E-mail:haiquan_yu@yahoo.com
