菊糖酶提高酿酒酵母利用菊糖产乙醇能力的研究进展
2016-09-10李丽丽侯英敏郭小宇李宪臻
李丽丽,侯英敏,郭小宇,杨 帆,李宪臻
(大连工业大学生物工程学院,辽宁大连 116034)
菊糖酶提高酿酒酵母利用菊糖产乙醇能力的研究进展
李丽丽,侯英敏,郭小宇,杨帆*,李宪臻
(大连工业大学生物工程学院,辽宁大连 116034)
化石能源的枯竭和生态环境的恶化,使乙醇等清洁能源的生产受到越来越多的关注。作为非粮作物的菊芋,因富含菊糖且生态适应能力极强,是生产乙醇的优良生物质原料。但因绝大多数酿酒酵母不具有菊糖降解能力,而无法直接利用菊糖生产乙醇,阻碍了利用菊芋生产乙醇的工业化进程。本文总结了近年来国内外学者采用发酵工程和基因工程手段优化菊糖生产乙醇的研究进展,探讨了菊糖酶在酿酒酵母生产乙醇中所起到的关键作用,并展望了利用菊糖降解元件的数据库挖掘及酿酒酵母菊糖酶的表面展示实现乙醇高效生产的前景。
生物乙醇,菊糖酶,发酵工程,基因工程
燃料乙醇是一种洁净的可再生能源,对于解决化石能源危机及环境污染意义重大[1]。鉴于我国人多地少、粮食生产压力大的基本国情,以甘蔗、玉米为原料的乙醇生产因争粮争地而限制了其发展空间[2];以纤维素为原料的乙醇生产,虽然容易获得且不与人争粮,但高成本的预处理过程严重阻碍了其工业化进程[3-4]。富含菊糖的菊芋具有很强的抗逆能力。研究发现,菊芋能够替代玉米淀粉生产乙醇[3],且生产1 t乙醇的原料成本比淀粉低1200~1500元,优势明显。
菊糖是由D-呋喃果糖通过β-2,1糖苷键相连而成的链状多聚果糖,其还原末端为葡萄糖残基,聚合度为2~60[4](图1)。目前,仅有少数几类微生物能够直接利用菊糖生产乙醇,但其乙醇生产能力有限[5]。酿酒酵母(Saccharomycescerevisiae)具有其他菌株无法比拟的优点,如乙醇生产能力强、针对该酵母的遗传操作平台成熟、操作简单,有利于基因工程改造[6],已成为利用菊糖生产乙醇的首选微生物。然而,大多数酿酒酵母菌株都不能有效利用菊糖生产乙醇,严重制约了菊糖基生物质生产乙醇的工业化进程[7]。

图1 菊糖的分子结构Fig.1 The molecular structure of inulin
目前,越来越多的新策略被用于酿酒酵母的菊糖发酵过程优化、菌种改良及代谢调控,以克服菌株自身局限性。本文从利用菊糖酶提高酿酒酵母的菊糖发酵性能角度出发,综合评述了国内外在强化酿酒酵母菊糖降解活性、提高乙醇生产能力等方面的研究进展,并展望了菊糖降解相关元件的进一步挖掘及细胞表面展示技术在改造酿酒酵母以提高其利用菊糖生产乙醇性能中的应用。
1 菊糖酶及其生产菌辅助酿酒酵母利用菊糖生产乙醇
传统乙醇生产工艺是先将菊糖基生物质酸水解为果糖,再由酿酒酵母发酵转化为乙醇。然而,酸水解处理菊糖基生物质常伴有抑制剂产生[8],且环境不友好。因此,研究者则致力于利用微生物或酶辅助酿酒酵母直接利用菊糖生产乙醇。
1.1菊糖酶辅助酿酒酵母生产乙醇
研究发现,有多种微生物能够合成菊糖酶,如真菌类的黑曲霉(Aspergillusniger)、米曲霉(Aspergillusoryzae)、青霉(Penicilliumsp.),酵母类的脆壁克鲁维酵母(Kluyveromycesfragilis)、马克斯克鲁维酵母(Kluyveromycesmarxianus)、拟热带假丝酵母等(Candidapseudotropicalis),细菌类的节杆菌(Athrobactersp.)、假单胞菌(Pseudomonassp.)、黄单胞菌(Xanthomonassp.)等[9]。不同来源的菊糖酶,其酶切方式和酶学特征各不相同,表1总结了近些年已报道菊糖酶的酶学特性。这些研究为国内外学者利用菊糖酶辅助酿酒酵母生产乙醇打下基础。
周正等[3]利用克鲁维酵母产菊糖酶将菊糖水解后,将浓度为150 g/L的酶解产物提供给酿酒酵母BY4742生产乙醇,其产量与以葡萄糖或果糖为碳源的对照组相比并无明显差异,该结果说明酿酒酵母能够高效地将酶水解后的菊糖转化为乙醇。Zhang等[21]利用重组毕赤酵母(PichiapastorisX-33/pPICZa-INU1)高表达季也蒙毕赤酵母(Pichiaguilliermondii)的菊糖酶,并用粗酶液水解菊糖,提供给酿酒酵母W0进行乙醇生产,发酵120 h时后的总糖利用率为98.9%、乙醇产量为0.384 g/g菊糖,该工作进一步证明了利用菊糖酶预处理菊糖的产物进行乙醇发酵的可行性。
Tatcha等[8]比较了硫酸和菊糖酶水解菊芋汁后发酵生产乙醇的效果,结果表明,当用硫酸水解菊芋汁后进行乙醇发酵时,其生产能力为0.65 g·L-1·h-1,而用菊糖酶水解菊芋汁后发酵生产乙醇的生产能力为0.39 g·L-1·h-1。酸水解菊芋生产乙醇技术的成本较低,但工艺较为繁琐、水解产物中常含有羟甲基糠醛等微生物生长抑制剂且环境不友好;菊糖酶水解菊芋生产乙醇技术的乙醇产量低于酸水解法,但其反应条件温和、毒副产物少,因而更具有研究及发展价值。
1.2菊糖酶生产菌与酿酒酵母共发酵生产乙醇
虽然菊糖酶辅助酿酒酵母生产乙醇是一种环境友好技术,但所形成的果糖会对菊糖酶形成产物抑制作用,因此,研究者又发展了共发酵乙醇生产技术,将菊糖酶生产菌与酿酒酵母混合培养,使酿酒酵母即时利用产酶微生物形成的菊糖降解糖进行乙醇发酵,以解除产物抑制作用。
Ge等[22]采用诱变技术获得一株产外切菊糖酶的黑曲霉SL-09,并将该菌与高乙醇耐受性酿酒酵母Z-06共培养。经48 h发酵菊芋汁后,菊糖利用率达到98%,通过菊糖补料使乙醇浓度最终达到19.6%(v/v),糖醇转化效率达到理论值的90%。常保磊等[23]比较了自絮凝颗粒酵母(S.cerevisiaeflo)共发酵菊芋汁与菊糖酶辅助自絮凝酵母生产乙醇的效果,结果表明共发酵菊芋汁的乙醇得率更高、发酵时间更短。当菊芋汁中的总糖浓度分别为105 g/L和179 g/L时,乙醇产量可达到50 g/L和82.5 g/L,比菊糖酶辅助酵母发酵生产乙醇分别高6.4%和13.8%。而在连续发酵实验中,当稀释率为0.02 h-1时,乙醇产量约为90 g/L,乙醇得率达到理论值的90%,生产能力达到2.12 g·L-1·h-1。
采用菊糖酶生产菌与酿酒酵母共发酵技术生产乙醇时,虽然乙醇产量和糖醇转化率都比菊糖酶辅助酿酒酵母生产乙醇时高,但仍受很多因素制约,比如,产酶微生物是否具有较好的乙醇耐受性、共培养微生物的生长条件是否一致、产酶微生物达到最大产酶量的时间是否与乙醇生产菌达到最高乙醇产量的时间一致等。因此,需要进一步优化微生物菌种和工艺优化,使共培养微生物发酵参数达到一致,最大程度提高菊糖生产乙醇效率。
2 菊糖酶自分泌酿酒酵母直接利用菊糖生产乙醇
利用外加菊糖酶或产酶微生物策略能在一定程度上弥补酿酒酵母自身缺少菊糖酶的缺陷,但由于工艺繁琐、发酵参数不易协调,阻碍了利用该策略进行工业化生产乙醇的进程。考虑到酿酒酵母遗传背景清晰、相应的遗传操作平台成熟,越来越多的研究者将注意力集中在利用基因工程策略构建重组有菊糖酶基因的酿酒酵母,直接利用菊糖生产乙醇。
2.1酿酒酵母表达异源外切菊糖酶时的乙醇生产
外切菊糖酶(EC 3.2.1.80)能够催化菊糖从非还原末端水解并释放果糖,以供酿酒酵母生产乙醇。研究者已将多种不同来源的外切菊糖酶转化至酿酒酵母中,并结合工艺优化、酶学特性比较、优良宿主与启动子选择等手段,提高酿酒酵母利用菊糖生产乙醇的效率。
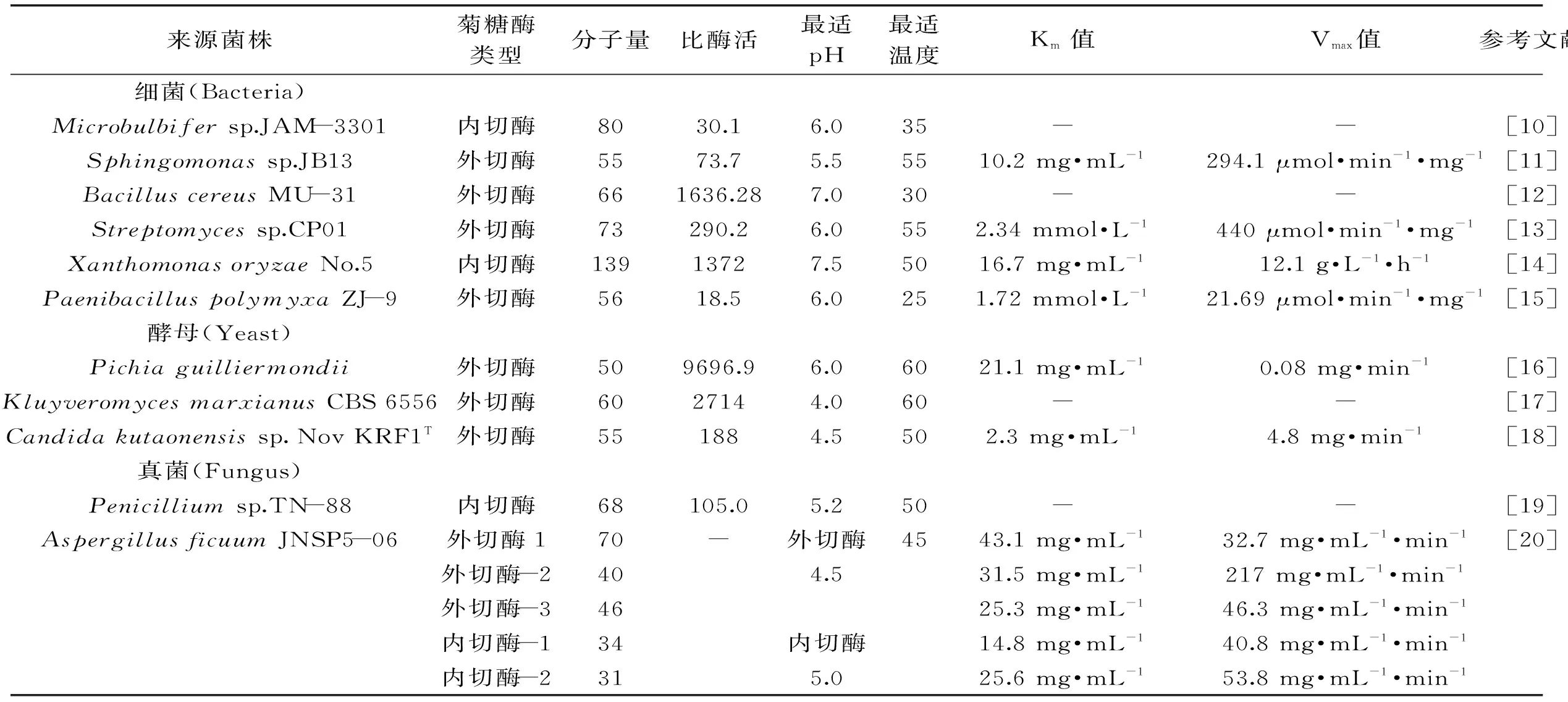
表1 不同微生物来源菊糖酶的酶学性质
Yuan等[24]将马克斯克鲁维酵母外切菊糖酶基因重组到酿酒酵母中,使其具有菊糖糖化能力,并采用联合生物加工工艺优化了乙醇发酵条件,当以总糖浓度为200 g/L的菊芋汁为原料时,经48 h发酵后的乙醇产量为72.5 g/L,生产能力为1.12~1.15 g·L-1·h-1。Du等[25]以自表达青霉菌(P.janthinellum)外切菊糖酶基因的酿酒酵母6525重组子为生产菌,利用响应面法进行菊芋粉培养基优化,结果表明,当接种量为39.8 mL/L、培养基中的酵母浸粉为9.24 g/L、MgSO4·7H2O为0.45 g/L时,获得最高乙醇产量为91.1 g/L。
Yuan等[26]比较了重组有马克斯克鲁维酵母和假丝酵母外切菊糖酶基因的酿酒酵母重组菌的乙醇生产性能,结果发现,含有假丝酵母外切菊糖酶基因的酿酒酵母重组子利用菊糖生产乙醇的性能优于含有马克斯克鲁维酵母外切菊糖酶基因的重组子,其中前者发酵菊糖的乙醇产量及生产强度分别为0.38 g/g和1.35 g·L-1·h-1,而表达有马克斯克鲁维酵母外切菊糖酶的重组菌菊糖的乙醇产量及生产强度分别为0.34 g/g和1.22 g·L-1·h-1。因此,来自于假丝酵母的外切菊糖酶基因更适于构建利用菊糖生产乙醇的酿酒酵母工程菌。
酿酒酵母W0由于其优秀的乙醇生产能力,逐渐成为研究者们进行基因工程改造的热点宿主菌。Zhang等[27]将海洋季也蒙毕赤酵母菌的外切菊糖酶基因INU1在W0菌中进行表达,重组菌在2 L发酵罐中培养96 h后,乙醇产量为14.9%(v/v)。为了使携带外源INU1基因的重组子传代性能更加稳定,Wang等[28]将外切菊糖酶基因INU1整合至W0菌的染色体上,重组子在含30%菊糖的5 L发酵罐中发酵,得到的乙醇浓度为14.7%(v/v)。随后,Zhang等[29]将海洋酵母(C.membranifacienssub sp.flavinogenie)W14-3的外切菊糖酶基因INU1转化至W0菌中,在250 g/L菊糖中培养168 h后的乙醇产量为12.5%(v/v)。为了进一步提高菊糖酶活力,Liu等[30]对季也蒙酵母(Meyerozymaguilliermondii)的外切菊糖酶基因INU1进行密码子优化并在W0菌中异源表达,所获重组子在发酵300 g/L菊糖时的乙醇产量达到126.3 g/L,乙醇生产强度为2.25 g·L-1·h-1。
绝大多数酿酒酵母在表达外源菊糖酶基因时均以基底水平进行转录,为了高效表达菊糖酶基因,必须提高酿酒酵母的基底转录水平,而酵母启动子在酵母表达系统中起着至关重要的作用[31],因此,选择高强度启动子是提高酿酒酵母菊糖酶活力的关键。Hong等[32]将马克斯克鲁维酵母外切菊糖酶基因分别与三种不同启动子(GPD PGK1、HXT7)及分泌信号肽(KmINU,MFα1,SUC2)融合,构建出9种不同表达盒并转化至酿酒酵母D452-2中。结果表明,含有PGK1启动子和MFα1分泌信号肽的酿酒酵母表现出最高酶活力和发酵性能,其发酵188.2 g/L菊糖时的乙醇产量为80.2 g/L,生产强度为1.22 g·L-1·h-1。
2.2酿酒酵母自表达内源外切菊糖酶时的乙醇生产
研究发现,自然界中有部分酿酒酵母,在不需要菊糖预处理条件下能够直接利用低聚合度的菊糖生产乙醇,如酿酒酵母NCYC 625能够有效利用聚合度在6~7之间的菊糖生产乙醇[33]。酿酒酵母KCCM50549在5 L发酵罐中,利用180 g/L菊芋粉(菊糖聚合度小于15)生产乙醇时的乙醇产量为36.2 g/L[7],而另一株酿酒酵母JZ1C利用200 g/L菊糖进行乙醇发酵时的生产强度为0.91 g·L-1·h-1[34]。作者实验室[5]也从菊芋根际土壤中分离到一株能够直接利用菊糖生产乙醇的酿酒酵母L610,离子色谱分析表明,L610能够有效利用聚合度小于20的菊糖,其发酵110 g/L菊糖时的乙醇产量为37.2 g/L。然而,所有这些酿酒酵母的基因组中都没有注释的菊糖酶基因。那么,酿酒酵母利用菊糖生产乙醇的分子机制是什么?Wang等[35]将酿酒酵母JZ1C发酵上清液中具有外切菊糖酶活性的蛋白质进行分离纯化,并通过质谱数据及序列比对证明,分离得到的蛋白质为蔗糖转化酶SUC2,是酿酒酵母利用菊糖的关键酶,但由于其蛋白质分子中缺少一段菊糖结合基序(SVEVF),导致该酶的菊糖降解能力较低。
随后,作者实验室[36]利用实时荧光定量PCR技术考察了不同酿酒酵母菌中SUC2的转录水平,发现酿酒酵母L610细胞内的SUC2表达水平远远高于非菊糖利用酿酒酵母BY4741,说明酿酒酵母的菊糖利用能力及菊糖酶活性与细胞内SUC2的表达水平直接相关。作者进一步构建了过表达SUC2 的BY4741重组菌,并对其利用菊糖进行乙醇发酵的性能进行考察。结果表明,与出发菌株BY4741相比(乙醇产量16 g/L),过表达SUC2菌的乙醇产量有显著提高,达到50 g/L。然而,与其他来源的外切菊糖酶相比,SUC2的活力有限,过表达SUC2酿酒酵母重组子的乙醇产量提高程度亦有限。
2.3酿酒酵母共表达内源外切菊糖酶与异源内切菊糖酶时的乙醇生产
酿酒酵母细胞中的内源性外切菊糖酶SUC2由于只能水解低聚合度的菊糖,无法释放出大分子菊糖中的果糖为酵母利用。而内切菊糖酶能够水解菊糖分子内糖苷键,将高聚合度菊糖分解成低聚合度菊糖如三糖、四糖和五糖等。因此,如果能够在酿酒酵母中异源表达内切菊糖酶,使菊糖部分水解以降低聚合度,再利用酿酒酵母自身的SUC2继续水解低聚合度菊糖,有望实现重组酿酒酵母高效利用菊糖生产乙醇。为了证明这一设想,Yuan等[37]将黑曲霉的菊糖内切酶基因INUA整合至酿酒酵母JZ1C染色体DNA的Ty1位点上,得到的重组菌的菊糖酶活力显著提高,乙醇生产强度达到1.15 g·L-1·h-1。随后,利用高效阴离子交换色谱HPAEC-PAD对菊糖发酵上清液进行糖组分分析,结果表明,较之野生型酿酒酵母JZ1C,重组菌发酵上清液中的高聚合度菊糖含量大幅降低甚至消失,而新增加了一些低聚合度菊糖组分,说明黑曲霉中的内切菊糖酶在酿酒酵母中的表达,能够有效水解高聚合度菊糖,但所形成的低聚合度菊糖中仍有一部分不能被酿酒酵母JZ1C的SUC2有效分解。
Li等[38]在Yuan等的研究基础上做了一定改进,他们将节杆菌中的内切菊糖酶基因整合到另一株性能优良的酿酒酵母W0染色体DNA的δ序列上,在含有30%菊糖培养基的3 L发酵罐中,重组酵母D5发酵菊糖所得乙醇产量为106 g/L。随后,Li等[38]又考察了D5直接发酵未灭菌菊芋粉的能力,乙醇产量为78.7 g/L。
虽然共表达外源内切菊糖酶与酿酒酵母SUC2对菊糖的协同降解能力仍然低于酿酒酵母单独表达异源外切菊糖酶所获得的菊糖降解能力,但这种研究为继续探究酿酒酵母细通过胞内内切菊糖酶基因与外切菊糖酶基因的协同作用,进一步提高酿酒酵母降解菊糖速率、缩短发酵周期、降低发酵成本、以实现乙醇工业化生产奠定基础。
3 展望
迄今为止,酿酒酵母基因工程重组菌还无法满足菊糖基乙醇工业化生产需要。因此,如何借助综合策略对酿酒酵母进行理性改造以获得性能优良的工程菌将成为今后研究的重点,而以下两方面的新型技术和理念为上述研究提供了崭新的视角。
首先,以Illumina公司的HiSeq2000,ABI公司的SOLiD和Roche公司的454 技术为代表的第二代高通量测序技术[39]及Helicos Heliscope和Pacific Biosciences推出的单分子测序技术[40],将生物信息学相关研究带入了一个崭新的时代,大大推动菊糖降解微生物基因组、转录组的研究,未来将有更多、更有效的菊糖降解相关酶基因及转录调控因子陆续得到挖掘。其次,酿酒酵母表面展示技术由于具有糖基化作用及蛋白翻译后折叠等优势,更利于基因工程操作,近年来有大量研究集中在利用该技术构建能够利用多种生物质生产乙醇的重组菌[41]。Fujita等[42]在酿酒酵母细胞表面展示表达了内切葡聚糖酶II和章鱼(Abdopusaculeatus)的β-葡糖苷酶I,重组菌能利用β-葡聚糖生产乙醇,产量为0.48 g/g,且发酵培养基中的β-葡聚糖能在酵母细胞表面连续水解为葡萄糖后,进入胞内被立即转化为乙醇。因此,该技术为今后进行酿酒酵母理性改造以实现有效生物质转化提供了有力的技术支持。
综上所述,借助综合策略,从基因组、转录组数据库中甄选性能优良的外切/内切菊糖酶,并分析挖掘高强度启动子元件,有效启动下游高活性菊糖酶转录,随后利用酿酒酵母表面展示技术,实现外切/内切菊糖酶在细胞表面的共表达,有望最终获得适于工业化生产的发酵菊糖生产乙醇的优良工程菌。
[1]程序. 现代生物能源第二波研发和产业化浪潮[J]. 中外能源,2014,19(4):16-22.
[2]Cardona CA,Sánchez óJ. Fuel ethanol production:process design trends and integration opportunities[J]. Bioresource technology,2007,98(12):2415-2457.
[3]周正,曹海龙,朱豫,等. 菊芋替代玉米发酵生产乙醇的初步研究[J]. 西北农业学报,2008,17(4):297-301.
[4]华艳艳,赵鑫,赵金,等. 圆红冬孢酵母发酵菊芋块茎产油脂的研究[J]. 中国生物工程杂志,2007,27(10):59-63.
[5]Yang,F,Liu Z,Dong W,et al. Ethanol production using a newly isolated Saccharomyces cerevisiae strain directly assimilating intact inulin with a high degree of polymerization[J]. Biotechnology and applied biochemistry,2013,61(4):418-425.
[6]何秀萍. 国内酿酒酵母分子遗传与育种研究40年[J]. 微生物学通报,2014,41(3):450-458.
[7]Lim SH,Ryu JM,Lee H,et al. Ethanol fermentation from Jerusalem artichoke powder using Saccharomyces cerevisiae KCCM50549 without pretreatment for inulin hydrolysis[J]. Bioresource technology,2011,102(2):2109-2111.
[8]Onsoy T,Thanonkeo P,Thanonkeo S,et al. Ethanol production from Jerusalem artichoke by Zymomonas mobilis in batch fermentation[J]. KMITL Science and Technology Journal,2007,7:55-60.
[9]Chi Z,Chi Z,Zhang T,et al. Inulinase-expressing microorganisms and applications of inulinases[J]. Applied Microbiology and Biotechnology,2009,82(2):211-220.
[10]Kobayashi T,Uchimura K,Deguchi S,et al. Cloning and sequencing of inulinase andβ-fructofuranosidase genes of a deep-sea microbulbifer species and properties of recombinant enzymes[J]. Applied and environmental microbiology,2012,78(7):2493-2495.
[11]Zhou J,Peng M,Zhang R, et al. Characterization of Sphingomonas sp. JB13 exo-inulinase:a novel detergent-,salt-,and protease-tolerant exo-inulinase[J]. Extremophiles,2015,19(2):383-393.
[12]Arumugam M,Manivasagam P,Balasubramanian T,et al. Purification and characterization of inulinase from marine bacterium Bacillus cereus MU-31[J]. Indian Journal of Geo-Marine Sciences,2013,42(4):510-515.
[13]Laowklom N,Chantanaphan R,Pinphanichakarn P. Production,purification and characterization of inulinase from a newly isolated Streptomyces sp. CP01[J]. Natural Resources,2012,3(3):137-144.
[14]Cho YJ,Yun JW. Purification and characterization of an endoinulinase from Xanthomonas oryzae No. 5. Process Biochemistry,2002,37(11):1325-1331.
[15]Gao J,Xu YY,Yang HM, et al. Gene Cloning,Expression,and characterization of an exo-inulinase from Paenibacillus polymyxa ZJ-9. Applied Biochemistry and Biotechnology,2014,173(6):1419-1430.
[16]Gong F,Zhang T,Chi Z, et al. Purification and characterization of extracellular inulinase from a marine yeast Pichia guilliermondii and inulin hydrolysis by the purified inulinase[J]. Biotechnology and Bioprocess Engineering,2008,13(5):533-539.
[17]Zhang S,Yang F,Wang Q,et al. High-level secretory expression and characterization of the recombinant Kluyveromyces marxianus inulinase[J]. Process Biochemistry,2012,47(1):151-155.
[18]Yuan B,Hu N,Sun J,et al. Purification and characterization of a novel extracellular inulinase from a new yeast species Candida kutaonensis sp. nov. KRF1T[J]. Applied Microbiology and Biotechnology,2012,96(6):1517-1526.
[19]Nakamura T,Shitara A,Matsuda S,et al. Production,purification and properties of an endoinulinase of Penicillium sp. TN-88 that liberates inulotriose[J]. Journal of fermentation and bioengineering,1997,84(97):313-318
[20]Chen HQ,Chen XM,Li Y,et al. Purification and characterisation of exo-and endo-inulinase from Aspergillus ficuum JNSP5-06[J]. Food Chemistry,2009,115(4):1206-1212.
[21]Zhang T,Chi Z,Zhao CH,et al. Bioethanol production from hydrolysates of inulin and the tuber meal of Jerusalem artichoke by Saccharomyces sp. W0[J]. Bioresource Technology,2010,101(21):8166-8170.
[22]Ge XY,Zhang WG. A shortcut to the production of high ethanol concentration from Jerusalem artichoke tubers[J]. Food Technology and Biotechnology,2005,43(3):241-246.
[23]常宝磊,袁文杰,赵心清,等. 自絮凝颗粒酵母发酵菊芋汁生产乙醇[J]. 过程工程学报,2009,9(4):796-800.
[24]Yuan WJ,Li NN,Zhao XQ, et al. Engineering an industrial Saccharomyces cerevisiae strain with the inulinase gene for more efficient ethanol production from Jerusalem artichoke tubers[J]. Engineering in Life Sciences,2013,13(5):472-278.
[25]Du YC,Long XH,Liu ZP,et al. Optimizing medium for producing ethanol from industrial crop Jerusalem artichoke by one-step fermentation and recombinant Saccharomyces cerevisiae[J]. Plant Biosystems,2014,148(1):118-126.
[26]Yuan B,Wang SA,Li FL. Expression of exoinulinase genes in Saccharomyces cerevisiae to improve ethanol production from inulin sources[J]. Biotechnology Letters,2013,35(10):1589-1592.
[27]Zhang T,Chi Z,Chi Z,et al. Expression of the inulinase gene from the marine-derived Pichia guilliermondii in Saccharomyces sp. W0 and ethanol production from inulin[J]. Microbial Biotechnology,2010,3(5):576-582.
[28]Wang JM,Zhang T,Chi Z,et al. 18S rDNA integration of the exo-inulinase gene into chromosomes of the high ethanol producing yeast Saccharomyces sp. W0 for direct conversion of inulin to bioethanol[J]. Biomass and Bioenergy,2011,35(7):3032-3039.
[29]Zhang LL,Tan MJ,Liu GL,et al. Cloning and characterization of an inulinase gene from the marine yeast Candida membranifaciens subsp. flavinogenie W14-3 and its expression in Saccharomyces sp. W0 for ethanol production[J]. Molecular biotechnology,2015,57(4):337-347.
[30]Liu G.L,Fu G.Y,Chi Z,et al. Enhanced expression of the codon-optimized exo-inulinase gene from the yeast Meyerozyma guilliermondii in Saccharomyces sp. W0 and bioethanol production from inulin[J]. Applied Microbiology and Biotechnology,2014,98(2):9129-9138.
[31]杨华军,邹少兰,刘成,等.纤维素酶在酿酒酵母中的表达研究[J]. 中国生物工程杂志,2014,34(6):75-83.
[32]Hong SJ,Kim,HJ,Kim JW,et al. Optimizing promoters and secretory signal sequences for producing ethanol from inulin by recombinant Saccharomyces cerevisiae carrying Kluyveromyces marxianus inulinase[J]. Bioprocess and Biosystems Engineering,2015,38(2):263-272.
[33]Schorr-Galindo S,Ghommidh C,Guiraud JP. Influence of yeast flocculation on the rate of Jerusalem Artichoke extract fermentation[J]. Current Microbiology,2000,41(2):89-95.
[34]Hu N,Yuan B,Sun J,et al. Thermotolerant Kluyveromyces marxianus and Saccharomyces cerevisiae strains representing potentials for bioethanol production from Jerusalem artichoke by consolidated bioprocessing[J]. Applied Microbiology and Biotechnology,2012,95(5):1359-1368.
[35]Wang SA,Li FL. Invertase SUC2 is the key hydrolase for
inulin degradation in Saccharomyces cerevisiae[J]. Applied and environmental microbiology,2013,79(1):403-406.
[36]Yang F,Liu ZC,Wang X,et al. Invertase Suc2-mediated inulin catabolism is regulated at the transcript level in Saccharomyces cerevisiae[J]. Microbial cell factories,2015,14(1):59
[37]Yuan B,Wang SA,Li FL. Improved ethanol fermentation by heterologous endoinulinase and inherent invertase from inulin by Saccharomyces cerevisiae[J]. Bioresource Technology,2013,139:402-405.
[38]Li Y,Liu G.L,Chi ZM. Ethanol production from inulin and unsterilized meal of Jerusalem artichoke tubers by Saccharomyces sp. W0 expressing the endo-inulinase gene from Arthrobacter sp[J]. Bioresource Technology,2013,147:254-259.
[39]王兴春,杨致荣,王敏,等. 高通量测序技术及其应用[J]. 中国生物工程杂志,2012,32(1):109-114.
[40]柳延虎,王璐,于黎. 单分子实时测序技术的原理与应用[J]. 遗传,2015,03(3):259-268.
[41]杨非,曹萌,金怡,等. 酿酒酵母细胞表面展示技术在燃料乙醇生产中的应用及研究进展[J]. 生物工程学报,2012,28(8):901-911.
[42]Fujita Y,Ito J,Ueda M,et al. Synergistic saccharification,and direct fermentation to ethanol,of amorphous cellulose by use of an engineered yeast strain codisplaying three types of cellulolytic enzyme[J]. Applied and environmental microbiology,2004,70(2):1207-1212.
Advance in the improvement ethanol production from Jerusalem artichoke flour bySaccharomycescerevisiaeusing inulinase
LI Li-li,HOU Ying-min,GUO Xiao-yu,YANG Fan*,LI Xian-zhen
(School of Biological Engineering,Dalian Polytechnic University,Dalian 116034,China)
The exhaustion of fossil fuels and the serious environmental problems lead to the continued concerning on the ethanol production. Jerusalem artichoke,as non-food grain crop,is suitable for ethanol production because of its high inulin content and excellent ecological adaptability. However,ethanol-producingSaccharomycescerevisiaecould not directly assimilate inulin into ethanol due to lacking inulinase,which delays its application in the industrial production of inulin-based ethanol. This review summarized the advance in studying ethanol production from inulin using fermentation engineering and genetic engineering approaches,as well as the key role of inulinase for ethanol production byS.cerevisiae. A comprehensive strategy based on both omics data mining and yeast surface display technology was discussed for the rational construction ofS.cerevisiaeproducing ethanol from inulin.
ethanol;inulinase;fermentation engineering;genetic engineering
2015-11-16
李丽丽(1990-),女,硕士研究生,研究方向:分子微生物学,E-mail:13591829130@163.com。
杨帆(1982-),博士,副教授,研究方向:酿酒酵母代谢工程方面的研究,E-mail:yangfan@d/pu.edu.cn。
国家自然科学基金项目(31201302);辽宁省高等学校优秀人才支持计划(LJQ2015009)。
TS261.1
A
1002-0306(2016)11-0367-06
10.13386/j.issn1002-0306.2016.11.067
