生物传感器在代谢工程中的应用
2016-08-30张楠楠王金保董润安高海军北京理工大学生命学院北京100081
张楠楠 王金保 董润安 高海军(北京理工大学生命学院,北京,100081)
生物传感器在代谢工程中的应用
张楠楠 王金保 董润安 高海军*
(北京理工大学生命学院,北京,100081)
摘要:近年来,合成生物学、代谢工程的发展推动了精细化学品和新一代能源的生物合成研究,为高效能酶或菌株的构建和筛选带来了新的契机。生物传感器由分子识别元件和信号转换器组成。作为一种新的生物信号检测技术手段,具有选择性高、分析速度快、操作简单等特点。主要有荧光共振能量转移传感器、核糖开关传感器和转录因子传感器三种类型。其中,转录因子传感器已广泛应用于酶或菌株等的代谢工程研究中。文章综述了这些生物传感器的基本原理和应用。
关键词:生物传感器;代谢工程;应用;生物合成
近年来,合成生物学在基因组编辑[1],DNA组装[2],标准化基因线路的构建及逻辑线路的设计[3,4]等方面,取得了巨大的进步。特别是Gibson等温组装、Gold-gate 组装、连接酶链反应(LCR)[5]和自动基因编辑(MAGE)[6,7]等DNA组装技术的发展推动了基因型文库的理论与应用研究。基因型文库的构建作为一种优化菌株代谢系统的工具,在高产菌株的构建过程中具有关键作用。然而,构建基因型文库仅仅是获得高产菌株的基础,还需进一步从瀚如烟海的文库中筛选到最好的表型。目前而言,表型筛选技术远远落后于文库构建技术[8,9],限制了高产菌株的获取。生物传感器作为一种新的生物信号检测技术,具有选择性高、分析速度快、操作简单等特点,在食品检测、环境监测、新药筛选、工业微生物菌株改造等研究领域受到广泛重视[10]。
1 生物传感器(Biosensor)原理与类型
生物传感器由分子识别元件和信号转换器两部分组成,分子识别元件可接受外界某一信号刺激,接着利用转换器把外界信号转变为另一种可被检测的电信号或光信号,进而实现对外界环境的持续检测(图 1)。生物传感器往往针对某一特定配体如葡萄糖、脂肪酸、氨基酸[11]、NAD(P)H/NAD(P)+[12]等,或环境因子如pH、温度[11]等,通过转换器将强弱程度不同的信号转变为可见或可检测的表型。至今为止,生物传感器主要有三种类型,即荧光共振能量转移(Forster resonance energy transfer,FRET)传感器、核糖开关(Riboswitches)传感器和转录因子(Transcription factors ,TF)传感器。

图1 生物传感器原理
1.1FRET传感器
F R E T传感器包含一对供体和受体荧光素(fluorophores),及两荧光素间的配体结合肽(ligandbinding peptide)[13]。当目标配体与多肽结合,使该多肽的构象发生变化,进而受体和供体荧光素发生复杂的变化,产生荧光共振能量转移现象。这种类型传感器主要依赖于配体结合肽构象的变化,而与胞内复杂的转录和翻译过程无关,早些年被科研工作者广泛使用[14]。菌体中大量发现的可结合小分子代谢物的蛋白支架(metabolite-binding protein scaffolds)为该传感器提供了源源不断的资源,可用于检测菌体内某一代谢物如磷酸葡萄糖、氨基酸、羧酸[15](carboxylic acids)、辅因子和金属离子[16]的积累情况。此外,该传感器还能用于检测胞内氧化还原状态,及难于检测的胞内大分子物质。尽管FRET传感器存在诸多优点,如好的正交性、高的灵敏度和简易的构建过程,但由于其动态范围太窄,现在主要用于胞内某一代谢物的动态监测。
1.2核糖开关传感器
核糖开关传感器是一种新型的传感器。核糖开关是位于mRNA链上的一种特殊结构,与特异性配体结合后发生构象改变,最终调节该mRNA的转录及翻译过程[17]。核糖开关传感器不存在“蛋白与蛋白”或“蛋白与代谢物”的相互作用,而是代谢物小分子直接作用于mRNA链,其响应相比其他转录因子传感器更加迅速[18]。核糖开关传感器主要依赖mRNA键连区域,即适配体(Aptamer),胞内适配体文库决定该传感器的应用范围。适配体文库的构建可采用多种方法进行,如Ceres等[19]在大肠杆菌中构建了一个平台表达系统,用于寻找可键连小分子的适配体。此外,也可利用软件进行设计,从头合成针对某一小分子的适配体[20]。
现今为止,已研究的核糖开关大多存在于细菌中,而对酵母中的研究较少。Michener 等[21]利用核酶(ribozyme)依赖的核糖开关,筛选了酵母中的咖啡因脱甲基酶文库。如图2,在文献中,作者使用了一种咖啡因类似物茶碱响应的核酶(theophylline- responsive ribozyme),其可控制上游GFP的表达。当培养基中不存在茶碱,GFP基因可正常翻译,酵母有绿色荧光产生;当茶碱存在时,其与适配体结合,激活核酶切割GFP,该情况下不存在荧光。总体来说,核糖开关传感器拥有很好的发展前景,未来可被广泛应用于代谢工程表型筛选。
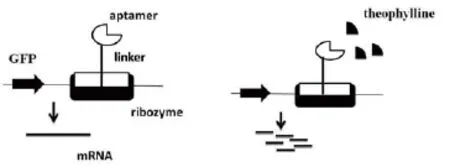
图2 核酶依赖的核糖开关传感器
1.3转录因子(TF)传感器
转录因子是广泛存在的一类感知蛋白,可以响应环境因子的改变,通过识别和结合基因启动子区的顺式作用元件进而控制下游基因的表达水平[22]。标准的TF传感器包括三部分,(1)能对某一信号响应的转录因子;(2)能被转录因子调节的启动子;(3)被启动子调节的报告基因。胞内存在的大量转录因子,为TF传感器提供了广泛的来源。如今,TF传感器已成为使用最为广泛的大通量筛选工具。以下综述的生物传感器的应用也主要指TF传感器。
2 TF生物传感器在代谢工程中的应用
2.1高活性酶的筛选
野生类型的酶由于受到胞内的严密调节,往往活性受到限制。定向进化技术是酶活性的改造的新策略,广泛应用在酶分子改造中。改造过程通常包括两步,首先进行定点突变或易错PCR获得酶的突变体文库,然后在庞大的突变体文库中筛选改性的酶突变体。针对某一小分子的TF传感器,已被用于胞内酶活性的筛选。如van Sint Fiet S等[23]使用来自假单胞杆菌(Pseudomonas putida)的转录因子NhaR对苯甲醛脱氢酶活性进行筛选,NhaR可有效监测胞内苯甲酸或2-羟基-苯甲酸浓度,通过诱导表达tetA(四环素转运蛋白),将苯甲醛脱氢酶活性与菌体生长相偶联,即在四环素抗性平板上,拥有高活性的酶的细菌生长更迅速。Uchiyama等[24]利用转录因子BenR,对大肠杆菌的苯酰胺酶进行活性筛选,通过将荧光强度于酶活性相偶联,成功从96000多个克隆中筛选到11种不同活性的酰胺酶。Tang等[25]利用改造过的转录因子AraC感知胞内的三乙酸内酯(TAL),实现了高活性2-吡喃酮合成酶的筛选,最终获得了提高20倍活性的目标酶。此外,利用生物传感器对高活性NADPH/NADH-依赖酶筛选的研究也较多[26,27]。该类传感器主要基于大肠杆菌中固有的SoxRS“双组份”调节系统,大肠杆菌可以通过该系统对外界或胞内氧化环境作出应激反应[28]。SoxR是由两个同源的二聚体构成的转录激活因子。结构中包含两个铁-硫簇([2Fe-2S]2-),可用于感知胞内氧化信号。当铁-硫簇呈现还原态时,SoxR为非活性状态;当铁-硫簇被氧化剂氧化后,SoxR呈现活性状态。活化的SoxR可激活SoxS的转录,SoxS可进一步激活一些酶的表达,如超氧化物歧化酶(sodA),6-磷酸葡萄糖脱氢酶(zwf)和延胡索酸酶(fumC)等。早在1992年,Liochev 和 Fridovich[29]利用SoxRS系统发明了一种可用于检测胞内NADPH浓度的传感器。具体传感器结构如图3,包括完整的SoxR基因,SoxS基因的启动子,黄色荧光蛋白基因。当胞内NADPH呈现较高浓度时,SoxR因其铁-硫簇未被氧化而表现为非活化状态,不能激活下游基因(荧光蛋白)的表达,因而,无黄色荧光产生;当胞内NADPH呈现低浓度时,SoxR被氧化激活,进而激活SoxS启动子,促进荧光蛋白的表达,最终产生出黄色荧光。

图3 依赖soxRS的NADPH传感器原理图
此外,Knudsen等[30]在酵母中构建了能对NADH/NAD+比率响应的TF传感器。文献中,作者使用GPD2启动子,GPD2为三磷酸甘油脱氢酶,负责酵母胞内NADH与NAD+的平衡。传感器报告基因为GFP,当胞内NADH较低时,启动子被激活,有绿色荧光产生。该传感器是首次被报道的可检测NADH浓度的传感器。
2.2高产菌的筛选
高产菌的获取是代谢工程所追求的主要目标之一。许多针对末端产物的TF传感器广泛应用于高产菌的直接筛选:Chen等[31]在大肠杆菌中构建了能感应四氢嘧啶(ectoine)的传感器。由于不存在能对四氢嘧啶响应的天然转录因子,作者通过改造AraC的疏水区域改变其配体分子类型,获得了能对四氢嘧啶响应的突变AraC。利用该传感器,四氢嘧啶产量明显提升;Dietrich等[32]利用来自丁醇假单胞杆菌(Pseudomonas butanovora)的转录因子BmoR及其诱导型启动子PBMO在大肠杆菌中构建了能对正丁醇响应的传感器。通过融合表达tetA和GFP,将胞内正丁醇浓度与菌体生长情况及荧光强度相偶联,最终大幅度提高了正丁醇产量;Parales等[33]利用来自恶臭假单胞菌的转录因子PcaR,及其诱导型启动子PpcaIJ,在大肠杆菌中构建了能对己二酸响应的传感器,通过测定荧光强度,获得了己二酸高产菌;此外,Davies等[34]利用大肠杆菌中固有的DcuR/DcuS双组份转录因子,及其诱导型启动子dctA,构建了能对胞内琥珀酸检测的传感器,优化了琥珀酸合成途径,提高了琥珀酸产量。
2.3动态调控蛋白表达
生物传感器中的报告基因除了常见的抗生素抗性基因、荧光蛋白和LacZ外,还可以是特殊基因或调控线路。Zhang等[36]在大肠杆菌中构建了可调节的脂肪酸代谢通路。通过使用能对脂肪酸响应的转录因子FadR构建了传感器,可有效监测胞内脂肪酸或脂肪酰辅酶A浓度。如图 4所示,当脂肪酸不积累时,FadR表达能抑制PmodB和PmodC,使乙醇不会积累;当脂肪酸积累时,脂肪酸或脂肪酰辅酶A与FadR结合,促进PmodB和PmodC表达,大量合成终产物FAEE。该类传感器实现了对生物合成途径的调控,胞内既可以避免积累有毒性的乙醇和脂肪酸,也同时实现产量大幅度的提升;Rogers等[37]利用来自Rhodobacter sphaeroides的转录因子acuR设计了可对3-羟基丙酸(3HP) 浓度响应的传感器,通过实时监控胞内3HP浓度的变化,优化了发酵条件,3HP的产量最终提高了23倍。

图4 动态脂肪酸代谢传感器[36]
2.4其他应用
Hwang等[38]在大肠杆菌中设计了可杀灭铜绿假单胞菌的生物传感器。利用能对高丝氨酸内酯(HSL)响应的转录因子LasR及其诱导型启动子,控制杀菌肽mcsS和DnasesI基因表达。大肠杆菌的LasR可以捕获培养基中铜绿假单胞菌生长时释放的群体效应分子HSL,进而激活大肠杆菌中杀菌肽和DNA酶的表达,达到杀灭病原菌的目的。Jonathan等[39]在肠道共生大肠杆菌中构建了四环素传感器,通过喂食小鼠,有效监测了小鼠肠道中四环素的含量。
3 展望
随着生物技术的不断进步,可实现许多具有高价值的化学品和新一代能源的生物合成。工业规模生物合成过程的建立主要受限于过低的底物转化率和生产效率。近年来,生物传感器已广泛应用于生物合成途径的系统优化和实时监测中。可以预料,生物传感器在高效能的酶或菌株的构建与筛选中的巨大潜力将推动生物技术的工业化进程。此外,随着社会的进一步信息化,生物传感器也必将获得越来越广泛的应用。
参考文献
[1] Gibson D G, Benders G A, Andrews P C, et al. Complete chemical synthesis, assembly, and cloning of a Mycoplasma genitalium genome[J]. science, 2008, 319(5867): 1215-1220.
[2] Ellis T, Adie T, Baldwin G S. DNA assembly for synthetic biology: from parts to pathways and beyond[J]. Integrative Biology, 2011, 3(2): 109-118.
[3] Lapique N, Benenson Y. Digital switching in a biosensor circuit via programmable timing of gene availability[J].Nature chemical biology, 2014, 10(12): 1020-1027.
[4] Nandagopal N, Elowitz M B. Synthetic biology: integrated gene circuits[J]. Sience, 2011, 333(6047): 1244-1248.
[5] Roth T L, Milenkovic L, Scott M P. A rapid and simple method for DNA engineering using cycled ligation assembly[J]. PloS one, 2014, 9(9): e107329.
[6] Wang H H, Isaacs F J, Carr P A, et al. Programming cells by multiplegenome engineering and accelerated evolution[J]. Nature, 2009, 460(7257): 894-898.
[7] Jeong J, Cho N, Jung D, et al. Genome-scale genetic engineering in Escherichia coli[J]. Biotechnology advances, 2013,31(6): 804-810.
[8] Gibson D G, Young L, Chuang R Y, et al. Enzymatic assembly of DNA molecules up to several hundred kilobases[J]. Nature methods, 2009, 6(5): 343-345.
[9] Shivange A V, Marienhagen J, Mundhada H, et al. Advances in generating functional diversity for directed protein evolution[J]. Current opinion in chemical biology, 2009, 13(1): 19-25.
[10] Vigneshvar S, Sudhakumari C C, Senthilkumaran B,et al. Recent Advances in Biosensor Technology for Potential Applications-An Overview[J]. Frontiers in bioengineering and biotechnology, 2016, 4.
[11] Mohsin M, Ahmad A. Genetically-encoded nanosensor for quantitative monitoring of methionine in bacterial and yeast cells[J]. Biosensors and Bioelectronics, 2014, 59: 358-364.
[12] Tang J L, Li C Y, Li Y F, et al. A ratiometric fl uorescent probe with unexpected high selectivity for ATP and its application in cell imaging[J]. Chemical Communications, 2014, 50(97): 15411-15414.
[13] Bermejo C, Haerizadeh F, Takanaga H, et al. Optical sensors for measuring dynamic changes of cytosolic metabolite levels in yeast[J]. Nature Protocols, 2011, 6(11):1806-1817.
[14] San Martín A, Ceballo S, Baeza L F, et al. Imaging mitochondrial fluin single cells with a FRET sensor for pyruvate[J]. PLoS One, 2014, 9(1): e85780.
[15] Peroza E A, Ewald J C, Parakkal G, et al. A genetically encoded Förster resonance energy transfer sensor for monitoring in vivo trehalose-6-phosphate dynamics[J]. Analytical biochemistry,2015, 474: 1-7.
[16] Vinkenborg J L, Nicolson T J, Bellomo E A, et al. Genetically encoded FRET sensors to monitor intracellular Zn2+ homeostasis[J]. Nature methods, 2009, 6(10): 737-740.
[17] Serganov A, Nudler E. A decade of riboswitches[J]. Cell, 2013, 152(1): 17-24.
[18] Berens C, Suess B. Riboswitch engineering-making the all-important second and third steps[J]. Current opinion in biotechnology, 2015, 31: 10-15.
[19] Ceres P, Garst A D, Marcano V J G, et al. Modularity of select riboswitch expression platforms enables facile engineering of novel genetic regulatory devices[J]. ACS synthetic biology,2013, 2(8): 463-472.
[20] Wachsmuth M, Findei B S, Weissheimer N, et al. De novo design of a synthetic riboswitch that regulates transcription termination[J]. Nucleic acids research, 2013, 41(4): 2541-2551.
[21] Michener J K, Smolke C D. High-throughput enzyme evolution in Saccharomyces cerevisiae using a synthetic RNA switch[J]. Metabolic engineering, 2012, 14(4): 306-316.
[22] Dietrich J A, Shis D L, Alikhani A, et al. Transcription factor-based screens and synthetic selections for microbial smallmolecule biosynthesis[J]. ACS synthetic biology, 2012, 2(1): 47-58.
[23] Van S F S, Van B J B, van Beilen J B, Witholt B. Selection of biocatalysts for chemical synthesis[J]. Proceedings of the National Academy of Sciences of the United States of America, 2006, 103(6): 1693-1698.
[24] Uchiyama T, Miyazaki K. Product-induced gene expression, a product-responsive reporter assay used to screen metagenomic libraries for enzyme-encoding genes[J]. Applied and environmental microbiology, 2010, 76(21): 7029-7035.
[25] Tang S Y, Qian S, Akinterinwa O, et al. Screening for enhanced triacetic acid lactone production by recombinant Escherichia coli expressing a designed triacetic acid lactone reporter[J]. Journal of the American Chemical Society, 2013,135(27): 10099-10103.
[26] Siedler S, Schendzielorz G, Binder S, et al. SoxR as a single-cell biosensor for NADPH-consuming enzymes in Escherichia coli[J]. ACS synthetic biology, 2013, 3(1): 41-47.
[27] Zhao F L, Zhang C, Zhang C, et al. A genetically encoded biosensor for in vitro and in vivo detection of NADP+[J]. Biosensors and Bioelectronics, 2016, 77: 901-906.
[28] Watanabe S, Kita A, Kobayashi K, et al. Crystal structure of the [2Fe-2S] oxidative-stress sensor SoxR bound to DNA[J]. Proceedings of the National Academy of Sciences, 2008,105(11): 4121-4126.
[29] Liochev S I, Fridovich I. Fumarase C, the stable fumarase of Escherichia coli, is controlled by the soxRS regulon[J]. Proceedings of the National Academy of Sciences,1992, 89(13): 5892-5896.
[30] Knudsen J D, Carlquist M, Gorwa GM. NADH-dependent biosensor in Saccharomyces cerevisiae: principle and validation at the single cell level[J]. AMB Express, 2014, 4(1): 81.
[31] Chen W, Zhang S, Jiang P, et al. Design of an ectoineresponsive AraC mutant and its application in metabolic engineering of ectoine biosynthesis[J]. Metabolic engineering,2015, 30: 149-155.
[32] Dietrich J A, Shis D L, Alikhani A, et al. Transcription factor-based screens and synthetic selections for microbial smallmolecule biosynthesis[J]. ACS synthetic biology, 2012, 2(1): 47-58.
[33] Parales R E, Harwood C S. Regulation of the pcaIJ genes for aromatic acid degradation in Pseudomonas putida[J]. Journal of bacteriology, 1993, 175(18): 5829-5838.
[34] Davies S J, Golby P, Omrani D, et al. Inactivation and regulation of the aerobic C4-dicarboxylate transport (dctA) gene of Escherichia coli[J]. Journal of bacteriology, 1999, 181(18): 5624-5635.
[35] Rogers J K, Taylor N D, Church G M. Biosensor-based engineering of biosynthetic pathways[J]. Current Opinion in Biotechnology, 2016, 42: 84-91.
[36] Zhang F, Carothers J M, Keasling J D. Design of a dynamic sensor-regulator system for production of chemicals and fuels derived from fatty acids[J]. Nature biotechnology, 2012,30(4): 354-359.
[37] Rogers J K, Church G M. Genetically encoded sensors enable real-time observation of metabolite production[J]. Proceedings of the National Academy of Sciences, 2016, 113(9): 2388-2393.
[38] Hwang I Y, Tan M H, Koh E, et al. Reprogramming microbes to be pathogen-seeking killers[J]. ACS synthetic biology, 2013, 3(4): 228-237.
[39] Kotula J W, Kerns S J, Shaket L A, et al. Programmable bacteria detect and record an environmental signal in the mammalian gut[J]. Proceedings of the National Academy of Sciences, 2014, 111(13): 4838-4843.
⋆国家重大科学仪器专项(No. 2012YQ0401400802)
中图分类号:Q81
文献标识码:A
DOI:[CLC Number] Q81[Document Code] A10.11967/2016140203 10.11967/2016140303
作者简介:张楠楠,女,理学硕士,Email:zhangnannan1216@126.com
通讯作者:高海军,男,副教授,硕士生导师,Email:hj_gao@bit.edu.cn
Application of Biosensor in Metabolic Engineering
Nannan Zhang, Jinbao Wang, Runan Dong, Haijun Gao*
( School of Life Science, Beijing Institute of Technology, Beijing, 100081 )
Abstract:Nanobiomedicine is a newly born subject with particularly rapid development. It is formed with the cross by biology, medicine and science. Nanobiomedicine achieved the precisely control ofmaterial structure from the scale of nanometer. Accordingly, itprovides a new medical thinking andtechnology for Modern Biomedicine. This article mainly reviews the current state application status and progress ofnano biomedical technology in disease diagnosis and treatment. Understanding the nanotechnology application status in this field has very important theoretical significance and practical value.
Key words:Nanotechnology; Nanobiomedicine; Nanodiagnosis; Nanotoxicology
