利用统计语言模型对GenoCAD设计结果进行优化
2016-08-29张社民
方 刚,张社民
(1.西安文理学院 生物与环境工程学院, 陕西 西安 710065; 2.陕西理工大学 管理学院, 陕西 汉中 723001)
利用统计语言模型对GenoCAD设计结果进行优化
方刚1,张社民2
(1.西安文理学院 生物与环境工程学院, 陕西 西安 710065; 2.陕西理工大学 管理学院, 陕西 汉中 723001)
GenoCAD(www.genocad.com)是一种基于Web的免费合成生物学设计软件,使用它可以进行表达载体及人工基因网络设计。不断地点击代表各种合成生物学标准“零件”的图标,以一种语法进行设计,最后就可以得到由数十个功能片段组成的复杂质粒载体。但是一般来讲在GenoCAD中,每一类的合成生物学标准“零件”数量众多。随着这些标准“零件”的不断开发,其数量也在进一步增加,目前选择合适的“零件”组装成功能性的质粒载体费时费力并且容易发生错误。在进行载体设计的最后阶段,从众多的“零件”中选择合适的往往比较困难。为解决这一问题,采用自然语言处理的统计语言模型,并以该模型为基础应用动态规划算法优化质粒载体设计,从众多的选项中找出最优者。利用这一方法可以减少进行生物学实验的冗余操作,从而减少载体构建过程中的花费。
合成生物学;统计语言模型;动态规划算法;GenoCAD
一、引 言
由美国弗吉尼亚理工大学生物信息研究所开发维护的合成生物学设计工具GenoCAD是基于网络环境的而且免费使用[1]。这一工具软件不仅仅用于表达载体的设计,而且还可以用于基因及代谢网络研究[2-3]。它通过编制一定的“语法”,将每一个合成生物学标准“零件”看做一个词,用来设计所需的合成生物学构件[4]。在GenoCAD的设计中,启动子、核糖体结合位点、基因及终止子都属于各自的“词类”,然后依据特殊编制的语法来设计分子生物学组件[5-6]。设计者们往往将已有的生物序列拆成“零件”来作为合成生物学的标准“零件”[7],而当设计者将这些属于不同词类的标准“零件”进行组装时,其过程通常耗时、费力而且容易发生错误。为了克服这些问题,研究者们引入了一些组装标准,比如BioBrick基金(BioBrickFoundation,BBF)倡导的一些标准。属于同一组装标准的“零件”两侧序列包含相应的限制性酶切位点,使用同一组限制性内切酶和连接酶可以将同一标准的“零件”连接起来[8-9]。在一个设计软件中,也会模拟使用这些组装标准。对这些合成生物学标准“零件”的模拟组装可以在计算机中完全自动化[10],但是这样只是考虑了最基本的“语法”,也忽略了以往的组装经验。在GenoCAD中,使用者可以根据自己需要编制“语法”,再根据一些具有生物学意义的设计规则变换设计结构,最后选择“零件”完成设计[11]。但是越来越多的“零件”被输入数据库,在设计的最后一步,设计者往往不知道选择哪个“零件”更为合适。为了解决这个问题,统计语言模型(statisticallanguagemodel,SLM)被引入设计中。统计语言模型(SLM)最初用于自然语言识别[12],它用来估算一组词串成为一个正确语句的概率的大小。它最初也是目前最主要的应用是语音识别,除此之外还应用于机器翻译、分词、智能输入法及文本语音转换。本文介绍的统计语音模型通过统计BioBrick合成生物学标准件的一些参数,将BioBrick的组装过程转化为统计语音模型,然后使用动态规划算法找出合适的“零件”组装成最后的表达载体而完成设计。这一算法将iGEM(InternationalGeneticallyEngineeredMachineCompetition)竞赛设计的经验考虑进来,以减少时间和花费。这一方法不仅可以用来优化其它工具软件的设计方案,而且可以独立使用设计新的基因表达载体及分子生物学组件。
二、材料方法
通过链接http://parts.igem.org/das/parts/entry_points/ 下载BioBrick合成生物学组件信息,2014年1月的版本包含7 242个合成生物学组件。编写一个Perl脚本通过链接http://parts.igem.org/das/parts/features/?segment=part# 分析并提取每个组件的信息。将每个组件的信息分解排列成特有的结构输入MySQL数据库。输入数据库后共分解提取出 75 744个“零件”,这些“零件”包括基础“零件” (启动子、核糖体结合位点、基因编码序列、终止子及质粒序列) 和复合“零件”。这些复合“零件”依然由基础“零件”组装而成。通过查询MySQL数据库提取出这些基础“零件”并统计它们的使用频数。同时编写一个Perl脚本和一些SQL语句分析复合“零件”,统计出相邻接的两个基础“零件”的使用频数。通过查询数据库,共提取出1 682个基础“零件”并符合RFC23组装标准[13]。这意味着这些基础“零件”序列中(除了两侧的连接序列)不会包含这一组装标准使用的限制性酶切位点。这1 682个基础“零件”包含405个启动子,42个核糖体结合位点,57个终止子以及1 178个基因序列。这些基础“零件”将被用来设计基因表达载体。同时每一个基础“零件”的使用频率和每一对基础“零件”的使用频率都可以被计算出来。
(一)语法模型
如何编制设计合成生物学组件的语法在以前的文献中已予以详细描述[14],可以根据不同的目的编制不同的语法。本文使用的语法与文献[14]使用的上下文无关的语法类似(见表1),其中规则1表示开始设计,规则2表示将一个表达盒变成两个,规则3表示翻转一个表达盒,规则4表示将一个表达盒变成一个启动子加一个阅读框加一个终止子,规则6表示将一个阅读框变成两个,规则7表示将一个阅读框变成一个核糖体结合位点与一个基因,规则8表示将一个终止子变成两个,规则9表示将一个基因变成两个。与文献[14]不同的是增加了第5条规则以表达融合蛋白,它使一个表达盒变成一个启动子加一个阅读框,以便在一个表达盒里让两个蛋白融合表达。表1给出了本文使用的详细语法。
(二)数学模型
在一些设计软件中,比如GenoCAD在完成设计的最后一步设计者往往要从大量的基础“零件”中选择合适的“零件”以完成设计(图1),这一过程往往是困难的。为解决这一问题,本文引入了在语音识别、机器翻译、智能输入法中广泛应用的统计语言模型。

表1 详细语法表

图1 在设计软件GenoCAD最后一步的多个选项
在这一模型中,一个句子(sentence,S)是否有意义并且合理基于其出现的概率。一个句子由一系列的词组成,在本文中一个“句子”S就是一个由基础“零件”构成的生物学组件,这些基础“零件”就是组成“句子”的词,一个基础“零件”part就是一个词。因此,S=part1,part2,…,partn需要知道其发生概率P(S)的大小:
P(S)=P(part1,part2,…,partn)
(1)
根据条件概率公式有:
P=(part1,part2,…,partn)
=P(part1)P(part2|part1)P(part3|part1,part2)…P(partn|part1,part2,…,partn-1)
(2)
式(2)中,P(part1) 指一个基础“零件”在一个设计中出现的概率。P(part2︱part1)指part1出现在part2之前part2出现的概率。根据式(2),partn出现的概率由所有出现在它之前的基础“零件”确定。其中 P(part1)andP(part2︱part1) 容易计算,但是计算P(part3︱part1,part2)的难度较大,而计算P(partn︱part1,part2,…,partn-1) 将非常困难,因为牵扯的变量太多导致条件过于复杂而难以估算。基于马尔科夫假设,可以认为一个“零件”在一个设计中出现的概率仅仅与它相邻的前一个“零件”相关。因此式(2)可以简化为:
P=(S)
=P(part1)P(part2|part1)P(part3|,part2)…P(parti|parti-1)…P(partn|partn-1)
(3)
现在P(S)即一个“句子”发生的概率就可以被计算出来了。式(3)就是统计语言模型中的二元模型(BigramModel)。因此,根据条件概率公式:
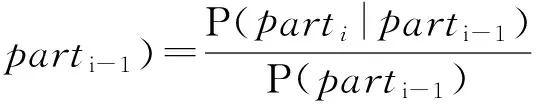
(4)
两个相邻基础“零件”出现的频率及单个基础“零件”出现的频率是可以被计算出来的,我们用它们来估计式(4)的条件概率。可得:

(5)
根据式(5),式(3)中任何一个成份都可以被计算出来。
设计的最后一步(图1),可以有很多个基础“零件”组合而成最后的设计。但是哪一种组合最合理且最有意义?根据统计语言模型理论,概率最大那一个将是最合理且最有意义的。在如图1所示网格结构中,可以有很多个候选路径产生“句子”,一条路径产生一个“句子”,一个“句子”就是一个设计(apath=a S=part1,part2,…,partn)。 最优的路径由PATH表示:

为避免计算时内存溢出,我们对P(S)取对数值:
PATH

(6)
根据式(5),得到了式(7)和式(8)
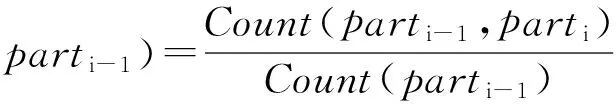
(7)
(8)
因为我们从一个相对稀疏的语料库中提取参数,零概率问题不可避免。为克服这一问题我们采用卡茨退避法进行数据平滑[15]。式(7)和(8)将被用来计算式(6)中各个成分的值从而得出最优路径。最优路径PATH将是在所有路径中具有最大概率的那一个。这里使用动态规划算法找出最优路径PATH。
(三)算法
在如图1的网格结构中找出最优的路径,这一路径将由一系列基础“零件”构成而它具有最大的出现概率。找最优路径的过程就是如何解式(6),具体的算法还是源于维特比算法[16],由三个步骤组成:
第一步,建立候选网格。每一类基础“零件”对应一列,而每列中的每一个节点对应一个基础“零件”。在网格的开始和结束添加BEG和END列。在这两列中两个虚拟节点B和E被添加进来(图 2), 每一个节点是一个三元组
全区设立有自治区级、市级、县级、乡镇四级社会保险征收服务机构,而税务部门根据税源变化的特点,已收缩乡镇征收机构,大多集中到县级,对城乡居民养老保险和医疗保险,尤其乡镇居民保险的征收管理带来一定程度的不便。
第二步,填充网格。从左至右填充网格,对于每一个三元组
1.对第一列,节点B使V=0且P=NULL。
2.对第二列每一节点三元组
V=VB+logP(part)=logP(part)
P=address_of_B
3.对第三列每一节点三元组
V=max{Vprior+logP(part|partprior)}
P=address_where_V_comes_from
4.重复3,在当前列中的每一个节点都与前一列的每一个节点组合计算其V值和P。
5.在END列,E节点的V将由选自前一列的最大值填充,P将存储前一列那个最大值节点的地址信息。
第三步,回溯找出最优路径PATH。从节点E开始不断找出前面节点的P (图 2),最终最优路径PATH将是具有最大概率的那一个,其产生的“句子”S就是最后设计的具有生物学意义的分子生物学组件。如果S的长度是L而一列中节点的个数最多是D,该算法的算法复杂度是O(L·D2),而穷举算法的算法复杂度是O(DL)。
三、结 果
为展示如何将BioBrick基础“零件”组装成功能性的合成生物学组件,我们挑选了一个可以产生香蕉气味的质粒 (http://parts.igem.org/Part:BBa_J45900) 。该质粒由麻省理工学院参加2006年iGEM竞赛的参赛队设计并实施。该组件包含两个表达盒:一个盒子包含BAT2 和THI3 基因,另一个表达盒的产物催化前面基因的产物而使大肠杆菌发出香蕉的气味,最后的设计如图1所示。
当确定了我们要表达的基因,该装配算法由一个Perl脚本执行,首次执行算法它给出序列R0040-B0034-J45008-B0030-J45009-R0040-B0034-J45014-B0010-B0012。
第二次执行算法,排除核糖体结合位点B0034,算法给出序列R0040-B0030-J45008-B0030-J45009-R0040-B0030-J45014-B0010-B0012。
第三次执行算法,当在第一列排除启动子R0040,算法给出序列R0011-B0030-J45008-B0030-J45009-R0040-B0030-J45014-B0010-B0012。
由这些基础“零件”构成的序列就是产生香蕉气味这一合成生物学组件的真实组成。如果进行其它的设计并执行算法,该方法将给出一个优化的结果,这一结果采用了以往设计的经验。如果需要更多的选项,我们可以排除一些“零件”并重新执行算法,它将给出其它一些优化的结果供选择。如果已知某些“零件”是确定相邻接的,使用者可以先确定这些连接然后执行算法。

图2 建立网格、填充网格及回溯过程图
四、讨 论
本文通过将BioBrick基础“零件”的装配过程转化成一个二元统计语言模型,然后执行动态规划算法找出最优的装配结果。算法可被迭代从而给出不同的优化结果供选择。这一方法不仅可以用来优化其它合成生物学软件设计结果,而且可以独立使用来模拟装配合成生物学基因片段产生表达载体。依据一定的语法输入不同类的合成生物学基础“零件”,算法依据以往经验自动选择合适的“零件”装配成合成生物学组件。采用这一方法可以减少真实装配过程的冗余操作,从而节省时间和费用。如前所述,该方法采用二元统计语言模型,这意味着每一个“零件”只与它前面一个相邻的“零件”有联系。然而在真实的分子生物学环境中,一个基因能否高效表达不仅与其核糖体结合位点有关,而且与其启动子有关。考虑N元模型,这意味着每一个“零件”与它前面N-1个“零件”有关系,但是这时条件概率是非常难以计算的。当N=3或4时,尽管在其它统计语音模型应用范例中(如机器翻译、分词、智能输入法)准确率会大大提高,但是计算量也大大增加,这时需要功能强大的计算机[12]121-122。下一步,我们将开发一个3元模型并且将质粒序列考虑进来,从而更高效地模拟合成生物学基因片段的组装过程并使真实的组装变得更为方便快捷。
当计算条件概率时,本文使用了卡茨退避数据平滑技术解决零概率问题。目前统计语言模型在合成生物学中鲜有应用,我们无从知道哪一种数据平滑技术更有效。下一步开发3、4元模型时,我们将扩大语料库,并考虑Good-Turing估计、线性插值法等数据平滑技术以提高准确率[17-18]。正如前述,我们从iGEM(InternationalGeneticallyEngineeredMachineCompetition)网站下载了一个相对稀疏的语料库。目前我们考虑将语料库扩展到常用的、商业化的表达载体上统计相应的参数,这样统计语言模型可以在合成生物学中更广泛地应用并得到检验。但是对这些合成生物学片段进行描述的术语还没有完全统一,因此发展合成生物学开放语言(SyntheticBiologyOpenLanguage,SBOL)变得十分必要。
感谢美国弗吉尼亚理工大学生物信息研究所(VirginiaBioinformaticsInstitute)的JeanPeccoud教授和MandyWilson指导作者搜集数据。
[1]CzarMJ,CaiY,PeccoudJ.WritingDNAwithGenoCAD[J].NucleicAcidsRes, 2009, 37(4).
[2]GolerJA,BramlettBW,PeccoudJ.GeneticDesign:RisingAbovetheSequence[J].TrendsBiotechnol, 2008, 26(6).
[3]GraslundS,NordlundP,WeigeltJ,HallbergBM,BrayJ,GileadiO,KnappS,OppermannU,ArrowsmithC,HuiR.etal.ProteinProductionandPurification[J].Nat.Methods, 2008,39(5).
[4]CaiY,WilsonML,PeccoudJ.GenoCADforiGEM:AGrammaticalApproachtotheDesignofStandard-CompliantConstructs[J].NucleicAcidsRes, 2010, 38(6).
[5]IsaacsFJ,DwyerDJ,DingC,Pervouchine,DD,CantorCR.EngineeredRiboregulatorsEnablePosttranscriptionalControlofGeneExpression[J].Nat.Biotechnol, 2004, 22(1).
[6]GardnerTS,CantorCR,CollinsJJ.ConstructionofaGeneticToggleSwitchinEscherichiaColi[J].Nature, 2000, 403(8).
[7]AdamesNR,WilsonML,FangG,LuxMW,GlickBS,PeccoudJ.GenoLIB:ADatabaseofBiologicalPartsDerivedfromaLibraryofCommonPlasmidFeatures[J].NucleicAcidsRes, 2015, 43(6).
[8]ArkinA.SettingtheStandardinSyntheticBiology[J].Nat.Biotechnol, 2008, 26(2).
[9]CantonB,LabnoA.EndyD.RefinementandStandardizationofSyntheticBiologicalPartsandDevices[J].Nat.Biotechnol, 2008, 26(2).
[10]DensmoreD,HsiauTHC,BattenC,KittlesonJT,DeLoacheW.AlgorithmsforAutomatedDNAAssembly[J].NucleicAcidsRes, 2010, 38(6).
[11]CollA,WilsonML,GrudenK,PeccoudJ.Rule-BasedDesignofPlantExpressionVectorsUsingGenoCAD[J].PLoSONE, 2015, 10(7).
[12]JelinekF.StatisticalMethodsforSpeechRecognition(Language,Speech,andCommunication)[M].Combridge:MITPress,1998.
[13]PhillipsIE,SliverPA.ANewBiobrickAssemblyStrategyDesignedforFacileProteinEngineering[EB/OL].DSpace@MIT, 2006,http://dspace.mit.edu/handle/1721.1/32535.
[14]CaiY,HartnettB,GustafssonC.PeccoudJ.ASyntacticModeltoDesignandVerifySyntheticGeneticConstructsDerivedfromStandardBiologicalParts[J].Bioinformatics, 2007, 23(1).
[15]ChenSF,GoodmanG.AnEmpiricalStudyofSmoothingTechniquesforLanguageModeling[J].ComputerSpeechandLanguage, 1999, 13(2).
[16]ViterbiAJ.APersonalHistoryoftheViterbiAlgorithm[J].IEEESignalProcessingMagazine,2006, 23(4).
[17]HuangFL,YuMS,HwangCY.AnEmpiricalStudyofGood-TuringSmoothingforLanguageModelsonDifferentSizeCorporaofChinese[J].JournalofComputerandCommunications, 2013, 22(1).
[18]KatzSM.EstimationofProbabilitiesfromSparseDatafortheLanguageModelComponentofaSpeechRecogniser[J].IEEETransactionsonAcoustics,Speech,andSignalProcessing, 1987, 35(6).
OptimizingGenoCADDesignbyUsingStatisticalLanguageModel
FANGGang1,ZHANGShe-min2
(1.SchoolofBiologicalandEnvironmentalEngineering,Xi'anUniversity,Xi'an710065,China;2.SchoolofManagement,ShaanxiSci-TechUniversity,Hanzhong723001,China)
GenoCAD(www.genocad.com)isafreeweb-basedapplicationthatguidesuserstodesignproteinexpressionvector,artificialgenenetworksandothergeneticconstructscomposedofgeneticparts.Bysuccessivelyclickingiconsrepresentingactualgeneticpartsaccordingtoagrammaticalmodel,complexgeneticconstructscomposedofdozensoffunctionalblockscanbedesigned.Butatthelaststepofdesign,usuallyeveryiconrepresentinggeneticpartshasitsoption.Withtheincreasingofgeneticpartsdatabase,moreandmorepartsareimportedintoGenoCADlibrary.Theprocessofassemblingmorethanafewsetsofgeneticpartscanbecostly,timeconsuminganderrorprone,anditissomewhatdifficulttomakedecisionwhichpartshouldbeselected.Basedonstatisticallanguagemodel,adynamicprogrammingalgorithmisdesignedtosolvetheproblemandoptimizestheresultsofGenoCADdesign.Inthisway,redundantoperationscanbereducedandthetimeandcostrequiredforconductingbiologicalexperimentcanbeminimized.
syntheticbiology;statisticallanguagemodel;dynamicprogrammingalgorithm;GenoCAD
2016-03-25;修复日期:2016-04-26
国家自然科学基金项目《蛋白质介导的核酸自组装体系及其计算问题研究与探索》(61173113)
方刚,男,陕西咸阳人,理学博士,副教授,研究方向:生物信息学;
F222∶TN912.3
A
1007-3116(2016)08-0020-06
(责任编辑:马慧)
张社民,男,陕西商洛人,理学博士,教授,研究方向:图与组合优化。
【统计理论与方法】
