主要作物中PAL基因家族的鉴定和序列分析
2016-07-25盖江涛沈建凯王鹏
盖江涛+++沈建凯++王鹏

摘要:苯丙氨酸解氨酶(PAL,phenylalanine ammonia-lyase [EC:4.3.1.24])存在于各种植物和部分微生物中,是与植物抗病性相关的关键酶,具有重要的植物生理意义。采用BLASTP的方法,依托全基因组数据库,获得了拟南芥[Arabidopsis thaliana (L.) Heynh.]、水稻(Oryza sativa L.)、玉米(Zea mays Linn.)、小麦(Triticum aestivum Linn.)、谷子[Setaria italica (L.) P. Beauv.]、大豆[Glycine max (Linn.) Merr.]6种植物中的PAL基因家族共45条序列,对其进行了系统进化分析、生物信息学分析等。分析结果显示:单子叶植物与双子叶植物分别聚集在不同的分支,说明单子叶植物水稻、玉米、小麦、谷子亲缘关系较近,双子叶植物拟南芥、大豆亲缘关系较近,而单子叶植物与双子叶植物亲缘关系较远,也说明PAL基因的分化在单子叶植物和双子叶植物分化前形成;酸性蛋白质占93.3%,稳定性蛋白占97.8%,有信号肽的蛋白占2.2%,有导肽的蛋白占4.4%,所有蛋白均为有明显跨膜现象的亲水性蛋白;所有PAL基因亚细胞定位于细胞质中,都有蛋白活性位点。分析结果为进一步研究作物中PAL代谢机理提供理论支持。
关键词:苯丙氨酸解氨酶;PAL;基因家族;氨基酸
中图分类号: Q78文献标志码: A
文章编号:1002-1302(2016)06-0045-05
苯丙氨酸解氨酶(phenylalanine ammonia-lyase,PAL,EC:4.3.1.24)存在于各种植物和少数微生物中,是目前研究较多的与植物抗病性相关的酶,与植物抗毒素及酚类化合物的形成密切相关[1-2]。PAL不能直接抵御病虫害,但在植物体内通过苯丙烷类途径进一步转化为木质素、黄酮、异黄酮、生物碱等次生代谢产物来增强植株的抗性[3-4]。
1961年,Koukol等首次记录了高等植物中PAL酶的提取分离[5],此后关于这种酶的研究迅速展开,目前PAL已从水稻[6]、小麦[6]等多种植物中得到分离纯化,并对拟南芥[7]、水稻[8]等植物PAL基因进行了cDNA克隆和序列分析。
研究发现,不同种植物中PAL活性不同,如水稻叶的PAL活性远比小麦的高[6]。在同一株植物中,不同的组织部位PAL活性也不同,一般来说越嫩的部位PAL活性越高[9],如杨树PAL活性在正在发育的木质部、嫩茎和嫩叶中最高[10]。免疫细胞化学研究显示PAL合成于栅栏细胞和海绵细胞,主要在细胞质和叶绿体内[11]。组织印迹显示PAL的mRNA常出现在表皮和微管附近的组织细胞中[12]。
PAL基因参与复杂的植物防御体系[13],比如受伤害、病原菌侵染及光照(红光、白光等)时,PAL与植物抗性表现出明显的相关性。玉米大斑病[14]、小斑病[15]、叶斑病[16],小麦赤霉病[17],水稻抗UV-B辐射及稻瘟病[18]、大豆疫霉根腐病[19]等的研究中都证实感染病原菌的植物PAL活性增强。
在拟南芥中,基因组编码4个PAL基因,分别为PAL1 (AT2G37040)、PAL2(AT3G53260)、PAL3(AT5G04230)、PAL4(AT3G10340)。用过量的Cu处理植物显示PAL活性增加,当同时再增加Si的含量时,PAL1、PAL2、PAL3呈现出相似的基因表达模式,表达水平均降低;而PAL4无论是否增加Si的含量,都没有引起PAL4表达水平降低。说明PAL基因对植物的非生物胁迫有作用,但是4个基因表达模式有区别[20]。
本研究以已知PAL基因功能的拟南芥[Arabidopsis thaliana (Linnaeus) Heynhold]的PAL基因为参考,以禾本科(Poaceae)植物水稻(Oryza sativa Linnaeus)、玉米(Zea mays Linnaeus)、小麦(Triticum aestivum Linnaeus)、谷子[Setaria italica (Linnaeus) P. de Beauvois]、豆科植物大豆[(Glycine max (Linnaeus) Merrill]作为研究对象,以其全基因组数据库为依托,通过BLAST的方法获得了这5种植物PAL基因家族的全部序列,通过系统发育分析、理化性质分析、结构分析等生物信息学方法分析PAL基因家族,比较主要作物中PAL基因的特性,为推动主要作物中PAL基因功能研究提供理论支持。
1材料与方法
1.1数据库搜索
从phytozome V9.1[21] (http://www.phytozome.net)中搜索得到拟南芥、水稻、玉米、谷子、大豆的PAL基因信息,从gramene(http://ensembl.gramene.org/Triticum_aestivum/Info/Index)中搜索得到小麦的PAL基因信息,并下载数据库中的氨基酸序列。
1.2多序列比对及系统发育分析
用Probcons[22]软件对所得数据进行多序列比对,并于MEGA6.0[23]中对比对结果进行手动调整。用MrBayes 3.2.2[24]对调整后的序列进行系统聚类分析,设置1 000 000代检测,取样频率为1 000,4条Markov链,其余参数均为软件默认值,运行2次,分裂频率(split frequencies)小于0.01时终止运行。所得的系统发育进化树在Figtree v1.4.0软件中进行查看、编辑。
1.3蛋白质的生物信息学分析
采用ProtParam tool[25] (http://web.expasy.org/protparam/)在线工具预测分析蛋白质的理化性质,应用TMpred[26]程序(http://www.ch.embnet.org/software/TMpred-form.html)在线分析来预测蛋白质跨膜区和跨膜方向,亚细胞定位应用Cell-PLoc 2.0 package[27]软件(http://www.csbio.sjtu.edu.cn/bioinf/plant-multi/)进行在线分析。用TargetP 1.1 Server[28] (http://www.cbs.dtu.dk/services/TargetP/)在线预测氨基酸序列导肽,应用signalP 4.1 server[29] (http://www.cbs.dtu.dk/services/SignalP/)中完成蛋白质信号肽的预测。活性位点运用在线分析软件Expasy[30] (http://prosite.expasy.org/)进行分析。
1.4拟南芥PAL基因生育期芯片数据表达模式的分析
从Plant Expression Database[31]网站的拟南芥数据库(http://www.plexdb.org/plex.php?database=Arabidopsis)中下载RMA文件,提取拟南芥的4条PAL基因(AT2G37040、AT3G53260、AT5G04230、AT3G10340)分别在各生育期组织[(子叶(7 d)、下胚轴(7 d)、幼苗(7 d)、幼叶(10 d)、根(7 d)、茎(17 d)、花(>21 d)、雄蕊(>21 d)、雌蕊(>21 d)、花粉(6周)、植株(21 d)]的表达情况,结果用gplots软件包中的heatmap.2软件绘制热图。
2结果与分析
2.1PAL基因家族的系统进化分析
通过BlastP的方法,去掉冗余序列,共得到45条氨基酸序列,其中拟南芥4条、水稻5条、玉米5条、谷子4条、小麦20条、大豆7条。对45条编码PAL的氨基酸序列进行系统发育分析(图1)。分析结果显示这45条PAL基因家族序列聚为明显的2支(Ⅰ、Ⅱ),单子叶植物纲禾本科的水稻、玉米、小麦、谷子聚为一支(Ⅰ),双子叶植物纲的拟南芥、大豆聚为一支(Ⅱ)。说明在进化过程中,PAL基因的分化在单子叶植物和双子叶植物分化前形成;且主要作物单子叶植物纲禾本科的水稻、玉米、小麦、谷子的亲缘关系较近,它们与双子叶植物纲的拟南芥、大豆亲缘关系较远。
在已知的拟南芥PAL基因功能的前提下,可以推测出与拟南芥最近的分支上的序列可能与拟南芥中PAL基因具有相似功能。小麦、大豆2个物种的PAL基因在分支上聚集得相对集中,推测他们在物种形成中具有重要的生理功能。
2.2PAL蛋白的理化性质分析
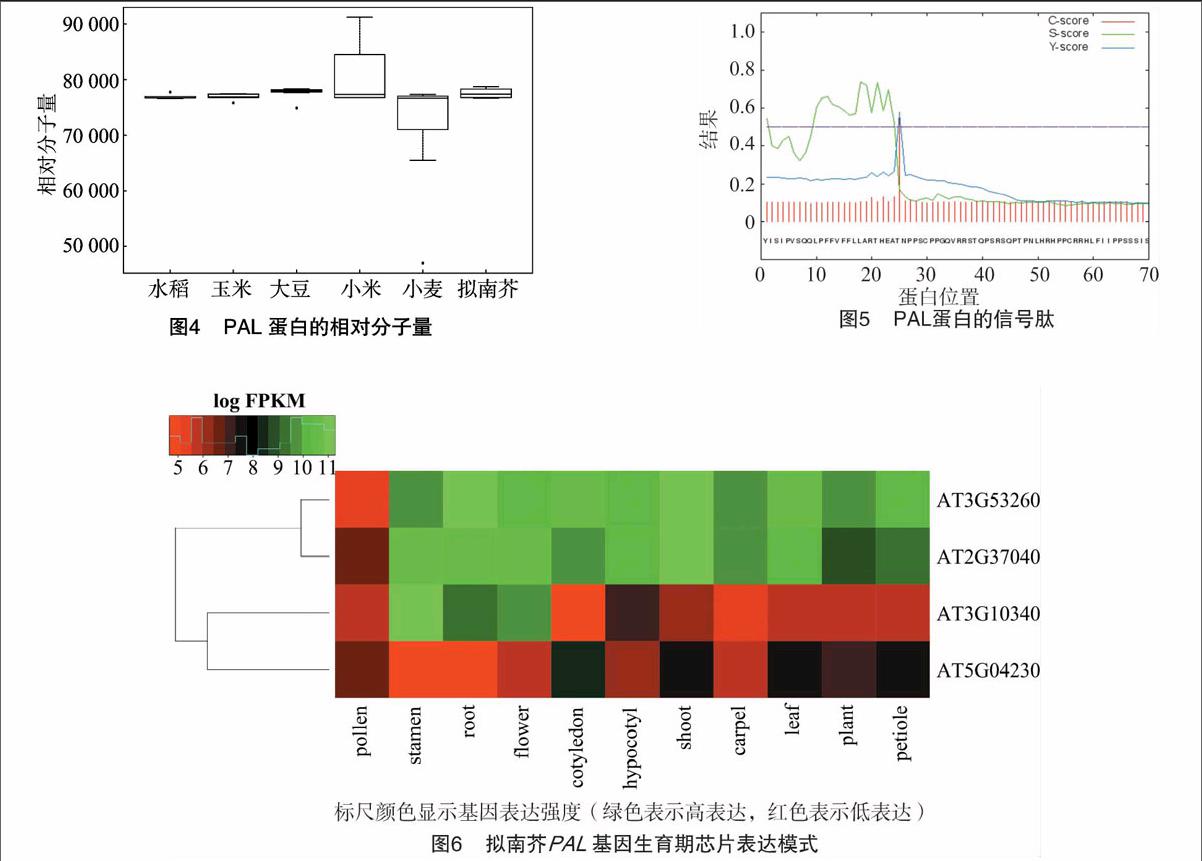
对45条氨基酸序列进行理化性质分析。结果显示:除Traes_1AS_6BDC65775、Traes_4AL_892C47ED5、Traes_2AS_EC4ADE04E为碱性蛋白(理论等电点pI>7)外,其余蛋白均为酸性蛋白质(理论等电点pI<7)(图2);根据Guruprasad等的方法[32],除Traes_1AS_6BDC65775为不稳定性蛋白(不稳定系数Ⅱ>40)外,其余蛋白均为稳定性蛋白(Ⅱ<40) (图3);相对分子量除Traes_1BS_A3B2A0DAD为46.97 ku、Si009345m为91.20 ku外,其余位于65.49~78.34 ku之间(图4),与之前报道的分子量一般在55~88 ku之间一致[33];所有蛋白都有明显跨膜现象;除Traes_1AS_6BDC65775有信号肽(图5)外,其余蛋白均无信号肽;仅小麦基因Traes_1AS_6BDC65775、Traes_2AS_EC4ADE04E具有叶绿体转运肽(Chloroplast transit peptide,CTP),其余氨基酸序列均没有导肽。
所有蛋白均为亲水性蛋白(GRAVY<0)。半衰期一致表现为:序列的N-端为甲硫氨酸(Met),在哺乳动物的活体中半衰期为30 h,在酵母活体中半衰期大于20 h,在大肠杆菌的活体中半衰期大于10 h。
2.3PAL的结构分析
所有PAL基因亚细胞定位于细胞质中。活性位点分析得出:所有基因都有活性位点,Glyma10g35381.1、Glyma20g32135.1的活性位点为GTITASGDLvPLSyvaG、LOC_Os02g41670.1、GRMZM2G170692_T01、GRMZM2G334660_T01,Si016475m、Si019385m的活性位点为GTVTASGDLvPLSyiaG,其余氨基酸序列的活性位点为GTITASGDLvPLSyiaG,具有活性位点的蛋白均属于PAL丝氨酸(Ser)酶,属于PAL基因家族的特征序列。
2.4拟南芥PAL基因生育期芯片中的表达模式
根据拟南芥各生育期芯片可以得出其4条PAL基因的表达模式。由图6可知,AT3G53260和AT2G37040在花粉(6周)中无明显表达,在其余组织中均有较强表达;AT3G10340主要在雄蕊(>21 d)、花(>21 d)、根(7 d)中有较强表达,在其他组织中无明显表达;AT5G04230在子叶(7 d)中有较弱表达,在其余各组织中均无明显表达。
3结论与讨论
PAL是连接初级代谢和苯丙烷类代谢、催化苯丙烷类代谢途径第一步反应的酶,是苯丙烷类代谢的关键酶和限速酶,苯丙烷类代谢途径的产物在植物生长发育过程中起着重要的作用,而这些物质的含量总是与PAL的活性密切相关,因此PAL对植物有着非常重要的生理意义。
本研究对拟南芥、水稻、玉米、小麦、谷子和大豆6种植物中共45条PAL基因家族蛋白进行了系统发育分析,结果显示,单子叶植物水稻、玉米、小麦、谷子的基因聚集在一支,双子叶植物拟南芥、大豆基因聚集在一支,单子叶植物与双子叶植物分别聚集在不同的分支,说明单子叶植物水稻、玉米、小麦、谷子亲缘关系较近,双子叶植物拟南芥、大豆亲缘关系较近,单子叶植物与双子叶植物亲缘关系相对较远,说明以PAL基因家族构建系统进化树能够准确地反映植物物种之间的亲缘关系及进化关系,也说明在进化过程中,PAL基因的分化在单子叶植物和双子叶植物分化前形成;同时,与拟南芥最近的分支上共有7条大豆基因和1条小麦基因,推测它们可能属于PAL基因家族;在已知的拟南芥的PAL基因功能的前提下,可以推测出与拟南芥最近的分支上的序列可能与拟南芥中PAL基因具有相似功能。
理化性质分析结果显示,酸性蛋白质占93.3%,与之前报[JP2]道的PAL是一种酸性蛋白一致[33-35]。稳定性蛋白占978%,[JP]有信号肽的蛋白占2.2%,有导肽的蛋白占4.4%,所有蛋白均为有明显跨膜现象的亲水性蛋白;仅小麦基因 Traes_1AS_6BDC65775、Traes_2AS_EC4ADE04E具有叶绿体转运肽,其余氨基酸序列均没有导肽。说明大多数蛋白为非分泌性蛋白,它们由游离的核糖体合成,在细胞质内参与生化反应,在细胞内代谢过程中发挥重要作用。
结构分析结果得出,所有氨基酸序列均有蛋白活性位点,均属于PAL丝氨酸(Ser)酶,具有PAL基因家族的特征序列,初步认为这些基因属于PAL基因家族。同时,亚细胞定位发现所有PAL基因都定位于细胞质中,与理化性质分析结果一致,进一步说明PAL基因在细胞质内发挥作用。
拟南芥PAL基因生育期芯片表达模式分析结果表明,AT3G53260和AT2G37040在子叶(7 d)、下胚轴(7 d)、幼苗(7 d)、幼叶(10 d)、根(7 d)、茎(17 d)、花(>21 d)、雄蕊(>21 d)、雌蕊(>21 d)等中均有较强表达,在花粉(6周)中无明显表达;AT3G10340主要在雄蕊(>21 d)、花(>21 d)、根(7 d)中有较强表达,在子叶(7 d)、下胚轴(7 d)、幼苗(7 d)、幼叶(10 d)、茎(17 d)、雌蕊(>21 d)、花粉(6周)中表达较弱;AT5G04230在子叶(7 d)中有较弱表达,在其余各组织中均表达较弱。说明拟南芥中PAL基因的表达模式呈现明显的组织特异性,为本研究中主要作物的PAL基因表达研究提供参考。
目前,虽有关于主要作物中PAL基因的研究,但都是对单个基因的克隆,尚未发现有从主要作物中PAL基因家族层面的报道。本研究选取了5种主要作物的PAL基因家族为研究对象,对其基因家族系统进化、理化性质、定位、结构等进行了分析,对今后在主要作物中研究PAL基因的时空表达模式及功能,增强多个基因的协同表达,更好地利用 PAL对主要作物进行改造,从而增强主要作物的抗病性有重要意义。
[HS2][HT8.5H]参考文献:[HT8.SS]
[1][ZK(#]Pellegrini L,Rohfritsch O,Fritig B,et al. Phenylalanine ammonia-lyase in tobacco (molecular cloning and gene expression during the hypersensitive reaction to tobacco mosaic virus and the response to a fungal elicitor) [J]. Plant Physiology,1994,106(3):877-886.
[2]Dorey S,Kopp M,Geoffroy P,et al. Hydrogen peroxide from the oxidative burst is neither necessary nor sufficient for hypersensitive cell death induction,phenylalanine ammonia lyase stimulation,salicylic acid accumulation,or scopoletin consumption in cultured tobacco cells [JP2]treated with elicitin [J]. Plant Physiology,1999,121(1):163-172.[JP]
[3]Hartley S E,Firn R D. Phenolic biosynthesis,leaf damage,and insect herbivory in birch (Betula pendula) [J]. Journal of Chemical Ecology,1989,15(1):275-283.
[4]江昌俊,余有本. 苯丙氨酸解氨酶的研究进展 (综述) [J]. 安徽农业大学学报,2001,28(4):425-430.
[5]Koukol J,Conn E E. The metabolism of aromatic compounds in higher plants IV. Purification and properties of the phenylalanine deaminase of Hordeum vulgare [J]. Journal of Biological Chemistry,1961,236(10):2692-2698.
[6]欧阳光察,应初衍,沃绍根,等. 植物苯丙氨酸解氨酶的研究——Ⅵ. 水稻、小麦 PAL 的纯化及基本特性[J]. 植物生理学报,1985,11(2):204-214.
[7]Mauch-Mani B,Slusarenko A J. Production of salicylic acid precursors is a major function of phenylalanine ammonia-lyase in the resistance of Arabidopsis to Peronospora parasitica [J]. The Plant Cell,1996,8(2):203-212.
[8]Minami E I,Ozeki Y,Matsuoka M,et al. Structure and some characterization of the gene for phenylalanine ammonia-lyase from rice plants [J]. European Journal of Biochemistry,1989,185(1):19-25.
[9] Ritter H,Schulz G E. Structural basis for the entrance into the phenylpropanoid metabolism catalyzed by phenylalanine ammonia-lyase [J]. The Plant Cell,2004,16(12):3426-3436.[ZK)]
[10][ZK(#]Kawamata S,Shimobarai K,Imura Y,et al. Temporal and spatial pattern of expression of the pea phenylalanine ammonia-lyase gene1 promoter in transgenic tobacco [J]. Plant and Cell Physiology,1997,38(7):792-803.
[11]Santiago L J M,Louro R P,de Oliveira D E. Compartmentation of phenolic compounds and phenylalanine ammonia-lyase in leaves of Phyllanthus tenellus Roxb. and their induction by copper sulphate [J]. Annals of Botany,2000,86(5):1023-1032.
[12]Nakazawa A,Nozue M,Yasuda H,et al. Expression pattern and gene structure of phenylalanine ammonia-lyase in Pharbitis nil[J]. Journal of Plant Research,2001,114(3):323-328.
[13]Raes J,Rohde A,Christensen J H,et al.Genome-wide characterization of the lignification toolbox in Arabidopsis[J]. Plant Physiology,2003,133(3):1051-1071.
[14]王光达,黄初女,吴委林,等. 不同玉米品种对大斑病的抗性与相关防御酶活性的关系研究[J]. 玉米科学,2014,22(5):146-152.
[15]马春红,翟彩霞,郑秋玲,等. 玉米小斑病菌 T 小种毒素诱导对玉米叶片苯丙氨酸解氨酶活性的影响[J]. 华中农业大学学报,2010,29(1):21-25.
[16]陈捷,蔺瑞明,高增贵,等. 玉米弯孢叶斑病菌毒素对寄主防御酶系活性的影响及诱导抗性效应[J]. 植物病理学报,2002,32(1):43-48.
[17]王雅平,吴兆苏,刘伊强. 小麦抗赤霉病性的生化研究及其机制的探讨[J]. 作物学报,1994,20(3):327-333.
[18]李元,高潇潇,高召华,等. UV-B 辐射和稻瘟病菌胁迫对水稻幼苗苯丙氨酸解氨酶活性和类黄酮含量的影响[J]. 中国生态农业学报,2010,18(4):856-860.
[19]张淑珍,徐鹏飞,吴俊江,等. 大豆疫霉根腐病菌毒素处理抗感不同大豆品种后苯丙氨酸解氨酶活性的变化[J]. 作物杂志,2008(1):47-49.[HJ1.7mm]
[20]Khandekar S,Leisner S. Soluble silicon modulates expression of Arabidopsis thaliana genes involved in copper stress [J]. Journal of Plant Physiology,2011,168(7):699-705.
[21]Goodstein D M,Shu S,Howson R,et al. Phytozome:a comparative platform for green plant genomics [J]. Nucleic Acids Research 2012,40(D1):D1178-D1186.
[22]Do C B,Mahabhashyam M S,Brudno M,et al. probCons:Probabilistic consistency-based multiple sequence alignment [J]. Genome Research,2005,15(2):330-340.
[23]Tamura K,Stecher G,Peterson D,et al. MEGA6:molecular evolutionary genetics analysis version 6.0 [J]. Molecular Biology and Evolution,2013,30(12):2725-2729.
[24]Ronquist F,Teslenko M,van der Mark P,et al. MrBayes 3.2:efficient Bayesian phylogenetic inference and model choice across a large model space [J]. Systematic Biology,2012,61(3):539-542.
[25]Walker J M. The proteomics protocols handbook [M]. Totowa,New Jersey:Humana Press,2005:571-607.
[26]Hofmann K,Stoffel W. Tmbase-a database of membrane spanning protein segments [J]. Biological Chemistry Hoppe-Seyler,1993,374:166.
[27]Chou K-C,Shen H-B. Plant-mPLoc:a top-down strategy to augment the power for predicting plant protein subcellular localization[J]. PLoS One,2010,5(6):e11335.
[28]Emanuelsson O,Brunak S,von Heijne G,et al. Locating proteins in the cell using TargetP,SignalP and related tools [J]. Nature Protocols,2007,2(4):953-971.
[29]Petersen T N,Brunak S,von Heijne G,et al. SignalP 4.0:discriminating signal peptides from transmembrane regions [J]. Nature Methods,2011,8(10):785-786.
[30]Sigrist C J,Cerutti L,Hulo N,et al. PROSITE:a documented database using patterns and profiles as motif descriptors [J]. Briefings in Bioinformatics,2002,3(3):265-274.
[31]Dash S,van Hemert J,Hong L,et al. PLEXdb:gene expression resources for plants and plant pathogens [J]. Nucleic Acids Research,2012,40(D1):D1194-D1201.
[32]Guruprasad K,Reddy B B,Pandit M W. Correlation between stability of a protein and its dipeptide composition:a novel approach for predicting in vivo stability of a protein from its primary sequence [J]. Protein Engineering,1990,4(2):155-161.
[34]刘邮洲,张摇磊,钱国良,等. 生防菌sf628对梨叶片苯丙氨酸解氨酶和过氧化物酶活性的影响[J]. 江苏农业学报,2014,30(2):268-274.
[35]王燕芳,吴瑛. 外源水杨酸对棉花相关抗虫酶活性诱导的时间和浓度效应[J]. 江苏农业科学,2015,43(4):138-140.
[36]崔建东,李艳,牟德华. 苯丙氨酸解氨酶 (PAL) 的研究进展[J]. 食品工业科技,2008,29(7):306-308.
