SH-SY5Y细胞中过表达RIPK3对ZFP36基因转录的影响
2016-07-14张国禄程世翔徐忠伟衣泰龙廖吉连涂悦张赛
张国禄,程世翔,徐忠伟,衣泰龙,廖吉连,涂悦,张赛
细胞与分子生物学
SH-SY5Y细胞中过表达RIPK3对ZFP36基因转录的影响
张国禄1,程世翔1,徐忠伟2,衣泰龙1,廖吉连1,涂悦1,张赛1
摘要:目的探讨SH-SY5Y细胞中受体相互作用蛋白激酶-3(RIPK3)下游信号通路及其中的关键信号分子的作用。方法通过质粒转染的方式在实验组SH-SY5Y细胞内表达外源性RIPK3蛋白,将转染空载质粒载体SHSY5Y细胞作为对照组。通过观察质粒所携带的绿色荧光蛋白(GFP)在细胞中的表达情况以验证转染结果,Western blot对外源性RIPK3表达进行验证。MTT法检测细胞增殖活性,观察过表达RIPK3对细胞活性的影响,以确认其在细胞内是否具有生物活性。运用转录组测序技术(RNAseq)分别检测2组细胞内基因转录丰度并应用Ingenuity Pathway Analysis(IPA)数据库进行分析,获得RIPK3下游信号通路及关键分子。通过微滴式数字化PCR(ddPCR)对部分RNAseq结果进行验证。结果外源性RIPK3在细胞内具有生物活性,能够抑制SH-SY5Y细胞的增殖。RNAseq数据经IPA分析后得出锌指蛋白36(ZFP36)为RIPK3下游效应分子,其相关效应分子包括血管内皮生长因子(VEGF)、人脱帽酶2(DCP2)、脑源性神经营养因子(BDNF)、肿瘤坏死因子(TNF)的转录丰度也发生了相应的变化。结论RIPK3能够参与对ZFP36以及与其相关效应分子的转录调控,进而在神经系统的发育、炎症和肿瘤发生等生理、病理过程中发挥重要作用。
关键词:神经母细胞瘤;细胞系,肿瘤;锌指;基因表达调控;血管内皮生长因子类;脑源性神经营养因子;肿瘤坏死因子类;受体相互作用蛋白激酶-3;锌指蛋白36;人脱帽酶2
受体相互作用蛋白激酶-3(receptor-interacting protein kinase 3,RIPK3)是受体相互作用蛋白家族(receptor-interacting protein kinases,RIPs)的重要成员。作为同族蛋白受体相互作用蛋白激酶-1(receptor-interacting protein kinase 1,RIPK1)重要的下游效应分子,激活状态下的RIPK3能够调控组织细胞的凋亡、程序性坏死及炎症的发生[1]。RIPK3在丘脑、黑质、胼胝体、壳核、脊髓和延髓中表达水平较高,提示RIPK3可能在神经系统的发育和疾病进程中发挥着特殊的作用。锌指蛋白36(Znic finger protein 36,ZFP36)是一种RNA结合蛋白,能够识别多种神经元转录产物,并调节mRNA的稳定性,在神经系统的发育、功能、炎症等各种生理、病理过程中发挥着重要的作用[2]。本实验中发现了RIPK3下游信号分子ZFP36以及相关信号通路,为RIPK3的研究开拓了新方向。
1 材料与方法
1.1材料 (1)细胞。人神经母细胞瘤细胞系SH-SY5Y购自ATCC。(2)主要仪器。倒置显微镜及激光共聚焦显微镜(Leica公司),Thermo Scientific CO2培养箱(Thermo公司),电子天平(METTLER TOLEDO公司),蛋白垂直电泳和转膜系统、QX200微滴式数字化PCR(droplet digital PCR,ddPCR)系统(Bio-Rad公司),凝胶成像系统(GE公司)。(3)主要试剂。DMEM/F12培养基、胎牛血清购自Gibco公司;pCMV6-ACGFP质粒购自OriGene公司;质粒提取试剂盒、cDNA合成试剂盒购自TIANGEN公司;Lipofectamine 3000购自Life公司;Western blot一抗兔抗GAPDH、小鼠抗RIPK3购自Abcam公司;羊抗兔、羊抗小鼠二抗购自KPL公司;G418、TRIzol购自Invitrogen公司;QX200 ddPCR EvaGreen Supermix购自Bio-Rad公司。
1.2方法
1.2.1细胞培养SH-SY5Y培养于含有10%FBS、100 U/mL氨苄青霉素、100 mg/L链霉素的DMEM∶F12=1∶1培养基中,培养环境为37℃、5%CO2、饱和湿度,0.25%胰酶-EDTA消化传代。当细胞处于对数生长期时用于实验。
1.2.2质粒扩增与细胞转染质粒转化DH5-α感受态大肠杆菌进行筛选和扩增,按照试剂盒所提供的标准实验步骤提取质粒,SH-SY5Y细胞汇合度约50%时采用Lipofectamine 3000进行转染,步骤参照所附说明书。转染48 h后用G418浓度为1 000 mg/L的完全培养基进行筛选,获得稳定过表达RIPK3的细胞株,继而使用G418浓度为800 mg/L的完全培养基维持培养,激光共聚焦显微镜观察,确认绿色荧光蛋白(green fluorescent protein,GFP)表达情况。实验组细胞转染携带有RIPK3过表达基因的质粒,对照组细胞转染空载质粒。
1.2.3Western blot取对数生长期的细胞,加入RIPA裂解后超声裂解,离心收集上清,加入Loading buffer后变性。每泳道25 μg加入到10%的SDS-PAGE凝胶中电泳,将蛋白转印于硝酸纤维素膜上,5%脱脂奶粉封闭1 h,一抗4℃孵育过夜,洗膜,二抗室温孵育2 h,再次洗膜。滴加ECL发光液在凝胶成像系统中显影。以GAPDH作为内参。
1.2.4MTT细胞增殖实验细胞以5×104/mL的密度接种于96孔板,每个时间点设5个复孔,常规培养。在检测时间点向培养孔中加入MTT,细胞培养箱中孵育4 h,检测光密度(OD)值,实验重复5次。计算过表达RIPK3的实验组相对细胞存活率=(实验组OD值/对照组OD值)×100%。
1.2.5转录组测序技术(RNAseq)及数据分析提取细胞RNA,质控检测确认符合实验要求。富集mRNA并合成cDNA,纯化、修复与修饰后,富集所需cDNA文库,定量并行RNAseq测序。计算出基因RPKM值(The value for reads per kilobase of coding sequence per million mapped reads)代表当前条件下基因转录丰度[3]。测序结果用Ingenuity Pathway Analysis(IPA)对基因表达差异进行分析,获得RIPK3下游信号通路及处于关键位置的效应分子转录丰度。转录丰度=(目的基因RPKM/GAPDH基因RPKM)×100%。
1.2.6微滴式数字化PCR(ddPCR) 将RNA反转录为cDNA。按照实验要求构建反应体系,微滴化后扩增,反应条件为:95℃预变性5 min;95℃变性15 s,50℃退火1 min,72℃延伸40 s,40个扩增循环;最后90℃维持5 min以稳定微滴。QX200微滴读取仪中对阴性/阳性微滴进行计数,用QuantaSoft(1.3.2.0)软件进行数据分析,得到目的基因的绝对浓度(copies/μL),以GAPDH为内参进行校正后计算实验组相对转录丰度=(实验组转录丰度/对照组转录丰度)×100%。引物序列参照表1。

Tab.1 The droplet digital PCR oligonucleotide sequences表1 ddPCR引物序列
1.3统计学方法采用IBM SPSS Statistics 19.0软件对数据进行统计学分析,实验结果用均数±标准差(x±s)表示,组间比较采用独立样本t检验,P<0.05为差异有统计学意义。
2 结果
2.1外源性RIPK3在实验组细胞内稳定表达转染后细胞在激光共聚焦显微镜下观察,2组细胞在激发光照射下发射绿色荧光,说明质粒序列成功插入到细胞基因组中,见图1。Western blot结果显示,实验组在RIPK3抗体的标记下出现2个条带,细胞内源性RIPK3分子质量为56 ku,实验组外源性RIPK3与GFP融合,分子质量为85 ku。质粒携带的外源性RIPK3蛋白在SH-SY5Y细胞中稳定表达,见图2。
Fig.1 The morphology of two groups of cells under confocal laser scanningmicroscope(×200)图1 激光共聚焦显微镜下观察2组细胞(×200)

Fig.2 The expression of RIPK3 in cells图2 RIPK3在细胞中表达情况
2.2外源性 RIPK3对SH-SY5Y细胞活性的影响MTT结果显示:12、20、28、36、44 h时点实验组细胞活性明显低于对照组(n=5,t分别为6.717、5.094、14.900、11.914、23.903,均P<0.01),见图3。

Fig.3 The effect of RIPK3 on cell viability图3 RIPK3对细胞活性的影响
2.3ZFP36是RIPK3的下游效应分子IPA对RNAseq测序数据分析得到RIPK3下游信号通路,以及其中的关键效应分子ZFP36。相对于对照组,实验组 ZFP36转录丰度发生上调(4.36%vs. 7.89%)。ddPCR检测cDNA样本中GAPDH与ZFP36基因绝对浓度,见图4,以GAPDH作为内参进行校正后计算得到实验组相对于对照组ZFP36转录丰度为(177.29±9.81)%(n=3,t=13.644,P<0.01)。ZFP36相对转录丰度差异与RNAseq结果基本一致。

Fig.4 Concentrations of GAPDH and ZFP36 in cDNA samples图4 cDNA样本中GAPDH与ZFP36浓度
2.4RIPK3对ZFP36下游效应分子的影响从IPA对RNAseq数据分析结果中遴选出ZFP36下游的效应分子,以GAPDH为内参进行校正,结果提示RIPK3对这些ZFP36下游效应分子血管内皮生长因子(vascular endothelial growth factor,VEGF)、人脱帽酶2(mRNA-decapping enzyme 2,DCP2)、脑源性神经营养因子(brain derived neurotrophic factor,BDNF)、肿瘤坏死因子(tumor necrosis factor,TNF)的转录丰度产生了明显的影响,见图5。
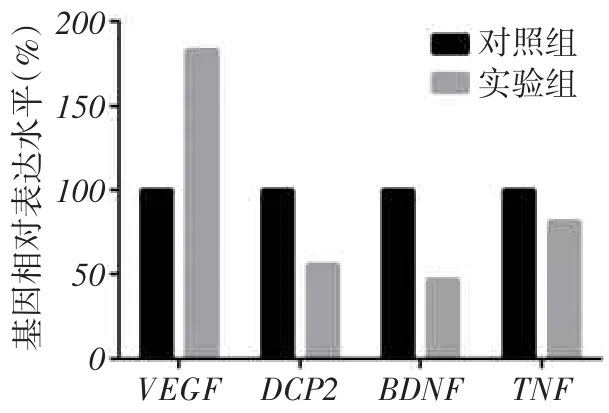
Fig.5 ZFP36 downstream signalingmolecule transcription levels图5 ZFP36下游效应分子的转录丰度
3 讨论
近年来,RIPK3越来越受到科研人员的重视,但研究方向主要局限于凋亡、坏死性凋亡及炎症发生等相关信号通路,RIPK3在神经系统中所发挥的作用及信号通路尚待探索。本实验以SH-SY5Y为基础建立RIPK3过表达神经元模型[4-5],MTT结果证明外源性的RIPK3在细胞内具有生物学活性,表现为抑制细胞活性,后续实验充分利用了RNAseq实验与IPA高通量、高敏感度的特点,对细胞基因的整个转录组mRNA进行检测及分析,实验过程中经过多重质控检测以确保数据准确可靠。本研究采用Bio-Rad公司最新的第3代PCR技术进行精确的绝对定量[6],实验结果与RNAseq结果保持高度一致,保证了实验具有较强的可重复性[7]。本研究经分析得出SH-SY5Y细胞内RIPK3下游存在以ZFP36为核心的信号通路。
ZFP36是一种RNA结合蛋白,能够促进3′端非编码区富含AU碱基(AU-rich elements,AREs)的mRNAs快速降解[8]。其自身未被发现具有mRNA酶活性,但是其与几种细胞mRNA降解机制中部分酶的活性相关,包括脱腺苷酶、脱帽因子及核酸外切酶,从而激活目标mRNA的降解[9]。mRNA的降解能够抑制蛋白质的过量表达、促进核苷酸的降解,在细胞代谢中发挥着核心作用。对含有ARE的mRNA的转录后调控是一个非常复杂的过程。预计哺乳动物体内超过8%的mRNA含有ARE,其中很大一部分编码的是具有高度调控作用的细胞因子,包括细胞因子、生长因子、转录因子以及早期反应基因[10]。
本研究不仅证实处于RIPK3下游潜在的重要信号分子ZFP36,更通过分析得出了部分转录丰度发生明显改变的信号分子。ZFP36在体内特别是神经系统中具有十分广泛的作用。在对恶性胶质瘤细胞的研究中,ZFP36诱导的mRNA降解能够导致明显的具有剂量依赖性的VEGF mRNA的改变,同时证实ZFP36的异位表达能够通过干扰VEGF从而发挥生长抑制作用[11]。ZFP36能够直接在mRNA水平调节BDNF,进而起到调节中枢神经系统的发育和功能、神经炎症、肌源性分化以及修复[12];同时,ZFP36能够直接动员并激活DCP2,并在其协助下完成对含有ARE结构的mRNA发挥脱帽作用,以促进其降解[13]。ZFP36的靶基因包括编码RNA结合蛋白(RNA-binding proteins,RBPs)的mRNA,包括Hu/Elavl和Nova家族,而这些蛋白在神经系统的发育和功能中具有重要的作用,同时也是重要的神经标志物[14]。在神经分化过程中减少ZFP36的表达有助于神经系统中3′端延长的mRNA多聚腺苷化亚型的产生[15]。综上,可以认为RIPK3能够通过对ZFP36的调节在神经系统肿瘤、炎症、修复以及发育过程中发挥重要的作用。
由于受到各种条件的约束,本研究仅验证了RIPK3过表达对ZFP36及其下游效应分子基因转录丰度的影响,并根据相关研究结果对其可能造成的下游效应进行合理的推测,并未对具体的相互作用关系进行进一步探究及阐述,需要在后续的实验中对其进行进一步完善。
参考文献
[1]Moriwaki K,Chan FK.RIP3:a molecular switch for necrosis and inflammation[J].Genes Dev,2013,27(15):1640-1649.doi:10.1101/gad.223321.113.
[2]Dai W,Li W,Hoque M,et al.A post-transcriptional mechanism pacing expression of neural genes with precursor cell differentiation status[J].Nat Commun,2015,6:7576.doi:10.1038/ncomms8576.
[3]Mortazavi A,Williams BA,McCue K,et al.Mapping and quantifying mammalian transcriptomes by RNA-Seq[J].Nat Methods,2008,5(7):621-628.doi:10.1038/nmeth.1226.
[4]Liu Y,Zhang RY,Zhao J,et al.Ginsenoside Rd protects SH-SY5Y cells against 1-Methyl-4-phenylpyridinium induced injury[J].Int J Mol Sci,2015,16(7):14395-14408.doi:10.3390/ijms160714395.
[5]Kwon B,Gamache T,Lee HK,et al.Synergistic effects of β-amyloid and ceramide-induced insulin resistance on mitochondrial metabolism in neuronal cells[J].Biochim Biophys Acta,2015,1852 (9):1810-1823.doi:10.1016/j.bbadis.2015.05.012.
[6]Pinheiro LB,Coleman VA,Hindson CM,et al.Evaluation of a droplet digital polymerase chain reaction format for DNA copy number quantification[J].Anal Chem,2012,84(2):1003-1011.doi:10.1021/ac202578x.
[7]Hindson CM,Chevillet JR,Briggs HA,et al.Absolute quantification by droplet digital PCR versus analog real-time PCR[J].Nat Methods,2013,10(10):1003-1005.doi:10.1038/nmeth.2633.
[8]Brooks SA,Blackshear PJ.Tristetraprolin(TTP):interactions withmRNA and proteins,and current thoughts on mechanisms of action [J].Biochim Biophys Acta,2013,1829(6/7):666-679.doi:10.1016/j.bbagrm.2013.02.003.
[9]Komnenov D,Scipione CA,Bazzi ZA,et al.Pro-inflammatory cytokines reduce human TAFI expression via tristetraprolin-mediated mRNA destabilisation and decreased binding of HuR[J].Thromb Haemost,2015,114(2):337-349.doi:10.1160/TH14-08-0653.
[10]Shu M,Taddeo B,Roizman B.Tristetraprolin recruits the herpes simplex virion host shutoff RNase to AU-rich elements in stress response mRNAs to enable their cleavage[J].J Virol,2015,89(10):5643-5650.doi:10.1128/JVI.00091-15.
[11]Xiao J,Gao H,Jin Y,et al.The abnormal expressions of tristetraprolin and the VEGF family in uraemic rats with peritoneal dialysis[J]. Mol Cell Biochem,2014,392(1/2):229-238.doi:10.1007/ s11010-014-2033-3.
[12]Kumar A,Varendi K,Peranen J,et al.Tristetraprolin is a novel regulator of BDNF[J].Springerplus,2014,3:502.doi:10.1186/2193-1801-3-502.
[13]Kim CW,Kim HK,Vo MT,et al.Tristetraprolin controls the stability of cIAP2 mRNA through binding to the 3′UTR of cIAP2 mRNA [J].Biochem Biophys Res Commun,2010,400(1):46-52.doi:10.1016/j.bbrc.2010.07.136.
[14]Ince-Dunn G,Okano HJ,Jensen KB,et al.Neuronal Elav-like (Hu)proteins regulate RNA splicing and abundance to control glutamate levels and neuronal excitability[J].Neuron,2012,75(6):1067-1080.doi:10.1016/j.neuron.2012.07.009.
[15]Miura P,Sanfilippo P,Shenker S,et al.Alternative polyadenylation in the nervous system:to what lengths will 3′UTR extensions take us?[J].Bioessays,2014,36(8):766-777.doi:10.1002/bies.201300174.
(2015-09-10收稿2015-10-28修回)
(本文编辑李国琪)
The effects of RIPK3 overexpression on the transcription of ZFP36 gene in SH-SY5Y cells
ZHANG Guolu1,CHENG Shixiang1,XU Zhongwei2,YI Tailong1,LIAO Jilian1,TU Yue1,ZHANG Sai1
1 Tianjin Key Laboratory of Neurotrauma Repair,Institute of Traumatic Brain Injury and Neuroscience of Chinese Armed Police Forces(CAPF),Neurology&Neurosurgery Hospital of CAFP,Tianjin 300162,China;2 Center Laboratory of Logistics University of CAPF Corresponding AuthorE-mail:ytumail@vip.126.com;zhangsai718@vip.126.com
Abstract:ObjectiveTo investigate the signalingpathway and the key signal molecules of protein kinase(RIPK)3 in SHSY5Y cells.MethodsSH-SY5Y cells were transfected with RIPK3 expression plasmid vector to upregulate intracellular RIPK3,while the SH-SY5Y cells were transfected with empty vector plasmid,which was considered as control group.Western blot assay was used to check the expression of exogenous RIPK3 in cells.The proliferation rate of SH-SY5Y cells was determined by MTT assay at designated time to detect exogenous RIPK3 activity.Whole transcriptome sequencing(RNAseq)was used to detect the transcription of genes.Whole-transcriptomic gene transcription was measured by following Ingenuity Pathway Analysis(IPA)to obtain downstream signaling pathways and the key molecule,which were partly confirmed by following droplet digital PCR (ddPCR).ResultsExogenous RIPK3 showed biological activity in SH-SY5Y,which inhibited the proliferation of cells.IPA showed that znic finger protein 36(ZFP36)was significantly up-regulated as compared with that of the control group.The transcription levels of ZFP36 downstream genes such as tumor necrosis factor(TNF),brain derived neurotrophic factor(BDNF),vascular endothelial growth factor(VEGF)and mRNA-decappingenzyme 2(DCP2)were affected at the same time.ConclusionWithin the limitations of this study,it seems that RIPK3 is notable for the development,inflammation and tumorigenesis of the nervous system as an independent regulator of ZFP36 gene and downstream effectors.
Key words:neuroblastoma;cell line,tumor;zinc fingers;gene expression regulation;vascular endothelial growth factors;brain-derived neurotrophic factor;tumor necrosis factors;receptor-interacting protein kinase 3;znic finger protein 36;mRNA-decappingenzyme 2
中图分类号:R34
文献标志码:A
DOI:10.11958/20150166
基金项目:国家自然科学基金资助项目(31200809);武警部队后勤科研项目(WJHQ2012-20);武警后勤学院科研创新团队(WHTD201306)
作者单位:1天津市神经创伤与修复重点实验室,武警部队脑创伤与神经疾病研究所,武警后勤学院附属医院脑科医院(邮编300162);2武警后勤学院中心实验室
作者简介:张国禄(1990),男,硕士在读,主要从事神经外科学研究
通讯作者E-mail:ytumail@vip.126.com;zhangsai718@vip.126.com
