采用6个微卫星位点研究6个绵羊品种遗传多样性
2016-06-23陈李鹏黄勇富赵永聚俄广鑫娜日苏
陈李鹏 黄勇富 赵永聚 俄广鑫 娜日苏
(西南大学动物科技学院,重庆 400716)
采用6个微卫星位点研究6个绵羊品种遗传多样性
陈李鹏 黄勇富 赵永聚 俄广鑫 娜日苏
(西南大学动物科技学院,重庆 400716)
利用6个微卫星座位对中国乌珠穆沁羊、小尾寒羊、滩羊、昭通绵羊、呼伦贝尔羊和甘肃高山细毛羊6个绵羊品种,共计280个个体进行遗传多样性分析。计算了6个绵羊品种间的多态信息含量、杂合度和遗传距离,并进行了主成分分析和UPGMA聚类。发现了101个等位基因,平均杂合度0.599-0.691,平均多态信息含量0.609-0.680;甘肃高山细毛羊与其他绵羊品种遗传分歧最大,而呼伦贝尔羊与乌珠穆沁羊间的分歧最小。结果表明6个绵羊品种均具有较高的遗传多样性,品种间的遗传关系与其形成历史、分化及地理分布基本一致。
微卫星;遗传多样性;绵羊
我国地方绵羊品种众多,遗传资源十分丰富,是世界少有的宝贵基因库[1],但受世界范围内商业品种的冲击,导致市场竞争力低的地方品种逐渐走向衰落。因此,对中国绵羊品种遗传多样性进行评估和保护,已是当务之急。
微卫星又称简单串连重复序列,一般由2-6 bp组成核心序列,重复10-20次,首尾相接而组成[2]。它广泛存在于真核生物的基因组中,可通过PCR扩增和电泳分型,具有数量多、多态性丰富、保守性好、共显性遗传以及检测方便快速等优点[3-5],在度量品种遗传多样性、估测品种间遗传距离及构建系统发生树等研究中显示出巨大的优势。目前,有很多学者应用微卫星DNA标记技术对绵羊的遗传多样性进行了研究。如Sun等[6]利用15个微卫星标记评估了5个绵羊品种的遗传多样性,结果表明群体遗传结构与群体间的遗传距离不一致。Agaviezor等[7]利用15个微卫星标记分析了4个尼日利亚绵羊品种共384只绵羊的遗传多样性,结果表明绵羊品种内遗传变异高于品种间遗传变异。Yilmaz等[8]利用18个微卫星标记分析了9个绵羊品种的遗传多样性,发现欧洲绵羊品种的遗传变异高于土耳其绵羊品种;Leroy等[9]利用21个微卫星标记分析了49个绵羊品种的遗传多样性,评估了法国部分绵羊品种的起源分化模式。Ćurković等[10]利用30个微卫星分析了18个绵羊品种的遗传多样性,评估了绵羊品种形成过程中自然和人工选择的重要性。
本研究选取呼伦贝尔羊、乌珠穆沁羊、小尾寒羊、滩羊和昭通绵羊等5个中国地方绵羊品种,以及甘肃高山细毛羊1个培育品种,在常染色体基因组水平上研究6个微卫星位点的遗传多样性和遗传分化关系,旨在为揭示其品种遗传结构和遗传背景,以及下一步开展资源保护与开发利用提供科学依据。
1 材料与方法
1.1 材料
1.1.1 实验样本 本实验样本采自6个绵羊品种分布的主产区,每个品种随机采集无亲缘关系个体40只或48只,共计280只。所采血样为8-10 mL/只,用柠檬酸葡萄糖抗凝,-20℃冻存。各品种的样本信息,见表1。

表1 6个绵羊品种的样本信息
1.1.2 微卫星引物 根据相关微卫星选择标准,并综合考虑微卫星位点在染色体中位置、引物序列组成特点、微卫星位点多态性和产物大小等多方面因素,最后确定了分布于5条不同常染色体上的6对微卫星引物,分别为 OarJMP29、OarAE129、MCM-527、ILSTS005、MAF209和OarFCB304(表2)。

表2 6个微卫星位点的信息
1.2 方法
1.2.1 DNA的提取 用基因组DNA提取试剂盒提取全血中的基因组DNA,-20℃保存。
1.2.2 PCR扩增 采用20 μL反应体系,其中各组分的终浓度分别为dNTPs 0.2 mmol/L、Mg2+1.5 mmol/L、混合上下游引物0.5 mmol/L、Taq酶5 U/μL、D NA模板1 μL(约60 ng)。PCR反应程序:94℃预变性5 min;94℃变性30 s,50-60℃退火30 s,72℃延伸30 s,经过35个循环;于72℃延伸7 min,4℃保存。PCR产物用ABI 3130 xl全自动基因分析仪进行分型检测,片段分析的上样操作参照曾盛诚[11]的文献报道。
1.2.3 数据的统计处理 利用Microsatellite Toolkit软件计算各微卫星座位的等位基因数、多态信息含量(PIC)、观察杂合度(HO)和期望杂合度(HE)等参数[12],Arlequin 3.5.1.3软件计算6个品种间遗传距离(DS)[13],GENEPOP 3.4软件进行哈德温伯格平衡检验[14],Fstat 2.9.3.2软件计算群体近交系数(FIS)[15],MVSP3.1软件进行品种间遗传分化的主成分分析和UPGMA聚类分析[16,17]。
2 结果
2.1 等位基因分布
6个绵羊品种微卫星座位的等位基因分布见表3。由表3可见 ,在OarFCB304座位检测到的等位基因数最多,达到24个,OarAE129座位检测到的等位基因数最少,为9个,6个绵羊品种在6个微卫星座位中共检测到101个等位基因,平均每个座位的等位基因数为16.8;同一个微卫星座位,在不同品种间的等位基因分布也不同,如在OarJMP29座位上,UQ、ZT、HBR、STH、TAN、GSH检测到的等位基因数分别为10、9、11、8、9、7。
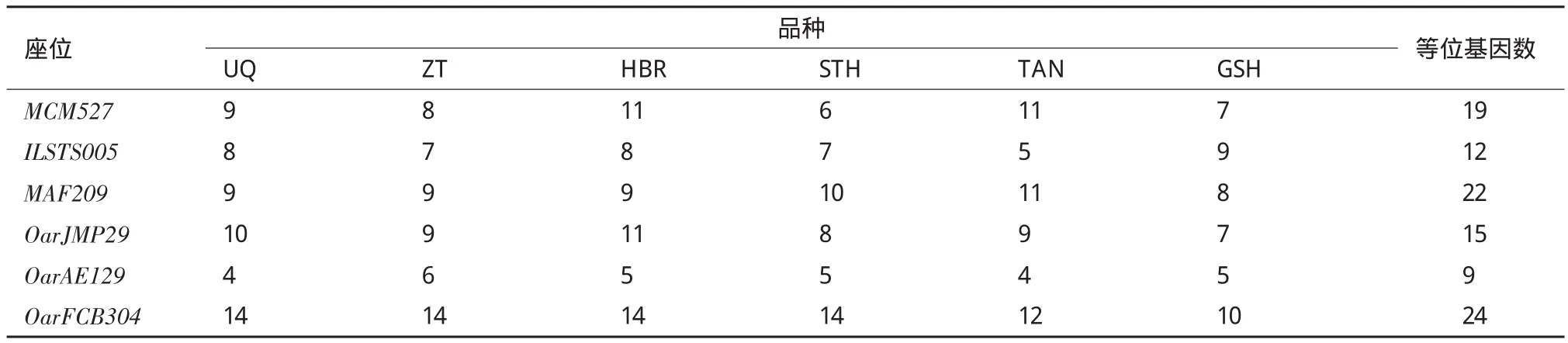
表3 6个微卫星座位在6个绵羊品种中的等位基因数
2.2 品种内的遗传变异检测
2.2.1 观测杂合度和期望杂合度 6个绵羊品种的观测杂合度(HO)和期望杂合度(HE)的检测结果见表4。就座位而言,MAF209 座位的平均HO最高,达到0.764;OarAE129 座位的平均HO最低。就品种而言,6个绵羊品种的群体杂合度均较高,除ZT的HO为0.599外,其余都在 0.6-0.7之间,属于高度杂合群体。
2.2.2 多态信息含量(PIC) 由表4可知,就座位而言,6个微卫星座位平均PIC值在0.505-0.755 之间,表现为高度多态,MCM527座位平均PIC值最大,达到 0.755。表明所选6个微卫星标记均具有较高的多态性,能够提供大量的遗传信息,可作为有效的分子遗传标记用于群体遗传多样性分析。就品种而言,6个绵羊品种PIC均高于0.5。GSH为培育品种,平均PIC值为0.672,其余5个地方绵羊品种平均PIC值在0.609-0.680间分布,表明这6个绵羊品种的遗传多样性较高,具有很高的育种潜力。
2.2.3 Hardy-Weinberg平衡检验 本研究中Hardy-Weinbery 平衡检验结果(表5)显示,GSH在2个基因座处于哈温不平衡、其余5个绵羊品种均在1个基因座处于不平衡状态。
2.2.4 群体内近交系数 6个绵羊品种的近交系数(FIS)测定值(表6)显示,6个绵羊品种的FIS范围是0.041-0.166,6个微卫星位点的平均FIS值在-0.005-0.254 7之间,矫正后的P值为0.001 4,除ZT的P值与校正后的P值相同外,其余5个品种的P值都大于校正后的P值,显示近交程度不明显。ZT的FIS值最大(FIS=0.166),其近交程度较高,通过校正其近交系数显著偏离0,这可能与尚未建立ZT的保护区和保种场和处于农户自繁自养状态有关。
2.3 品种间的遗传关系
2.3.1 遗传距离 基于SSR数据计算了6个绵羊品种间的Nei氏标准遗传距离(DS),结果见表7。由表7可见,UQ、ZT、HBR、STH和TAN 5个地方品种之间遗传距离在0.012-0.156之间,HBR与UQ遗传距离最小,STH与UQ和HBR两个品种间的遗传分歧较小,GSH与其他5个地方品种之间的遗传距离均较大。
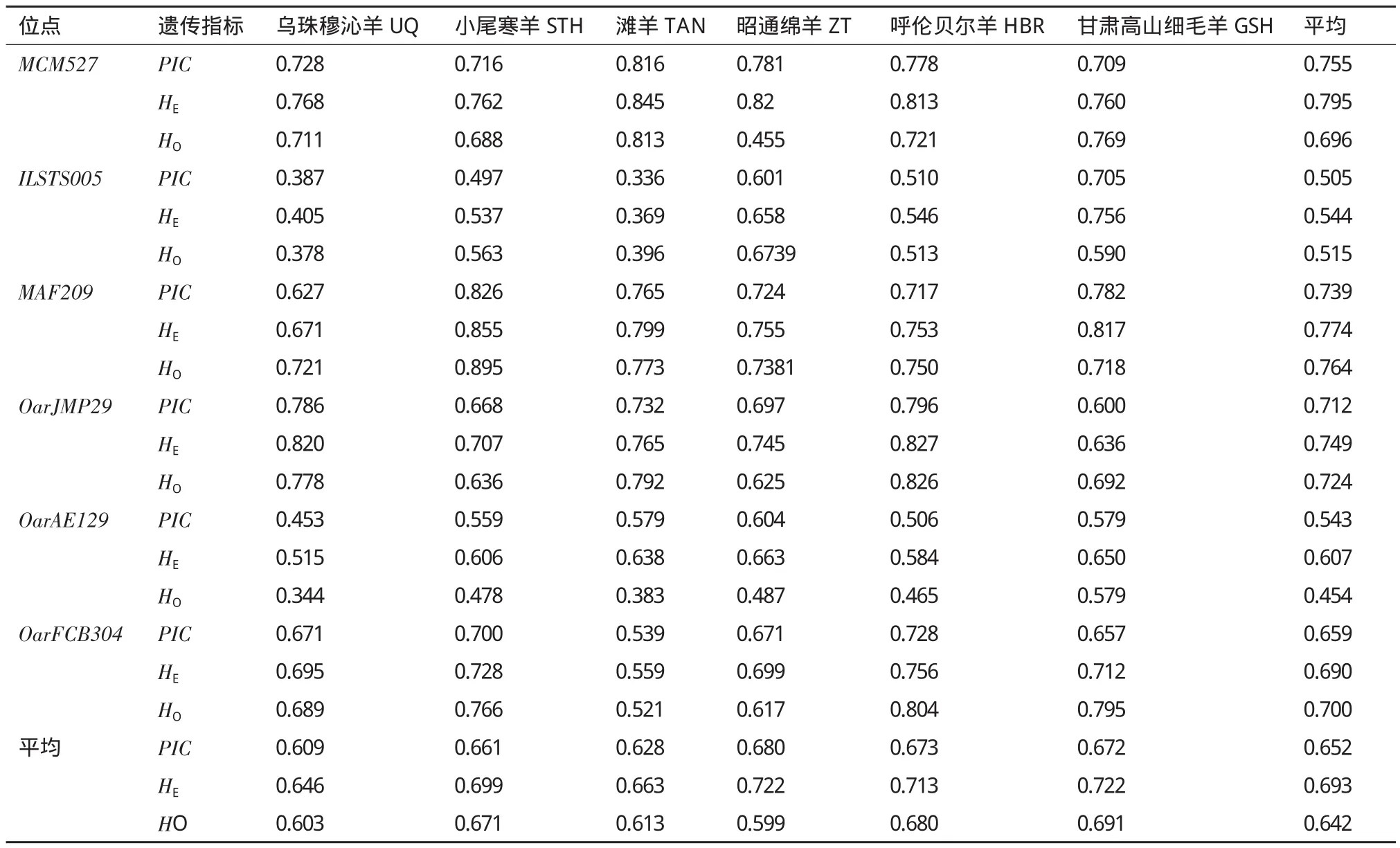
表 4 6个绵羊品种在6个微卫星位点上的遗传变异参数
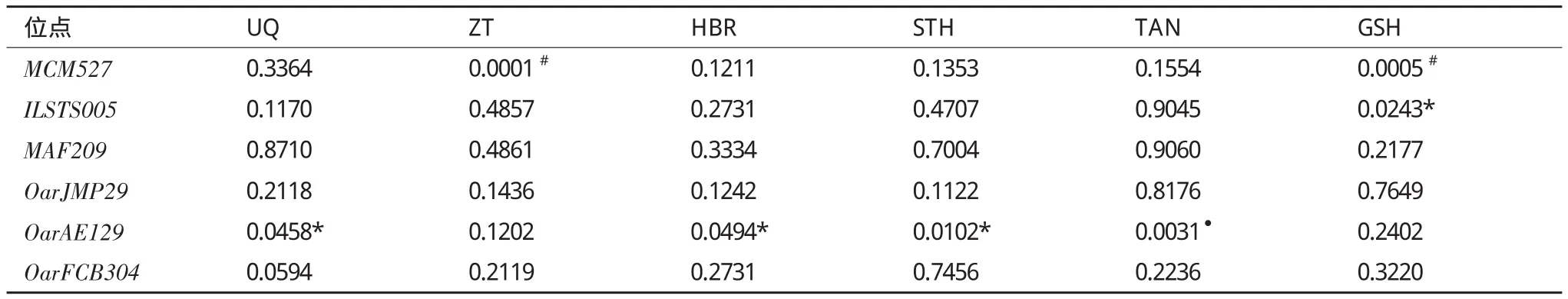
表5 哈温平衡检测各品种各基因座P值
2.3.2 聚类分析 主成分分析是揭示群体间遗传分化程度和遗传关系的一种聚类方法,研究群体间在多个标记座位上的差异性,具有多变量的特征[18]。基于微卫星数据,利用MVSP3.1软件进行了主成分分析,结果如表8所示,在5个主成分中前3个主成分累计贡献率达到76.795%,足以代表原始因子的大部分信息,据此绘制主成分分析散点图(图1)。其中第1主成分、第2主成分和第3主成分分别占总变异的38.02%、23.19%和15.58%。
在前3个主成分中,第1主成分将GSH与其他5个地方品种分开;第2主成分将ZT从5个地方品种中分出;第3主成分将UQ、HBR和STH分为一类,与TAN分开。同样利用MVSP3.1软件进行了UPGMA聚类分析,结果如表9所示,据此得到聚类图(图2),与主成分分析结果基本一致。
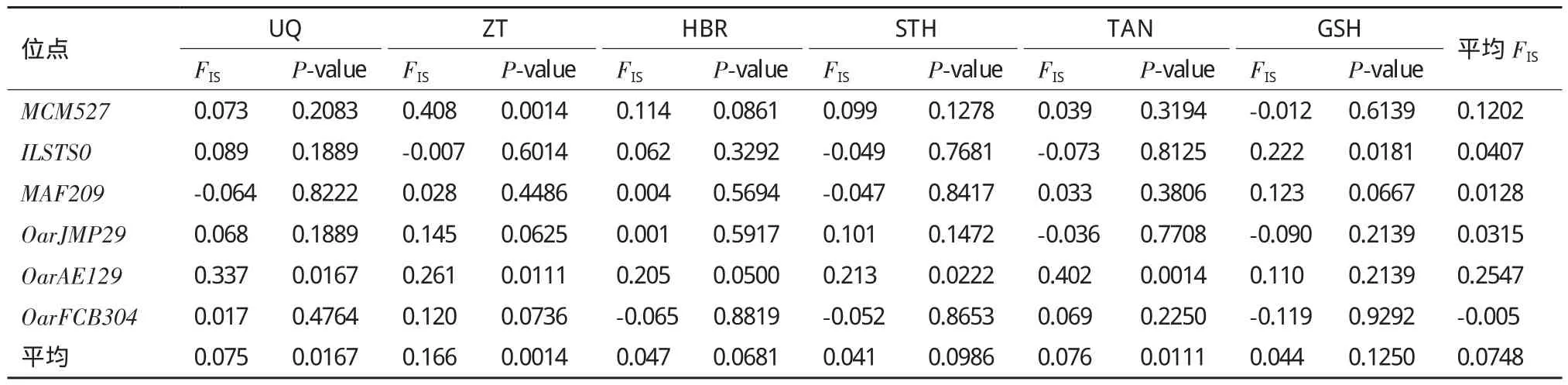
表 6 六个绵羊品种的近交系数
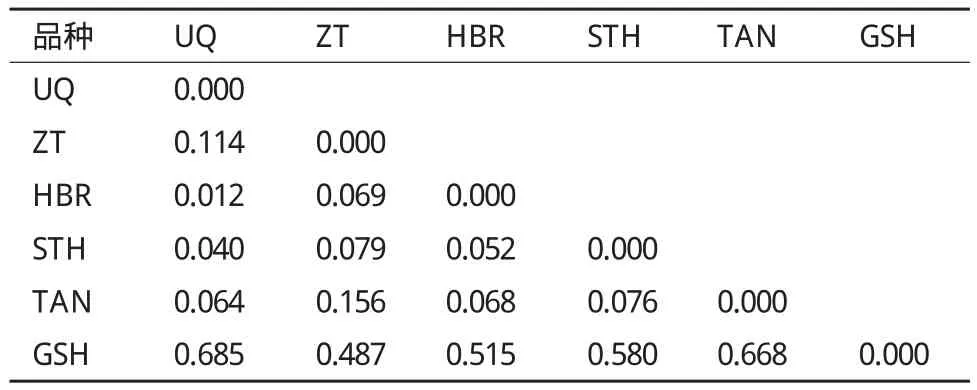
表7 6个绵羊品种间的遗传距离
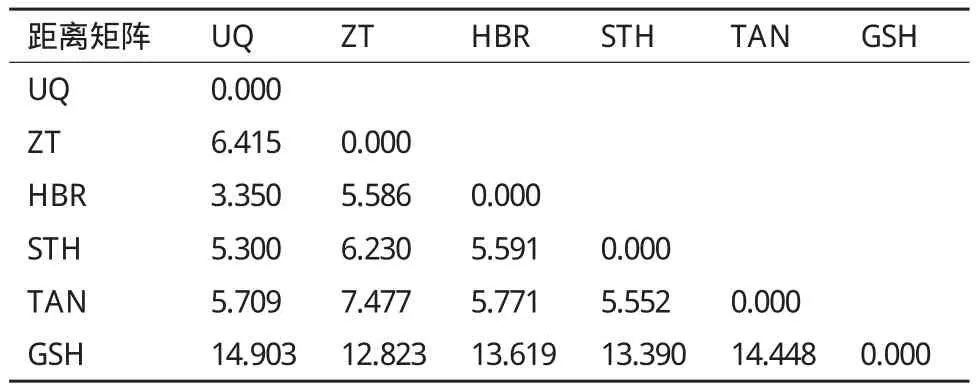
表9 UPGMA聚类结果
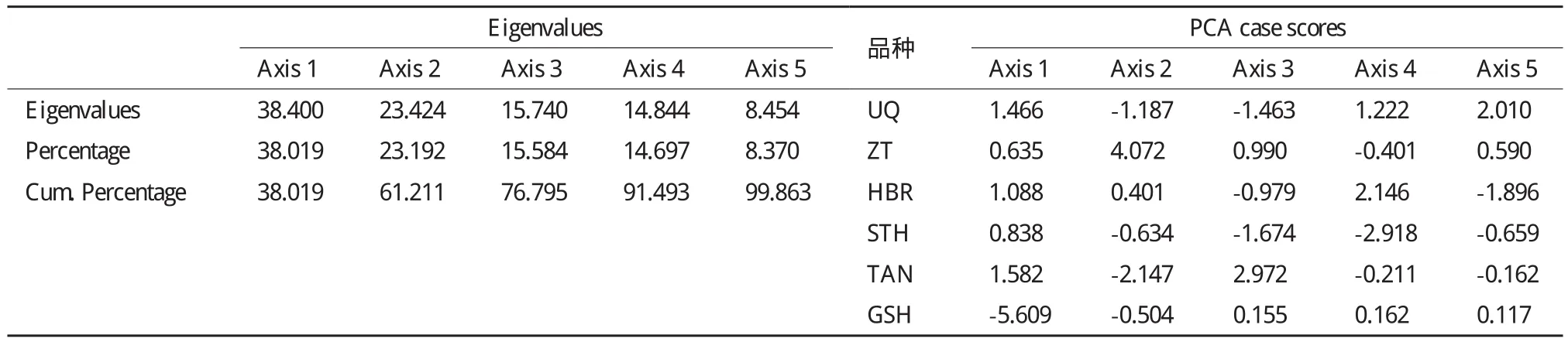
表8 MVSP主成分分析结果
3 讨论
3.1 遗传多样性和遗传分化分析
本研究所用的6个微卫星座位在6个绵羊品种中均检测到9个以上等位基因,各座位的多态信息含量都很高。根据Bolstein等[19]提出的基因变异程度衡量指标,本研究涉及的6个微卫星座位均属高度多态基因座。因此,初步认为这6个微卫星标记是普遍适合于绵羊遗传分化的多态标记。
多态信息含量(PIC)是群体内遗传变异的量度指标,可用来描述微卫星位点的变异程度。Ma等[20]利用30个微卫星标记分析了12个地方绵羊品种的遗传多样性,平均PIC在0.519-0.666之间;储明星等[21]利用2个微卫星标记对5个绵羊品种的研究结果得出平均PIC为0.60;本研究中6个微卫星均为高度多态,其中以MCM527的多态性最高,为0.755。
杂合度又称基因多样性,反映群体在多个位点上的遗传变异。一般认为它是度量群体遗传变异的一个重要参数。本实验所选取的6个微卫星平均HE在0.544-0.795之间,6个绵羊品种的平均HO为0.599-0.691,表明这6个绵羊品种具有丰富的遗传变异和较高的遗传多样性。吕慎金等[22]利用30个微卫星标记得出8个绵羊群体的平均杂合度为0.539-0.714。储明星等[23]用5个微卫星标记揭示的STH平均杂合度为0.456 9,而本研究中6个微卫星标记得到STH的平均杂合度为0.671,表明本研究中所选择的微卫星座位具有较高的多态性。
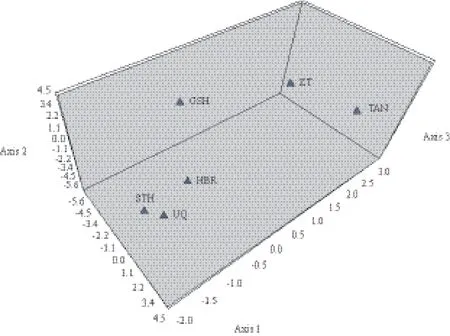
图 1 6个绵羊品种前3个主成分的三维散点图
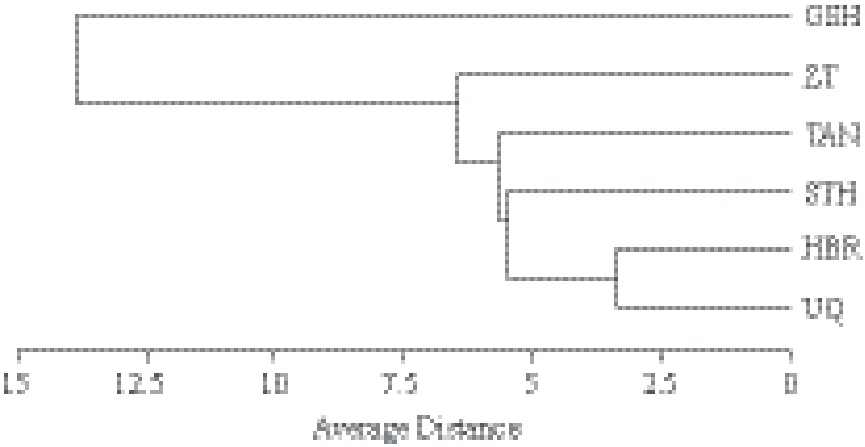
图 2 6个绵羊品种间的UPGMA聚类图
微卫星DNA本身是选择中性的,不受选择的压力,因此在一个理想群体中各等位基因在群体中的分布频率应该是稳定的[24]。通过哈代温伯格平衡检验6个座位,发现MAF209、OarJMP29和OarFCB304三个座位在6个品种都处于平衡状态,ILSTS005座位在GSH偏离平衡,MCM527座位在ZT和GSH偏离平衡,OarAE129在UQ、HBR、STH和TAN等4个品种中偏离哈代温伯格平衡。
3.2 遗传距离和聚类分析
该研究通过Arlequin软件计算得到了Nei 氏标准遗传距离,应用MVSP 3.1进行了主成分分析和UPGMA聚类。通过这两种方法,发现6个绵羊品种间的聚类结果与遗传距离基本一致。一般认为群体分化时间越短,遗传距离越小,HBR、UQ、STH和TAN等4个本地品种之间的遗传距离较小,ZT与这4个品种之间的遗传距离相对较大。而培育品种GSH与5个地方品种遗传距离均较大,独自聚为一类,聚类结果与品种的形成史、分化及地理分布基本一致。如HBR与UQ遗传距离最小,首先聚为一类,它们均来自内蒙古,生活环境极为相似,均是在当地长期的自然和人工选择情况下而逐渐育成的肉脂兼用型绵羊品种[25]。GSH是从1950年开始,采用复杂育成杂交方法进行培育,于1981年育成的培育品种[26],与其他5个地方绵羊品种遗传距离最大,亲缘关系较远。
HBR、UQ、STH和TAN等4个品种均隶属于蒙古羊系统,起源于共同的祖先群体而亲缘关系相对较近,有相关研究也证实了这一观点。如仲涛等[27]采用21个微卫星座位,运用DA遗传距离将9个地方绵羊品种进行了分类,其中属于蒙 古羊系的UQ、HBR、TAN和湖羊聚 为一类。郭小雅等[28]分析了我国6个绵(山)羊品种的遗传分化,进一步证明了UQ和STH、TAN、湖羊、同羊有较近的亲缘关系,共同归属于“蒙古羊”系。任春环等[29]分析了5个绵羊品种的遗传多态性,结果表明TAN与STH的亲缘关系较近。马月辉等[1]采用30个微卫星标记,对12个中国地方绵羊品种和一个外来品种进行遗传多样性研究,发现苏尼特羊和UQ首先聚为一类,TAN和STH聚为一类,然后这4个品种作为一个分支聚为一类。本研究中TAN与STH遗传距离较小为0.076,STH与UQ遗传距离最小为0.040,本研究中仅用了6个微卫星位点分析6个绵羊品种遗传多样性,但与先前研究结果基本一致,说明本研究采用的6个高度多态微卫星在一定程度上能够反映绵羊群体的遗传结构。
昭通绵羊的起源有可能是云南本土或是由西藏的野生绵羊驯化而来[27]。兰蓉等[30]对云南绵羊进行了线粒体DNA和血液蛋白多态性研究,结果发现昭通绵羊和印度绵羊、尼泊尔绵羊关系较近,而与西藏绵羊关系较远,但其起源还有待于深入研究。仲涛等[31]对中国部分绵羊品种的遗传多样性进行了研究,表明昭通绵羊与其他藏系绵羊间遗传分化较明显,支持了昭通绵羊起源于云南本土的说法。娄渊根等[32]认为昭通绵羊因位于云贵高原,地理隔离阻碍了其与其他品种的基因交流,使其在分类上自成一类。本研究中昭通绵羊与其他5个绵羊品种遗传距离较大,主成分分析和聚类分析也得到相同的结果,研究结果支持昭通绵羊起源于云南本土的说法。
4 结论
本研究选用的6个微卫星为高度多态性位点,在一定程度上可以反映绵羊群体的遗传结构。通过微卫星标记分析发现6个绵羊品种具有丰富的遗传变异和较高的遗传多样性。品种间遗传距离分析、主成分分析及UPGMA聚类,结果均一致说明培育品种GSH与其他5个地方品种的遗传分歧最大,HBR与UQ遗传分歧最小;STH、TAN、HBR和UQ共同归属于蒙古羊系,亲缘关系相对较近,进一步证实了STH与TAN亲缘关系较近的说法,各绵羊品种间的遗传关系与其形成史、分化及地理分布基本一致,昭通绵羊与其他绵羊品种间的遗传距离较大,支持了其起源于云南本地的观点。
致谢:感谢中国农业科学院北京畜牧兽医研究所马月辉研究员、四川农业大学动物科技学院仲涛副研究员在样品采集中给予的帮助。
[1]马月辉, 吕慎金, 侯冠或, 等. 中国部分绵羊品种遗传多样性研究[C]//中国畜牧兽医学会养羊学分会全国养羊生产与学术研究会议论文集. 中国草食动物杂志出版社, 2005.
[2]岳远瑞, 袁晓航, 周路, 等. 新疆7个绵羊品种MHC区段微卫星遗传多样性的分析[J]. 石河子大学学报:自然科学版, 2014, 32(4):438-443.
[3]Botstein D, White RL, Skolnick M, et al. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32:314-331.
[4]Lumsden JM, Loard EA, Montgomery GW. Characterisation and linkage mapping of ten microsatellite markers derived from a sheep x hamster cell hybrid[J]. Animal Genetics, 1996, 27:203-206.
[5]Chen HJ, Yue YS, Fan XZ, et al, Study comparatively on genetic distance and genus analysis measures in Shandong indigenous chicken breeds[J]. Journal of Farming and Veterinarian, 2004, 35(1):33-36.
[6]Sun W, Chang H, Tsunoda K, et al. Analysis of geographic and pairwise genetic distances among sheep populations[J]. Biochem Genet, 2010, 48(5-6):376-84.
[7]Agaviezor BO, Peters SO, Adefenwa MA, et al, Morphological and microsatellite DNA diversity of Nigerian indigenous sheep[J]. Anim Sci Biotechnol, 2012, (1):38.
[8]Yilmaz O, Cemal I, Karaca O. Genetic diversity in nine native Turkish sheep breeds based on microsatellite analysis[J]. Anim Genet, 2014, 45(4):604-608.
[9]Leroy G, Danchin-Burge C, Palhière I, et al. How do introgression events shape the partitioning of diversity among breeds:a case study in sheep[J]. Genet Sel Evol, 2015, 47(1):48.
[10]Ćurković M, Ramljak J, Ivanković S, et al. The genetic diversity and structure of 18 sheep breeds exposed to isolation and selection[J]. J Anim Breed Genet, 2016, 133(1):71-80.
[11]曾盛诚. 利用20个微卫星标记分析国内外11个鸡群体的遗传多样性[D]. 北京:中国农业科学院, 200 9.
[12]Park SDE. Trypanotolerance in West African Cattle and thepopulation genetic effects of selection[D]. Dublin:University of Dublin, 2001.
[13]Excoffier L, Lischer HEL. Arlequin 3.5:A new series of programs to perform population genetics analyses under Linux and Windows[J]. Molecular Ecology Resources, 2010, 10:564-567.
[14]Raymond M, Rousset F. GENEPOP(Version 1. 2):population genetics software for exact tests and ecumenicism[J]. Journal of Heredity, 1995, 86(3):248-249.
[15]Goudet J. STAT(Version 1. 2):A computer program to Calculate F-Statistic[J]. J Hered, 1995, 86:485-486.
[16]Nei M, Li WH. Mathematical model for studying genetic variation in terms of restriction endonucleases[J]. Proc Natl Acad Sci USA, 1979, 76:52-69.
[17]Nei M, Li WH. Analysis of gene diversity in subdivided populations[J]. Proc Nat l Acad Sci USA, 1973, 70:3321-3323.
[18]常爽. 9个绵羊群体遗传多样性的微卫星分析[D]. 郑州:河南农业大学, 2010.
[19]Botstein D, White RL, Skolnick MH, Davis R. Construction of a genetic linkage map in man using restriction fragment length polymorphisms[J]. Am J Hum Genet, 1980, 32:314-331.
[20]Ma YH, R ao SQ, Lu SJ, et al. Phylogeography and origin of sheep breeds in Northern China[J]. Conservation Genetics, 2006, 7(1):117-127.
[21]储明星, 程金华, 过纬. 微卫星标记OarAE101和BM1329在五个绵羊品种中的初步研究[J]. 遗传学报, 2001, 28(6):510- 517.
[22]吕慎金, 杨燕, 候冠玉, 等. 运用微卫星标记对中国地方绵羊品种的遗传多样性分析[J]. 畜牧兽医学报, 2008, 7:858-865.
[23]储明星, 王吉振, 王爱国, 等. 小尾寒羊五个微卫星基因座遗传多态性研究[J]. 遗传学报, 2002, 29(6):502-506.
[24]李延璐. 7个绵羊群体15个微卫星DNA座位遗传多样性分析[D]. 合肥:安徽农业大学, 2009.
[25]赵有璋. 中国养羊学[M]. 北京:中国农业出版社, 2011.
[26]杜立新. 中国畜禽遗传资源志. 羊志[M]. 北京:中国农业出版社, 2011.
[27]仲涛, 马月辉, 关伟军, 等. 10 个绵羊品种的微卫星 DNA 多态性研究[J]. 畜牧兽医学, 2008, 39(5):555-561.
[28]郭小雅, 杨章平, 常洪, 等. 13我国6个绵(山)羊群体遗传分化的微卫星分析[J]. 畜牧与兽医, 2007, 39(5):16-20.
[29]任春环, 张子军, 吴建平, 等. 5个绵羊群体微卫星标记的遗传多态性分析[J]. 安徽农业大学学报, 2010, 37(3):436-439 .
[30]兰蓉, 洪琼花, 张俊, 等. 云南不同地区绵羊血液蛋白多态性研究[J]. 草食家畜, 2002, 3(1):43-47.
[31]仲涛. 中国部分绵羊品种的遗传多样性研究[D]. 北京:中国农业科学院, 2007.
[32]娄渊根, 刘丑生, 刘建学, 等. 9个绵羊品种微卫星DNA遗传多样性分析[J]. 中国畜牧兽医, 2011, 38(10):106-112.
(责任编辑 李楠)
The Genetic Diversity of Six Chinese Sheep Breeds by Six Microsatellite Markers
CHEN Li-peng HUANG Yong-fu ZHAO Yong-ju E Guang-xin NA Ri-su
(College of Animal Science and Technology,Southwest University,Chongqing 400716)
Six microsatellite loci were used to analyze the genetic diversity of 280 sheep from 6 Chinese bre eds of Ujumqin sheep,Smalltailed Han sheep,Tan sheep,Zhaotong sheep,Hulun Buir sheep,and Gansu Alpine Merino. We calculated the polymorphism information content,heterozygosity and genetic distance,analyzed the principal components and clustered UPGMA from these 6 sheep breeds. Altogether,101 alleles were found,the mean heterozygosity was 0.599-0.691,and mean polymorphism information content was 0.609-0.680. The Gansu Alpine Merino sheep had the most genetic divergence compared to other sheep breeds,while the Hulun Buir and Ujumqin ones were the least in genetic divergence. The above results showed that these 6 breeds had relatively high genetic diversity,and their genetic relationships were consistent with their formation history,differentiation and geographical distribution.
microsatellite;genetic diversity;sheep
10.13560/j.cnki.biotech.bull.1985.2016.05.012
2015-07-28
中央高校基本科研业务费专项资金(SWU114023)
陈李鹏,男,硕士,研究方向:动物遗传;E-mail:clpshanxi@126.com
黄勇富,男,研究员,研究方向:动物遗传多样性、遗传资源评价与创新利用;E-mail:hyf65@163.com
