中国新疆小反刍兽疫病毒F基因序列分析
2016-06-03马海璐刘永宏曹胜波刘学锋王海国李岳洋马世强蒋秀梅张保军萨依甫加玛卡依萨尔努尔古丽图尔贡廖秋萍焦海宏
马海璐,刘永宏,赵 丽,曹胜波,刘学锋,王海国,李岳洋,马世强,蒋秀梅,张保军,萨依甫加玛·卡依萨尔,努尔古丽·图尔贡,廖秋萍,焦海宏
(1.塔里木大学 动物科学学院,新疆生产建设兵团塔里木畜牧科技重点实验室,新疆阿拉尔 843300;2.华中农业大学 动物医学院,武汉 430070;3.新疆生产兵团 第十四师一牧场,新疆和田 848306;4.新疆生产建设兵团 第三师红旗农场生产科,新疆阿图什 845350)
中国新疆小反刍兽疫病毒F基因序列分析
马海璐1,刘永宏1,赵丽1,曹胜波2,刘学锋3,王海国4,李岳洋1,马世强1,蒋秀梅1,张保军1,萨依甫加玛·卡依萨尔1,努尔古丽·图尔贡1,廖秋萍1,焦海宏1
(1.塔里木大学 动物科学学院,新疆生产建设兵团塔里木畜牧科技重点实验室,新疆阿拉尔843300;2.华中农业大学 动物医学院,武汉430070;3.新疆生产兵团 第十四师一牧场,新疆和田848306;4.新疆生产建设兵团 第三师红旗农场生产科,新疆阿图什845350)
摘要为分析中国新疆小反刍兽疫病毒(PPRV)毒株分子演变特点和疫情传播路线,从GenBank数据库下载所有国家全部的PPRV F基因全序列,应用DNAStar软件,结合疫情毒株时空分布进行序列分析。结果显示,中国新疆PPRV株F基因全序列,与2014年小反刍兽疫情中河南和北京毒株核苷酸的同源性最高,为99.8%~99.9%,与中国西藏3个毒株核苷酸的同源性为97.7%,与中国采用的疫苗株核苷酸同源性为92.9%,核苷酸同源性最低的是肯尼亚毒株,与国外毒株核苷酸同源性最高的是印度毒株。F基因系统进化关系分析显示,中国新疆PPRV株属于基因Ⅳ系。F蛋白氨基酸同源性结果显示,除与科特迪瓦1989年毒株ICV89氨基酸同源性最低外,与其他氨基酸同源性比对结果均与核苷酸的比对结果一致。中国新疆PPRV株存在一定程度的变异,可能是由周边国家传入。
关键词中国;新疆;小反刍兽疫;F基因
小反刍兽疫(Peste des petits ruminants,PPR),是由小反刍兽疫病毒(Peste des petits ruminants virus,PPRV)引起的一种小反刍动物急性接触性传染疾病。有报道[1-2]显示,牛、水牛、骆驼和狮均可感染该病毒,表现为发热、眼鼻分泌物增多、口炎、肠炎和肺炎等。
1942年,西非科特迪瓦首次报道PPR[3],之后PPR在多个国家和地区均有发生,呈全球蔓延趋势。据报道,目前 至少有49个国家发生PPR疫情[2,4],其中亚洲有23个国家,中国周边有12个国家[4-5]。
2005年,通过发病情况和血清学调查发现,中国西藏发生小反刍兽疫疫情;随后,通过分子流行病学调查确证西藏于2007年7月发生PPR[6-8]。2008年和2010年,西藏至少发生3次PPR疫情[7-9]。2013年年底,新疆伊犁地区成为中国第2个发生PPR疫情的省区,随后,新疆多地暴发该疫情[10]。2014年,PPR疫情呈大流行趋势,在中国的至少22省(市、自治区)256个县发生,其中,云南、江苏、安徽、湖北和贵州等省为重灾区,并于4月达到发病高峰。截止2014年9月,中国约有7万多只绵羊和山羊因患PPR被扑杀,损失巨大[11]。
PPRV为副粘病毒科麻疹病毒属的成员[12],只有1个血清型[13],该病毒核酸类型为单股负链RNA,基因组约为15 948 nt,从3′到5′依次为N、P、M、F、H和L6个基因,分别编码核衣壳蛋白N、磷蛋白P、基质蛋白M、融合蛋白F、血凝蛋白H、大蛋白L等6个结构蛋白[14]。
2013年11月,新疆伊犁哈萨克自治州霍城县三宫乡某村发生PPR疫情,提取该村患病山羊淋巴结组织样品,通过病毒RNA提取、RT-PCR、PCR产物纯化测序及末端RACE法,获得完整PPRV基因组序列,命名为PPRV China/XJYL/2013,为新奇变异株,GenBank登录号为KM091959[15]。根据文献[1,14]报道,以N或F基因序列可进行遗传演化分析。
本研究从GenBank数据库下载各国PPRVF基因全序列,并结合每次疫情发生的时空分布,对中国新疆小反刍兽疫疫情毒株分子演变特点和疫情传播路线进行系统分析,以期为该病防治及疫病消灭提供依据。
1材料与方法
1.1材 料
中国新疆仅有的一个PPRV China/XJYL/2013株F基因全序列,中国其他地区和其他国家的全部PPRVF基因全序列,均来源于GenBank数据库,各毒株的具体信息见表1。
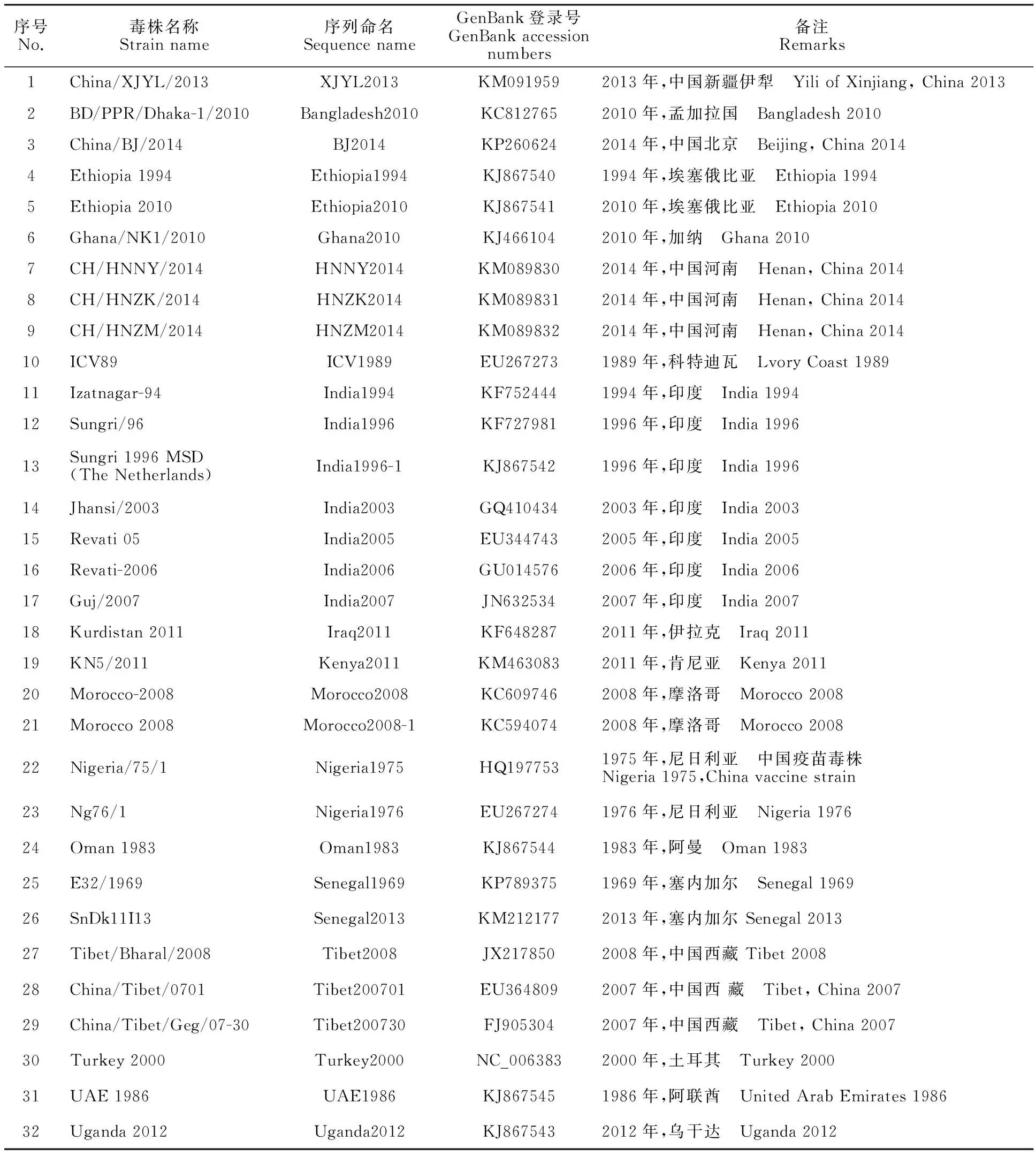
表1 参考毒株
1.2方 法
搜索GenBank数据库全部的PPRVF基因全序列,运用DNAStar软件的EditSeq程序将其核苷酸和氨基酸序列保存为*.Seq和*.Pro格式文件,运用该软件MegAlign程序的Clustal W法进行比对,然后在Veiw菜单中通过Sequence Distance和Phylogenetic Tree提取序列比对结果,并绘制进化树图。同时,结合毒株时空分布,分析中国新疆伊犁地区PPRV来源及演变特征。
2结果与分析
2.1中国新疆PPRV F基因核苷酸序列分析
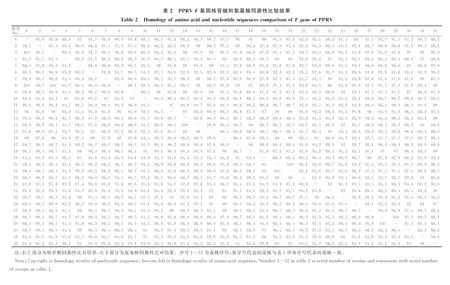
对不同时空分布的32个PPRVF基因全序列进行核苷酸同源性分析,结果显示,2013年新疆伊犁地区PPRV株China/XJYL/2013F基因与2014年PPP疫情大流行中河南和北京PPRV毒株F基因核苷酸同源性最高,达到99.8%~99.9%;与中国西藏阿里地区和那曲地区3个PPRV毒株F基因核苷酸同源性均达到97.7%;与中国疫苗株Nigeria/75/1F基因核苷酸同源性为92.9%;核苷酸同源性最低的是肯尼亚2011年KN5/2011株,与其他国家毒株核苷酸同源性最高的是印度1994年Izatnagar-94株;中国毒株与其他国家毒株核苷酸同源性为87.8%~98.3%(表2右上)。
采用PPRVF基因核苷酸全序列绘制基因进化树(图1),结果显示,1989年西非科特迪瓦的1个毒株和1969年塞内加尔的1个毒株构成1个小分支(Ⅱ);2个西非尼日利亚的毒株、1个塞内加尔2013年毒株和1个加纳2010年毒株构成1个小分支(Ⅰ);亚洲邻国阿联酋和阿曼各1个毒株、非洲东北部埃塞俄比亚1994年1个毒株、非洲东部的肯尼亚和乌干达各1个毒株构成为1个分支(Ⅲ);亚洲18个毒株、非洲东北部埃塞俄比亚2010年1个毒株和非洲西北部摩洛哥2个毒株构成1个分支(Ⅳ)。F基因系统进化树显示,埃塞俄比亚和塞内加尔不同年代的毒株被划分到不同的分支;2013年中国新疆伊犁地区毒株与中国西藏毒株和2014年PPP疫情中国其他毒株被划分在同一个大分支(Ⅳ),但2013-2014年毒株与中国西藏毒株被划分为2个不同的小分支,与孟加拉国2010年毒株系统进化关系最近。
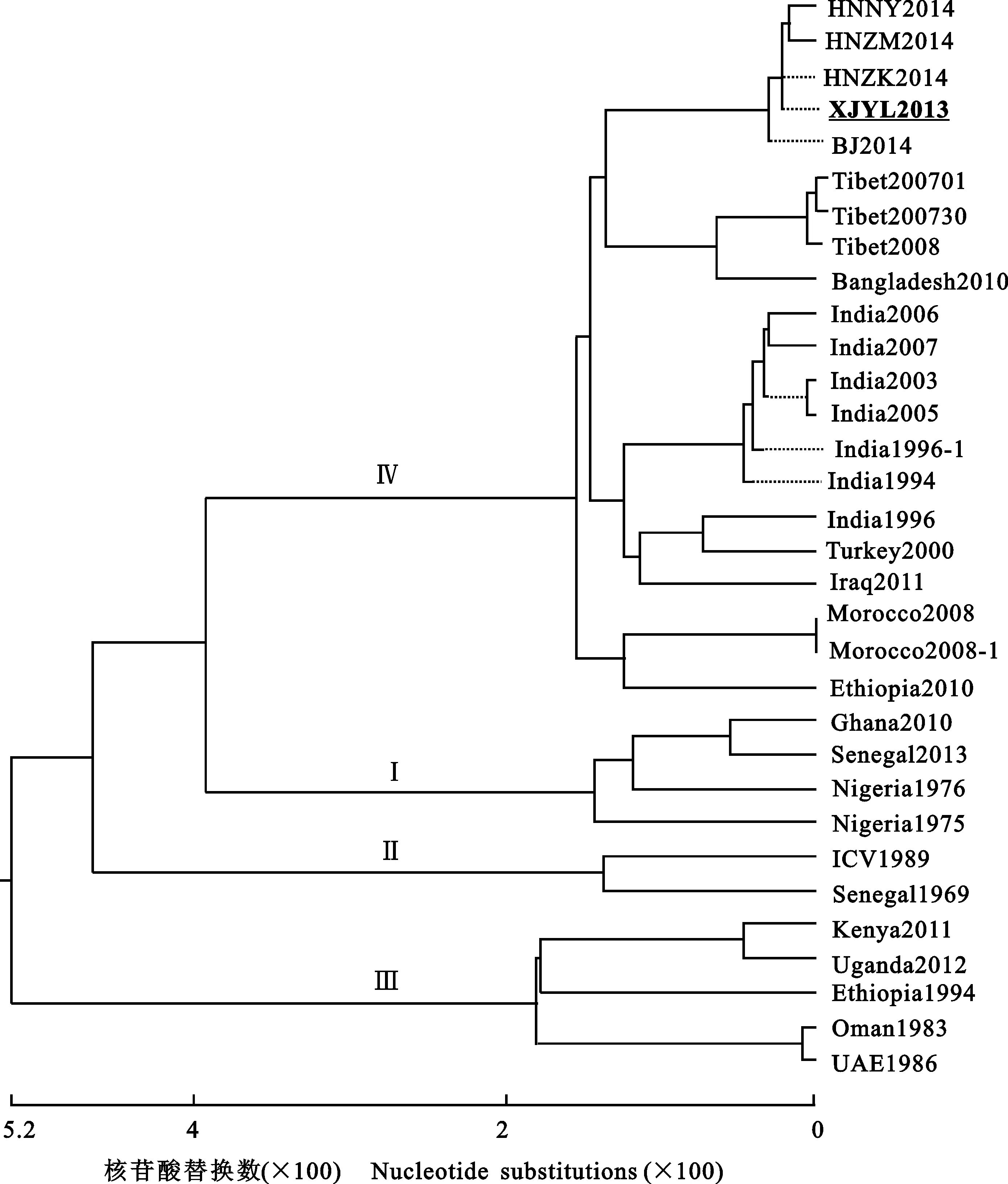
图1 不同地区和不同年代的小反刍兽疫病毒毒株F基因系统进化树
2.2中国新疆PPRV F蛋白氨基酸序列分析
对不同时空分布的32个PPRV F蛋白全序列进行氨基酸同源性分析,结果显示,2013年中国新疆伊犁地区PPRV株China/XJYL/2013 F蛋白与2014年PPP疫情大流行中国河南和北京PPRV毒株F蛋白氨基酸同源性最高,达到99.8%~100%;与中国西藏阿里地区和那曲地区3个PPRV毒株F蛋白氨基酸同源性为98.7%~98.9%;与中国疫苗株Nigeria/75/1 F蛋白氨基酸同源性为96.7%;与科特迪瓦1989年毒株ICV89氨基酸同源性最低,与国外毒株氨基酸同源性最高为印度1994年Izatnagar-94株、1996年Sungri/96株和2003年Jhansi/2003株,同源性均为99.1%;中国毒株与其他国家毒株氨基酸同源性为94.0%~99.3%(表2左下)。
3讨 论
3.12014年中国PPR疫情来源分析
通过对F基因核苷酸序列进行遗传演化分析[1],可反映出PPRV更多的以时间为轴地演化过程,从而为该病毒在不同地理区域间的传播途径提供线索[12]。研究者们用PPRVF基因部分核苷酸序列进行分析,将不同来源的PPRV毒株划分为4系,其中,Ⅰ系由塞内加尔和尼日利亚的毒株组成,Ⅱ系由几内亚和科特迪瓦的毒株组成,Ⅲ系包括埃塞俄比亚、也门、苏丹以及1株1992年分离自印度南部的毒株,Ⅳ系均为亚洲毒株[16]。
本研究采用PPRVF基因全序列进行系统进化分析,将GenBank数据库的32个PPRV流行株F基因全序列进行分系,结果显示,新疆China/XJYL/2013株与西藏3个毒株均划分为Ⅳ系,与前人报道相符[12,17]。但是,中国2014年PPP疫情中的PPRV毒株与早前中国西藏毒株未划分在同一个小分支,且基因序列也有差异。由于2010-2013年11月中国未发生过小反刍兽疫疫情;同时,考虑新疆少数民族生活习惯及消费羊产品的群体、新疆丰富的野生小反刍兽资源、新疆的地理位置和国家对新疆大力发展养羊业的政策等导致羊只流动频繁、接触性大等因素,分析得出,2014年PPP疫情为西藏疫情的延续或者是西藏毒株为适应中国环境和易感动物而发生一些基因变异的可能性极小。
另外,China/XJYL/2013株与国外毒株中的2010年孟加拉国毒株遗传演化关系最近,这与王志亮等[17]报道不一致,可能与该文献PPRV进化分析中采用的是F基因的部分序列有关,采用具有代表性基因的全序列分析遗传发生关系,可能更具代表性;若能结合宿主、发生时间和地点进行分析则更有意义。
F蛋白氨基酸同源性分析显示,2013年中国新疆PPRV毒株F蛋白与2014年PPR疫情大流行中国其他毒株氨基酸同源性最高,与之前中国西藏毒株氨基酸同源性也较高,与国外毒株氨基酸同源性最高为印度不同年代的3个毒株,且同源性高于其与中国西藏毒株的同源性。F蛋白氨基酸同源性比对结果与核苷酸比对结果相似。
综合分析,此次PPR大流行疫情可能是周边国家毒株的跨境传入。至于更全面更准确的结果,有待于不同时空分布的更多PPRV毒株F基因全序列的报道。
3.2国外PPR疫情特征
本研究显示,Ⅰ系和Ⅱ系毒株分类与前人报道相符[1,16];Ⅲ系为亚洲邻国阿联酋和阿曼各1个毒株、非洲东北部埃塞俄比亚1994年1个毒株、非洲东部的肯尼亚和乌干达各1个毒株构成;Ⅳ系包括21个毒株,其中18个亚洲毒株,1个非洲东北部和2个非洲西北部毒株。由此可知,在系统分类中出现一个地区或一个国家存在多个PRRV基因系的情况。若一些国家出现多个基因系毒株或新奇毒株,可能与其在自身进化中受到独特的易感动物、生存环境等的影响有关。
综上所述,周边国家疫情、养殖特点、兽医卫生制度、动物及动物产品生产条件等均对中国畜牧业发展和疫情发生有一定影响,尤其对新疆等周临多国的中国边境地区有明显影响。此外,中国的引种制度、活畜频繁交易而检疫实际操作不严格或不作为等,均可能与中国多种疫情的发生有至关重要的关系。
4结 论
2013年中国新疆伊犁地区PPRV株China/XJYL/2013F基因与2014年PPR疫情大流行中国河南和北京PPRV毒株F基因核苷酸同源性最高,与肯尼亚2011年毒株KN5/2011核苷酸同源性最低,与国外毒株核苷酸同源性最高为印度1994年Izatnagar-94株。2013年中国新疆伊犁地区毒株属于Ⅳ系毒株,该毒株可能是由周边国家传入。
参考文献Reference:
[1]BALAMURUGAN V,SEN A,VENKATESAN G,etal.Peste des petits ruminants virus detected in tissues from an Asiatic lion (Panthera Leo Persica) belongs to Asian lineage iv[J].JournalofVeterinaryScience,2012,13(2):203-206.
[2]刘永宏,曹胜波,赵丽,等.中国小反刍兽疫疫情分析[J].西北农业学报,2014,23(9):19-26.
LIU Y H,CAO SH B,ZHAO L,etal.Analysis of epidemic Chinese Peste des Petits Ruminants[J].ActaAgriculturaeBoreali-OccidentalisSinica,2014,23(9):19-26(in Chinese with English abstract).
[3]GARGADENNEC L,LALANNE A.La peste des petits ruminants[J].BulletindesServicesZooTechniqueetdesEpizootiedel’AfriqueOccidentaleFranfiaise,1942,5:16-21.
[4]罗静,何宏轩.小反刍兽疫病毒的分子生物学特性及其在全球的流行[J].河北师范大学学报(自然科学版),2009,3(4):533-540.
LUO J,HE H X.The molecular biological characterization of peste des petits ruminants virus and its prevalence in the world [J].JournalofHebeiNormalUniversity(NaturalScienceEdition),2009,3(4):533-540(in Chinese with English abstract).
[5]蒋梅,杨仕标,张念祖.小反刍兽疫的流行趋势与防控[J].动物医学进展,2007,28(S1):88-91.
JIANG M,YANG SH B,ZHANG N Z.Epidemic trend and control of peste des petits ruminants[J].ProgressinVeterinaryMedicine,2007,28(S1):88-91(in Chinese).
[6]BAO J,LI L,WANG Z,etal.Development of one-step real-time RT-PCR assay for detection and quantitation of peste des petits ruminants virus[J].JournalofVirologicalMethods,2008,148(1-2):232-236.
[7]次仁罗布.西藏阿里地区小反刍兽疫的流行情况与综合防控[J].山东畜牧兽医,2013,5(34):44-45.
CIRLB.The epidemic situation and comprehensive prevention and control of Tibet Ali region of peste des petits ruminants[J].ShandongJournalofAnimalScienceandVeterinaryMedicine,2013,5(34):44-45(in Chinese).
[8]WANG Z,BAO J,WU X,etal.Peste des petits ruminants virus in Tibet,China[J].EmergingInfectiousDiseases,2009,15(2):299-301.
[9]王乐元,次真,吴国珍,等.中国西藏小反刍兽疫的发生状况与防控[J].畜牧兽医学报,2011,42(5):717-720.
WANG L Y,TSED,WU G ZH,etal.Occurrence and control strategy of peste des petits ruminants in Tibet Autonomous region of China[J].ChineseJournalofAnimalandVeterinarySciences,2011,42(5):717-720(in Chinese with English abstract).
[10]刘永宏,赵丽,曹胜波,等.小反刍兽疫在中国的流行趋势及应对措施[J].动物医学进展,2015,36(1):110-113.
LIU Y H,ZHAO L,CAO SH B,etal.Popular trends and countermeasures of peste des petits ruminants in China[J].ProgressinVeterinaryMedicine,2015,36(1):110-113(in Chinese with English abstract).
[11]WU X,LI L,LI J,etal.Peste des petits ruminants viruses re-emerging in China,2013-2014[J].Transboundary&EmergingDiseases,2015.
[12]包静月,王志亮,李林,等.我国西藏小反刍兽疫病毒China/Tib/Gej/07-30核衣壳蛋白基因和基因组启动子区的分子特征分析[J].病毒学报,2008,24(6):464-471.
BAO J Y,WANG ZH L,LI L,etal.Sequence analysis of the nucleocapsid gene and genome promoter region of peste des petits ruminants virus of Chinese origin[J].ChineseJournalofVirology,2008,24(6):464-471(in Chinese with English abstract).
[13]包静月,李林,王志亮,等.一步法实时定量RT-PCR检测小反刍兽疫病毒方法的建立[J].中国动物检疫,2007,24(8):27-29.
BAO J Y,LI L,WANG ZH L,etal.One-step real-time quantitative reverse transcriptase PCR (qRT- PCR) for the detection of peste des petits ruminants virus[J].ChineseJournalofAnimalHealthInspection,2007,24(8):27-29(in Chinese with English abstract).
[14]BAILEY D,BANYARD A,DASH P,etal.Full genome sequence of peste des petits ruminants virus,a member of the Morbillivirus genus[J].VirusResearch,2005,110(1/2):119-124.
[15]BAO J Y,WANG Q H,ZHANG Y Q,etal.Complete genome sequence of a novel variant strain of peste des petits ruminants virus,China/XJYL/2013[J].GenomeAnnouncements,2014,2(5):1-2.
[16]SHAILA M S,SHAMAKI D,FORSYTH M A,etal.Geographic distribution and epidemiology of peste des petits ruminants virus[J].VirusResearch,1996,43 (2):149-153.
[17]王志亮,包静月,吴晓东,等.我国首例小反刍兽疫诊断报告[J].中国动物检疫,2007,24(8):24-26.
WANG ZH L,BAO J Y,WU X D,etal.Diagnosis of the first outbreak of peste des petits ruminants in Tibet [J].ChineseJournalofAnimalHealthInspection,2007,24(8):24-26(in Chinese).
Received 2015-07-30Returned2015-11-20
Foundation itemCollege Students Innovation and Entrepreneurship Training Program of China (No.201510757011); College Students Innovation Program of College of Animal Science, Tarim University (No.DKY2014004,No.DKY2014009); Joint Fund Project of Huangzhou Agricultural University and Tarim University (No.HNTDLH1404).
First authorMA Hailu, male,undergraduate student.Research area:infectious diseases and immune pathology.E-mail: 420723205@qq.com
ZHAO Li, female,Ph.D,associate professor.Research area:parasitic diseases and immune pathology.E-mail: zhaolidky@126.com
(责任编辑:郭柏寿Responsible editor:GUO Baishou)
Sequence Analysis ofFGene of Peste Des Petits Ruminants in Xinjiang
MA Hailu1, LIU Yonghong1, ZHAO Li1, CAO Shengbo2, LIU Xuefeng3,WANG Haiguo4, LI Yueyang1, MA Shiqiang1,JIANG Xiumei1, ZHANG Baojun1,Sayifujiama Kayisaer1, Nuerguli Tuergong1, LIAO Qiuping1and JIAO Haihong1
(1.College of Animal Science, Tarim University, Key Laboratory of Tarim Animanl Husbandry Science and Technology of Xinjiang Production & Construction Corps, Alar Xinjiang843300, China; 2.College of Veterinary Medicine,Huazhong Agricultural University, Wuhan430070, China; 3.The 1st Pasturage Farm, Agricultural Division 14 of Xinjiang Production and Construction Corps, Hetian Xinjiang848306, China; 4.Hongqi Farm Production Department, Third Divisions, Xinjiang Production and Construction Corps, Artush Xinjiang845350, China)
AbstractIn order to analyze the molecular evolution characteristic of peste des petits ruminants virus (PPRV) and the epidemic disseminated route in Xinjiang.We downloaded the complete sequences of F gene of PPRV from GenBank of all the countries in the world.We used DNAStar software and combined it with spatial and temporal distribution of strains from NCBI to analyze its sequence.The results showed that the nucleotide homology of Xinjiang strain was the highest with the epidemic strainsof other places in China, the nucleotide homology with Tibet strain which was epidemic before, and with Chinese vaccine strain used were 97.7% and 92.9% respectively.The nucleotide homology with Kenya strain was the lowest, with India strain was the highest among foreign strains.The phylogenetic analysis of F gene showed that Xinjiang strain belongs to the gene series Ⅳ.The amino acid homology of F protein was consistent nearly with the nucleotide homology, but was the lowest with ICV89 strain from Ivory Coast.There are certain variation in the PPRV strain, it may be introduced into the neighboring countries.
Key wordsChina; Xinjiang; PPR; F gene
Corresponding authorLIU Yonghong,male,associate professor.Research area: infectious diseases and immune pathology.E-mail: lyhdky@126.com
中图分类号S855.3
文献标志码A
文章编号1004-1389(2016)03-0328-07
通信作者:刘永宏,男,副教授,研究方向为传染病与免疫病理学。E-mail: lyhdky@126.com
基金项目:国家级大学生创新创业训练计划(201510757011);塔里木大学动物科学学院大学生创新项目(DKY2014004,DKY2014009);华中农业大学塔里木大学科研联合基金(HNTDLH1404)。
收稿日期:2015-07-30修回日期:2015-11-20
网络出版日期:2016-03-06
网络出版地址:http://www.cnki.net/kcms/detail/61.1220.S.20160306.1610.004.html
第一作者:马海璐,男,本科生,研究方向为传染病与免疫病理学。E-mail:420723205@qq.com
赵丽,女,博士,副教授,研究方向为寄生虫病与免疫病理学。E-mail: zhaolidky@126.com
