2009-2014年国家冬小麦区域试验品系的遗传多样性及群体结构分析
2016-05-27刘丽华庞斌双刘阳娜李宏博赵昌平
刘丽华,庞斌双,刘阳娜,邱 军,李宏博,张 欣,王 娜,赵昌平
(1.北京市农林科学院杂交小麦工程技术研究中心/杂交小麦分子遗传北京市重点实验室,北京 100097; 2.农业部全国农业技术推广服务中心,北京 100026)
2009-2014年国家冬小麦区域试验品系的遗传多样性及群体结构分析
刘丽华1,庞斌双1,刘阳娜1,邱 军2,李宏博1,张 欣1,王 娜1,赵昌平1
(1.北京市农林科学院杂交小麦工程技术研究中心/杂交小麦分子遗传北京市重点实验室,北京 100097; 2.农业部全国农业技术推广服务中心,北京 100026)
摘要:为了解我国小麦的育种水平、发展趋势及存在的问题,利用21对核心SSR标记对2009-2014年参加国家冬小麦区域试验的430个品系进行遗传多样性和群体结构分析。结果显示,21个SSR位点共检测到236个等位变异,平均每个位点11.24个,平均基因多样性和多态性信息量(PIC)分别为0.73和0.70;从不同年份分析,参试品系的遗传多样性从2012年开始略有下降;从不同生态种植区域分析,参试品系的遗传多样性自北向南(北部冬麦区组至长江流域冬麦区组)呈明显下降趋势;UPGMA聚类、主坐标和群体结构分析均表明,长江上游和长江中下游组的参试品系与其他区组的参试品系明显分开,且分别归属于不同亚类,显示出独特的生态区域类型;此外,群体结构分析结果还揭示,71.9%的参试品系群体结构比较单一,其中长江流域组的参试品系最为单一。
关键词:小麦;区域试验;SSR标记;遗传多样性;群体结构
小麦是世界谷物贸易量最大的粮食作物,在全球粮食安全和农产品贸易中占有极其重要的地位。《中华人民共和国种子法》的颁布实施大大提高了小麦种子生产者、经营者和育种者的积极性,我国小麦新品种选育和种子产业得以快速发展,品种数量明显增加。截止2014年国家审(认)定小麦品种428个,每年参加国家区域试验的品系数以百计。区域试验是小麦品种审定和新品种推广的重要环节,是推动小麦品种更新换代的源动力,代表了当前我国小麦育种的水平和发展趋势。因此构建国家区域试验品种的DNA指纹图谱,揭示其遗传多样性,探明其群体结构,可以客观、全面的了解当前我国小麦育种的现状,对于品种选育、品种管理以及种质资源收集评价与利用均具有重要意义。
随着分子生物学的发展,分子标记技术为评估品种的遗传多样性和群体结构提供了重要手段。众多标记中,SSR和SNP标记因具有共显性遗传、标记数量多、易实现高通量等优点,已被广泛应用于玉米[1-3]、水稻[4-6]、大豆[7-8]、棉花[9]、马铃薯[10]和小麦[11-15]等作物的遗传多样性和群体结构分析中。迄今国内外关于小麦遗传多样性和群体结构分析的报道,主要集中在小麦种质资源、育种亲本和省审定品种等材料的分析[11-15],如Wang等[11]利用SSR标记对云南、西藏和新疆的小麦品种进行了遗传多样性分析,结果显示,我国云南和西藏小麦品种的遗传多样性高于新疆小麦品种;Hao等[12]利用SSR标记分析了我国核心种质的遗传多样性,结果显示,地方品种的遗传多样性明显高于育成品种;Cao等[13]利用SNP标记分析了近10年河南省审定小麦品种的遗传多样性,结果发现遗传多样性不够丰富,多数品种亲缘关系较近。
北京杂交小麦工程技术研究中心拥有国家冬小麦区域试验品种标准样品库,目前收集保存了1 300余份国家区域试验标准样品,并构建了基于SSR标记的DNA指纹图谱。本研究利用覆盖全基因组、用于中国小麦标准样品指纹构建的21对核心SSR标记对2009-2014年参加国家冬小麦区域试验的430个品系进行遗传多样性和群体结构分析,以期了解我国小麦的育种水平、发展趋势及存在的问题,为我国小麦品种改良和品种管理提供参考。
1材料与方法
1.1试验材料
供试材料为2009-2014年度参加国家冬小麦区域试验的430个品系。
1.2基因组DNA提取
采用简化的CTAB法[16]提取供试材料的基因组总DNA,每个品系提取20个个体DNA,用紫外分光光度计测量DNA浓度,总DNA用0.1×TE稀释至50 ng·μL-1,4 ℃保存备用。
1.3SSR标记分析
本研究所用的SSR标记为绘制中国小麦品种指纹图谱的21对核心SSR标记[17]。PCR反应体系为10 μL,含10×buffer 1.0 μL、10 mmol·L-1dNTP 0.2 μL、50 ng·μL-1模板DNA 3.0 μL、1.25 μmol·L-1引物2.0 μL、2 U·μL-1TaqDNA聚合酶0.25 μL(北京鼎国生物技术公司)及ddH2O 3.55 μL。PCR扩增程序:94 ℃预变性5 min;94 ℃变性30 s,50~65 ℃退火1 min,72 ℃延伸30 s,35个循环;72 ℃延伸5 min。PCR产物经6%聚丙烯酰胺变性凝胶分离(电压为2 000 V,电泳时间由PCR产物长度决定。)后,采用简化硝酸银染色法显带[16]。
1.4数据统计分析
1.4.1遗传多样性分析
按照王立新等[18]的方法记录430个供试材料的各种带型,并建立430×21个位点的DNA指纹数据库。分别基于不同年份及不同生态种植区域的材料,采用PowerMarker V3.25软件[19]对等位变异数、基因多样性(期望核苷酸杂合度)和多态性信息量(Polymorphism information content,PIC)等进行分析。采用PowerMarker V3.25软件[19]构建基于UPGMA(Unweighted pair-group method with arithmetic means)方法的Nei’s遗传距离聚类图。选用多变量统计分析软件MVSP ver.3.13(Kovach computing services),将进行国家冬小麦区域试验的9个区试组分别作为独立群体进行主坐标分析(Principal coordinate analysis,PCoA),直观反映9个区试组之间的关系。
1.4.2群体结构分析
利用软件Structure v2.3.4[20](http://pritch.bsd.uchicago.edu/ structure.html) 对供试材料进行贝叶斯聚类分析,计算各个材料相应的Q值(各个体归入某一类群的概率),并绘制群体结构图,以确定材料的遗传组成。具体分析过程如下:首先需要假定存在一个群体数目(K),并认为位点都是独立的,每个群体内都遵循Hardy-Weinberg平衡。设定K值为2~12,参数iterations设为100 000,burn-in period设为1 000 000。每个K值重复运行3次,依据似然值最大的原则选取合适的K值作为群体数目。根据所有品系在各个类群中的Q值分布,当某品系在某个类群中的Q值大于等于0.6时,推测该品系的群体结构相对比较单一,被划分到相应的类群中;当某品系在任何类群中的Q值均小于0.6 时,则推测该品系群体结构比较复杂。
2结果与分析
2.1SSR标记的多态性
21对SSR标记在430个品系中共检测到236个等位变异,每个位点等位变异数的变幅为6~20,平均等位变异数为11.24个;PIC值的变幅为0.57~0.85,平均0.70(图1)。在检测到的236个等位变异中, Xgwm610-09等位变异出现的频率最高(图2)。分析430个品系的DNA指纹数据,发现21对SSR引物能够区分430个品系中的408个品系,引物区分能力为95%。
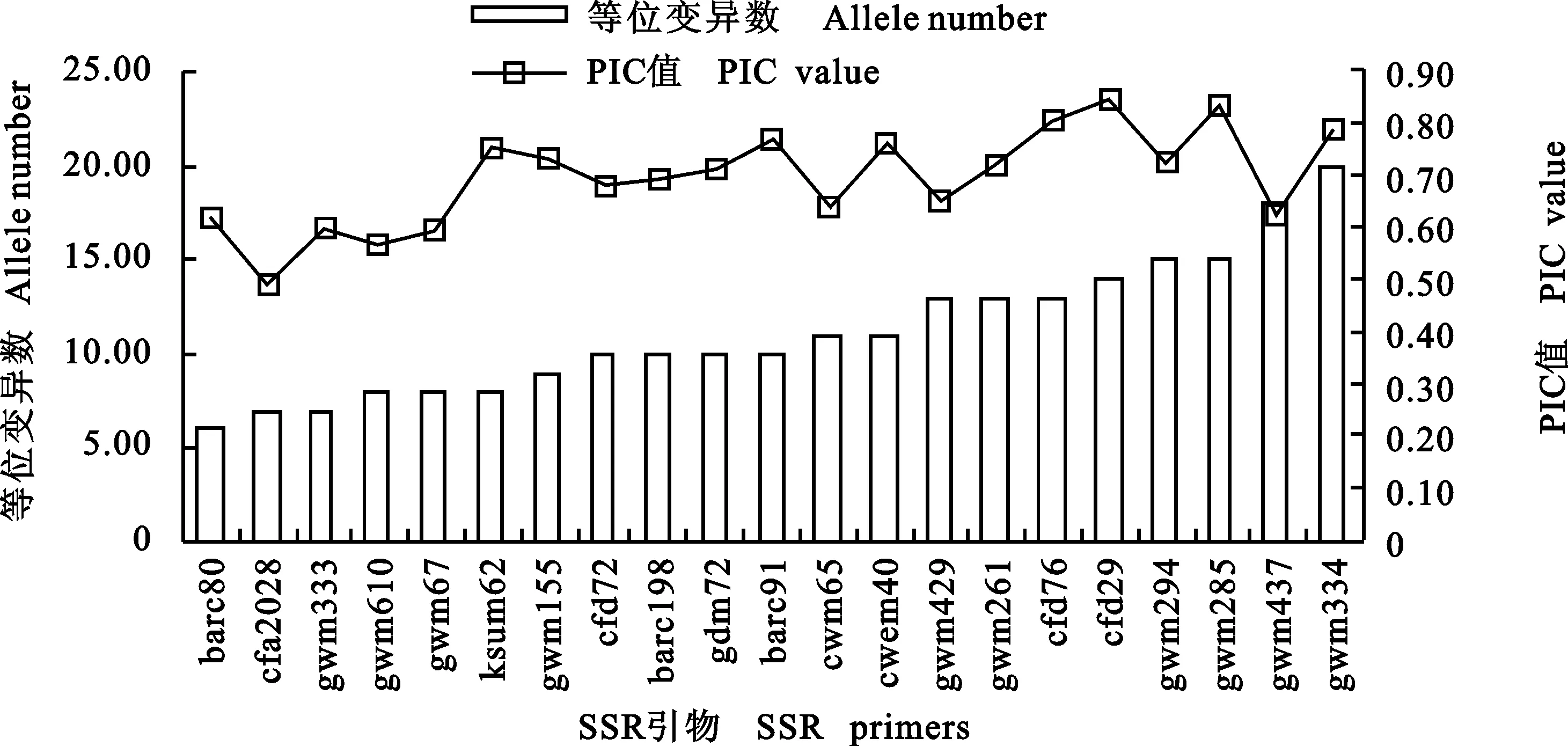
图1 21对SSR引物在430份品系中检测到的等位变异和PIC值
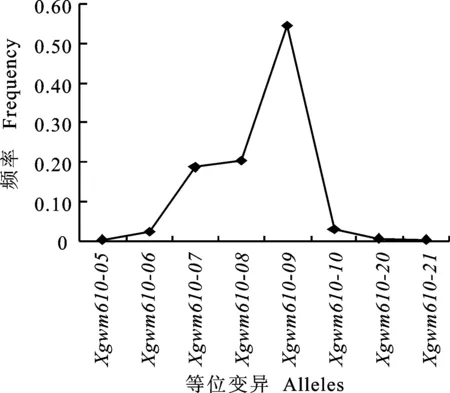
图2 Xgwm610位点检测到的等位变异及其频率
2.2参试品系的遗传多样性
2.2.1不同年份参试品系的遗传多样性
利用21对SSR引物分别对不同年份的区试品系进行遗传多样性分析,结果(表1)显示,2009-2011年品系的平均等位变异数、平均PIC值、平均基因多样性和平均遗传距离均略高于2012-2014年,表明近些年来参加国家小麦区域试验品系的遗传多样性从2012年开始略有下降。
2.2.2不同区试组别间品系的遗传多样性
利用21对SSR引物分别对不同区组试验品系进行了遗传多样性分析,结果(表2)显示,自北向南参试品种的遗传多样性呈明显下降趋势。如北部冬麦区组的参试品系平均基因多样性范围为0.73~0.74,黄淮海冬麦区组参试品系平均基因多样性范围为0.61~0.73,长江流域冬麦区组参试品系平均多样性范围为0.55~0.56。
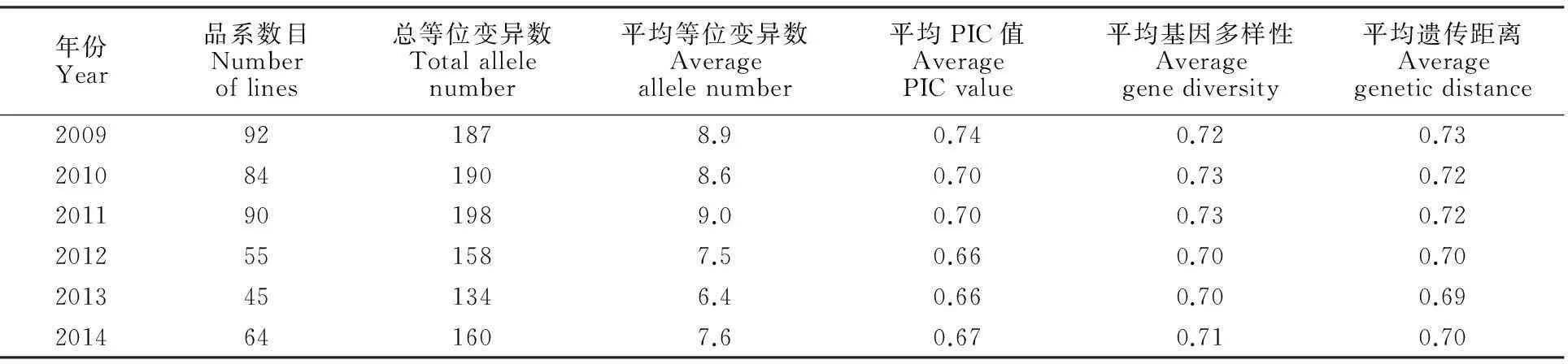
表1 不同年份间区域试验品系的遗传多样性比较
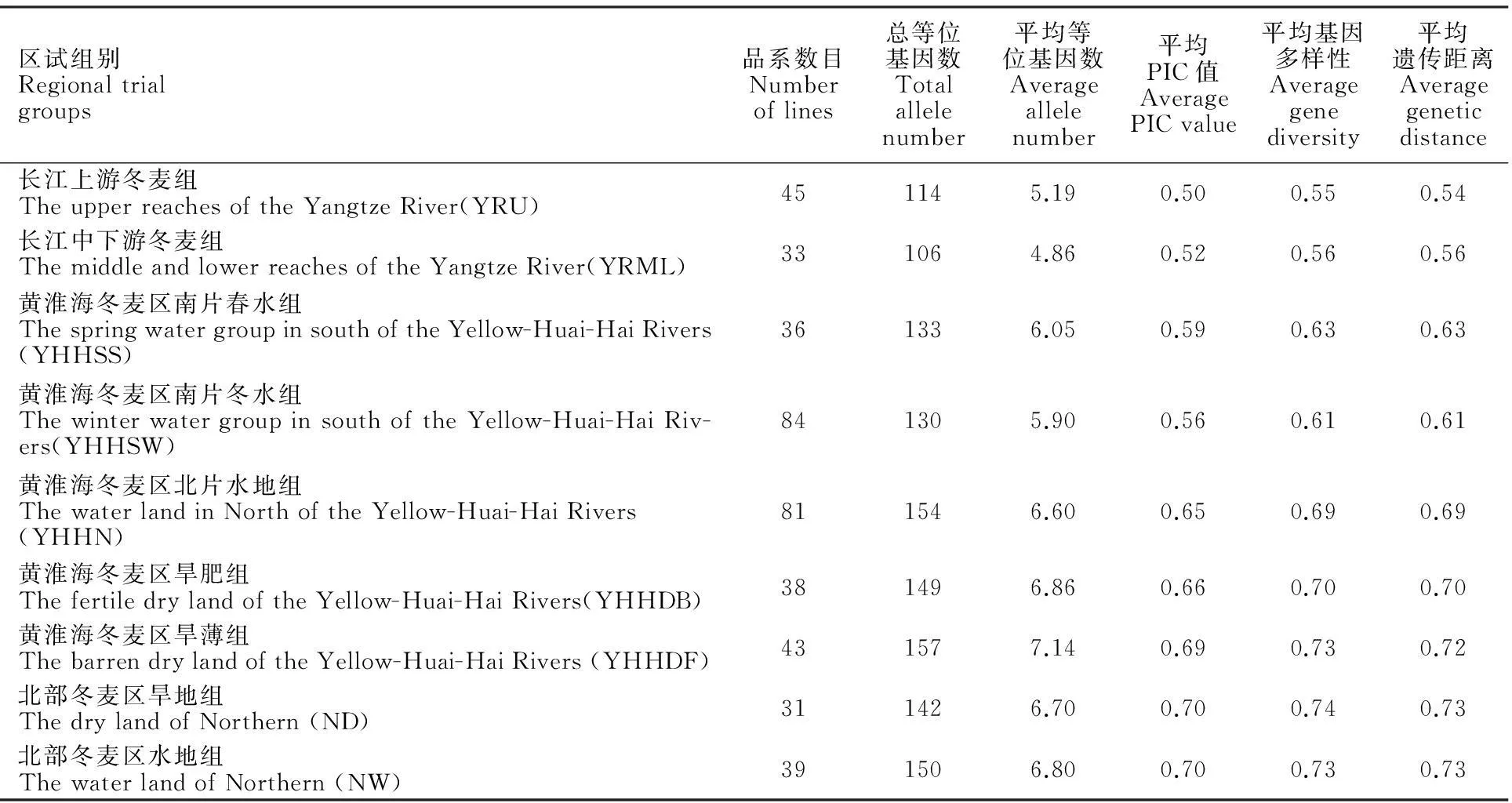
表2 不同区试组别间品系的遗传多样性比较
2.2.3聚类分析
利用21个SSR位点计算了430个国家区域试验品系之间的遗传距离(GD),变化幅度为0~0.9 762 ,平均0.7 300,表明参试品系间存在不同程度的遗传差异,遗传基础比较丰富。利用PowerMarker 3.25软件构建基于UPGMA法的Nei’s遗传距离聚类图,结果(图3)显示,430个品系被分成九大类,离散程度较好,与区组有一定的相关性。A类包括138个品系,占总群体的32.1%,以黄淮海南片春水组和黄淮海南片冬水组品系为主,且两区组品系相互渗透;B类包括64个品系,占总群体的14.9%,以长江上游组和长江中下游组品系为主,且两区组各自独立成群;C类包括18个品系,占总群体的4.2%,以黄淮海旱薄组品系为主;D类包括6个品系,占总群体的1.4%,以旱地组品系为主,其中2个来源于黄淮海旱肥组,2个来源于北部冬麦旱地组,1个来源于黄淮海旱薄组,1个来源于黄淮海南片冬水组;E类包括146个品系,占总群体的34.0%,以黄淮海北片水地组和北部冬麦区水地组为主;F类包括8个品系,占总群体的1.9%,均为旱地组品系,其中4个来源于北部冬麦旱地组,3个来源于黄淮海旱薄组,1个来源于黄淮海旱肥组;G类包括19个品系,占总群体的4.4%,以北部旱地组品系和黄淮海旱薄组品系为主;H类包括20个品系,占总群体的4.7%,以北部水地组和黄淮海旱薄组品系为主;I类包括11个品系,占总群体的2.6%,以北部水地组和北部旱地组品系为主。类群D~I的品系来源分布显示黄淮海北片水地组、黄淮海旱肥组、黄淮海旱薄组、北部冬麦区旱地组和北部冬麦区水地组品系之间相互渗透。
2.2.4主坐标分析
采用多变量统计分析软件MVSP ver. 3.13对9个不同区试组别的参试品系进行主坐标分析,结果(图4)显示,长江上游和长江中下游的大部分参试品系与其他组别的参试品系明显分开;黄淮海南片春水组和黄淮海南片冬水组的大部分材料聚集在一起,且与黄淮海北片水地组品系在图形的位置有部分重叠;黄淮海旱肥组、黄淮海旱薄组、北部旱地组、北部水地组品系与黄淮海北片水地组品系相互渗透,且在图中分布范围较广泛,表明这些区组的品系遗传背景较为宽广,遗传多样性较高。
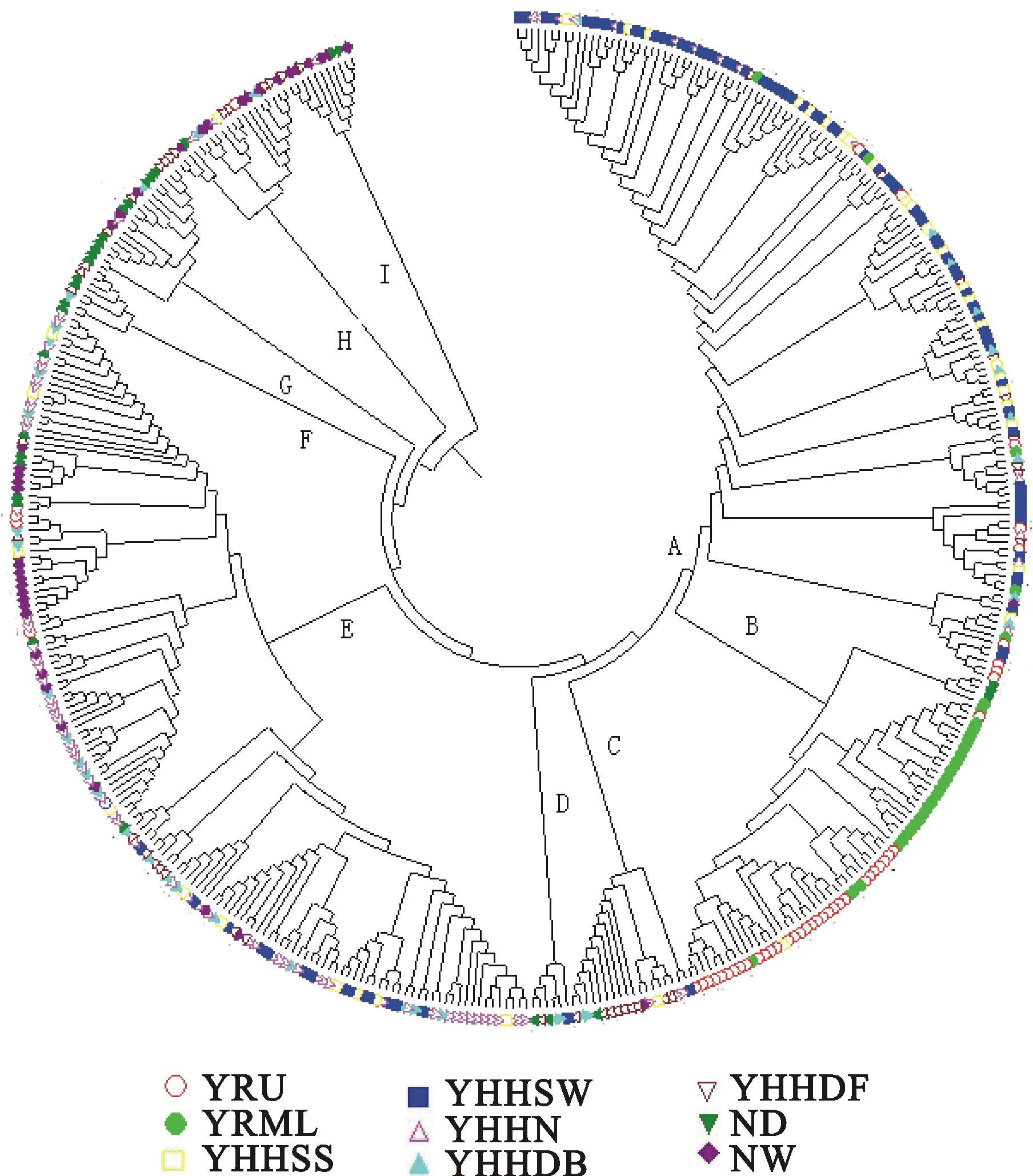
图3 基于UPGMA法的Nei’s遗传距离聚类图
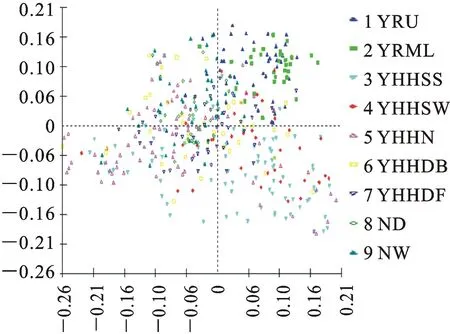
图4 国家区试品系主坐标分析结果
2.3群体结构分析
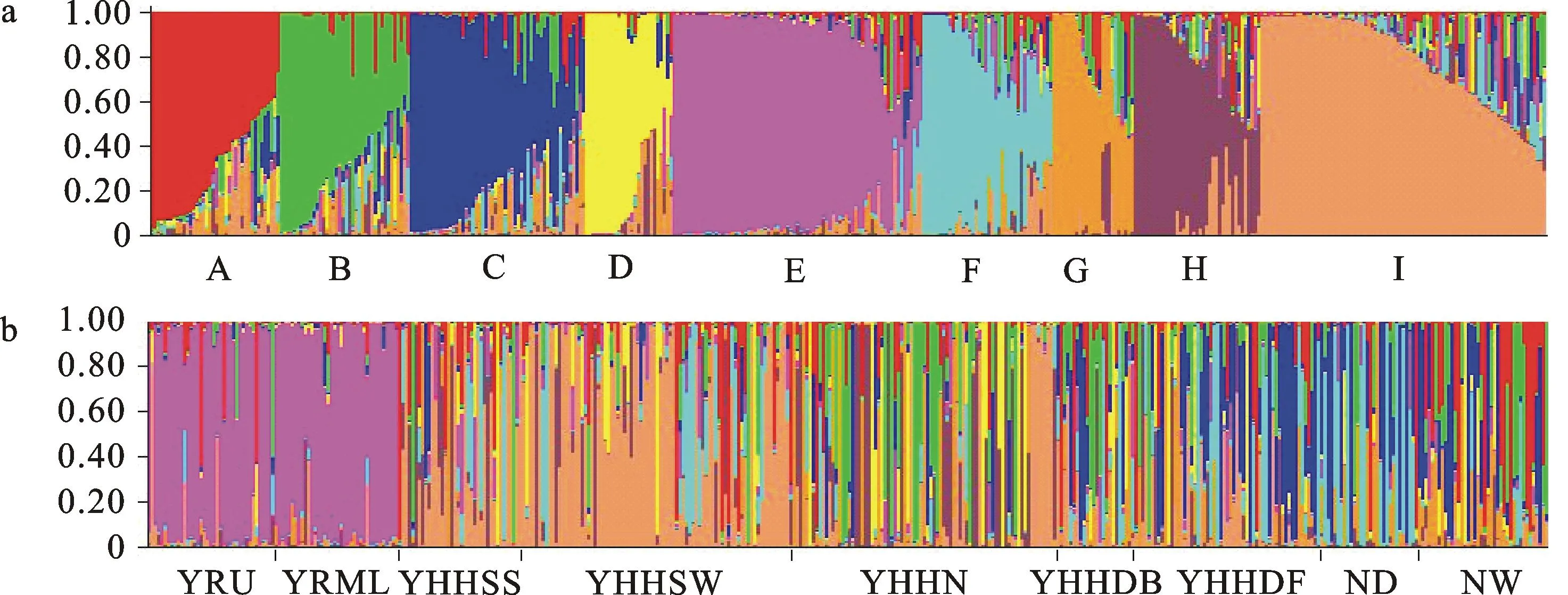
a:基于模型的9个类群; b: 9个类群在9个生态区的分布
a:Nine clusters based on model; b: The distribution of the nine clusters in nine ecological regions
图5430个品系的遗传结构
Fig.5The genetic structure of 430 lines based on model
利用Structure 2.3.4软件对供试材料进行群体结构分析,结果发现,当K=9时,似然值最大,即430个品系的遗传结构可分为9个类群(A~I)(图5a)。计算各品系在不同类群中的Q值,71.9%的品系群体结构相对比较单一,被划分到相应的9个类群中,28.1%的品系群体结构比较复杂。对9个类群在9个生态区的分布进行分析(图5b、表3),长江上游组和长江中下游组的品系主要分布在类群E中,群体结构单一,且与其他7个区组形成了相对独立的遗传结构;黄淮海南片春水组和黄淮海南片冬水组的品系主要分布在类群I中,且前者比后者群体结构复杂;黄淮海北片水地组的品系均匀分布在类群A~D、F~I中,比长江流域组的品系群体结构复杂;黄淮海旱肥组、黄淮海旱薄组、北部旱地组和北部水地组的品系主要分布在除类群E和I以外的7个类群中,群体结构复杂,趋向多元化。

表3 不同区试组别中群体结构单一品系的比例
3讨 论
3.1关于参试品系的SSR标记多态性问题
本研究所用的21对SSR标记,是北京杂交小麦工程技术研究中心在对审定小麦品种、国家及省市区域试验参试材料和小麦种质材料进行鉴定的基础上,经10余年实践基础上筛选出来的,是绘制中国小麦品种指纹图谱以及品种鉴定的核心SSR标记,不仅扩增稳定,重复性好,而且分辨率较高[17]。本研究结果显示,21对SSR引物的区分能力为95%,未区分出的22对品系中,指纹相同的品系来自于同一育种单位,甚至来源于相同的亲本。由此可见,21对SSR标记的多态性非常高,具有较高的代表性,可较好地用于小麦品种遗传多样性和群体结构分析。此外,研究结果还显示,位点 Xgwm610-09等位变异出现的频率很高,这可能与多数育种单位在品种选育过程中集中使用某些优良亲本有关,也可能该等位变异与某些重要优良农艺性状紧密连锁,在品种选育过程中,被育种者保留下来,具体原因还有待于进一步研究和验证。
3.2关于参试品系的遗传多样性问题
近6年来国家冬小麦区试品系的遗传多样性分析结果显示,参加国家小麦区域试验品系的遗传多样性从2012年开始有下降趋势。究其原因可能受参试品系数量的影响,也可能与多数育种单位频繁集中使用部分骨干亲本的育种模式有关。不同种植区域的国家冬小麦区试品系的遗传多样性分析结果显示,自北向南国家区域试验参试品系的遗传多样性呈逐渐降低的趋势,说明我国长江流域的小麦育种相对比较薄弱,北部冬麦区的小麦育种力量很强,这一结论与郝晨阳等[12]的研究结果相同。长江流域品系遗传多样性低、品系间亲缘关系近的原因可能与集中利用优良亲本以及骨干种质有关,在今后育种过程中应注意加强拓宽小麦亲本的遗传背景,丰富小麦育种资源,从而提高我国长江流域小麦品系的遗传多样性。此外,随着时代的发展,为了提升育种水平,培育适应性更广、产量潜力更高及抗逆性更强的小麦新品种,在今后的小麦育种过程中,应加大远缘种质、地方品种、外引品种等小麦种质的引入,提高我国冬小麦品种的遗传多样性,拓宽群体的遗传背景,增加不同地区间品系的基因交流,使群体结构复杂化,提高小麦品种的抗病性、抗逆性和广适性,提升小麦的产量水平。
对430个国家区域试验品系的UPGMA法聚类、二元主成分和群体结构分析的结果基本吻合,长江上游和长江中下游组的参试品系与其他区组的参试品系明显分开,独自聚成一类,且又分别归属于不同亚类。2个区组品系独自聚成一类是由于该区域的品系一般为春性品系,与其他区组品系之间遗传关系远,基因交流甚少;2个区组分别归属不同的亚类,与2个区组所涉及地理区域和生态区域差异性、适宜品种类型差异性及主要育种资源和模式差异性相关,进一步说明国家区域试验中将长江流域划分成长江上游组和长江中下游组从分子水平看也比较科学。黄淮海南片春水组和黄淮海南片冬水组的大部分材料相互渗透,前者属于弱春性品系区,后者属于半冬性品系区,能够划分在一起说明两区组间基因交流频繁。这2个区组的品系又与黄淮海北片水地组的品系有部分重叠,黄淮海北片水地组的品系一般为冬性品系,说明黄淮海南片和北片有互相引种的可能,基因交流比较频繁;黄淮海旱肥组、黄淮海旱薄组、北部旱地组和北部水地组的品系与长江流域、黄淮海南片组的品系未聚在一起,这些区域的品系一般为冬性品系,与长江流域、黄淮海南片组的种质交流甚少。总之,国家区试品系显示出了一定程度的区域特点,从分子水平验证了国家区域试验按照9个生态区划分的可行性。
3.3关于参试品系的群体结构问题
群体结构分析与基于遗传距离的聚类法、主坐标分析法相比,具有显示材料内在遗传结构的优势。本研究采用了基于模型的聚类方法来分析群体结构,并计算了品系的Q值,结果显示,430个品系中,71.9%的品系群体结构比较单一,其中长江上游组和长江中下游组的大部分品系群体结构最为单一,说明我国区域试验大部分品系的育种模式比较简单、育种亲本遗传基础狭窄,外引种质或各区组间相互引种偏少。今后应要加强国内外种质的交流,增加优异基因渗透,加强通过各种育种手段来改良小麦区域试验品系的遗传结构,拓宽遗传基础。
参考文献:
[1]刘志斋,吴 迅,刘海利,等.基于40个核心SSR 标记揭示的820份中国玉米重要自交系的遗传多样性与群体结构 [J].中国农业科学,2012,45(11):2107-2138.
Liu Z Z,Wu X,Liu H L,etal.Genetic diversity and population structure of important Chinese maize inbred lines revealed by 40 core simple sequence repeats(SSRs) [J].ScientiaAgriculturaSinica,2012,45(11):2107-2138.
[2]王凤格,田红丽,赵久然,等.中国328个玉米品种(组合)SSR标记遗传多样性分析 [J].中国农业科学,2014,47(5):856-864.
Wang F G,Tian H L,Zhao J R,etal.Genetic diversity analysis of 328 maize varieties(hybridized combinations) using SSR markers [J].ScientiaAgriculturaSinica,2014,47(5):856-864 .
[3]van Inghelandt D,Melchinger A E,Lebreton C,etal.Population structure and genetic diversity in a commercial maize breeding program assessed with SSR and SNP markers [J].TheoreticalandAppliedGenetics,2010,120:1289-1299.
[4]齐永文,张冬玲,张洪亮,等.中国水稻选育品种遗传多样性及其近50年变化趋势 [J].科学通报,2006,51(6):681-688.
Qi Y W,Zhang D L,Zhang H L,etal.Genetic diversity of rice cultivars(OryzaSativaL.) in China and the temporal trends in recent fifty years [J].ChineseScienceBulletin,2006,51(6): 681-688.
[5]Courtois B,Frouin J,Greco R,etal.Genetic diversity and population structure in a European collection of rice [J].CropScience,2012,52:1663-1675.
[6]Singh N,Choudhury D R,Singh A K,etal.Comparison of SSR and SNP markers in estimation of genetic diversity and population structure of Indian rice varieties [J].PloSOne,2013,8(12):e84136.
[7]张 军,赵团结,盖钧镒.中国东北大豆育成品种遗传多样性和群体遗传结构分析 [J].作物学报,2008,34(9):1529-1536.
Zhang J,Zhao T J,Gai J Y.Genetic diversity and genetic structure of soybean cultivar population released in northeast China [J].ActaAgronomicaSinica,2008,34(9):1529-1536.
[8]文自翔,赵团结,丁艳来,等.中国栽培及野生大豆的遗传多样性、地理分化和演化关系研究 [J].科学通报,2009,54(23):4393-4403.
Wen Z X,Zhao T J,Ding Y L,etal.Genetic diversity,geographic differentiation and evolutionary relationship among ecotypes ofGlycinemaxandG.sojain China [J].ChineseScienceBulletin,2009,54(23):4393-4403.
[9]匡 猛,杨伟华,许红霞,等.中国棉花主栽品种DNA 指纹图谱构建及SSR标记遗传多样性分析 [J].中国农业科学,2011,44(1):20-27.
Kuang M,Yang W H,Xu H X,etal.Construction of DNA fingerprinting and analysis of genetic diversity with SSR markers for cotton major cultivars in China [J].ScientiaAgriculturaSinica,2011,44(1):20-27.
[10]段艳凤,刘 杰,卞春松,等.中国88 个马铃薯审定品种SSR指纹图谱构建与遗传多样性分析 [J].作物学报,2009,35(8):1451-1457.
Duan Y F,Liu J,Bian C S,etal.Construction of fingerprinting and analysis of genetic diversity with SSR markers for eighty-eight approved potato cultivars(SolanumtuberosumL.) in China [J].ActaAgronomicaSinica,2009,35(8):1451-1457.
[11]Wang H Y,Wang X,Chen P D,etal.Assessment of genetic diversity of Yunnan,Tibetan,and Xinjiang wheat using SSR markers [J].JournalofGeneticsandGenomics,2007,34:623-633.
[12]郝晨阳,董玉琛,王兰芬,等.我国普通小麦核心种质的构建及遗传多样性分析 [J].科学通报,2008,53(10):908-915.
Hao C Y,Dong Y C,Wang L F,etal.Genetic diversity and construction of core collection in Chinese wheat genetic resources [J].ChineseScienceBulletin,2008,53(10):908-915.
[13]曹廷杰,谢菁忠,吴秋红,等.河南省近年审定小麦品种基于系谱和SNP标记的遗传多样性分析 [J].作物学报,2015,41(2):197-206.
Cao T J,Xie J Z,Wu Q H,etal.Genetic diversity of registered wheat varieties in Henan province based on pedigree and single-nucleotide polymorphism [J].ActaAgronomicaSinica,2015,41(2):197-206.
[14]Wurschum T,Langer S M,Longin C F,etal.Population structure,genetic diversity and linkage disequilibrium in elite winter wheat assessed with SNP and SSR markers [J].TheoreticalandAppliedGenetics,2013,126:1477-1486.
[15]王升星,朱玉磊,张海萍,等. 小麦育种亲本材料SSR标记遗传多样性及其亲缘关系分析 [J].麦类作物学报,2014,34(5):621-627.
Wang S X,Zhu Y L,Zhang H P,etal.Analysis of genetic diversity and relationship among wheat breeding parents by SSR marker [J].JournalofTriticeaeCrops,2014,34(5):621-627.
[16]季 伟,王立新,孙 辉,等.小麦SSR分析体系的简化 [J].农业生物技术学报,2007,15(5):907-908.
Ji W,Wang L X,Sun H,etal.Predigestion of wheat SSR analysis protocol [J].JournalofAgriculturalBiotechnology,2007,15(5):907-908.
[17]王立新,常利芳,李宏博,等.小麦区试品系DUS测试的分子标记 [J].作物学报,2010,36(7):1114-1125.
Wang L X,Chang L F,Li H B,etal.Molecular markers for estimating distinctness,uniformity,and stability of wheat lines in regional trials [J].ActaAgronomicaSinica,2010,36(7):1114-1125.
[18]王立新,赵昌平,邱 军,等. 记录小麦SSR带型的快捷方法 [J].麦类作物学报,2006,26(4):164-168.
Wang L X,Zhao C P,Qiu J,etal.A new scoring method of SSR patterns for wheat [J].JournalofTriticeaeCrops,2006,26(4):164-168.
[19]Liu K,Muse S V.PowerMarker: An integrated analysis environment for genetic marker analysis [J].Bioinformatics,2005,21(9):2128-2129.
[20]Pritchard J K,Stephens M,Donnelly P.Inference of population structure using multilocus genotype data [J].Genetics,2000,155(2):945-959.
Genetic Diversity and Population Structure Analysis of Winter Wheat Lines from Recent National Regional Trials in China
LIU Lihua1,PANG Binshuang1,LIU Yangna1,QIU Jun2,LI Hongbo1,ZHANG Xin1,WANG Na1,ZHAO Changping1
(1.Beijing Engineering and Technique Research Center for Hybrid Wheat,Beijing Academy of Agriculture and Forestry Sciences,Beijing Key Laboratory of Molecular Genetics in Hybrid Wheat,Beijing 100097,China; 2.National Agriculture Technology Extension Center,Agriculture Ministry of China,Beijing 100026,China)
Abstract:To understand the wheat breeding level,development trend and existing problems,twenty-one core SSRs covering the entire wheat genome were used to analyze the genetic diversity and population structure of 430 winter wheat lines from national regional trials during 2009-2014 in China. 236 allelic variations were detected with an average allele number of 11.24. The average gene diversity values and PIC(Polymorphism information content) were determined as 0.73 and 0.70,respectively. Based on the analysis on different breeding years,genetic diversity decreased slightly since 2012. Based on the analysis from different appropriate growing regions,genetic diversity decreased gradually from north to south. UPGMA clustering,principal coordinate analysis(PCoA) and population structure analysis showed that the testing lines from the upper reaches of Yangtze river,the middle and lower reaches of the Yangtze river were obviously distinguished from other varieties of district group,and respectively belonged to different subpopulation,showing a certain degree of regional characteristic. In addition,population structure analysis also revealed that the population structure of 71.9% of the varieties was relatively simple,and that of varieties at the Yangtze river region was most simple.
Key words:Wheat; Regional trials; SSR marker; Genetic diversity; Population structure
中图分类号:S512.1;S330
文献标识码:A
文章编号:1009-1041(2016)02-0165-07
通讯作者:赵昌平(E-mail: cp_zhao@vip.sohu.com)
基金项目:北京市农林科学院科技创新能力建设专项(KJCX20140202; KJCX20140421);北京市农林科学院青年基金项目(QNJJ201509)
收稿日期:2015-10-26修回日期:2015-11-20
网络出版时间:2016-01-26
网络出版地址:http://www.cnki.net/kcms/detail/61.1359.S.20160126.1944.012.html
第一作者E-mail: llh216@.163.com(刘丽华);pangbinshuang1122@aliyun.com(庞斌双,与第一作者同等贡献)
