基础医学模式生物酿酒酵母接合型的鉴定—PCR法
2016-05-18刘超韩雅婷杨冰孙琰王玺
刘超,韩雅婷,杨冰,孙琰,王玺
(天津医科大学基础医学院细胞生物学系,天津300070)
论著
基础医学模式生物酿酒酵母接合型的鉴定—PCR法
刘超,韩雅婷,杨冰,孙琰,王玺
(天津医科大学基础医学院细胞生物学系,天津300070)
目的:利用PCR法鉴定酿酒酵母a、α单倍体或a/α双倍体三种接合型。方法:根据酿酒酵母a、α或a/α三种接合型MAT基因座及附近序列信息,分别设计特异的寡核苷酸引物。提取酿酒酵母DNA作为模版和利用单菌落PCR法,进行特异性PCR扩增。结果:MATa基因座特异性引物能在a和a/α接合型中扩增出特异性条带;MATα基因座特异性引物则能在α和a/α接合型中扩增出特异性条带。结论:利用PCR鉴定酿酒酵母接合型是一种操作简单、准确灵敏的方法,为基础医学单细胞真核模式生物的研究提供方便。
酿酒酵母;接合型;MAT基因座;PCR鉴定;单菌落PCR
在基础医学研究中,酿酒酵母(Saccharomyces cerevisiae)是一种低等的单细胞真核生物,具有易培养、生长速度快、遗传背景清楚、基因工程操作简便等特点,广泛用于真核细胞的研究,包括细胞代谢、细胞周期、染色体结构与功能、DNA复制与修复、基因表达与信号通路调控、自噬等生物过程以及线粒体相关疾病、抗癌药物的研发和筛选等[1-5]。因此,酿酒酵母是基础医学中重要的研究工具。酿酒酵母具有单倍体a或α、二倍体a/α三种接合型,由位于III号染色体长臂上的MAT基因座决定,不同接合型的酿酒酵母具有性别特异的基因表达。传统检测酵母接合型的方法是通过观察其是否能与已知接合型的测试菌株杂交。但是这种方法操作繁琐,耗时要至少1 d,结果准确率低,易受细胞状态、环境因素和遗传背景影响[6]。MAT基因座位于III号染色体长臂上,距中心粒约100 kb,分为5个区域(W,X,Y,Z1和Z2),Ya和Yα分别包含有性别特异基因的启动子和开放读码框[7-8]。本研究计划根据基因组中性别决定的MAT基因座及附近序列信息分别设计特异性引物,通过PCR法鉴定菌株的接合型。
1 材料与方法
1.1 材料与仪器 酿酒酵母菌种YPH499(MATa)、YPH500(MATα)和YPH501(MATa/α)(天津医科大学发育与肿瘤发生表观遗传学实验室),含2%葡萄糖YPD培养基,Taq DNA聚合酶、dNTP(美国Thermo Fisher Scientific公司),DNA HyperLadder I(美国Gentuar公司)。
311系列 CO2培养箱(美国 Thermo Fisher Scientific公司),ZWY-240全温型多振幅轨道摇床(上海智城公司),S1000梯度PCR仪(美国Bio-Rad公司),D30核酸蛋白测定仪、Centrifuge 5427R冷冻高速离心机(德国Eppendorf公司),SynGene Gene Genius全自动凝胶成像系统(美国SynGene公司),EPS 200电泳仪(上海天能公司)。
1.2 方法
1.2.1 引物的设计与合成 根据GenBank中酿酒酵母性别决定的MATa和MATα基因座及附近基因序列,设计并委托生物工程(上海)有限公司合成引物(表1)。
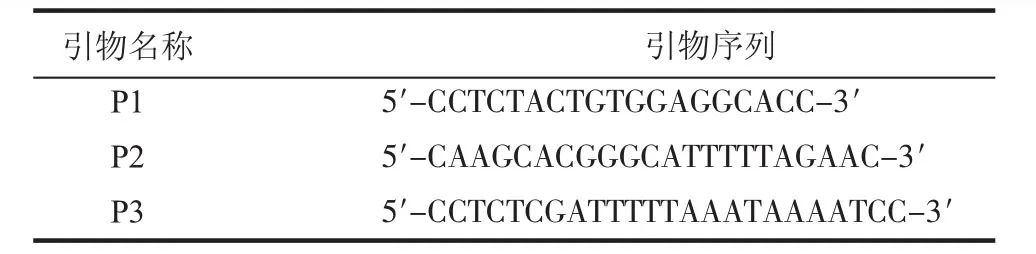
表1 PCR引物Tab 1 PCR primers
1.2.2 酵母基因组的提取 提取酵母基因组DNA方法参照文献[9]。接种酵母到2.5 mL酵母试管培养基中(YPD培养基),30℃培养16~18 h(OD600=0.4);取200μL酵母培养液,室温下,1500×g离心5~10min收集菌体;菌体用灭菌水洗1次,1500×g离心5min。用100 μL CH3COOLi(0.2 mol/L醋酸锂,1%SDS)重悬细胞,70℃孵育5 min,加入300 μL无水乙醇,充分混匀,15 000×g离心3 min,将沉淀用70%的酒精洗涤1次后风干。风干后的DNA溶于100 μL H2O或TE缓冲液中,15 000×g离心15 s去除细胞碎片,转移上清到新的1.5 mL离心管中,-20℃保存。
1.2.3 单菌落PCR模板制备 取直径为1 mm的单菌落,用5 μL水悬浮,95℃处理5 min[10]。
1.2.4 PCR扩增 反应体系为25 μL,包括:10× PCR缓冲液(含Mg2+)2.5 μL,提取的酵母基因组或单菌落PCR模板DNA 1 μL,Taq DNA聚合酶0.25 μL,dNTP(2.5 mmol/L)2 μL,上下游引物(2.5 μmol/L)各2.5 μL,其余用ddH2O补足。
PCR扩增条件:95℃预变性5 min,95℃变性45 s,55℃复性45 s,72℃延伸1.5 min,21个循环,最后72℃延伸10 min;4℃保存。PCR扩增产物在含溴化乙锭的2%琼脂糖凝胶中进行电泳,在凝胶成像系统中进行观察,并照相保存。
2 结果
位于III号染色体长臂上的MAT基因座,长约2 500 bp,其中Ya长约700 bp,Yα长约850 bp,含有各自性别特异的基因序列。III号染色体上还具有另外两个具有Y区的基因座,分别为位于短臂上的HMLα基因座和长臂上MAT基因座与端粒之间的HMRa基因座,这两个基因座通常是沉默的,在接合型突变中作为基因重组的供体(图1)。根据MAT基因座Ya和Yα的序列差异分别设计接合型特异的引物,P1为MAT基因座下游公共的下游引物,P2和P3分别为Ya和Yα序列特异的上游引物(图1)。引物序列见表1。
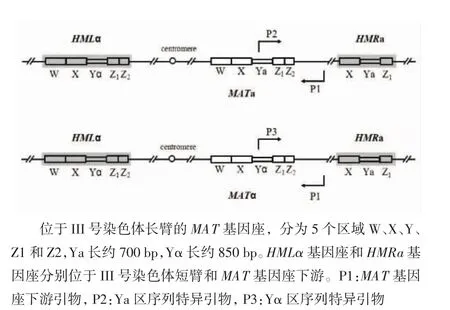
图1 III号染色体上的MAT基因座Fig 1 The MAT locus in chromosome III
从电泳图谱可以看出,常规PCR和单菌落PCR中,阴性对照无条带,P1和P2作上下游引物时能够在a接合型和a/α接合型酵母中扩增出约864 bp的条带,而在α接合型酵母中则无条带(图2);P1和P3作上下游引物时能够在α接合型和a/α接合型酵母中扩增出约864 bp的条带,而在a接合型酵母中则无条带(图3)。两组引物扩增出的条带均与期望的条带大小一致。
单菌落PCR和提取基因组进行常规PCR的结果相似,都获得了大小一致且清晰的条带,证明设计的引物扩增效率高、特异性好。由于单菌落PCR的方法成本低、操作快速简便,实用性更强。
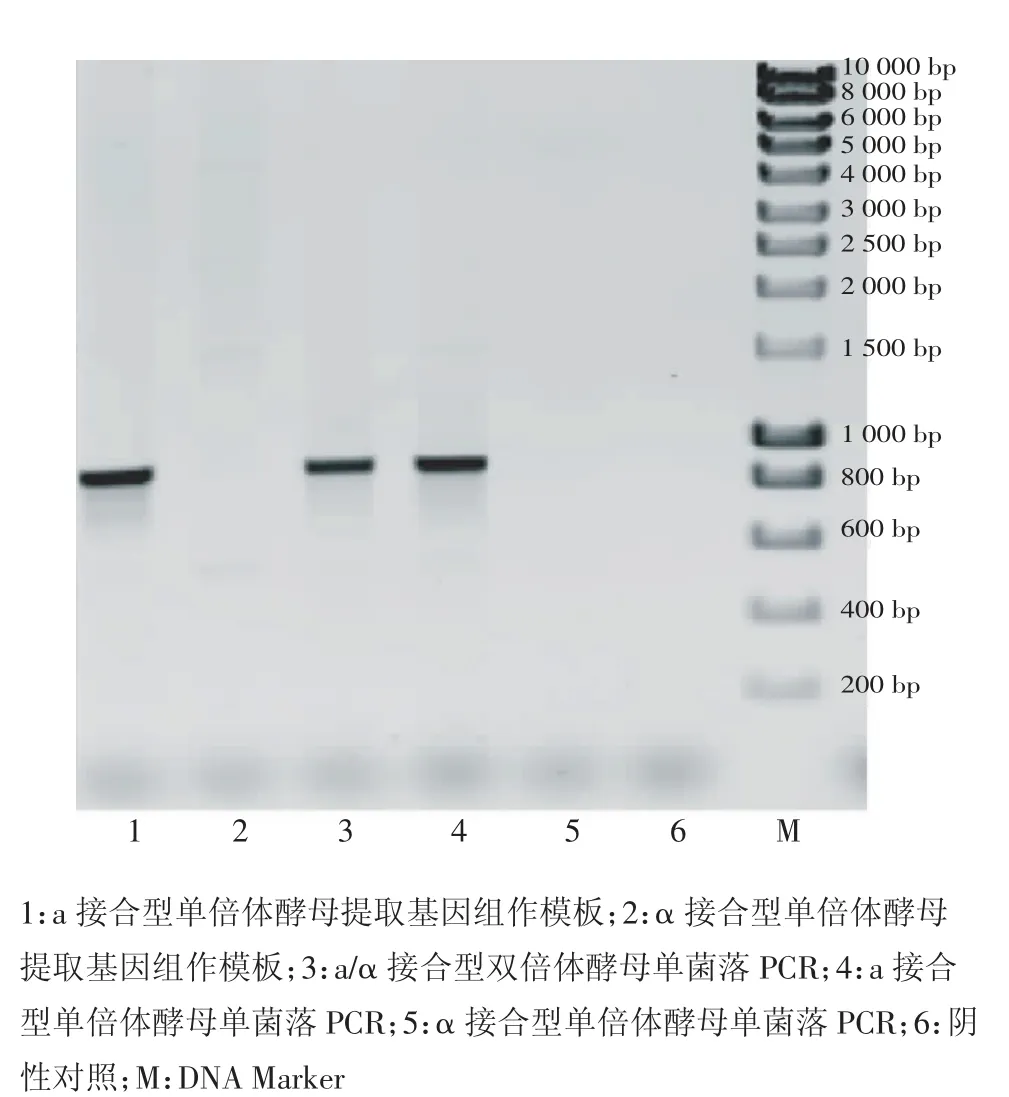
图2 P1和P2作引物PCR检测MATaFig 2 Identification of MATa by PCR with P1 and P2
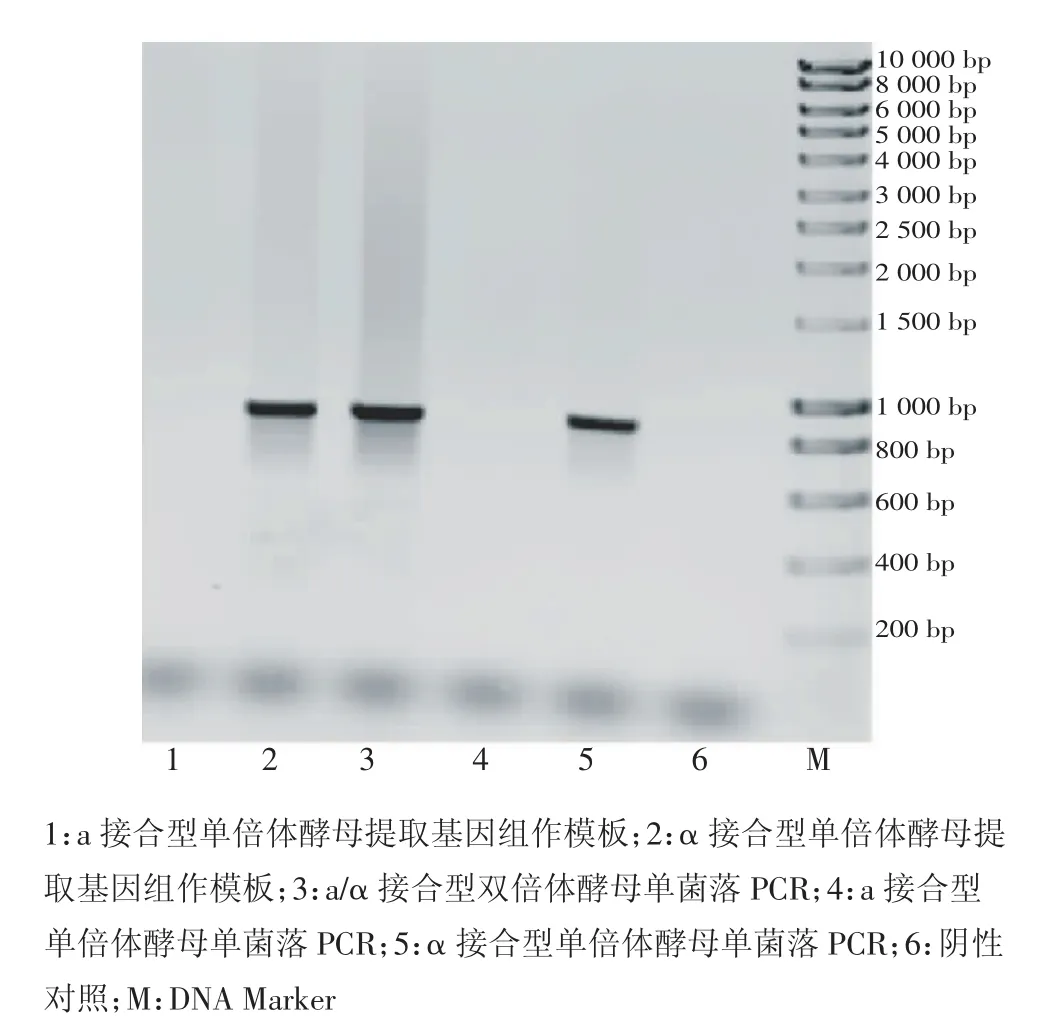
图3 P1和P3作引物PCR检测MATαFig 3 Identification of MATα by PCR with P1 and P3
3 讨论
酿酒酵母是主要的生物乙醇和酿酒业发酵菌株,而且作为研究真核细胞的模式生物,在阐明许多生物过程和机制中发挥了重要作用,对于细胞自噬作用分子机制、基因表达调控机制、染色质结构、基因重组机制、衰老与凋亡机制等的了解,几乎都来源于对酵母的研究。例如,对于酿酒酵母接合型转换的研究,从这一高度精细调控的生物过程中,了解了基因表达调控、染色体结构、同源重组机制的许多方面,如Sir蛋白家族通过严格调控HO基因的表达,并以HMLα或HMRa作为同源重组的供体,调节接合型之间的转换[11]。在利用酵母进行研究的过程中,经常需要测定菌株接合型。传统测定酵母接合型的方法主要是通过观察待测菌株是否能与已知接合型的单一营养缺陷型测试菌株杂交。例如以MATa(thr4-)或MATα(thr4-)作为测试菌株,而待测菌株通常为野生型,可能具有多种营养缺陷但不包含thr4-,这样测试菌株和待测菌株都不能单独在基本培养基上生长,只有发生异性接合相互弥补各自的营养缺陷所形成的二倍体菌体,才能在基本培养基上生长,由此实现待测菌株接合型的鉴定。但是这种方法操作过程繁琐,耗时长,结果易受多种因素影响,出现假阳性或假阴性结果。例如细胞状态、培养板和温度等环境因素可能会影响杂交的效率,菌株间的遗传背景可能会发生营养缺陷并不互补情况等[6]。而且有时为了获得合适的酵母菌株,往往需要筛选数十个甚至上百个重组子,使得鉴定酿酒酵母接合型的工作量增加、实验成本升高。
本研究利用PCR方法,根据酵母单倍体a、α或二倍体a/α三种接合型中III号染色体性别决定的MATa和MATα基因座及其附近序列信息,分别设计特异性寡核苷酸引物,进行PCR扩增。MATa的特异性引物能在a和a/α接合型中扩增出864 bp特异性条带;MATα的特异性引物则能在α和a/α接合型中扩增出864 bp特异性条带。通过使用不同引物进行PCR并观察结果的条带位置,一般不超过6 h,便能鉴定出酿酒酵母的接合型。本方法的特点是:首先,本研究是根据酿酒酵母基因组序列设计并合成的寡核苷酸引物,具有序列特异性;第二,这是一种新建的利用PCR鉴定酿酒酵母结合型的方法,与传统方法相比,具有耗时少,结果准确,不受细胞状态、遗传背景、环境因素等影响的优点。第三,本研究采用的单菌落PCR法,其实验结果与提取酵母基因组作为模板的常规PCR法的实验结果相同,也有效地扩增出了目的条带。但是单菌落PCR省去了提取基因组步骤,不仅实验成本低,而且进一步节省了约18 h的实验时间,具有更加简单、经济、高效的特点[12-13]。
综上所述,本文提供利用设计的特异性寡核苷酸引物新建的PCR法能够直接快速地鉴定酿酒酵母的接合型,在酿酒酵母研究中具有较大的应用前景。这种设计并利用特异性寡核苷酸引物进行PCR用于鉴定单细胞真核模式生物性别的方法为基础医学研究提供了方便。
[1] Wang R,Mozziconacci J,Bancaud A,et al.Principles of chromatin organization in yeast:relevance of polymer models to describe nuclear organization and dynamics[J].Curr Opin Cell Biol,2015,34: 54
[2] Ren J,Wang C L,Sternglanz R.Promoter strength influences the S phase requirement for establishment of silencing at the Saccharomyces cerevisiae silent mating type Loci[J].Genetics,2010, 186(2):551
[3] Feng Y,He D,Yao Z,et al.The machinery of macroautophagy[J]. Cell Res,2014,24(1):24
[4] Lasserre J P,Dautant A,Aiyar R S,et al.Yeast as a system for modeling mitochondrialdisease mechanismsand discovering therapies[J].Dis Model Mech,2015,8(6):509
[5] Matuo R,Sousa F G,Soares D G,et al.Saccharomyces cerevisiae as a model system to study the response to anticancer agents[J]. Cancer Chemother Pharmacol,2012,70(4):491
[6]Sherman F.Getting started with yeast[J].Methods Enzymol,1991, 194:3
[7] Astell C R,Ahlstrom-Jonasson L,Smith M,et al.The sequence ofthe DNAs coding for the mating-type loci of Saccharomyces cerevisiae[J].Cell,1981,27(1 Pt 2):15
[8] Haber J E.Mating-type genes and MAT switching in Saccharomyces cerevisiae[J].Genetics,2012,191(1):33
[9] Löoke M,Kristjuhan K,Kristjuhan A.Extraction of genomic DNA from yeasts for PCR-based applications[J].Biotechniques,2011,50 (5):325
[10]陈忠翔,房志家,陈婷,等.一种简单高效的酵母单菌落PCR方法[J].生物技术通讯,2013(2):225
[11]Li J,Coïc E,Lee K,et al.Regulation of budding yeast matingtype switching donor preference by the FHA domain of Fkh1[J]. PLoS Genet,2012,8(4):e1002630
[12]Güssow D,Clackson T.Direct clone characterization from plaques and colonies by the polymerase chain reaction[J].Nucleic Acids Res,1989,17(10):4000
[13]徐丽,蔡俊鹏.菌落PCR方法的建立及其与常规PCR方法的比较[J].华南理工大学学报:自然科学版,2004,32(5):51
(2015-10-27收稿)
Identification of the mating-type of model organism S.cerevisiae in basic medical science by PCR
LIU Chao,HAN Ya-ting,YANG Bing,SUN Yan,WANG Xi
(Department of Cell Biology,School of Basic Medical Sciences,Tianjin Medical University,Tianjin 300070,China)
Objective:To identify S.cerevisiae mating-type to be a,α haploid or a/α diploid by PCR.Methods:Specific PCR primers were designed according to the MAT locus and its nearby sequence of the three mating-types,and applied in specific PCR reaction using genome DNA as template or the method of colony PCR.Results:A specific band was obtained in a or a/α mating-type when specific primers were used on MATa,while a specific band was obtained in α or a/α mating-type by using specific primers on MATα.Conclusion:With specific primers,PCR may be a convenient,fast and precise method to identify S.cerevisiae mating-type,which could facilitate the study of monocellular eukaryotes model organism in basic medical science.
S.cerevisiae;mating-type;MAT locus;PCR identification;colony PCR
Q2
A
1006-8147(2016)03-0256-03
刘超(1990-),男,硕士在读,研究方向:肿瘤表观遗传学;通信作者:王玺,E-mail:wangxi@tijmu.edu.cn。
