头颈部肿瘤PET图像分割随机游走方法*
2016-04-25刘国才胡泽田朱苏雨张九堂
刘国才,胡泽田,朱苏雨,袁 媛,刘 科,吴 峥,张九堂,莫 逸
(1. 湖南大学 电气与信息工程学院,湖南 长沙 410082;2. 中南大学湘雅医学院 附属湖南省肿瘤医院放疗,科放疗中心,PET/CT中心,湖南 长沙 410013)
头颈部肿瘤PET图像分割随机游走方法*
刘国才1†,胡泽田1,朱苏雨2,袁媛2,刘科2,吴峥2,张九堂2,莫逸2
(1. 湖南大学 电气与信息工程学院,湖南 长沙410082;2. 中南大学湘雅医学院 附属湖南省肿瘤医院放疗,科放疗中心,PET/CT中心,湖南 长沙410013)
摘要:针对肿瘤放疗生物靶区高精度勾画难题,根据头颈部肿瘤PET(positron emission computed tomography)影像特点,提出了肿瘤PET图像分割随机游走方法.首先,根据PET SUV(standardized uptake value)影像,采用三维自适应区域生长和数学形态学膨胀方法确定随机游走方法的种子点,将包含肿瘤的感兴趣区域分为核心肿瘤区域(标记为前景种子点)、正常组织区域(标记为背景种子点)和待定区域.然后,利用头颈部肿瘤和周围正常组织PET图像具有不同的对比度纹理特征,将PET SUV及其对比度纹理值作为随机游走方法中边的权值计算依据.实验结果表明,该法不仅比传统随机游走方法平均提速9.34倍,而且,以临床医生手工勾画的大体肿瘤区作为参考标准,相似度平均提高32.5%(P<0.05).本文方法能够有效地自动勾画头颈部肿瘤放疗生物靶区.
关键词:医学图像分割;随机游走;区域生长;生物靶区;头颈癌
18F-脱氧葡萄糖(18FDG)正电子发射断层扫描(positron emission computed tomography, PET)是一种在分子水平上非侵入性的生物学特性成像方法,是核医学影像最先进的分子影像技术之一,目前已被广泛应用于恶性肿瘤的检测和分期,并已成为当前肿瘤生物调强放疗计划和疗效评估研究的重要影像技术[1-3].基于PET影像的高精度肿瘤生物靶区勾画是肿瘤放疗计划和疗效评估的基础,但肿瘤PET影像不仅信噪比低,而且空间分辨率低、部分容积效应严重,头颈部肿瘤周围正常组织的18FDG标准摄取值(standardized uptake value, SUV)通常还和肿瘤的SUV非常接近,因此,PET影像中的肿瘤边界通常模糊,且与毗邻正常组织的分界不清晰.肿瘤内部还具有各向异性的生物学特性,因此,高精度的肿瘤生物靶区勾画非常困难.
肿瘤生物靶区勾画本质上是肿瘤PET图像分割问题.随机游走(random walk, RW)是一种基于图论的交互式图像分割方法[4-6],它将图像的分割问题转化为图的Dirichlet最优化问题[5].随机游走图像分割算法在图像像素数量比较少的情况下,分割速度快,分割效果好,特别是在处理含有强噪声、弱边界或缺失边界、有模棱两可的图像区域时显示出了突出优势.这些特性使得随机游走算法非常适合处理PET图像.Bagci等[7]和Onoma等[8]最先将随机游走方法应用在肺部肿瘤PET图像分割中,并取得不错的效果.但由于头颈部解剖结构复杂,目前尚未见有关将随机游走方法应用于三维头颈癌PET图像分割的文献.
随机游走分割结果一方面依赖于种子点的位置和数量,另一方面依赖于顶点间边的权值计算方法.种子点越多,提供的前景与背景区域信息就越多,相应的分割结果就越令人满意[9]. 我们也发现肿瘤PET图像分割随机游走问题的求解不仅需要提供正确标记的肿瘤和非肿瘤种子点,而且种子点的数目和位置对图像分割结果有很大影响.不合适的种子点标记,可使随机游走问题无法有效求解;种子点数目不充足,可导致分割结果不理想.但是,在三维PET情况下,人工选择种子点不仅效率十分低下,而且种子点不足难以得到令人满意的分割结果,尤其对于PET SUV图像中生物特性各向异性的肿瘤分割更是如此.因此,我们根据头颈癌PET SUV分布特点,提出采用三维自适应区域生长和数学形态学膨胀方法,将包含肿瘤的感兴趣区域分为核心肿瘤区域、正常组织区域和待定区域三类,并将核心肿瘤区域全部体素点标记为肿瘤分割随机游走问题的生物靶区种子点,同时将正常组织区域全部体素点标记为非生物靶区种子点,这样只需要对待定区域的体素点进行随机游走分割.这不仅使随机游走问题的求解速度显著提高,而且大量的种子点使肿瘤分割结果更准确.另一方面,我们发现头颈部肿瘤和其周围正常组织PET SUV图像具有不同的对比度纹理特征,提出一种基于PET SUV及其对比度纹理特征量的肿瘤分割随机游走顶点间边的权值计算方法,以便更好地区分肿瘤和其周围高SUV的正常组织,进一步提高肿瘤分割结果精度.
1经典随机游走方法
首先,由图像构造一个无向加权图G=(V,E,W),其中V是顶点集,E是边集,W是相邻顶点间边的权值集合.图像的每个像素定义为图G的一个顶点vi∈V;顶点vi与vj间定义一条边eij∈E⊆V×V;每条边被赋予一个权值wij∈W.若两顶点不相邻,则两点间的权值定义为0,表示随机游走者不可能经过这条边.权函数有很多不同的定义形式[10],随机游走方法通常采用高斯权函数来定义相邻顶点间的相似性:
wij=exp(-β(Ii-Ij)2).
(1)
式中:Ii和Ij分别对应顶点vi与vj的图像灰度值.wij大小直接表明了像素点vi和vj之间的相似关系,对于无向图而言wij=wji.β>0为自由参数.
其次,通过求解图像分割随机游走Dirichlet最小值问题(2)的最优解x,获得随机游走者从任何一个顶点vi首次到达某类已标记顶点的概率xi:

(2)
其中,拉普拉斯矩阵L定义为:
(3)
而di=∑wij称为顶点vi的度,定义为与顶点vi所有邻边eij上的权重之和.为求解Dirichlet最小值问题(2),将所有顶点分为两类,即已标记为种子点的顶点集合VM和未标记的顶点集合VU,且满足VM∪VU=V,VM∩VU=∅.根据顶点所属集合不同,可以将拉普拉斯矩阵分解为如下形式:

(4)
则Dirichlet最小值问题(2)可以写成:
(5)
式中:xU表示随机游走者从任何一个未标记顶点首次到达某类已标记顶点的概率向量,每个分量对应一个顶点;xM表示随机游走者从任何一个已标记顶点首次到达某类已标记顶点的概率向量.对两类分割问题,已标记前景种子点的概率值设定为1,而已标记背景种子点的概率值设定为0;LM为已标记顶点之间边对应的权值矩阵;LU为未标记点之间边的权值矩阵;BT为未标记点和已标记点之间边的权值矩阵.
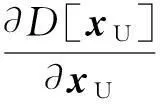
LUxU=-BTxM.
(6)
因为已标记种子点的多少与位置决定了xM,而顶点间边的权值计算方法决定了线性方程(6)的系数矩阵.所以,已标记种子点越多,求解线性方程(6)的速度就越快,所需要的计算机内存就越少.而且,种子点的选择方法和顶点间边的权值计算方法决定了方程(6)的解xU,因此,也决定了最终图像分割结果的正确性.
基于以上理论分析,我们提出了以下新的肿瘤PET图像分割随机游走种子点选择方法和顶点间边的权值计算方法.
2 随机游走方法的改进
2.1种子点的自动选取
肿瘤区域葡萄糖代谢活跃,因此肿瘤区域的18FDGPETSUV较高,这为肿瘤PET图像分割随机游走种子点的选择提供了方便.但肿瘤内部具有各向异性的生物学特性,且肿瘤PET图像空间分辨率低、噪声大、部分容积效应强.头颈部肿瘤周围正常组织的18FDGSUV通常还和肿瘤的SUV非常接近,这使得自动选择数目充足、分布合理种子点的工作变得困难.受文献[11]启发,本文采用三维自适应区域生长方法来自动选取合适的随机游走肿瘤和正常组织的种子点.图1为本文种子点的自动选取原理图,详细步骤如下:
1)为了减少计算量,我们粗略地在PET图像序列中选择了一个大小完全包含恶性肿瘤组织的三维感兴趣区域(regionofinterest,ROI),根据PET图像SUV的特点以及临床肿瘤专家知识,选择肿瘤区SUV值最大的体素点作为初始种子点,并通过算法自动获得后添加进种子区域列表中.
2)搜索种子点三维6-邻域(见图2所示)内未处理过的全部体素点,将其添加进邻域列表中.
3)计算当前种子区域列表中所有体素点的SUV平均值,记为SUVmean.
4)计算邻域列表中所有体素点与SUVmean的差值,选取差值最小的体素点作为区域生长的候选种子点.
5)若候选种子点的特征值满足式(7),则将该候选种子点添加进区域生长的种子区域列表,同时将该点从邻域列表删除,并转步骤2.
SUV(x,y,z)>T×SUVmean.
(7)
其中,自适应区域生长阈值T确定方法如下:设定阈值从1→0,每次以0.1的幅度递减,根据阈值与区域生长结果总体素数的关系,获得一个突变点t,即当阈值再减小时,满足生长准则的体素点个数突然增大60%以上,如图1(c)所示.同样,可以继续对t→t-0.1进行细分,每次以0.01的幅度递减,获得最优阈值T.
6)若候选种子点的特征值不满足式(7),则生长完成.
7) 考虑到PET图像自身的部分容积效应,将以上得到的自适应区域生长结果逐片进行数学形态学上的膨胀操作,为确保膨胀后的区域外围都是正常组织,结构元素可以取大一点,考虑到裕量,本文所有实验结构元素大小均取25×25,该大小适用于本文实验中所有大小不一的分割目标.标记自适应区域生长结果内的所有点为肿瘤区域种子点,膨胀后所得区域外的所有点为正常组织区域种子点,其余的点为待定点,如图1(d)所示.

图1 自动种子点选取方法

图2 6-邻域连通性示意图
2.2权值函数的改进
传统的随机游走方法仅考虑像素之间的灰度和位置关系,没有考虑其邻域的灰度信息.恶性肿瘤PET图像具有显著的各向异性特点,表现出肿瘤区域内部具有不均匀的SUV分布.头颈癌的亚临床区域通常与其周围代谢活跃的正常脑部组织表现出非常相近的SUV分布,这使得仅基于PET图像SUV特征的分割方法得不到令人满意的肿瘤分割精度.为解决该问题,本文提出联合利用PET图像的纹理特征改进随机游走权函数的构造,以弥补仅用SUV进行分割的不足.
纹理是图像表现出的一种重要的视觉信息,是图像中普遍存在而又难以描述的特征.通常采用纹理分析的方法研究像素间的空间分布关系.纹理特征是量化肿瘤内部各向异质性的一种有效度量.宏观上可以把各种纹理描述的方法分为四大类:模型家族、结构家族、统计家族和信号处理家族[12].统计家族这类纹理描述的方法和思想简单,易于实现.统计家族的方法是基于像素及其邻域的灰度属性,研究纹理区域中的统计特性,如一阶、二阶或高阶统计特性.一阶统计特性常用的有均值、中值、标准偏差、偏斜和峰度等统计特征量.二阶统计特性常用的有基于灰度共生矩阵统计得到的熵、相关性、和平均及相异性等特征量.高阶统计特性主要是基于图像的邻域灰度差异矩阵(neighbourhoodgrey-tonedifferencematrix,NGTDM)统计得到.图像的灰度差异矩阵反映的是灰度关于间隔以及变化幅度的综合信息,它提供的是图像的高阶信息,更符合人类的视觉特性.Amadasun[13]等于1989年提取了基于邻域灰度差异矩阵的5个影像纹理特征:粗糙度、对比度、频繁度、复杂度、纹理强度.受文献[14-15]启发,我们发现PETSUV的对比度纹理特征有利于区分头颈癌及其周围高SUV的正常组织.本文中,PETSUV的对比度纹理特征值基于三维邻域灰度差异矩阵提取,如式(8)-(10):
(8)
其中
Ai=A(k,l,s)=
(9)
N=d3-1,m,n,t≠0.
Ni表示肿瘤ROIPET以体素点(x,y,z)为中心、D为半径的邻域内所有SUV值为i的全部体素点集{(k,l,s)}.Ai表示以体素点(k,l,s)为中心、d为半径的邻域内各体素点的SUV均值.
进一步,PETSUV的对比度纹理特征量定义为
(10)
式中:Gh表示肿瘤PETROI中以点(x,y,z)为中心,半径为D的邻域(sROI)中最大SUV值;Ng是sROI中不同SUV值的总数.Pi=Ni/N,其中Ni表示sROI中SUV值为i的体素点总数,N表示sROI中体素点总数.
将PET图像对比度纹理特征引入随机游走方法中顶点间边的权值计算方法:
wij=exp (-β1(Ii-Ij)2-β2(Ci-Cj)2).
(11)
式中:Ii是PET图像像素点i的SUV值;Ci是对应点PETSUV的对比度纹理特征值;β1和β2是自由参数.该权值计算方法自适应地融合了PETSUV特征与PET图像对比度纹理特征信息.在图3所示矩形区域内的肿瘤边缘处,肿瘤和正常组织的PETSUV很相近,(Ii-Ij)2趋近为0,而相应的肿瘤和正常组织对比度纹理则有明显区别,(Ci-Cj)2非0.两者融合后的相应边权值wij将比仅考虑SUV时减小,从而有利于正确区分肿瘤和高SUV正常组织.(图中右侧虚线箭头所指为肿瘤区域,左侧点画线箭头所指为正常组织)

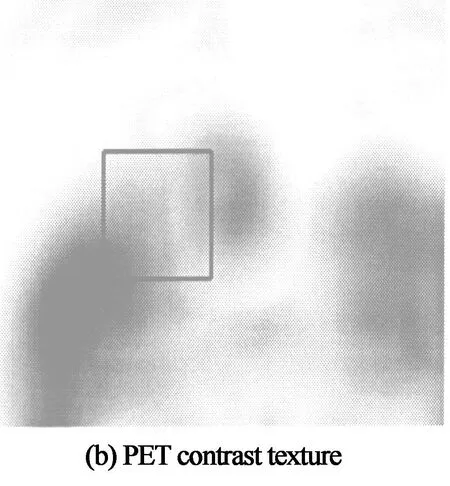
图3 PET SUV及其对比度纹理
3实验结果与分析
为了验证本方法的有效性,本文以7例头颈癌临床PET影像数据作为研究对象.这些PET影像数据由中南大学湘雅医学院附属湖南省肿瘤医院PET/CT中心通过GEDiscoverySTPET/CT扫描系统采集获得,每床位采集3min;对所采集的PET数据使用相应CT信息进行衰减校正,之后采用三维傅立叶重组迭代重建法进行三维重建.这7例PET影像数据大小均为128×128×110,体素大小为4.687 5mm×4.687 5mm×3.27mm,CT图像大小均为512×512×110,体素大小为0.976 6mm×0.976 6mm×3.75mm.
为了减少存储空间和计算量,我们根据不同的肿瘤位置和大小情况选取相应包含肿瘤的区域ROI进行处理,详情见表1.并在计算PET图像的纹理前,将SUV值进行三线性插值,使其体素大小成为0.976 6mm×0.976 6mm×0.976 6mm,并将SUV值转化为0~255的整数级.本文对所有实验都选取D=9和d=4,通过大量实验发现这个邻域大小能较好地提取PETSUV的对比度纹理特征.
在三维肿瘤PET图像分割中,若采用传统随机游走方法手工选择少量种子点,则求解线性方程(6)时需要占用极大的计算机内存,在普通个人计算机运行时会出现内存泄露,导致分割失败.采用本文方法可以自适应地选择大量种子点,因此本方法可以在普通个人计算机上运行,同时能够保证速度很快,这是本文方法的一大改进.但是为了便于与传统随机游走方法比较,体现本文所提算法的优越性,本文选择在如下实验环境下进行对比实验,尽管本文方法根本无需这么大的内存环境:IntelXeonCPUE5-2643 0 @ 3.30GHz,16GB内存,Linux操作系统,MATLAB2014a.

表1 本文方法和传统RW分割图像速度对比
受条件限制,我们无法获取每个病例的病理标本作为金标准.因此,我们邀请3位经验丰富的放疗科医生独立地对每个病例分别进行手工勾画大体肿瘤靶区(gross tumor volume,GTV),然后由另外一位经验丰富的放疗科主任医师综合以上3位医生勾画结果给出最终的GTV,我们将这个最终的GTV作为参考标准(ground truth, GT).为了验证提出方法的性能,本文采用Dice相似性(similarity, SIM)、灵敏性(sensitivity, SEN)和特异性(specificity, SPE)作为定量评价指标.
3.1性能指标
定义1相似性是指实验方法所得勾画结果与金标准的重合程度.SIM值越大表明两者几何形状重合得更好.定义为:
(12)
定义2灵敏性定义为GTV金标准被正确勾画部分所占的比例.
(13)
定义3特异性定义为非GTV金标准(实际正常组织器官)被正确地判定为正常组织部分所占的比例.
(14)
在公式(12)(13)和(14)中,Q,Q1与Q2分别代表实验选取的ROI、临床医生手工勾画的金标准和实验方法勾画结果.单一的灵敏性高而特异性低,说明分割结果存在一定的假阳性;单一的特异性高而灵敏性低,说明分割结果存在一定的假阴性;只有当两者同时较高时才能够说明分割方法的准确性.
3.2与传统随机游走方法性能比较
有研究[16]指出:在三维下的随机游走分割算法中,采取三维26-邻域来构造图所取得的分割效果最佳.因此,本文采用三维26-邻域来描述三维空间内体素与其相邻体素的关系.为了便于比较,且根据文献[5],本文对于所有随机游走方法选取β,β1和β2值均为90.为了简便起见,我们将手工选择种子点和未将纹理引入权值函数(即β2=0)的随机游走算法记为传统随机游走方法.图4给出了不同种子点选择方法所得到的随机游走分割结果.图4(b)和图4(j)是手工选取的种子点,图4(c)和图4(k)是本文基于三维自适应区域生长方法自动选取的种子点.从分割结果来看,用手工选取种子点的随机游走方法分割效果并不理想,图4(f)和图4(n)显示结果都出现了欠分割.这是因为在三维图像分割中,医学图像情况复杂、数量较多,手工选取种子点费时费力且带有很大的主观性,种子点太少不足以表征前景和背景区域的特征,造成分割结果不理想.本文提出的自动选择种子点方法是通过大量实验,在保证较好的分割结果和较快的分割速度前提下选择得到的.本文方法在执行过程中不需要人工干预选择种子点,从而在保证分割结果精确度的前提下大大提高了方法的执行效率.
图4(a)中黑色方框内的肿瘤和正常组织的PET SUV很相近,肿瘤组织与其右侧腮腺毗邻,边界没有明显的界限,此时(11)式中(Ii-Ij)2趋近为0,而相应的肿瘤和正常组织对比度纹理则有明显界限(见图4(d)黑色框内),此时(Ci-Cj)2非0.图4(i)与图4(l)也是类似情况,图4(i)中肿瘤不仅与其左侧的腮腺边界模糊,而且与其下方的小脑组织也没有明显的界限.本文提出的改进的权值函数,将PET图像的SUV信息与PET图像对比度纹理联合起来,两者融合后的相应边权值wij将比仅考虑SUV时减小,从而有利于正确区分肿瘤和高SUV正常组织.(外围十字点集:背景种子点,中间星号点集:前景种子点,封闭曲线:肿瘤轮廓,前两行为本实验的一例病例,后两行为另外一例病例的实验结果)

图4 不同随机游走分割结果
图5,图6和图7给出了本文自动选取得到种子点的分割结果与传统手工选取种子点随机游走分割结果定量比较.从图5可以看出,本文方法性能明显优于传统随机游走方法.本文方法分割结果比传统随机游走方法分割结果与参考标准的相似度平均提高32.5%,差异具有统计学意义(t检验:H=1,P<0.05).传统手工选取种子点数量不足造成了传统随机游走三维图像分割中出现欠分割(见图4(f)和图4(n)所示).因此,这种情况在特异性指标上会高于本文方法,但是灵敏性却很低.单一的特异性高没有实际意义,而本文方法分割结果却能同时具有较高的灵敏度和特异性,如图6和图7所示.
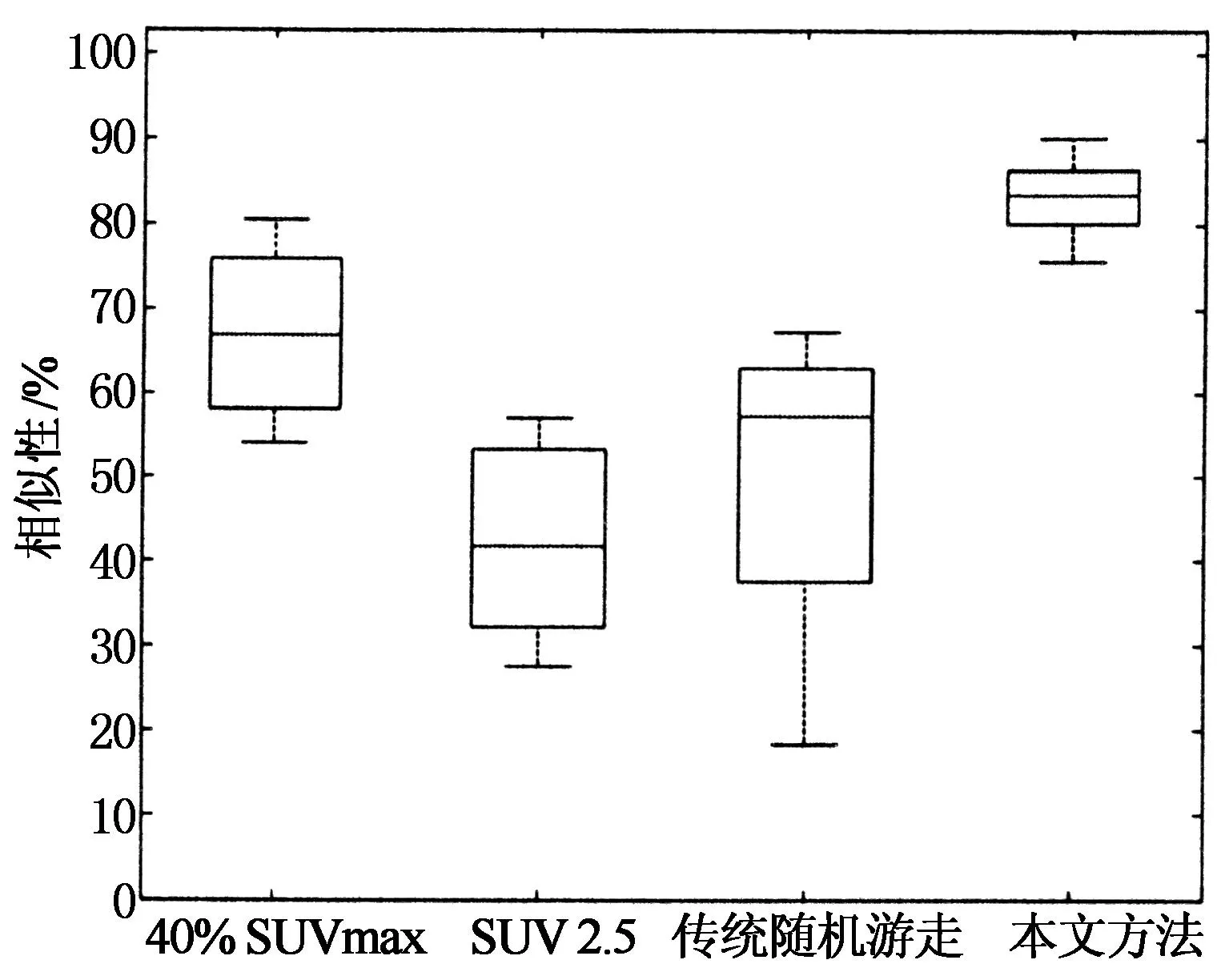
图5 不同方法相似性指标比较

图6 不同方法灵敏性指标比较

图7 不同方法特异性指标比较
表1给出了本文方法与传统随机游走方法运行的CPU时间对比结果.从表1可以看出,本文方法的运行时间要明显少于传统随机游走方法的运行时间,本文提出的方法比传统随机游走方法平均提速9.34倍左右,差异具有统计学意义(t检验:H=1,P<0.05).
3.3与传统阈值法比较
为进一步检验方法的性能,我们还将本文方法的分割结果同时与经典的40%最大SUV值(记为40% SUVmax)阈值法和SUV 2.5阈值法(记为SUV 2.5)的分割结果进行了比较.从图8可以直观地看到40% SUVmax阈值法和SUV 2.5阈值法都将与肿瘤毗邻的正常组织包括进来了(见图8(c)(d)中箭头所指区域).传统手工选取种子点数量不足造成了传统随机游走三维图像分割效果并不理想,图5,图6和图7中显示,对于有些病例,传统随机游走分割效果甚至不如40% SUVmax阈值法和SUV 2.5阈值法的分割效果好,这也证实了随机游走算法依赖种子点的数量和位置,种子点数量不足导致传统随机游走方法分割效果并不理想.而本文提出的方法却能够得到较好的分割结果,如图8(b)所示.本文所提方法的分割结果光滑圆润,没有离散点,充分显示了本文方法的优越性.

图8 本文方法与阈值方法分割结果
4结论
本文在三维自适应区域生长方法的基础上提出一种新颖的自动选取肿瘤PET分割随机游走种子点的方法.利用PET图像SUV的分布特点,通过三维自适应区域生长方法结果来确定随机游走种子点.由此得到的种子点,不仅数目充足,减少了图中未标记节点的数目,使计算时间大大减少,计算速度明显提高;而且位置合理,充分地利用PET图像的全信息,可以很好地表示肿瘤区域和正常区域的特征.本研究还将PET图像基于NGTDM提取的对比度纹理特征引入随机游走分割方法的权函数中,改进后的权函数自适应地融合了PET SUV特点与PET图像对比度纹理特征,进一步提高了PET图像分割精度.
实验结果表明,针对不同头颈癌PET图像分割任务,本文提出的分割方法比传统随机游走方法均表现出更好的分割效果和更快的分割速度,差异具有统计学意义.而且在与肿瘤毗邻的正常组织具有与肿瘤区域相似SUV值等较难分割情况下,该方法利用PET图像纹理信息改进了肿瘤分割的准确性.该方法还有待大规模的临床研究,以确定其临床应用价值.
参考文献
[1]NAQA I E. The role of quantitative PET in predicting cancer treatment outcomes[J]. Clinical and Translational Imaging, 2014, 2(4): 305-320.
[2]SCHWARTZ D L, FORD E, RAJENDRAN J,etal. FDG-PET/CT imaging for preradiotherapy staging of head-and-neck squamous cell carcinoma[J]. International Journal of Radiation Oncology Biology Physics, 2005, 61(1): 129-136.
[4]WECHSLER H, KIDODE M. A random walk procedure for texture discrimination[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 1979,1 (3): 272-280.
[5]GRADY L. Random walks for image segmentation[J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2006, 28(11): 1768-1783.
[6]MEILA M, SHI J. Learning segmentation by random walks[C]//Proceedings of Advances in Neural Information Processing Systems. Cambridge, Massachusetts: MIT Press,2001.
[7]BAGCI U, YAO J, CABAN J,etal. A graph-theoretic approach for segmentation of PET images[C]//Proceedings of 2011 Annual International Conference of the IEEE Engineering in Medicine and Biology Society. New York: IEEE, 2011: 8479-8482.
[8]ONOMA D P, RUAN S, GARDIN I,etal. 3D random walk based segmentation for lung tumor delineation in PET imaging[C]//Proceedings of 2012 9th IEEE International Symposium on Biomedical Imaging. New York: IEEE, 2012: 1260-1263.
[9]GRADY L, SCHIWIETZ T, AHARON S,etal. Random walks for interactive organ segmentation in two and three dimensions: Implementation and validation[M]//Medical Image Computing and Computer-Assisted Intervention-MICCAI 2005. Berlin: Springer Berlin Heidelberg, 2005: 773-780.
[10]SHI J, MALIK J. Normalized cuts and image segmentation [J]. IEEE Transactions on Pattern Analysis and Machine Intelligence, 2000, 22(8): 888-905.
[11]LIU Guo-cai, ZHU Su-yu, WANG Yao-nang,etal. Hierarchical and adaptive volume-growing methods for the PET based biologic target volume delineation for radiotherapy treatment planning[C] //Proceedings of 2011 IEEE Nuclear Science Symposium and Medical Imaging Conference (NSS/MIC). New York: IEEE, 2011: 4293-4295.
[12]REED T R, DUBUF J M H. A review of recent texture segmentation and feature extraction techniques[J]. CVGIP: Image Understanding, 1993, 57(3): 359-372.
[13]AMADASUN M, KING R. Textural features corresponding to textural properties[J]. IEEE Transactions on Systems, Man and Cybernetics, 1989, 19(5): 1264-1274.
[14]CUI H, WANG X, FENG D. Automated localization and segmentation of lung tumor from PET-CT thorax volumes based on image feature analysis[C]//Proceedings of 2012 Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC). New York: IEEE, 2012: 5384-5387.
[15]LIU Guo-cai, YANG We-li, ZHU Su-yu,etal. PET/CT image textures for the recognition of tumors and organs at risk for radiotherapy treatment planning[C]//Proceedings of 2013 IEEE Nuclear Science Symposium and Medical Imaging Conference (NSS/MIC). New York: IEEE, 2013: 1-3.
[16]GRADY L, JOLLY M P. Weights and topology: a study of the effects of graph construction on 3D image segmentation[M]// Medical Image Computing and Computer-Assisted Intervention-MICCAI 2008. Berlin: Springer Berlin Heidelberg, 2008: 153-161.
Random Walk Method for PET Image Segmentation of Head and Neck Cancer
LIU Guo-cai1†, HU Ze-tian1, ZHU Su-yu2, YUAN Yuan2, LIU Ke2, WU Zheng2, ZHANG Jiu-tang2, MO Yi2
(1. College of Electrical and Information Engineering, Hunan Univ, Changsha, Hunan410082, China;2. Depts of Radiation Oncology, Medical Physics and PET/CT Center of the Cancer Hospital of Hunan Province, Xiangya School of Medicine of Central South Univ, Changsha, Hunan 410013, China)
Abstract:In order to solve the problem of the high accuracy delineation of biological target volume (BTV) for the radiotherapy of head and neck cancer, a random walk method was proposed by using PET (positron emission computed tomography) image features of tumors. Firstly, the selected region of interest (ROI) was segmented into the primary tumor (labeled as foreground seeds), normal tissue (labeled as background seeds) and pending region by three-dimensional adaptive region growing and morphological dilation based on PET SUV images. Secondly, due to the differences of contrast texture feature of head and neck tumor and surrounding normal tissues in PET images, the contrast texture feature was incorporated into the weights of random walk(RW) to further improve the accuracy of tumor segmentation results. Clinical PET image segmentations of head and neck cancer have shown that the improved RW is 9.34 times faster than the traditional RW on average. And the similarity is increased by 32.5% on average if the gross tumor volume delineated by clinicians is considered as the ground truth (P<0.05). The proposed method is an efficient and accurate method for the delineation of the BTV corresponding to head and neck tumors.
Key words:medical image segmentation; random walk; region growing; biological target volume; head and neck cancer
中图分类号:TP391.4
文献标识码:A
作者简介:刘国才(1963-),男,湖南华容人,湖南大学教授,博士生导师†通讯联系人,E-mail:lgc630819@hnu.edu.cn
基金项目:国家自然科学基金资助项目(61271382,61301254,61471166), National Natural Science Foundation of China(61271382);湖南省肿瘤医院科研平台建设
*收稿日期:2015-04-15
文章编号:1674-2974(2016)02-0141-09
