重金属协同选择环境细菌抗生素抗性及其机制研究进展
2016-04-21张佳奇毛大庆
张佳奇,徐 艳,罗 义,毛大庆*
(1.天津大学环境科学与工程学院,天津300072;2.南开大学环境科学与工程学院教育部环境污染过程与基准重点实验室,天津300071)
重金属协同选择环境细菌抗生素抗性及其机制研究进展
张佳奇1,徐艳2,罗义2,毛大庆1*
(1.天津大学环境科学与工程学院,天津300072;2.南开大学环境科学与工程学院教育部环境污染过程与基准重点实验室,天津300071)
摘要:抗生素的长期滥用,引起环境细菌耐药性不断增强,加速了抗生素抗性基因(Antibiotic resistance genes, ARGs)在环境中的传播扩散。在重金属污染的环境中,细菌不仅具备重金属抗性,并且具备多种抗生素抗性,抗生素抗性基因的污染水平也随之升高。在介绍重金属与抗生素抗性最新研究进展的基础上,阐述了环境细菌的抗生素抗性、重金属抗性及其相关抗性机制,并着重论述重金属和抗生素协同选择环境细菌耐药性及其机制。
关键词:抗生素抗性;重金属抗性;协同选择抗性;协同选择抗性机制
张佳奇,徐艳,罗义,等.重金属协同选择环境细菌抗生素抗性及其机制研究进展[J].农业环境科学学报, 2016, 35(3):409-418.
ZHANG Jia-qi, XU Yan, LUO Yi, et al. Co-selection mechanisms of bacterial resistance to heavy metals and antibiotics[J]. Journal of Agro-Environment Science, 2016, 35(3): 409-418.
近年来,由于抗生素的滥用引起细菌耐药问题越来越受到关注。在抗生素长期作用下抗生素抗性基因(Antibiotic resistance genes,ARGs)在环境中以及细菌中被不断筛选和富集,从而表现为耐药性整体水平升高,进而导致抗生素类药物的治疗效力大大降低,迫使抗生素不断升级换代。通常抗生素在环境中的含量极低(通常在10-9~10-6数量级),远低于最低抑菌浓度(MIC),在低浓度抗生素长期选择压力作用下,细菌逐渐产生耐药性且携带的抗性基因持久存在,并在各种环境介质中传播扩散。尤其是最近出现的“超级细菌”被赋予了能够抵抗几乎所有抗生素的能力,其抗性根源在于该细菌携带的NDM-1(New Delhi metallo-β-lactamase-1)抗性基因,该基因自首次发现以来已经在全球迅速蔓延[1]。据此,抗生素抗性基因作为一个新的全球性污染问题已经引起广泛关注,世界卫生组织(WHO)已将“细菌耐药问题”作为21世纪威胁人类健康最大的挑战之一,并将在全球范围内对抗性基因的控制做出战略部署。
1 环境中抗生素抗性与重金属抗性的研究进展
目前人们认为抗生素的滥用是引起环境细菌耐药性增加的主要因素,Luo等[2]对我国海河流域表层水和沉积物中抗生素抗性基因的定量研究发现,两种磺胺类抗生素抗性基因sul1和sul2的相对丰度与磺胺类抗生素的含量存在显著的正相关性。沈群辉等[3]的研究也获得了类似的发现,黄浦江流域江水中sul3 和tetW以及底泥中的sul2丰度与对应的磺胺类抗生素含量显著正相关,江水中sulA与水中的氯霉素、四环素的含量显著相关。这些研究表明,环境中的残留抗生素作为抗生素抗性基因富集的环境选择压力促进了环境中抗性基因水平升高。然而近期研究发现,在临床和自然环境中,抗生素使用量的减少有时并不能有效控制抗生素抗性在环境中的传播和扩散[4]。Enne 等[5]研究结果表明,尽管临床上磺胺类抗生素的使用量已经减少了97%,但是大肠杆菌的磺胺抗性丰度却依然保持不变,对于这种现象,有研究认为磺胺抗性基因与其他的抗性基因连锁在同一个可移动的遗传元件上。Dantas等[6]从土壤中提取了480种链霉菌菌株,发现平均每种菌株对7~8种抗生素有耐药性,且其耐药对象还包括新开发出来的抗生素。细菌抗性基因对全新抗生素也具有耐药性,说明抗生素的存在并不是抗性基因发展的唯一影响因子。因此,环境中其他因素同样可能影响抗性基因的浓度水平及其传播扩散[7-8]。
在自然环境中,某些重金属离子作为细菌生长所必需的微量元素(如Zn、Cu、Mn、Mg、Ca、K等),当处于痕量水平时可促进微生物生长,并且能通过各种生理代谢机制维持体内所需金属离子的动态平衡;然而,当上述金属离子超过一定浓度时,就会阻碍细菌生长,降低多样性,甚至杀死菌体。与抗生素相比较,重金属化学性质较稳定,在环境中不易降解,其作为典型的环境污染物,在我国水/土环境介质中具有较高的背景值,已引起广泛关注。细菌可以通过相应机制对重金属毒性产生耐受或抗性。目前,一些金属如Zn、Cu等微量元素作为饲料添加剂已被广泛添加到饲料中,造成养殖业的重金属污染问题,同时为了实现增产,多种抗生素也被添加到畜禽养殖饲料中,我们已经在养殖动物的粪便中发现高浓度的抗生素和重金属[7]。高浓度的抗生素和重金属导致畜禽养殖厂及其周边环境抗生素和重金属交叉污染,重金属和抗生素对细菌产生抗生素和重金属抗性的协同、交叉等机制,加剧了抗生素抗性基因的污染[8]。
越来越多的研究表明抗生素抗性基因的丰度与环境中的重金属污染密切相关。De la lglesia等[9]研究认为,抗生素抗性基因的丰度与抗生素以及As、Cu等重金属污染程度显著相关,表明As、Cu等重金属和抗生素的复合污染可以增加环境中抗性基因的丰度。同时,越来越多的研究发现重金属与细菌耐药性的关系。在重金属污染地区,细菌不仅对重金属具备抗性,还能对多种抗生素产生抗性,即抗生素抗性基因水平随着重金属污染水平增加而增加[10-25]。
Kolpin等[10]研究发现,地表水与城市水循环系统中含有大量多重抗性细菌,在重金属污染的位点会导致抗药性的增强。Gao等[11]对上海一个污水厂的水样研究也发现,样品中Zn和Pb含量大小对红霉素抗性基因具有重要的选择作用。Stepanauskas等[12]在实验室模拟的微宇宙实验中发现水中抗生素抗性检出频率随重金属暴露浓度的升高而升高,在金属Cd的影响下,氨苄青霉素抗性菌株(Ralstonia mannitolilytica)富集。重金属引起微生物抗药性水平升高在野外研究中也有报道。Berg等[13]对高Cu污染地区的土壤土著菌的研究表明,在这些环境中的微生物不仅对Cu具有很高的耐受性,对四环素和万古霉素的耐药性也显著增加,同时发现土壤中的Cu不仅筛选出对其具有抗性的土著菌,也筛选出了对抗生素具有抗性的土著菌。Knapp等[14]对苏格兰地区多处土壤样品的研究分析表明,土壤中Cu的含量与土壤中多种抗性基因(tetM、tetW、blaOXA、ermB、ermF)的丰度有着显著的正相关性,而Cr、Ni、Pb等金属的含量与一些特定的抗性基因丰度具有显著正相关性。其中与金属Cr相关的基因型有tetM、blaCTX-M、blaOXA,与金属Ni相关的基因型是tetW,而tetM基因型的丰度同时受到金属Cu、Ni和Pb的含量水平的影响。在农业土壤中Cu的广泛富集不仅能增强土著菌对Cu的耐受性,而且能够增强其抗生素抗性[15]。Wardwell等[16]研究表明,在美国缅因州中心沼泽泥样品中,Hg含量的增加不仅能增强环境中细菌对Hg的抗性,而且能同时增加细菌的多重抗药性。Alonso等[17]研究也发现,土壤中Cu的暴露特异性能够选择出对Cu具有抗性的菌株,同时能够协同选择抗生素抗性菌株。Ji等[18]对上海多个畜牧养殖场的动物粪便和周边土壤样品的研究发现,样品中Cu、Zn和Hg的含量与磺胺类抗性基因,特别是sul2基因的丰度具有显著的正相关性,同时发现重金属As的含量与tetB基因丰度也具有显著正相关性。研究结果说明,粪肥和施用过粪肥的土壤中的重金属能协同选择部分抗性基因。不仅在土壤介质,水环境中也有重金属污染引起微生物抗药性增强的报道。McKinney等[19]对畜牧养殖场泄湖水样的研究发现,样品中重金属含量与抗生素抗性基因丰度存在相关性。Lu等[20]对大辽河和辽河入海口的水样研究发现,在大辽河样品中,金属Cr、Co、Ni、Cu、Zn和Pb的含量与sul1、sul2和sul3基因丰度具有显著正相关性,同时金属Mn、Ni和Zn的含量与int1丰度显著相关。Icgen等[21]研究了土耳其克泽尔河水中重金属与抗生素抗性共生微生物,对290株分离菌株的研究发现,24株对重金属与抗生素同时具有抗性,其中50%~92%的分离菌株对重金属Pb、Sn、Ag、Ni、Ba、Li具有抗性,超过50%的菌株抗喹诺酮类、磺胺类、氨基糖苷类抗生素。Devarajan等[22]对瑞士日内瓦湖不同位置底泥沉积物样品的定性定量研究表明,样品中抗性基因(blaTEM、blaSHV、blaCTX-M、blaNDM和aadA)和大肠杆菌属、肠球菌属、假单胞菌属等指示细菌的丰度、含量与有机物质含量、重金属含量密切相关。Wu等[23]对上海虎林地区和溆浦地区两个垃圾中转站和一个垃圾填埋场的垃圾渗滤液样品研究表明,sul1、sul2、tetM、tetQ、ermB及mefA等抗性基因丰度与Cr、Cd、Ni和As等金属含量水平有着显著相关性。此外,在临床研究中Alonso等[24]发现,临床分离的革兰式阴性菌(Stenotrophomonas maltophilia)中包含抗生素抗性基因和重金属抗性基因,在该研究中发现的抗性基因中包括抗大环内酯类基因(mphB)和抗镉外流泵基因(cadA),及其转录调节编码基因(cadC)。Ahmad等[25]研究发现,从炼油厂的废水中分离出的菌株至少抗一种重金属(>200 μg·mL-1),在研究的13种抗菌类药物中,这些菌株也至少抗一种抗菌类药物。
上述研究表明,环境中的重金属污染对抗生素抗性基因的筛选具有促进作用。
2 细菌抗生素抗性与重金属抗性的协同选择机制
目前,公认的细菌耐药性机制包括:细菌生理活动使抗生素分解或失去活性;细菌生理活动使抗生素作用的靶点发生改变;抗生素的存在改变细菌细胞特性;细菌产生药泵将进入细胞的抗生素泵出细胞。细菌的重金属抗性机制有:细菌细胞阻止金属离子内流作用;细胞内的隔离作用和解毒作用;细胞促进金属离子外流;进入细胞内金属的转化作用等。
随着微生物抗性的提高和微生物耐药性的普遍存在,加上新型合成和复合金属材料以及重金属抗菌剂的广泛应用,重金属与抗生素的协同选择抗性问题越来越普遍,并且引起广泛的关注。
2.1细菌抗生素抗性与重金属抗性的协同抗性机制
协同抗性指细菌携带的多种特定抗性基因位于同一遗传元件上,如质粒、整合子或者转座子等[8,10](图1)。研究发现重金属抗性基因和抗生素抗性基因可以存在于质粒上[21,26],利用转化、质粒消除及质粒测序等方法可证实两者的协同抗性。
Rasmussen等[26]对丹麦海港水域的研究发现,从重金属污染的水域中分离出来的菌株较之非重金属污染水域分离出来的菌株含有更多的抗性质粒。Davison等[27]研究同样发现,抗生素的选择压力的存在对于抗生素抗性基因的选择并不一定是必需的。从自然环境中分离出来的菌株,通常发现抗生素抗性基因和重金属抗性基因是相关联的。在临床样本中,重金属银与磺胺类抗生素抗性的协同选择是因为银和磺胺类抗生素的抗性基因位于同一个复制子上[28]。临床上,用0.5% AgNO3溶液处理患者的烧伤表面,对处理后烧伤表面分离得到鼠伤寒沙门氏菌(Salmonella typhimurium)进行研究,结果表明Ag离子可以选择出对Ag和多种抗生素同时具有抗性的菌株,这些菌株携带有这两种抗性基因的抗性质粒[29]。Summers 等[30]和Edlund等[31]的研究指出,牙科上使用的牙科Hg合金可以筛选出口腔和肠道内抗生素抗性增强的菌株。Davis等[32]对牙科Hg合金的研究也发现,从空腔中分离得到的屎肠杆菌中发现一种新的抗Hg操纵子位于转座子上(Tnmer1在ISL3家族),且在该转座子上发现抗链霉素基因aadK。除了在临床样本上发现抗生素抗性基因与重金属抗性基因在同一个基因遗传元件上有相关性外,在人工养殖环境样本中也发现有这两类基因协同存在的报道。Hasman等[33]发现从养猪场分离出的屎肠杆菌(E. faecium)对铜和大环内酯类、糖肽类抗生素具有抗性,同时发现抗Cu基因tcrB与抗大环内酯类基因ermB、抗糖肽类抗生基因vanA存在相关性,原因可能由于它们同时位于同一接合质粒上。McIntosh等[34]从水产养殖环境中分离得到杀鲑气单胞菌(Aeromonas salmonicida),并在其中一个IncA/C的质粒上鉴定出抗Hg操纵子(mer operon)和多种抗生素抗性基因,包括氟洛芬抗生素的抗性基因floR。Wright等[35]和Rosewarne等[36]发现受重金属污染的水环境中,Ⅰ类整合子的丰度有显著的提升。此外,通过病原体伤寒沙门氏菌血清型CT18基因组测序,发现接合质粒(pHCM1)上存在抗甲氧苄氨嘧啶(dhfr1b)、磺胺类药(sul2)、氨苄青霉素(bla)和链霉素(strAB)基因以及一个抗Hg操纵子[10]。
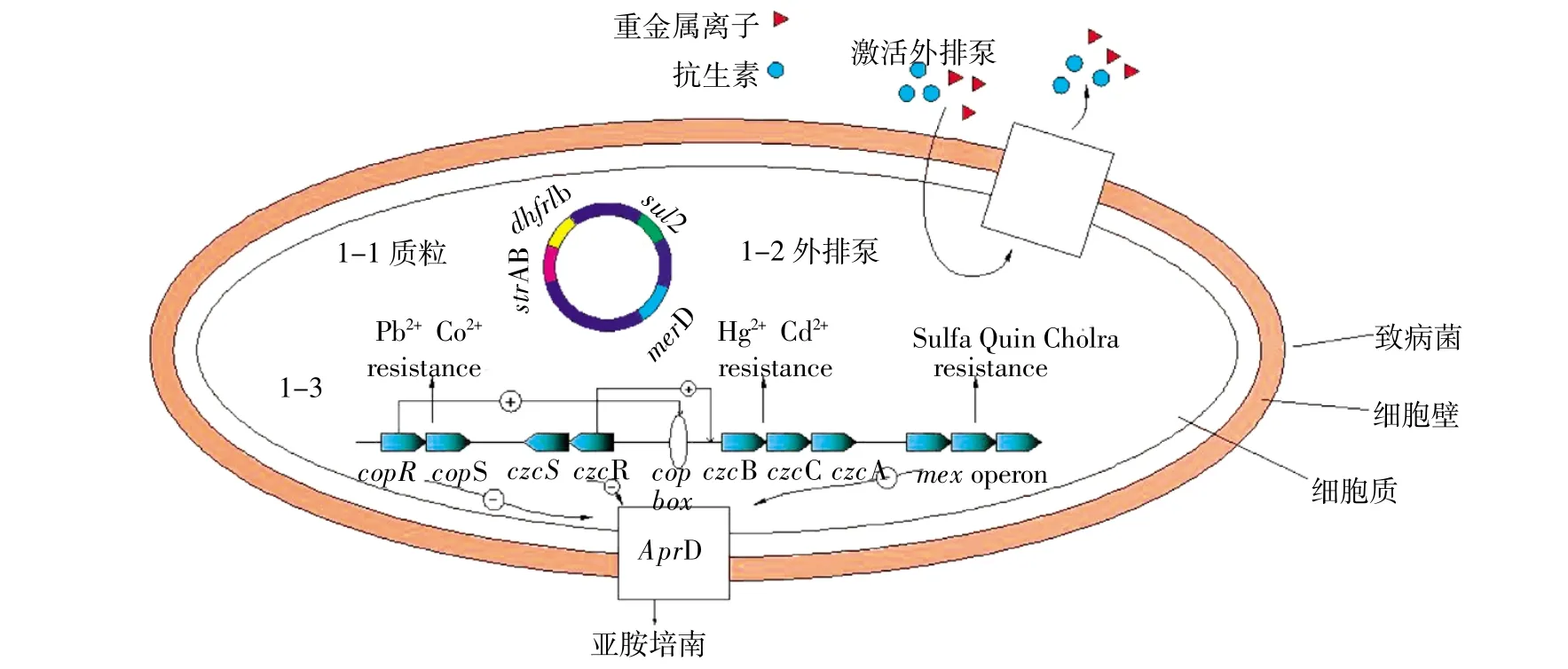
图1 细菌抗生素与重金属协同选择抗性机制图示Figure 1 Co-selection mechanisms for bacterial antibiotic and heavy metal resistance
综上所述,细菌协同抗性产生的本质原因是抗生素抗性基因和重金属抗性基因位于同一个抗性遗传载体上。细菌抗生素抗性和重金属抗性连锁传播的主要载体有接合性质粒、整合子及转座子[37]。整合子作为基因捕获系统,位于染色体和质粒或转座子上,通过质粒或作为转座子的一部分在细菌间进行传播[38]。其中一类整合子被认定对抗性连锁传播有重要作用,它们通常含有耐药基因盒,并且在长期重金属压力下,这类整合子介导的耐药细菌具有选择性优势。
诸多研究发现,在多种细菌体内存在可移动的抗性连锁传播载体。Zhang等[37]在香港沙田的五个污水处理厂的活性污泥样本中都发现一类整合酶基因intⅠ1与抗生素和重金属抗性基因同时存在,从香港两座污水厂活性污泥中分离出来的109株肠杆菌Enterobacteriaceae中,33%的菌株中同样也发现一类整合子。除了污水厂活性污泥样本,一类整合子普遍存在于环境污水中、自然河流及土壤介质中,在介导抗生素和重金属抗性中起重要作用[35-36]。上述研究表明,在特定环境中,这些可移动抗性遗传载体在细菌抗生素抗性基因和重金属抗性基因连锁传播过程中发挥重要作用,并对重金属和抗生素复合污染环境下细菌协同抗性的产生起到决定性作用。
2.2细菌抗生素抗性与重金属抗性的交叉抗性机制
交叉抗性是指细菌细胞利用同一种抗性系统对抗生素和重金属同时产生抗性,例如外排泵系统(图1)。这种间接的选择过程是因为抗生素和重金属的抗性机制的耦合作用,这种抗性机制在生理学上的耦合作用称为交叉抗性;而如图中1-1部分在基因上的耦合作用称为协同抗性。在交叉抗性的作用情形下,菌株对多种抗菌剂具有抗性,这些抗菌剂包括多种抗生素和重金属离子[39]。当不同的抗生素攻击同一靶点时,可启动细胞共同通路或者共享一个共同的途径获得各自的作用目标。
之前研究发现,伤寒沙门菌(S.enterica serovar)具有多种药物外排泵,其中外排泵mdtABC和AcrD使其对β-内酰胺、新生霉素、Cu和Zn离子产生抗性。Aendekerk等[ 40]对铜绿脓假单胞菌(Pseudomonas aeruginosa)中MexGHI-OpmD外排泵的研究发现,整个外排泵的反式运输机制增强了对金属V和抗生素替卡西林的抗性。Gillings等[41]和Stokes等[42]研究发现,从淡水中分离出来的细菌Ⅰ类整合子靠近编码多重抗药性的外排泵系统czcA,而这个外排泵系统能够外排Zn2+、Cd2+和Co2+等离子。Hayashi等[43]的研究发现,洋葱假单胞菌(Burkholderia cepacia)是具有多重耐药性的菌株,其中B.cepacia KF1菌株不仅对多种β-内酰胺类药物具有抗性,而且对卡那霉素、红霉素、四环素、新生霉素、利福平等也具有抗性。而B. cepacia KF1的这些抗性表型是由OprM和OprA的外排泵系统作用的结果。Hernanadez等[44]从炼油厂污染土壤中分离出两种肠杆菌(Escherichia hermannii 和Enterobacter cloacae),发现这两种菌在钒酸存在条件下,不仅能够很好地蓄积重金属,同时对更多抗生素产生抗性。原因可能是钒酸刺激了多重抗性表型,促进这些菌种中膜结合外流泵系统发挥作用,对其精确的机制尚需要进一步研究。
2.3细菌抗生素抗性与重金属抗性的协同调控机制
协同调控是指细菌在抗生素或重金属任何一种压力下,细菌体内的一系列转录和翻译应答系统均会对其作出反应的作用过程(图1)。例如细菌可通过双组分系统进行协同调控,对环境变化作出应答[45-49]。
为应对不同的环境变化,细菌演化出多种细胞信号转导途径,其中磷酸化和去磷酸化在生物体中广泛存在,其过程由不同的激酶催化完成。几乎所有的细胞都利用磷酸化介导的信号转导机制来应对代谢以及环境和细胞周期的变化[45]。双组分系统在细菌、古生菌和真菌中均有发现,其在细菌中存在最为广泛,参与对环境刺激作出反应的多种信号转导过程[46-49]。双组分系统有多个“靶点”,如感应外界刺激的位点、激酶自主磷酸化位点和反应调控蛋白磷酸化位点等。双组分系统能感应不同环境刺激并作出应答反应。
重金属污染的环境能够触发菌株中协同调控机制的运行,通过协同调控抗生素抗性基因的表达增强菌株对抗生素的耐受性。重金属离子能够协同调控抗生素抗性基因,进而降低菌株对抗生素的敏感度[50]。Harrison等[51]研究发现,在大肠杆菌体内SoxS蛋白就是AcrAB外排泵系统的调控子。在由CrO-7和Cu2+引起的细胞氧化应激作用下,SoxS蛋白增加,使得AcrAB外排系统的表达增强,细菌对氯霉素、四环素、新生霉素、新青霉素Ⅲ等抗生素的耐受性增强。
绿脓假单胞菌(P.aeruginosa)中copRS和czcRS双组分系统分别调控对Cu、Zn、Cd和Co的抗性,然而负调控OprD基因对抗生素产生抗性;用Cu处理该菌,czcRS双组分系统操纵子和czcCBA操纵子的转录水平均提高,同时引起膜孔蛋白OprD减少,最终导致对亚胺培南产生抗性,即Cu激活copR,后者参与调控czcRS双重组分系统,进而控制抗生素的进入和重金属的排出[52]。微阵列分析恒温培养下的大肠杆菌(E. coli MG1655)表明过量的Zn会使mdtABC操纵子上调,从而对新生霉素、脱氧胆酸盐和Zn离子产生抗性[53]。随后研究也表明,在过量的Zn条件压力下,与环境和临床相关的mtd系统会上调,大肠杆菌的质粒基因robA(为DNA结合蛋白质XylS-AraC的一员)的超表达分析表明这个基因增加了突变体的抗性谱,使得大肠杆菌对一系列不同的抗生素和金属Ag、Cd等也产生抗性[54]。
除细菌的双组分系统以外,细菌抗生素和重金属的协同调控对菌群生物膜的形成也有重大影响。细菌生物膜被认为是工业、临床和生态环境系统中细菌的主要生长表型(图2),会导致一些在临床上顽固的感染,包括绿脓杆菌、肺炎克雷伯菌和表皮葡萄球菌。生物膜表型的表达是细菌群体感应的结果。细菌之间的空间狭小,为基因的横向转移提供了理想的环境。自然条件下,微生物以浮游和生物膜两种生长状态存在,形成生物膜的细菌对金属和抗生素的耐受性远远高于浮游细菌。研究表明向浮游细菌中加入金属或抗生素会刺激产生胞外多糖(EPS),导致细胞黏附,最终形成生物膜。刺激产生的胞外多糖对生物膜的形成和生物膜三维结构的稳定起着重要的作用[50]。
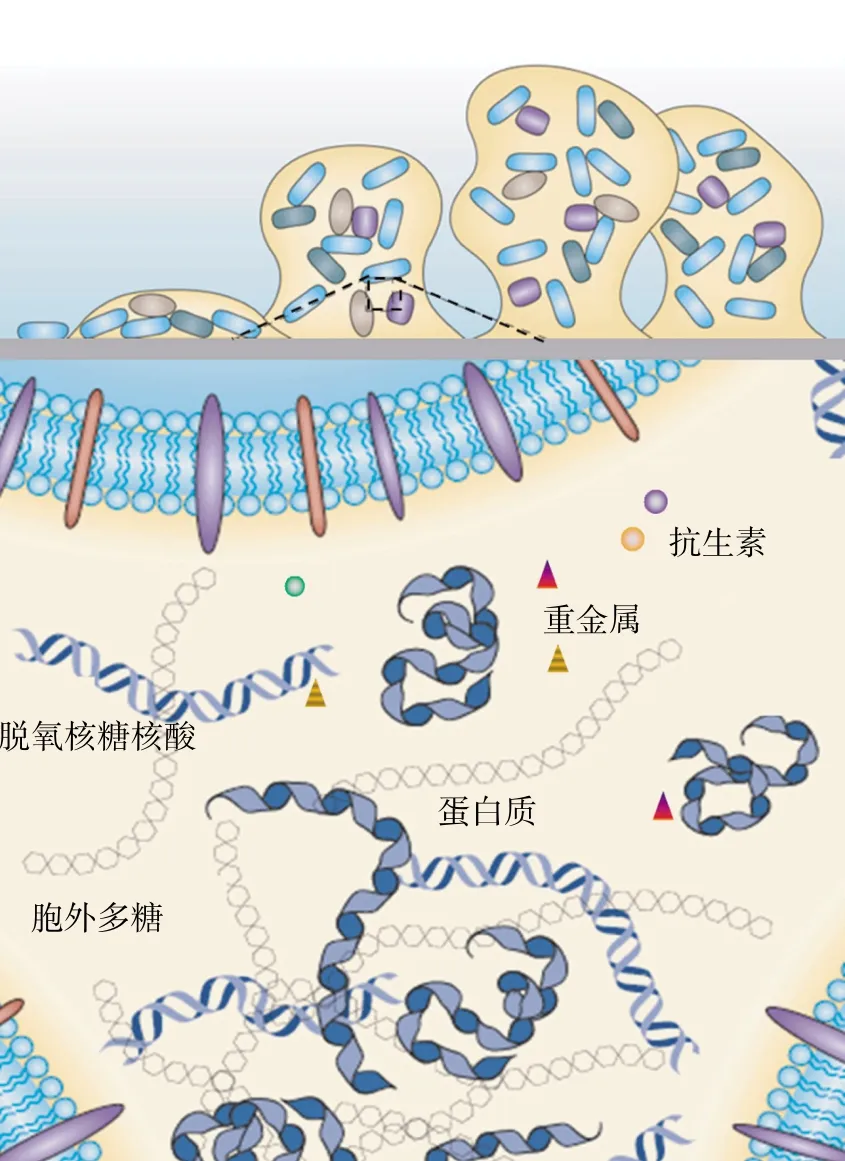
图2 菌群生物膜诱导作用图示[55]Figure 2 Mechanisms induced by bacterial biofilm[55]
研究证实,发现古细菌、蓝藻和假单胞菌在重金属暴露下容易刺激EPS的产生。与未形成生物膜的浮游绿脓假单胞菌P.aeruginosa相比,形成生物膜的绿脓假胞单菌对Pb和Cu离子的耐受性分别提高了2倍和600倍[56]。因此,重金属与抗生素的共调控和交叉抗性对生物膜生成机制的研究十分重要,如果细菌暴露于任何重金属存在的环境中,将诱导生物膜的增长,同时导致细菌抗生素抗性的提高。
3 细菌抗生素抗性基因的其他影响因素
其他一些环境地球化学因素对抗性基因的影响不容忽视,例如包括温度、pH值、溶解氧、离子(重金属)、浊度、氮、磷、细菌总数在内的水质因子,以及光照和氧气等因素。其中温度、pH值、溶解氧、浊度、氮、磷及细菌总数代表了水环境水质状况,这些指标的变化将导致整体微生物的丰度与种群结构的差异。Liao 等[57]对我国南部未开采的铜矿红土中的微生物量、微生物呼吸作用、土壤基质利用模型进行研究,结果表明土壤微生物群落在很大程度上受到重金属的影响,在相对高浓度的重金属污染环境下,微生物量和呼吸作用受到负面影响。研究还发现,两个重要的微生物生理生化参数——微生物C/N比和代谢熵与重金属压力显著相关,随着重金属浓度的升高,C/N比会明显下降而代谢熵增加,唯一碳源利用模型多变量数据分析表明,重金属的污染对微生物的群落结构和功能差异有重要影响[57]。微生物群落组成结构是抗生素抗性基因产生和传播的原因。有研究指出,土著菌本身群落结构的不同和多样性也会影响到它们自身对Cu污染的敏感性和抗性的不同。所以,菌群自身的差异就部分控制了Cu对它们的影响[58-59]。Novo等[60]的研究表明,在污水处理过程中,微生物群落结构组成对四环素类抗生素的降解具有一定的影响,并且污水中四环素浓度与Epsilonproteobacteria具有正相关关系,与Gammaproteobacteria、Betaproteobacteria和Firmicutes具有负相关关系。Forsberg等[61]就一系列农业土壤和草地土壤对18种抗生素的抵抗力进行功能元基因组的筛选,发现土壤细菌很少拥有物种之间抗生素抗性基因交换的序列特征,认为土壤中细菌群落的组成结构是促进抗生素抗性基因变化的重要因素。Tom-Petersen等[62]在不同的土壤条件下研究发现,Cu本身的生物有效性会有很大的差异,所以铜在不同的实验中对微生物群落结构的影响是不同的[62]。不同的土壤组成成分会通过不同的物理化学反应,例如吸附作用、络合作用和沉淀作用等,影响到金属离子的固定,除了微生物群落结构差异的影响,土壤本身的条件也会影响抗生素抗性基因的传播。郑加玉等[63]研究表明,人工湿地处理3种四环素类抗生素抗性基因(tetM、tetO、tetW)的结果发现沸石湿地对3种抗生素抗性基因的去除效果好于火山岩湿地。腐殖土中抗生素抗性基因绝对含量高于红壤土,说明腐殖土对抗生素抗性基因具有较好的累积能力,植物的存在减少了土壤中tetM和tetO累积量,但是增加了tetW累积量。
Sara等[64]对污水处理系统的研究发现,在对四环素类抗生素药物的去除过程中,温度对其去除率的影响主要是通过影响微生物的活性实现的。温度是微生物的一个重要生态因子,在最佳温度范围内,微生物可以达到最大的生长速度和最佳的代谢效率,从而提高对四环素类抗生素的去除效率。四环素在不同体系pH值条件下会发生质子化和去质子化反应,改变其在溶液中的存在形式而呈现不同价态的离子属性,从而影响其在污水处理过程中与活性微生物之间的相互作用,进而影响其去除过程机制[65]。另有研究发现,在污水厂中抗生素抗性基因的浓度有明显的季节特征,冬季高于夏季[66]。原因不仅是冬夏两季污水中收纳的抗生素种类不同,更是因为两季节污水中微生物的群落结构组成不同。城市污水是一个多组分的复杂体系,往往含有多种抗生素和其他污染物质,如重金属、杀菌剂等,这些物质的共同作用也会对细菌微生物产生共选择效应和交叉抗性作用[67]。重金属浓度与抗生素抗性之间有着复杂的关系,重金属和抗生素的交叉作用主要表现为协同抗性和抗性杀菌,浓度对交叉抗性组合的影响主要分为3种类型。在低浓度表现为协同抗性、高浓度表现为协同杀菌的抗性组合有Cr6+或Zn2+与红霉素,Cu2+与头孢拉定;在低浓度表现协同杀菌、高浓度表现为协同抗性的抗性组合有Cr6+或Zn2+与阿莫西林;菌体的抗生素抗性变化只与重金属种类相关的抗性组合有Cu2+与四环素、阿莫西林或红霉素,Cr6+与头孢拉定[68]。
抗生素在自然环境中的残留对抗生素抗性基因具有直接的选择压力。上述研究发现,微生物群落结构对四环素类抗生素的降解和富集产生影响,且温度通过影响环境中微生物的群落生长状态对四环素类抗生素发挥重要的影响作用。另外,由于抗生素自身结构性质的不同,大多抗生素具有光降解特性。在强光照下会加速抗生素的降解,减少对相应抗性的选择性压力,诱导产生的抗性基因也就相应减少。抗生素光降解过程中受到pH、催化剂、初始浓度等因素的影响,研究发现盐酸四环素的光催化降解与pH、TiO2投加量和四环素的初始浓度有关。改变影响因子会使多种抗生素的共同降解产生选择性,例如四环素与磺胺甲恶唑共同降解时,碱性条件下会优先降解四环素[69]。水环境和土壤环境中抗生素的降解情况不同,这与水环境和土壤环境中的光照、温度、水分等条件差异有关,且与抗生素种类有关。在土壤环境中,Schlusener等[70]研究了大环内脂类抗生素在土壤中的有氧生物降解,实验结果表明红霉素在土壤中的降解半衰期是20 d,而罗红霉素在整个实验过程中几乎不降解。另有研究发现,水环境中氧气是否充足对抗生素的降解也造成很大的影响,在可吸收礁湖肥料区域的地下水中测不到莫能菌素,表明莫能菌素在地表有氧层的降解比在无氧条件下快[71]。Pei等[72]在研究生物处理对废水中抗生素抗性基因的作用时发现,厌氧和光照处理后,四环素类抗性基因tetO、tetW和磺胺类抗性基因sul1、sul2有明显的降低趋势。研究还发现,在好氧处理过程中,tetO基因在4℃时增长到丰度的最高值;在厌氧处理过程中,tetO基因在20℃时增长到浓度的最大值[72]。Toes等[73]对大肠杆菌中两种Cu抗性基因copA和cusA的研究发现,前者在好氧与厌氧环境下低、中、高的Cu选择压力下都有表达,后者只在厌氧和好氧环境中的高浓度Cu选择压力下表达;然而,对奥奈达希瓦式菌(Shewanella oneidensis)的研究发现,在好氧和厌氧的环境中Cu的选择压力下会优先引起cusA基因的表达。
综上所述,自然环境中抗生素、抗生素抗性基因的影响因素往往不是单独发挥作用。自然环境条件下的温度、pH值、溶解氧、重金属离子浓度对微生物群落组成结构产生影响,微生物群落组成结构和自然光照条件对环境中抗生素的降解和富集产生影响,进而影响到环境中抗生素抗性基因的产生与传播。于此同时,重金属与抗生素的交叉污染对抗性基因的协同选择与传播产生复杂的综合影响。
4 重金属引起的细菌抗药性未来研究展望
目前研究发现,在抗菌药物的过度消费和抗药细菌的频繁检出之间存在明显的相关性。除了抗菌药外,其他污染物也可能成为驱使环境中抗性基因的选择性的重要因素。在农业领域中对含有毒金属杀菌剂的使用,在食品行业和医疗领域含Hg和Ag的防腐剂、药剂的广泛使用,以及动物饲料中重金属添加剂的添加都会造成重金属污染物直接被吸收进入人体,并可能长期蓄积造成严重的健康危害,而且还能引起环境中细菌耐药性水平总体升高,最终造成更大的健康危害。因此,开展微生物抗重金属及抗生素抗性机制的研究,对于研究重金属污染作为一种环境选择压力对病原微生物携带抗性基因的维持机制非常重要,对降低医疗领域抗生素耐药性以及消除环境中的细菌耐药性均具有重要意义。
目前对环境抗生素抗性的研究主要集中在抗性基因的定性、定量以及一些相应抗性菌株的检测等,大多数研究比较单一,缺乏整体性和系统性。现实环境的污染情况却日趋复杂,存在多种抗生素污染、抗生素-重金属复合型污染等。因此,相关研究在考虑抗生素污染物的前提下,还应该考虑复合型污染所带来的环境效应,在以后的研究中还需开展以下关于环境中抗性基因的研究:
开展抗生素抗性基因在环境中的传播、扩散机制以及控制对策的研究。对抗性基因从污染源向地表水迁移、地下水渗漏以及在土壤介质中的传播规律应予特别重视,同时应对渔业、畜牧环境中抗生素抗性基因、重金属抗性基因的传播、扩散机制开展深入的研究,以期更加有效地遏制抗生素抗性基因在环境中的扩散。
开展环境中抗生素和重金属的复合污染、多重抗性以及抗性基因组学的研究。许多抗生素抗性基因常与一些可移动遗传元件相关联,而这些元件往往还携带大量的其他种类的抗性基因,如重金属抗性基因、抗杀虫剂基因等,从而使微生物具有多重抗性,对环境和人类生活都造成更大的潜在危害。因此,需开展对各环境介质中多重抗性的污染水平、抗性基因种类以及基因水平迁移规律的研究,评估其生态健康风险,为国家制定相关政策提供理论依据。
参考文献:
[1]徐丽娟,黄敏.“超级细菌”NDM-1的研究现状[J].动物医学进展, 2010, 31(11):100-103. XU Li-juan, HUANG Min. Research advances on the "superbug" NDM-1[J]. Progress in Veterinary Medicine, 2010, 31(11):100-103.
[2] Luo Y, Mao D Q, Rysz M, et al. Trends in antibiotic resistance genes occurrence in the Haihe River, China[J]. Environmental Science & Technology, 2010, 44(19):7220-7225.
[3]沈群辉,冀秀玲,傅淑珺,等.黄浦江水域抗生素及抗性基因污染初步研究[J].生态环境学报, 2012, 21(10):1717-1723. SHEN Qun-hui, JI Xiu-Ling, FU Shu-jun, et al. A preliminary study on the Huangpu River antibiotics and resistance genes pollution situation [J]. Ecology and Environmental Sciences, 2012, 21(10):1717-1723.
[4] Salyers A A, Amabile-cuevas C F. Why are antibiotic resistance genes so resistant to elimination?[J]. Antimicrobial Agents and Chemotherapy, 1997, 41(11):2321-2325.
[5] Enne V I, Bennette P M, Livermore D M, et al. Enhancement of host fitness by the sul2-coding plasmid p9123 in the absence of selective pressure[J]. Journal of Antimicrobial Chemotherapy, 2004, 53(6):958-963.
[6] Dantas G, Sommer M O A, Oluwasegun R D, et al. Bacteria subsisting on antibiotics[J]. Science, 2008, 320(5872):100-103.
[7] Mu Q H, Luo Y, Sun Y X, et al. Occurrence of sulfonamide-, tetracycline-, plasmid-mediated quinolone- and macrolide-resistance genes in livestock feedlots in Northern China[J]. Environmental Science and Pollution Research, 2015, 22:6932-6940.
[8]张俊亚,魏源送,陈梅雪,等.畜禽粪便生物处理与土地利用全过程中抗生素和重金属抗性基因的赋存与转归特征研究进展[J].环境科学学报, 2015, 35(4):935-946. ZHANG Jun-ya, WEI Yuan-song, CHEN Mei-xue, et al. Occurrence and fate of antibiotic and heavy metal resistance genes in the total process of biological treatment and land application of animal manure:A review[J]. Acta Scientiae Circumstantiae, 2015, 35(4):935-946.
[9] De la lglesia R, Valenzuela-Heredia D, Pavissich J P, et al. Novel polymerase chain reaction primers for the specific detection of bacterial copper P-type ATPases gene sequences in environmental isolates and metagenomic DNA[J]. Letters in Applied Microbiology, 2010, 50(6):552-562.
[10] Kolpin D W, Furlong E T, Meyer M T, et al. Pharmaceuticals, hormones, and other organic wastewater contaminants in U.S. streams, 1999-2000:A national reconnaissance[J]. Environmental Science & Technology, 2002, 36(6):1202-1211.
[11] Gao P, He S, Huang S L, et al. Impacts of coexisting antibiotics, antibacterial residues, and heavy metals on the occurrence of ery -thromycin resistancegenesinurbanwastewater[J].Applied Microbiology andBiotechnology, 2015, 99(9):3971-3980.
[12] Stepanauskas R, Glenn T C, Jagoe C H, et al. Coselection for microbial resistance to metals and antibiotics in freshwater microcosms[J]. Environmental Microbiology, 2006, 8(9):1510-1514.
[13] Berg J, Thorsen K M, Holm P E, et al. Cu exposure under field conditions coselects for antibiotic resistance as determined by a novel cultivation-independent bacterial community tolerance assay[J]. Environ Sci Technol, 2010, 44(22):8724-8728.
[14] Knapp C W, McCluskey S M, Singh B K, et al. Antibiotic resistance gene abundances correlate with metal and geochemical conditions in Archived Scottish soils[J]. Plos One, 2011, 6(11): e 27300.
[15] Berg J, Tom-Peterson A, Nybroe O. Copper amendment of agricultural soil selects for bacterial antibiotic resistance in the field[J]. Letters in Applied Microbiology, 2005, 40:146-151.
[16] Wardwell L H, Jude B A, Moody J P, et al. Co-selection of mercury and antibiotic resistance in sphagnum core samples dating back 2000 years [J]. Geomicrobiology Journal, 2009, 26(4):238-247.
[17] Alonso A, Sanchez P, Martinez J L. Environmental selection of antibiotic resistance genes[J]. Environmental Microbiology, 2001, 3(1):1-9.
[18] Ji X L, Shen Q H, Liu F, et al. Antibiotic resistance gene abundances associated with antibiotics and heavy metals in animal manures and agricultural soils adjacent to feedlots in Shanghai, China[J]. Journal of Hazardous Materials, 2012, 235:178-185.
[19] McKinney C W, Loftin K A, Meyer M T, et al. Tet and sul antibiotic resistance genes in livestock lagoons of various operation type, configuration, and antibiotic occurrence[J]. Environmental Science & Technology, 2010, 44(16):6102-6109.
[20] Lu Z H, Na G S, Gao H, et al. Fate of sulfonamide resistance genes in estuary environment and effect of anthropogenic activities[J]. Science of the Total Environment, 2015, 527:429-438.
[21] Icgen B, Yilmaz F. Co-occurrence of antibiotic and heavy metal resistance in Kizilirmak river isolates[J]. Bulletin of Environmental Contamination and Toxicology, 2014, 93(6):735-743.
[22] Devarajan N, Laffite A, Graham N D, et al. Accumulation of clinically relevant antibiotic-resistance genes, bacterial load, and metals in freshwater lake sediments in Central Europe[J]. Environmental Science & Technology, 2015, 49(11):6528-6537.
[23] Wu D, Huang Z T, Yang K, et al. Relationships between antibiotics and antibiotic resistance gene levels in municipal solid waste leachates in Shanghai, China[J]. Environmental Science & Technology, 2015, 49 (7):4122-4128.
[24] Alonso A, Sanchez P, Martinez J L. Stenotrophomonas maltophilia D457R contains a cluster of genes from gram-positive bacteria involved in antibiotic and heavy metal resistance[J]. Antimicrobial Agents and Chemotherapy, 2000, 44(7):1778-1782.
[25] Ahmad I, Hayat S, Ahmad A, et al. Metal and antibiotic resistance traits in Bradyrhizobium sp.(cajanus)isloated from soil receiving oil refinery wastewater[J]. World Journal of Microbiology & Biotechnology, 2001, 17(4):379-384.
[26] Rasmussen L D, Sorensen S J. The effect of longterm exposure to mercury on the bacterial community in marine sediment[J]. Current Microbiology, 1998, 36:291-297.
[27] Davison J. Genetic exchange between bacteria in the environment[J]. Plasmid, 1999, 42:73-91.
[28] Klasen H J. A historical review of the use of silver in the treatment of burns[J]. Burns, 2000, 26(2):131-138.
[29] McHugh G L, Moellering R C, Hopkins C C, et al. Salmonella typhimurium resistant to silver nitrate, chloramphenicol, and ampicillin [J]. The Lancent, 1975, 305(7901):235-240.
[30] Summers A O, Wireman J, Vimy M J, et al. Mercury released from dental "Silver" fillings provokes an increase in mercury- and antibioticresistant bacteria in oral and intestinal floras of primates[J]. Antimicrobial Agents and Chemotherapy, 1993, 37(4):825-834.
[31] Edlund C, Bjorkman L, Ekstrand J, et al. Resistance of the normal human microflora to mercury and antimicrobials after exposure to mercury from dental amalgam fillings[J]. Clinical Infectious Diseases, 1995, 22:944-950.
[32] Davis I J, Richards H, Mullany P. Isolation of silver- and antibioticresistant enterobacter cloacae from teeth[J]. Oral Microbiology and Immunology, 2005, 20(3):191-194.
[33] Hasman H, Aarestrup F M. TcrB, a gene conferring transferable copper resistance in Enterococcus faecium:Occurrence, transferability, and linkage to macrolide and glycopeptide resistance[J]. Antimicrobial A-gents and Chemotherapy, 2002, 46(5):1410-1416.
[34] McIntosh D, Cunningham M, Ji B, et al. Transferable, multiple antibiotic and mercury resistance in Atlantic Canadian isolates of Aeromonas salmonicida subsp. salmonicoda is associated with carriage of an IncA/ C plasmid similar to the Salmonella enterica plasmid pSN254[J]. Journal of Antimicrobial Chemotherapy, 2008, 61(6):1221-1228.
[35] Wright M S, Baker-Austin C, Lindell A H, et al. Influence of industrial contamination on mobile genetic element:Class 1 integron abundance and gene cassette structure in aquatic bacterial communities[J]. The ISME Journal, 2008, 2(4):417-428.
[36] Rosewarne C P, Pettigrove V, Stokes H W, et al. Class 1 integrons in benthic bacterial communities:Abundance, association with Tn402-like transposition modules and evidence for coselection with heavymetal resistance[J]. FEMS Microbiology Ecology, 2010, 72:35-46.
[37] Zhang X X, Zhang T, Zhang M, et al. Characterization and quantification of class 1 integrons and associated gene cassettes in sewage treatment plants[J]. Environmental Biotechnology, 2009, 82:1169-1177.
[38]杨凤霞,毛大庆,罗义,等.环境中抗生素抗性基因的水平传播扩散[J].应用生态学报, 2013, 24(10):2993-3002. YANG Feng-xia, MAO Da-qing, LUO Yi, et al. Horizontal transfer of antibiotic resistance genes in the environment[J]. Chinese Journal of Applied Ecology, 2013, 24(10):2993-3002.
[39] Chapman J S. Disinfectant resistance mechanisms, cross-resistance, and co-resistance[J]. International Biodeterioration & Biodegradation, 2003, 51:271-276.
[40] Aendekerk S, Ghysels B, Cornelis P, et al. Characterization of a new efflux pump, MexGHI-OpmD, from Pseudomonas aeruginosa that confers resistance to vanadium[J]. Microbiology, 2002, 148(8):2371-2381.
[41] Gillings M, Boucher Y, Labbate M, et al. The evolution of Class 1 integrons and the rise of antibiotic resistance[J]. Journal of Bacteriology, 2008, 190(14):5095-5100.
[42] Stokes H W, Nesbo C L, Holley M, et al. Class 1 integrons potentially predating the association with Tn402-like transposition genes are present in a sediment microbial community[J]. Journal of Bacteriology, 2006, 188(16):5722-5730.
[43] Hayashi S, Abe M, Kimoto M, et al. The DsbA-DsbB disulfide bond formation system of Burkhholderia cepacia is involved in the production of protease and alkaline phosphatase, motility, metal resistance, and multi-drug resistance[J]. Microbiology and Immunology, 2000, 44 (1):41-50.
[44] Hernandez A, Mellado R P, Martinez J L. Metal accumulation and vanadium-induced multidrug resistance by environmental isolates of Escherichia hermannii and Enterobacter cloacae [J]. Applied and Environmental Microbiology, 1998, 64(11):4317-4320.
[45]郝艳华,张维,陈明.细菌双组分系统的研究进展[J].中国农业科技导报, 2012, 14(2):67-72. HAO Yan-hua, ZHANG Wei, CHEN Ming. Research progress on two component system of bacteria[J]. Journal of Agricultural Science and Technology, 2012, 14(2):67-72.
[46] Mizuno T. Compilation of all genes encoding two-component phosphotransfer signal transducers in the genome of Escherichia coli[J]. DNA Research, 1997, 4(2):161-168.
[47] West A H, Stock A M. Histidine kinases and response regulator proteins in two-component signaling systems[J]. Trends in Biochemical Sciences, 2001, 26(6):369-376.
[48] Wuichet K, Cantwell B J, Zhulin I B. Evolution and phyletic distribution of two-component signal transduction systems[J]. Current Opinion in Microbiology, 2010, 13(2):219-225.
[49] Bourret R B, Silversmith R E. Two-component signal transduction[J]. Current Opinion in Microbiology, 2010, 13(2):113-115.
[50] Baker-Austin C, Wright M S, Stepanauskas R, et al. Co-selection of antibiotic and metal resistance[J]. Trents in Microbiology, 2006, 14(4):176-182.
[51] Harrison J J, Tremaroli V, Stan M A, et al. Chromosomal antibioxidant genes have metal ion-specific roles as determinants of bacterial metal tolerance[J]. Environmental Microbiology, 2009, 11(10):2491-2509.
[52] Caille O, Rossier C, Perron K. A copper-activated two-component system interacts with zinc and imipenem resistance in pseudomonas aeruginosa[J]. Journal of Bacteriology, 2007, 189(13):4561-4568.
[53] Lee L J, Barrett J A, Poole R K. Genome-wide transcriptional response of chemostat-cultured Escherichia coli to zinc[J]. Journal of Bacteriology, 2005, 187(3):1124-1134.
[54] Nakajima H, Kobayashi K, Kobayashi M, et al. Overexpression of the robA gene increases organic solvent tolerance and multiple antibiotic and heavy metal ion resistance in Escherichia coli[J]. Applied and environmental microbiology, 1995, 61(6):2302-2307.
[55] Flemming H C, Wingender J. The biofilm matrix[J]. Nature Reviews Microbiology, 2010, 8(9):623-633.
[56] Zhang L, Mah T F. Involvement of a novel efflux system in biofilm-specificresistancetoantibiotics[J].Journal of Bacteriology, 2008, 190(13):4447-4452.
[57] Liao M, Xie X M. Effect of heavy metals on substrate utilization pattern, biomass, and activity of microbial communities in a reclaimed mining wasteland of red soil area[J]. Ecotoxicology and Environmental Safety, 2007, 66(2):217-223.
[58] Girvan M S, Bullimore J, Ball A S, et al. Responses of active bacterial and fungal communities in soils under winter wheat to different fertilizer and pesticide regimens[J]. Applied and Environmental Microbiology, 2004, 70(5):2692-2701.
[59] Ranjard L, Echairi A, Nowak V, et al. Field and microcosm experiments to evaluate the effects of agricultural Cu treatment on the density and genetic structure of microbial communities in two different soils[J]. FEMS Microbiological Ecology, 2006, 58(2):303-315.
[60] Novo A, Andre S, Viana P, et al. Antibiotic resistance, antimicrobialresidues and bacterial community composition in urban wastewater[J]. Water Research, 2013, 47(5):1875-1887.
[61] Forsberg K J, Patel S, Gibson M K, et al. Bacterial phylogeny structures soil resistomes across habitats[J]. Nature, 2014, 509(7502):612-615.
[62] Tom-Petersen A, Leser T D, Marsh T L, et al. Effects of copper amendment on the bacterial community in agricultural soil analyzed by the TRFLP technique[J]. FEMS Microbiology Ecology, 2003, 46(1):53-62.
[63]郑加玉.养殖废水中抗生素及抗性基因在垂直流人工湿地中的去除[D].哈尔滨:东北林业大学, 2013. ZHENG Jia-yu. Elimination of veterinary antibiotics and antibiotic resistance genes from swine wastewater in the vertical flow constructed wetlands[D]. Harbin:Northeast Forestry University, 2013.
[64] Castiglioni S, Bagnati R, Fanelli R, et al. Removal of pharmaceuticals in sewage treatment plants in Italy[J]. Environmental Science & Technology, 2006, 40(1):357-363.
[65] Rivera-Utrilla J, Gomez-Pacheco C V, Sanchez-Polo M, et al. Tetracycline removal from water by adsorption/bioadsorption on activated carbons and sludge-derived adsorbents[J]. Journal of Environmental Management, 2013, 131:16-24.
[66] Yang Y, Li B, Ju F, et al. Exploring variation of antibiotic resistance genes in activated sludge over a four-year period through a metagenomic approach[J]. Environmental Science & Technology, 2013, 47 (18):10197-10205.
[67]黄圣琳,何势,魏欣,等.污水处理厂中四环素类抗生素残留及其抗性基因污染特征研究进展[J].化工进展, 2015, 34(6):1779-1785. HUANG Sheng-lin, HE Shi, WEI Xin, et al. Pollution characteristics of tetrarycline residues and tetrarycline resistance genes in sewage treatment plants:A review[J]. Chemical Industry and Engineering Progress, 2015, 34(6):1779-1785.
[68]温丽华,许燕滨,周艳,等.重金属胁迫对Pseudomonas alcaligenes LH7抗生素抗性的影响[J].生态学报, 2015, 35(10):3411-3418. WEN Li-hua, XU Yan-bin, ZHOU Yan, et al. Effects of heavy metal stress on antibiotics resistance of Pseudomonas alcaligenes LH7[J]. Acta Ecologica Sinica, 2015, 35(10):3411-3418.
[69]宋晨仪,尹大强.四环素光催化降解特性与选择性研究[J].环境科学, 2014, 35(2):619-625. SONG Chen-yi, YIN Da-qiang. Characteristics and selectivity of photocatalytic-degradation of tetracycline hydrochloride[J]. Environmental Science, 2014, 35(2):619-625.
[70] Schlusener M P, Bester K. Persistence of antibiotics such as macrolides, tiamulin and salinomycin in soil[J]. Environmental Pollution, 2006, 143(3):565-571.
[71] Watanabe N, Harter T H, Bergamaschi B A, et al. Environmental occurrence and shallow ground water detection of the antibiotic monensin from dairy farms[J]. Journal of Environmental Quality, 2008, 37(5):S78-S85.
[72] Pei R T, Cha J, Carlson K H, et al. Response of antibiotic resistance genes(ARG)to biological treatment in dairy lagoon water[J]. Environmental Science & Technology, 2007, 41(14):5108-5113.
[73] Toes A C M, Daleke M H, Kuenen J G, et al. Expression of copA and cusAin shewanella during copper stress[J]. Microbiology, 2008, 154 (9):2709-2718.
Co-selection mechanisms of bacterial resistance to heavy metals and antibiotics
ZHANG Jia-qi1, XU Yan2, LUO Yi2, MAO Da-qing1*
(1.College of Environmental Science and Engineering, Tianjin University, Tianjin 300072, China; 2.College of Environmental Science and Engineering, Ministry of Education Key Laboratory of Pollution Processes and Environmental Criteria, Nankai University, Tianjin 300071, China)
Abstract:Excessive uses of antibiotics and increasingly serious pollution of heavy metals are continuously contributing to the emergence, persistence and transmission of antibiotic resistant genes(ARGs)in the environment. This paper summarized the latest research on antibiotic resistance and heavy metals resistance, and illustrated pollution status of ARGs and heavy-metal resistance genes and its coincidence mechanisms in bacteria in various environment. Bacteria were resistant not only to heavy metals, but also to multiple antibiotics in heavymetal contaminated environment. Heavy-metal stresses caused elevated abundance of ARGs in natural rivers, sewage plants and livestock farms. Under heavy metal stresses, bacteria obtained antibiotic resistance through(a)co-resistance mechanism, i.e. antibiotic and heavymetal resistance genes located on a same vector such as plasmids, integrons, and transposons;(b)cross-resistance mechanism such as efflux pumps taking effect when stimulated by heavy metals;(c)co-regulation mechanism, which relies on a two-component system and takes effect when stimulated by heavy metals. Finally, heavy metal stresses in the environment were promoting bacteria to obtain antibiotic resistance and to propagate ARGs among and in bacterial community. Nowadays, human beings are facing not only a serious pollution of antibiotics and heavy metals but a challenge of ARGs pollution.
Keywords:antibiotic resistance;heavy-metal resistance; co-selection resistance; co-selection resistance mechanism
*通信作者:毛大庆E-mail:mao@tju.edu.cn
作者简介:张佳奇(1991—),男,湖北赤壁人,硕士生,研究方向为分子生态。E-mail:958759870@qq.com
基金项目:国家自然科学基金项目(31270542,41473085)
收稿日期:2015-10-13
中图分类号:X503
文献标志码:A
文章编号:1672-2043(2016)03-0409-10
doi:10.11654/jaes.2016.03.001
