基因表达谱大数据在抗癌药物研发中的应用
2016-04-08刘阳白卉伯晓晨
刘阳,白卉,伯晓晨
军事医学科学院放射与辐射医学研究所,北京 100850
基因表达谱大数据在抗癌药物研发中的应用
刘阳,白卉,伯晓晨
军事医学科学院放射与辐射医学研究所,北京 100850
作为功能基因组学中重要的组成部分,基因表达谱在生物学、医学和药物研发等多个领域发挥着重要作用。特别是随着精准医疗概念的提出,整合包括基因表达谱在内的多组学数据用于个性化医疗是未来的发展趋势。从抗癌药物研发背景出发,介绍了基于细胞扰动表达谱的大数据分析方法,并且总结了其在抗癌药物研发中的应用。
基因表达谱;关联图谱;整合网络细胞印记库;抗癌药物研发
1 引言
受中国老龄化程度加深和社会环境变迁,特别是环境污染等的影响,国内恶性肿瘤患者的发病率和死亡率逐年增加。恶性肿瘤逐渐成为最主要的公共卫生问题。根据2015年国家癌症中心在癌症研究顶级期刊《CA: A Cancer Journal for Clinicians》上发布的统计数据,2000—2011年,中国癌症发病率逐年增长(男0.2%,女2.2%),给癌症的治疗和抗癌药物的研发带来极大挑战[1]。未来癌症很可能代替心脑血管疾病成为人类健康的首要杀手。
目前,癌症的常规治疗仍然以手术、化疗和放疗3种手段互相辅助进行。这3种治疗方法没有先后主次之分,临床上往往根据癌症类型和其严重程度采用不同的治疗程序。新出现的靶向治疗药物和免疫治疗药物逐渐在癌症治疗中发挥重要作用。在全球药物研发市场中,抗癌药物研发一直是药物研究的热点,其研发量约占全球药物研发总量的1/3。虽然中途停研项目多、最终上市率低,但是各国政府和大型制药公司在抗癌药物研发的资金投入却逐年增长。2005—2014年的10年时间里,美国食品与药品管理局(FDA)批准了超过60种抗癌药物。目前全球抗癌药物销量最高的是单抗类靶向药物,如罗氏的美罗华(利妥昔单抗,Rituxan)、安维汀(贝伐珠单抗,Avastin)和赫赛汀(曲妥珠单抗,Herceptin)等。
癌症如何产生目前并没有定论,人们广泛接受的理论是突变累积学说[2],即癌症的形成是由基因突变带来的选择性生长优势造成的,正常细胞从良性增殖发展到恶性癌细胞会经历一系列的突变。其他学说则尝试从代谢和进化等角度解释癌症的发生过程[3]。Hanahan等人[4]总结了癌症的十大特征,其中给抗癌药物研发带来巨大挑战的是其高度基因异质性(tumor heterogeneity)[5-7],即其基因突变往往因时间点、肿瘤位置、病人而异。这种异质性造成对于同一种癌症类型,也需要不同的药物进行合理用药和精准治疗的现状。癌症异质性是造成抗癌药物药效不一致、研发成功率低的重要原因。
2015年1月,美国总统奥巴马提出精准医疗(precision medicine initiative)计划[8],该计划强调准确、准时、共享、个体化四大要素,要求根据病人的基因组学、表观遗传学以及临床病例等信息合理用药,提供最佳的联合方案和合适的药物剂量,减少药物的毒副作用,在取得最佳治疗方案的同时将费用降到最低,这在解决癌症异质性造成的疗效差异方面迈出了重要一步。美国精准医学计划由两个目标组成:近期目标关注癌症领域,着力解决精准癌症医学中的难题,如难以解释的耐药性问题、癌症基因组异质性、癌症反应监测与复发风险评估、组合用药问题[8];长期目标扩展到人类各种疾病的治疗,包括疾病风险评估、阐释疾病机制、预测各种疾病的最佳治疗方法等。
受美国“精准医疗”计划的影响,中国也在大力推进精准医疗[9]。2016年中国全面启动精准医疗计划,其规模比美国的精准医疗计划大很多,预计在2030年前将投入600亿元用于该计划。2016年3月,中华人民共和国科学技术部发布了中国精准医疗白皮书。“精准医学研究”专项的总体目标如下。
● 以我国常见疾病及若干流行率相对较高的罕见病为切入点,协同攻关,构建百万人以上的健康队列和重大疾病专病队列。
● 建立生物医学大数据共享平台,突破组学临床应用技术和生物医学大数据分析技术,建立疾病预警、诊断、治疗与疗效评价的生物标志物、靶标、制剂的实验和分析技术体系。
● 以临床应用为导向,形成重大疾病的风险评估、预测预警、早期筛查、分型分类、个体化治疗、疗效和安全性预测及监控等精准防诊治方案和临床决策系统,形成可用于精准医学应用全过程的生物医学大数据参考咨询、分析判断、快速计算和精准决策的系列分类应用技术平台。
● 建设中国人群典型疾病精准医学临床方案的示范、应用和推广体系,推动一批精准治疗药物和分子检测技术产品进入国家医保目录,为显著提升人口健康水平、减少无效和过度医疗、避免有害医疗、遏制医疗费用支出快速增长提供科技支撑,使精准医学成为经济社会发展新的增长点。
不同国家常见癌症类型和人群本身基因组存在差异,因此中国的精准医学计划有自己的侧重点。但是中国的精准医学计划也存在大量挑战,如国内医院研究转化效率低、临床样本共享难、自主研发药物少等问题。中国精准医学计划的实施还需要科研机构的广泛合作与国家的统筹协调。
随着高通量测序技术和全基因组DNA芯片检测技术的日臻成熟及成本大幅降低,与癌症相关的各种组学数据(基因组拷贝数变异、基因突变、甲基化数据以及转录组、蛋白质组、代谢组数据)正在快速积累。癌症相关多组学公共大数据为整合分析癌症异质性及其对抗癌药物影响、进而指导精准药物治疗提供了良好的数据基础。整合患者癌症细胞/组织的基因组、转录组、细胞药物敏感性基因组等多组学数据以及基因通路信息,指导合理(组合)用药,是实现个性化治疗并最终提高治疗有效性的重要方法和手段。
基因表达谱是后基因组时代最先发展起来的高通量技术[10],其能够从整体水平研究基因的结构与功能,揭示特定的生物学过程和疾病发生发展的分子机制,是目前识别和描述与特定表型或者扰动相关的基因表达模式最为有效、准确和高重复性的数据类型,在生物医学领域被广泛应用[11]。基因表达谱的典型应用包括:构建可能介导某个重要的生物过程或者疾病发生发展相关的基因调控网络[12-14];识别与特定表型或者扰动相关的基因表达模式;鉴定疾病诊断和预后判断的基因标志物[15];寻找潜在的药靶,用于疾病治疗;药物重定位等。
2 细胞扰动表达谱大数据分析方法
细胞扰动基因表达谱是在给定扰动的条件下(如基因沉默、基因过表达、不同浓度小分子化合物作用),采取多时间点测量细胞全基因组的转录水平表达量。2006年Lamb等人[16]首先测定了5个人类癌症细胞系上1309种药物作用后的全基因组表达谱,并利用此数据构建了关联图谱(connectivity map,CMap)数据集。CMap采用基因集富集分析(gene set enrichment analysis,GSEA)方法[17],通过与组织细胞在不同生理、病理、药物作用等条件下的基因表达谱印记进行对比分析,建立基因—疾病—药物之间的联系,成功用于药物重定位和发现药物新作用方式等[18]。该方法由3部分组成:参考基因表达谱数据库、查询表达谱印记、模式匹配算法。图1为CMap方法的原理。
图1左半部分为基因表达谱印记,代表诱导产生的目标细胞状态;中间部分为大量扰动的人体细胞系表达谱构成的参考数据库,采用模式匹配算法对查询的表达谱印记进行打分,评估富集程度和方向;右半部分根据关联打分排列扰动药物,排名置顶(正相关)和置底(负相关)的药物与查询的目标细胞状态表达谱印记建立联系。

图1 关联图谱原理[16]
参考基因表达谱数据库包含不同生物状态下的基因表达谱,如不同的扰动类型、细胞系、作用时间、浓度条件等。查询表达谱印记是由用户提供的基因集,用于描述特定的生物状态。根据研究目的的不同,其可以来自人体样本、疾病动物模型、小分子药物作用的细胞和组织等,一般通过相关表达谱数据进行差异表达分析得到。目前存在一些整理的表达谱印记集可用于CMap分析,如分子印记数据库(molecular signatures database,MSigDB)[17]是GSEA(基因集富集分析)附带的表达谱印记数据集。基因印记数据库(gene signature database,GeneSigDB)[19]是通过文献整理得到的表达谱印记数据库,最新第四版数据包含从1604篇文章整理得到的3515个表达谱印记。这些数据集可以提供不同生物状态下的参考表达谱印记,为相关领域的科学研究提供了极大便利。模式匹配算法用于评估查询表达谱印记与参考表达谱之间的相关性,GSEA基于Kolmogorov-Smirnov统计[20],是一种非参数、基于秩序列的模式匹配算法。
然而,CMap项目仅在5个细胞系测量了1300多种药物作用后的基因表达谱,作为比对关联的参考数据集,其数据规模大大限制了其潜在应用。2010年,美国国立卫生研究院(NIH)资助布罗德研究所(Broad Institute)启动了整合网络细胞印记库项目(Library of Integrated Network-based Cellular Signatures,LINCS)。该项目旨在在更多不同细胞系上测量不同扰动类型引起的转录组变化,获得更大规模的表达谱数据进行关联网络分析,推动新的生物医学发现。值得注意的是,LINCS项目表达谱测定采用L1000[21]技术,该技术考虑基因表达之间的相关性,将需要测量的基因数量大幅减少,达到控制成本的目的。L1000挑选978个标志基因进行测量,进一步通过构建模型外推出其他基因的表达量。实验数据表明这些标志基因的表达可以代表全基因组80%的信息。虽然存在一定误差,但是该技术可以将成本减小到全基因组基因芯片成本的1%,使得测量大规模基因表达谱可以实现。在降低成本、保证数据质量的前提下已获得并公开包括基因沉默(RNA interference)、基因过表达(overexpression)、小分子化合物(small-molecule compounds)这三大类扰动类型下不同种类细胞系的基因表达谱。截至2016年2月,LINCS计划已公开的表达谱数据规模已超过百万,涉及77个细胞系、4372个基因沉默、3124个基因过表达、20413个小分子化合物的多因素扰动的表达谱,且其主要的细胞扰动表达谱均是在不同癌症细胞系上测量获得的,主要包括乳腺癌(MCF7)、结肠癌(HT29)、肝癌(HEPG2)、肺癌(A549、HCC515)、黑色素瘤(A375)和前列腺癌(PC3、VCAP)。这些癌症细胞系的选择代表了美国每年新增癌症病例最多的癌症类型。
除了不同的扰动类型,LINCS细胞扰动表达谱还涉及不同序列(基因沉默)、不同时间、不同剂量(小分子化合物)的影响,如何综合考虑不同因素的作用,从数据产出有价值的信息,是LINCS细胞扰动表达谱大数据处理的难点。另外L1000的线性外推模型本身存在误差,其数据预处理方法也值得深入研究。Chen等人[22]采用深度学习算法挖掘基因表达之间的非线性关系,并宣称其将预测的基因表达量精度提高了15.33%。
在LINCS表达谱数据计算中,考虑到数据规模和关联图谱本身的计算复杂度,需要借助大规模并行计算才能在短时间内得到计算结果。事实上,笔者在天河高性能计算机上完成了相关计算流程的构建。同时也要看到,虽然LINCS细胞扰动大数据相比其他领域的大数据规模较小,但是其已经远远超出传统表达谱数据产出的规模,有效利用该数据对于生物医学和转化医学研究都具有重大潜在价值。
3 表达谱大数据在抗癌药物研发中的应用
与传统的基于单一药效指标的高通量筛选不同,关联图谱指导下的药物发现是建立在疾病表型(disease phenotype)特异的表达谱印记基础上的大规模关联筛选。具体来说,在抗癌药物研发中,其一般方法是测定多个临床癌症样本的表达谱并提取一组特征性差异表达的基因集合,即表达谱印记,然后在公共的大规模药物扰动细胞基因表达谱数据库中进行比对和关联分析,筛选出与之高度负相关的候选药物,进而通过体内外实验进行抗癌活性评价。
经过近10年的发展,以CMap为先导的表达谱印记比对及关联网络分析方法在基础和临床、系统生物学和药物发现之间架起了桥梁,尤其在抗癌药物发现领域得到了诸多成功应用[18]。Wang等人[23]根据基因芯片表达谱数据得到与肺腺癌高度相关的表达谱印记,然后采用该方法发现HSP90抑制剂、PPAR拮抗剂和PI3K抑制剂这几类药物与肺腺癌特异性表达谱印记高度负相关,并且实验证实了17-AAG(一种HSP90抑制剂)能够抑制肺腺癌细胞生长。Siavelis等人[24]整合了5个阿尔兹海默症相关的表达谱数据,采用CMap、SPIEDw[25]、sscMap[26]、LINCS-L10004个分析工具分别对药物作用细胞表达谱数据集进行分析,发现了27个阿尔兹海默症的潜在治疗药物,进一步的信号通路和蛋白质相互作用网络分析发现这些药物可能是通过影响表皮生长因子受体相关的通路来发挥作用的。表1列出了2013年至今CMap和LINCS数据应用于抗癌药物研发的应用案例,更多研究应用可以参考[18]。
LINCS细胞表达谱数据除了可以直接利用小分子化合物表达谱进行抗癌药物预测外,还可以结合基因沉默和过表达扰动的表达谱数据寻找潜在的抗癌药物药靶、确定小分子化合物的潜在靶点、构建某个生物过程的基因调控网络确定关键基因
等,可以从多角度服务于抗癌药物的研发,是转化医学研究的重要数据资源。
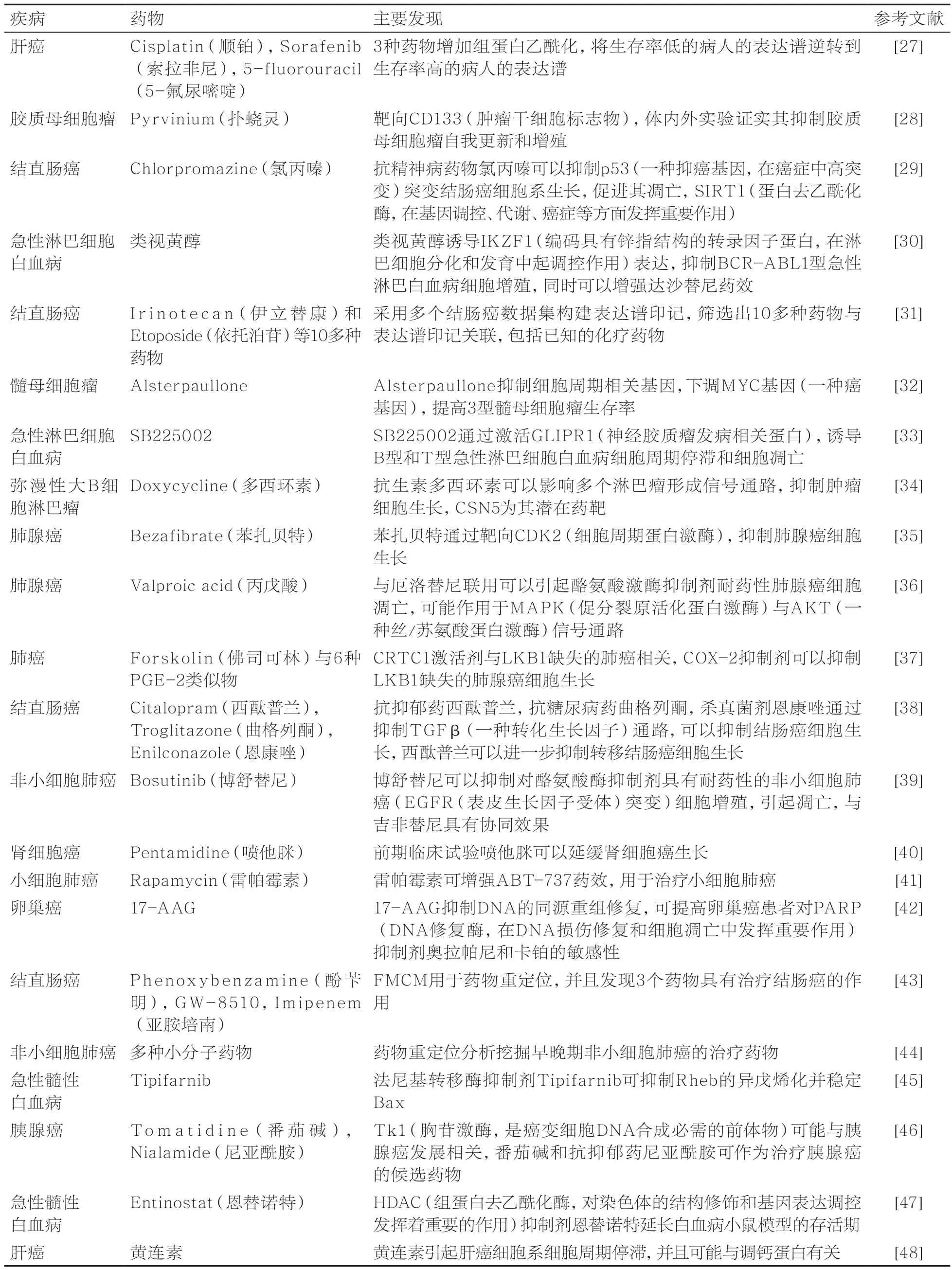
表1 CMap与LINCS用于抗癌药物研发研究汇总(2012年至今)
4 结束语
作为一种有效的技术方法,基因表达谱在抗癌药物的早期发现、后期研发以及临床用药指导都发挥着重要作用[49]。而随着测序技术的发展,转录组测序成本不断降低,将使得在成本可控的条件下测量大量不同条件下的基因表达谱数据可行,结合最新的基因表达谱分析方法,将为药物发现提供更为广阔的空间。实际上,LINCS第二阶段将大大增加数据类型和数据规模,随着相关研究的深入,其价值将逐渐凸显。
随着多组学时代的来临,融合包括基因表达谱在内的多组学数据是未来的发展趋势,而其应用不仅仅局限于疾病分型和药物重定位方向。结合最新的人工智能技术,其已经服务于精准医疗,如国际商业机器公司(IBM)的沃森(Waston)系统,可以将患者的组学数据与构建的疾病知识库比对,根据患者的遗传学特征提供最优的治疗方案。而组学数据的进一步增长,将进一步提高该系统的准确度。目前,美国和加拿大的多家肿瘤中心以及国内的多家医院已经部署沃森系统,并应用于癌症诊断与治疗中。
当然,基因表达谱分析也存在一定的不足,如样本少、特征太多引起的统计检验有效性问题。而LINCS的细胞扰动表达谱数据的复杂度和误差对数据分析方法提出更多挑战,如何降低预测结果的假阳性和假阴性,为临床医学研究提供更多有价值的信息需要进一步研究。相信随着转录组测序技术和相关数据分析技术的持续发展,未来基因表达谱在抗癌药物研发中将发挥更加重要的作用。
[1] CHEN W, ZHENG R, BAADE P D, et al.Cancer statistics in China, 2015[J].CA Cancer J Clin, 2016, 66(1): 115-132.
[2] NORDLING C O.A new theory on cancerinducing mechanism[J].British Journal of Cancer, 1953.7(1): 68-72.
[3] ROZHOK A I, DEGREGORI J.Toward an evolutionary model of cancer: Considering the mechanisms that govern the fate of somatic mutations[J].Proceedings of the National Academy of Sciences of the United States of America, 2015, 112(29): 8914-8921.
[4] HANAHA N D, WEINBERG R A.Hallmarks of cancer: the next generation[J].Cell, 2011, 144(5): 646-674.
[5] FISHER R, PUSZTAI L, SWANTON C.Cancer heterogeneity: implications for targeted therapeutics[J].British Journal of Cancer, 2013, 108(3): 479-485.
[6] SWANTON C.Intratumor heterogeneity: evolution through space and time[J].Cancer Research, 2012, 72(19): 4875-4882.
[7] YATES L R, CAMPBELL P J.Evolution of the cancer genome[J].Nature Reviews Genetics, 2012, 13(11): 795-806.
[8] COLLINS F S, H Varmus.A new initiative on precision medicine[J].New England Journal of Medicine, 2015, 372(9): 793-795.
[9] CYRANOSKI D.China embraces precision medicine on a massive scale[J].Nature, 2016, 529(7584): 9-10.
[10] LOCKHART D J, WINZELER E A.Genomics, gene expression and DNA arrays[J].Nature, 2000, 405(6788): 827-836.
[11] GLIGORIJEVIC V, MALOD-DOGNIN N, PRZULJ N.Integrative methods for analysing big data in precision medicine[J].Proteomics, 2015, 16(5): 7.
[12] MARGOLIN A A, NEMENMAN I, BASSO K,et al.ARACNE: an algorithm for the reconstruction of gene regulatory networks in a mammalian cellular context[J].BMC Bioinformatics, 2006, 7(2): S7.
[13] LEFEBVRE C, RAJBHANDARI P, ALVAREZ M J, et al.A human B-cell interactome identifies MYB and FOXM1 as master regulators of proliferation in germinal centers[J].Molecular Systems Biology, 2010, 6(1): 377.
[14] CHEN J C, ALVAREZ M, TALOS F, et al.Identification of causal genetic drivers of human disease through systems-level analysis of regulatory networks[J].Cell, 2014, 159(2): 402-414.
[15] D E H M E R M, M U E L L E R L A, EMMERTSTREIB F.Quantitative network measures as biomarkers for classifying prostate cancer disease states: a systems approach to diagnostic biomarkers[J].PLos One, 2013, 8(11): e77602.
[16] LAMB J, CRAWFORD E D, PECK D, et al.The connectivity map: using geneexpression signatures to connect small molecules, genes, and disease[J].Science, 2006, 313(5795): 1929-1935.
[17] SUBRAMANIAN A, TAMAYO P, MOOTHA V K, et al.Gene set enrichment analysis: a knowledge-based approach for interpreting genome-wide expression profiles[J].Proceedings of the National Academy of Sciences, 2005, 102(43): 15545-15550.
[18] QU X A, RAJPAL D K.Applications of connectivity map in drug discovery and development[J].Drug Discovery Today, 2012, 17(23-24): 1289-1298.
[19] CULHANE A C, SCHRÖDER M S, SU LTA NA R, et al.G ene Si g DB: a manually curated database and resource for analysis of gene expression signatures[J].Nucleic Acids Research, 2012, 40(Database Issue): 1060-1066.
[20] HOLLANDER M, WOLFE D A, CHICKEN E.Nonparametric statistical methods[M]// Biostatistics and Microbiology: A Survival Manual.New York: Springer New York, 2013: 121-162.
[21] PECK D, CRAWFORD E D, ROSS K N, et al.A method for high-throughput gene expression signature analysis[J].Genome Biology, 2006, 7(7): 91-96.
[22] CHEN Y, LI Y, NARAYAN R, et al.Gene expression inference with deep learning[J].Bioinformatics, 2016, 32(12): 1832-1839.
[23] WA NG G, Y E Y, YA NG X, et al.Expression-based in silico screening of candidate therapeutic compounds for lung adenocarcinoma[J].PLoS One, 2011, 6(1): e14573.
[24] SIAVELIS J C, BOURDAKOU M M, ATHANASIADIS E I, et al.Bioinformatics methods in drug repurposing for Alzheimer's disease[J].Briefings in Bioinformatics, 2015: 1-14.
[25] WILLIAMS G.SPIEDw: a searchable platform-independent expression database web tool[J].BMC Genomics, 2013, 14(1): 1-6.
[26] ZHANG S D, GANT T W.sscMap: an extensible Java application for connecting small-molecule drugs using gene-expression signatures[J].BMC Bioinformatics, 2009, 10(1): 236.
[27] GILLET J P, ANDERSEN J B, MADIGAN J P, et al.A gene expression signature associated with overall survival in patients with hepatocellular carcinoma suggests a new treatment strategy[J].Molecular Pharmacology, 2016, 89(2): 263-272.
[28] VENUGOPAL C, HALLETT R, VORA P, et al.Pyrvinium targets CD133 in human glioblastoma brain tumor-initiating cells[J].Clinical Cancer Research, 2015, 21(23): 5324-5337.
[29] LEE W Y, LEE W T, CHENG C H, et al.Repositioning antipsychotic chlorpromazine for treating colorectal cancer by inhibiting sirtuin 1[J].Oncotarget, 2015, 6(29): 27580-27595.
[30] CHURCHMAN M L, LOW J, QU C, et al.Efficacy of retinoids in IKZF1-mutated BCR-ABL1 acute lymphoblastic leukemia[J].Cancer Cell, 2015, 28(3): 343-356.
[31] WEN Q, O'REILLY P, DUNNE P D, et al.Connectivity mapping using a combined gene signature from multiple colorectal cancer datasets identified candidate drugs including existing chemotherapies[J].Bmc Systems Biology, 2015, 9(5): 1-11.
[32] FARIA C C, SAMEER A, MACK S C, et al.Identification of alsterpaullone as a novel small molecule inhibitor to target group 3 medulloblastoma[J].Oncotarget, 2015, 6(25): 21718-21729.
[33] VASCONCELLOS J F D, LEAL P C, BHASIN M K, et al.SB225002 induces cell death and cell cycle arrest in acute lymphoblastic leukemia cells through the activation of GLIPR1[J].Plos One, 2015, 10(8): 19.
[34] PULVINO M, CHEN L, OLEKSYNET D, et al.Inhibition of COP9-signalosome (CSN) deneddylating activity and tumor growth of diffuse large B-cell lymphomas by doxycycline[J].Oncotarget, 2015, 6(17): 14796-14813.
[35] LIU X, YANG X, CHEN X, et al.Expression profiling identifies bezafibrate as potential therapeutic drug for lung adenocarcinoma[J].Journal of Cancer, 2015, 6(12): 1214-1221.
[36] ZHUO W, ZHANG L, ZHU Y, et al.Valproic acid, an inhibitor of class I histone deacetylases, reverses acquired erlotinibresistance of lung adenocarcinoma cells: a connectivity mapping analysis and an experimental study[J].American Journal of Cancer Research, 2015, 5(7): 2202-2211.
[37] CAO C, GAO R, ZHANG M, et al.Role of LKB1-CRTC1 on glycosylated COX-2 and response to COX-2 inhibition in lung cancer[J].Jnci Journal of the National Cancer Institute, 2015, 107(1): 358.
[38] VAN N V, SCHÖLCH S, ISKAR M, et al.Novel drug candidates for the treatment of metastatic colorectal cancer through global inverse gene-expression profiling[J].Cancer Research, 2014, 74(20): 5690-5699.
[39] KIM J, VASU V T, MISHRA R, et al.Bioinformatics-driven discovery of rational combination for overcoming EGFR-mutant lung cancer resistance to EGFR therapy[J].Bioinformatics, 2014, 30(17): 2393-2398.
[40] ZERBINI L F, BHASIN M K, VASCONCELLOS J F D, et al.C omput ational repositioning and preclinical validation of pentamidine for renal cell cancer[J].Molecular Cancer Therapeutics, 2014, 13(7): 1929-1941.
[41] GARDNER E E, CONNIS N, POIRIER J T, et al.Rapamycin rescues ABT-737 efficacy in small cell lung cancer[J].Cancer Research, 2014, 74(10): 2846-2856.
[42] CHOI Y E, BATTELLI C, WATSON J, et al.Sublethal concentrations of 17-AAG suppress homologous recombination DNA repair and enhance sensitivity to carboplatin and olaparib in HR proficient ovarian cancer cells[J].Oncotarget, 2014, 5(9): 2678-2687.
[43] CHUNG F H, CHIANG Y R, TSENG A L, et al.Functional module connectivity map (FMCM): a framework for searching repurposed drug compounds for systems treatment of cancer and an application to colorectal adenocarcinoma[J].PLos One, 2014, 9(1): e86299.
[44] HUANG C H, CHANG M H, LIN Y J, et al.Drug repositioning discovery for early- and late-stage non-small-cell lung cancer[J].Biomed Research International, 2014(1): 309-322.
[45] DING H, MCDONALD J S, YUN S, et al.Farnesyltransferase inhibitor tipifarnib inhibits Rheb prenylation and stabilizes Bax in acute myelogenous leukemia cells[J].Haematologica, 2014, 99(1): 60-69.
[46] WANG B S, LIU Z, SUN S L, et al.Identification of genes and candidate agents associated with pancreatic cancer[J].Tumour Biology: the Journal of the International Society for Oncodevelopmental Biology and Medicine, 2014, 35(1): 81-88.
[47] RAMSEY J M, KETTYLE L M J, SHARPE D J, et al.Entinostat prevents leukemia maintenance in a collaborating oncogenedependent model of cytogenetically normal acute myeloid leukemia[J].Stem Cells, 2013, 31(7): 1434-1445.
[48] MA C, TANG K, LIU Q, et al.Calmodulin as a potential target by which berberine induces cell cycle arrest in human hepatoma Bel7402 cells[J].Chemical Biology & Drug Design, 2013, 81(6): 775-783.
[49] BAI J P, ALEKSEYENKO A V, S TATN I KOV A, et al.St rat eg ic applications of gene expression: from drug discovery/development to bedside[J].Aaps Journal, 2013, 15(2): 427-437.
Application of large scale gene expression profiles in anticancer drug development
LIU Yang, BAI Hui, BO Xiaochen
Institute of Radiation Medicine, Academy of Military Medical Sciences, Beijing 100850, China
As an important part of functional genomics, gene expression profile plays an important role in many fields, such as biology, medicine and drug discovery.With the advent of precision medicine, integration of multi-omics data including gene expression profile data for personalized health care is becoming the trend of future medicine.The advances of anticancer drug development were introduced firstly, and then the methods for perturbational gene expression profile analysis were illustrated, especially connectivity map idea.Finally applications of these data in anticancer drug development were summarized.
gene expression profile, connectivity map, library of integrated network-based cellular signatures, anticancer drug development
Q811.4,R9
A
10.11959/j.issn.2096-0271.2016051

刘阳(1988-),男,军事医学科学院放射与辐射医学研究所博士生,主要研究方向为生物医学大数据挖掘。

白卉(1982-),女,军事医学科学院放射与辐射医学研究所博士后,第四军医大学药理学博士,主要研究方向为反义抗菌核酸药物和网络药理学。先后承担各类国家和军队科研项目10余项,发表SCI期刊论文10篇,参编英文专业书稿3部,现为中国药理学会会员。

伯晓晨(1973-),男,军事医学科学院放射与辐射医学研究所研究员、博士生导师,主要研究方向为生物医学大数据处理与挖掘技术。先后承担各类国家和军队科研项目20余项,发表SCI 期刊论文50篇。现兼任北京自动化学会理事、中国药理学会网络药理学专业委员会常务委员、军队转化医学委员会委员、中国生化与分子生物学会青年委员。
2016-06-29
国家自然科学基金资助项目(No.U1435222)
Foundation Item: The National Natural Science Foundation of China (No.U1435222)
