面向国际的生命组学大数据管理体系建设
2016-04-07赵文明张思思唐碧霞陈婷婷郝丽丽桑健李茹姣肖景发章张
赵文明,张思思,唐碧霞,陈婷婷,郝丽丽,桑健,李茹姣,肖景发,章张
中国科学院北京基因组研究所生命与健康大数据中心,北京 100101
面向国际的生命组学大数据管理体系建设
赵文明,张思思,唐碧霞,陈婷婷,郝丽丽,桑健,李茹姣,肖景发,章张
中国科学院北京基因组研究所生命与健康大数据中心,北京 100101
组学数据是生命科学研究中的一类极其重要的大数据,特别是二代测序技术的发展推动了组学大数据的爆炸式增长。通过借鉴国际数据中心建设的成功经验,分析国内组学产出数据及数据库建设、数据管理现状及应用需求,构建了面向国际的生命组学大数据管理体系,涵盖组学原始序列归档库、基因组序列数据库、基因表达数据库、基因组变异数据库、DNA甲基化数据库系统等,初步形成中国组学数据资源的存储、共享与应用体系。
组学数据;大数据;数据共享;生物信息学;基因组
1 引言
从1999年我国正式加入人类基因组计划并承担1%测序任务以来,我国的基因组学研究已经历了16年,实现了我国基因组学乃至生命科学研究的跨越式发展,尤其是2005年前后第二代基因组测序技术的面世,推动了整个领域的飞速发展。然而,与本领域飞速发展极不相称的是,我国基因组学研究过程中产生的数据资源却没能很好地收集、存储与管理。10多年来,以郝柏林院士为代表的许多有识之士,呼吁建立国家生物医学信息中心,但至今杳无音信。国内的科学家甚至有受国际期刊及国际生物信息数据库“绑架”的被动局面,要向国际数据库提交数据才能发表文章。从科学的严谨性、公开性及结果经得起考验的角度讲,这种做法无可厚非,但从国家基础科研数据积累、方便我国科学家使用的角度来讲,急需建立我国自己的生物信息数据库系统。以国际相关数据库为借鉴对象及发展目标,跨越层层障碍,构建我国组学数据存储与管理体系,或许能为中国的组学数据积累与发展奠定基础。
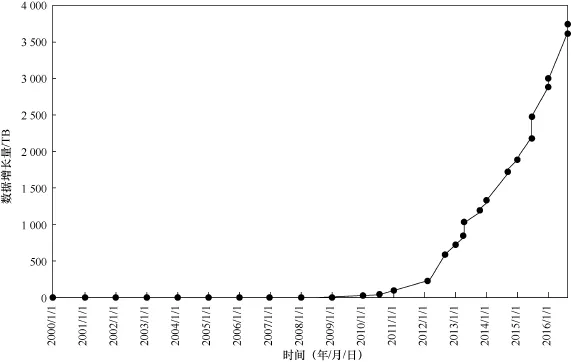
图1 NCBI-SRA数据增长情况①
2 国际组学数据库发展状况
近10多年来,生命科学研究进入了以生物组学数据研究为基础,以人口健康为主要落脚点,加速向临床医学转化并不断取得重大突破的高速发展时代,新的生物学技术和方法的出现,引发了生物学数据和信息的新一轮爆炸性增长,美国国立生物技术信息中心(Natioal Center of Biotechnology Institute,NCBI)—原始序列档案库(Sequence Read Archive, SRA)的数据增长情况如图1所示。近年来,1 000美元完成个人基因组重测序的目标已经实现[1,2],精准医学研究计划已在世界各国陆续启动,复杂而多层次的生物组学数据和信息的年产出量已经达到PB量级。生物医学和生物技术的发展和创新性研究越来越依赖于对生物信息数据的积累、管理、共享以及应用。
国际上已有的3个生物信息中心,收录了几乎所有科研用途产出的组学大数据,即美国国立生物技术信息中心、欧洲生物信息研究所(EMBL European Bioinformatics Institute,EMBL-EBI)和日本DNA数据库中心(DNA Data Bank of Japan,DDBJ)。这3个中心专注于存储、管理和共享科学家们产出的基因组序列信息,并形成了国际核苷酸序列数据库共享联盟(International Nucleotide Sequence Database Collaboration,INSDC)。时至今日,这3个中心已经在全球范围以遥遥领先之势形成组学数据资源垄断的局面。
2.1 美国国立生物技术信息中心
20世纪80年代后期,美国国会参议员Claude Pepper意识到计算机化信息处理方法对生物医学研究的重要性,倡导建立国家生物技术信息中心,NCBI因此于1988年成立,由美国国立卫生研究院(National Institute of Health,NIH)直接拨款资助。NCBI虽然在行政上隶属于美国国立医学图书馆(National Library of Medicine,NLM),但在业务与经费上相对独立。NCBI的主要任务就是利用计算机技术和国际互联网系统,收集、存储生物医学文献资料以及核酸、蛋白质等生物分子序列、结构等数据,开发生物信息应用软件和分析平台,为生物医学、生命科学和生物技术研究开发提供服务。
经过近30年的发展,NCBI已经成为全球数据资源最丰富的生物信息中心,主要数据及数据库覆盖了分子生物学、生物化学、遗传学、基因组学等多个方面,其中比较有特色的包括生物医学文献摘要数据库PubMed,核酸序列数据库GenBank,基因组、转录组、甲基化组等各种组学数据库以及二代测序原始序列档案库[3]。根据NCBI官方网站中公布的数据增长图显示②https://trace. ncbi.nlm.nih.gov,仅SRA数据库每年约有2 PB的数据增长量,即平均每天有超过5 TB的数据递交至SRA数据库。此外,NCBI还开发了一套功能强大的搜索引擎系统(Entrez),将NCBI内部数据库进行信息整合与汇总,免费为用户提供检索、浏览、下载和分析服务。
2.2 欧洲生物信息研究所
欧洲生物信息研究所是欧洲生物信息学研究和服务的中心,隶属于欧洲分子生物学实验室(European Molecular Biology Laboratory,EMBL)。EMBLEBI的前身是世界上第一个核苷酸序列数据库,即1980年位于德国海德堡的欧洲分子生物学实验室创建的核酸序列数据库—— EMBL[4]。1992年欧洲议会决定建立欧洲生物信息研究所,经费来源除欧盟各成员国外,也得到英国Wellcome基金会、美国国立卫生研究院、英国医学研究理事会等机构资助[5]。EMBL-EBI为欧洲及世界各地科学家提供公开、免费的生物信息资源,包括多个特色生物医学数据库和分析工具,其中最值得关注的是欧洲核苷酸档案库(European Nucleotide Archive,ENA)。
2.3 日本DNA数据库中心
日本DNA数据库中心是一个收集DNA序列的生物数据库[6,7],它位于日本静冈县的国家遗传学研究所(National Institute of Genetic,NIG)。DDBJ创立于1984年,1987年开始正式服务,并由日本国家遗传研究所维护和更新,目前仍是亚洲唯一的核苷酸序列数据库。DDBJ主要向研究者收集DNA序列信息并赋予其数据存取号,数据来源主要是日本的研究机构,它也可以接受来自任何其他国家科学家的数据。数据库通过环球网、匿名FTP、E-mail或Gopher方式为广大研究人员服务。
2.4 国际核苷酸序列数据库共享联盟
国际核苷酸序列数据库共享联盟③http://insdc.orgINSDC是由NCBI、EMBL-EBI和DDBJ共同发起并建立的一个国际性的核苷酸序列数据库共享联盟。联盟的成员约定了数据交换与共享的原则、数据的命名规范、交换的标准及数据交换的范围等,同时,坚持每日同步共享各数据库收集的信息(见表1),确保为全世界的科研工作者提供最新的核苷酸序列数据应用服务[8]。
3 国内组学数据及数据库系统发展现状
3.1 我国组学数据产出现状
纵观全球,组学数据大规模产出与发展是随着国际人类基因组研究计划而快速起步的,在2005年后,新一代测序技术(第二代测序技术)的出现及技术的不断革新、测序通量的提高与成本的大幅降低,将以基因组测序为主要手段的生命科学研究推向新的高潮,基因组、转录组等组学数据以井喷之势爆发,生命科学的研究进入组学大数据时代。
由于拥有世界最多的人口及丰富的生物样本资源,我国很快成为组学数据产出大国。据不完全统计,近几年,国内从事新一代高通量测序的相关企业蓬勃发展。据粗略估算,我国约有1 700台第二代高通量测序仪,绝大部分设备来源于美国的3家公司:Illumina测序仪约1 000台(其中Hiseq X10共8套),Thermo Fisher测序仪约600台,Pacific Biosciences约50台,其他约50台。设备主要集中在一线城市,如北京约460台,上海约450台,深圳约410台。这些设备若全负荷运转,组学数据年产出量高达10 PB。随着国家在人口健康领域的研究部署,越来越多的大型人群队列研究正在陆续建设,如“国家大型健康队列”,将要收集数十万甚至百万人群的健康数据。这些海量的数据将会成为未来医学模式转变的重要基础。
3.2 国内组学数据库建设情况
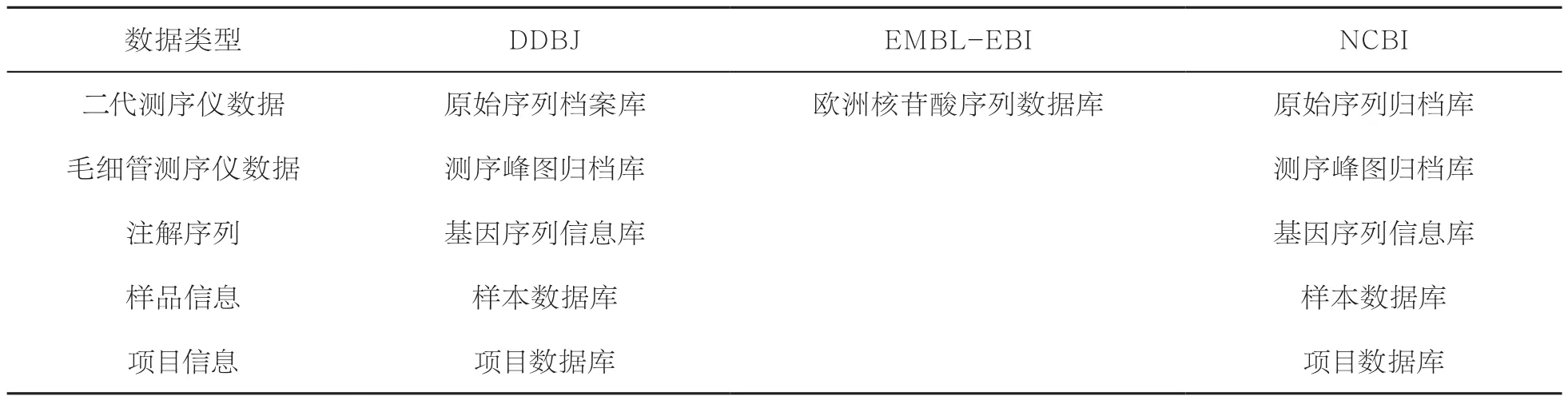
表1 INSDC成员数据交换内容
按照领域内对数据库类别的划分方法,组学数据库通常分为一级数据库、二级数据库[9]。一级数据库的数据直接来源于实验获得的原始数据,只是经过简单的归纳、整理和注释;二级数据库一般是指对原始生物分子数据库进行分类、整理的结果,是在一级数据库数据的深度加工和分析的基础之上形成的具有特定目的的数据库。一级数据库中的数据需要再分析和加工以获取更多信息,二级数据库可直接为科学家提供某些特定的信息。
中国组学数据库的建设更多是集中于二级数据库建设,一方面是由于二级数据库大部分是科研成果的直接展示形式,另一方面是二级数据库开发、运行和维护成本相对较低,课题组或实验室便可承担二级数据库的建设与维护。表2列出了从Database Commons④http://bigd. big.ac.cn/ databasecommons收录的自2005年开始由国内科研人员公开发表的公共数据库情况,从表2可以看出,国内二级组学数据资源已涵盖从DNA、RNA、蛋白质到表达、表型、药物等多种数据类型,呈现出多样化的数据资源情况。同时有一批数据库还广泛得到国际同行的认可,并具有较高的国际引用率(见表3)。
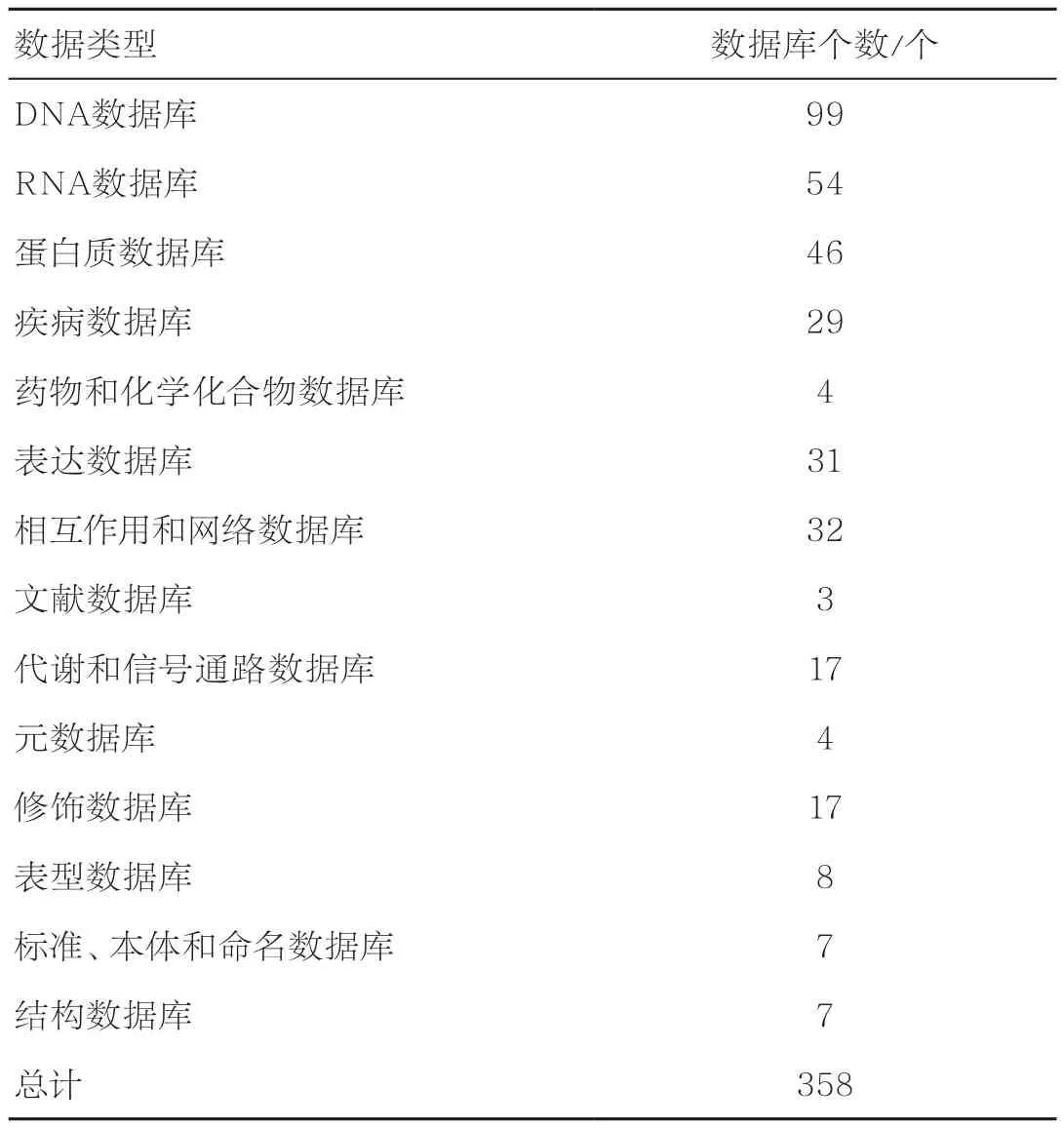
表2 国内组学二级数据库统计(数据截至2016年9月)
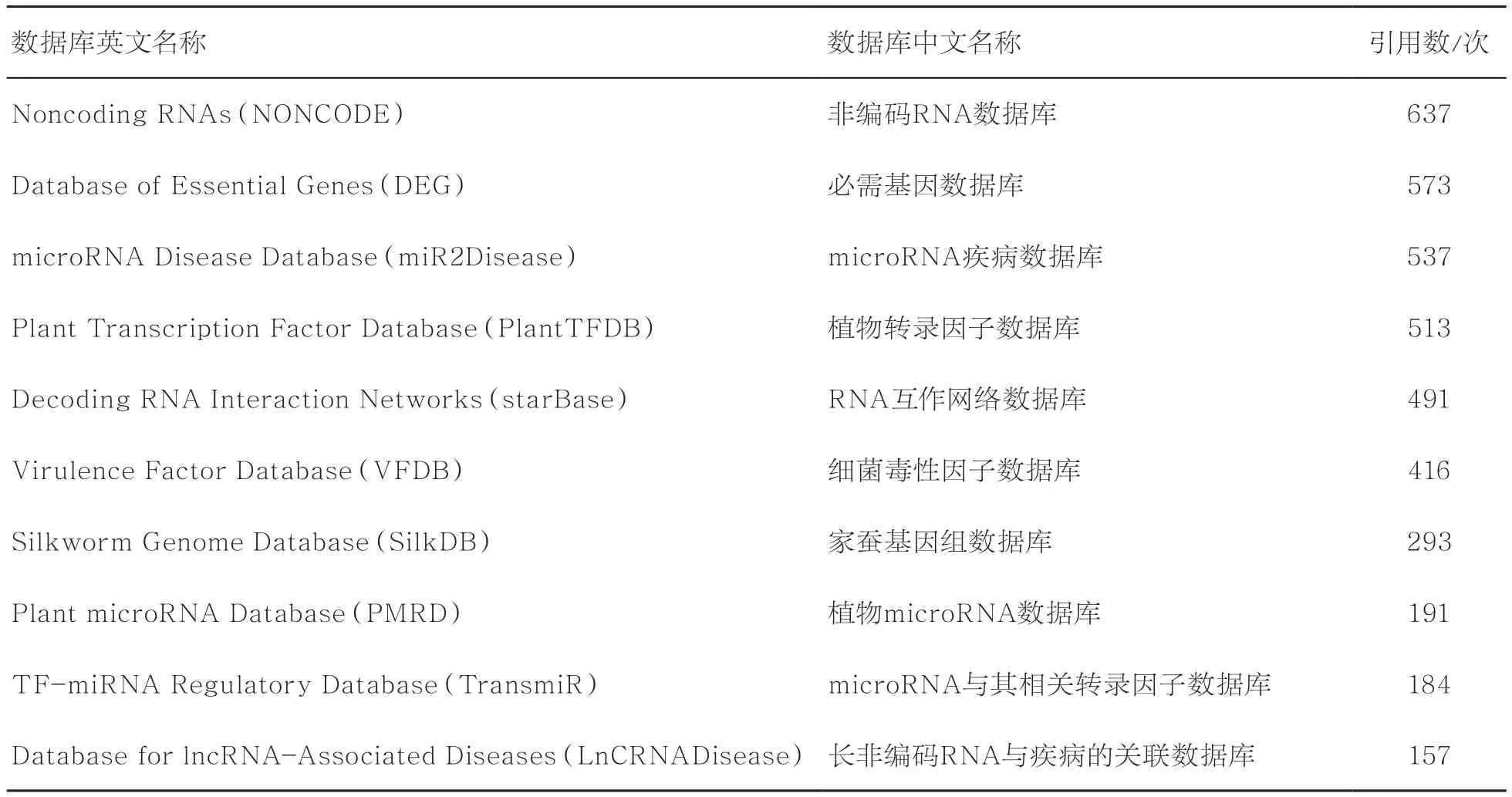
表3 国内组学二级数据库引用率前10位(数据截至2016年9月)
相对于蓬勃发展的二级数据库,国内在一级数据库系统建设方面相对较晚或较弱,主要原因是一级数据库建设工程大、周期长、耗资高、运行维护成本高,且缺乏相应的政策支持及稳定的经费投入。因此早在多年前,虽有一些成形的一级数据库系统,但其运行状况不佳,未能形成国内组学数据集中汇交、存储与共享体系,也没有获得国际及国内同行的认可。这一状况导致中国产出的绝大部分组学数据需要提交到NCBI、EMBLEBI或DDBJ等数据库平台进行管理和发布。从某种意义上讲,缺乏一级组学数据集中汇交、存储、管理与共享的数据库体系,使中国失去了对所产组学数据的管理权。
4 组学数据管理体系建设
为了改变国内组学数据被动外流的现状,积累中国产出的组学大数据,并为国内从事生命科学研究的科学家提供本地化的数据服务体系,中国科学院北京基因组研究所于2016年初成立“生命与健康大数据中心(Big Data Center,BIGD)”,旨在立足本地建立国际化的组学数据管理平台,提供组学数据汇交、共享、发布及数据应用服务。同时,构建了涵盖不同数据类型及应用方向的综合性的数据库系统,包括组学原始序列归档库(Genome Sequence Archive,GSA)系统、基因组序列数据库(Genome Warehouse,GWH)系统、基因表达数据库(Gene Expression Nebulas,GEN)系统、基因组变异数据库(Genome Variation Map,GVM)系统和DNA甲基化数据库(MethBank)系统,初步形成了继NCBI、EMBL-EBI、DDBJ国际数据库之后的第四个国际组学数据管理系统。
4.1 组学原始序列归档库
GSA⑤http://gsa.big. ac.cn是一个基因组数据的集中存储与数据管理数据库,存储数据包括基因组、转录组、表观组等其他组学原始测序数据。GSA接收常见的多种测序平台产出的原始数据,包括Illumina、PacBio SMRT、Complete Genomics等,并且除了原始测序数据,GSA也可接收二级分析的数据,如BAM、VCF格式的数据。
GSA的数据元素包括数据本体及其元数据,其中数据本体为数据文件或测序文件,以文件形式存储于文件系统;元数据为数据本体的描述信息,以记录的形式保存于数据库表中,包括“项目信息”“样本信息”“实验信息”“测序信息”。元数据按照由大到小的逻辑顺序,即从“项目”“样本”“实验”到“测序”,建立一对多的关联关系,确保信息的完整性。GSA中各元素的数据编码规范采用国际同行编码规则,并使用字母“C”表示中国,如PRJCA00001、SAMC00001等数据元素。GSA支持数据在线提交,也支持数据的离线复制,并针对每一个递交的数据项(包括数据本体和元数据)均具有内部审核的机制,从而确保数据质量。GSA为每一个用户递交的每一组数据分配唯一的存取号,且当数据为发布状态时,使这些数据在全球范围内公开可用。
GSA系统是一个面向国际的公共数据管理平台,可以长期保存科学家产出的原始测序数据,并可以帮助科学家实现数据的共享与发布,因此除中国之外,可接收来自世界各地的用户提交的数据本体和元数据。目前,GSA已经获得国际10余家期刊(包括PNAS、Cell Research等)的认可,并允许作为其刊发论文的数据存储与共享平台。
4.2 基因组序列数据库
基因组数据及其注释信息是从事某物种研究的最基本的信息,GWH⑥http://bigd.big. ac.cn/gwh则是一个综合性的收集、整理与展示基因组及其注释信息的数据库系统,该数据库系统建设的主要目标是为科学家提供一个方便、高效的数据检索、获取以及发布的平台。
GWH已收录了27个物种的基因组序列数据,涉及的数据内容既包含了丰富的序列资源(如基因序列、蛋白质序列、非编RNA序列、基因的位置及功能注释信息等),也包含了某一物种基因组的元信息概述(如染色体的大小和数目、拼接质量、所发表的论文信息等),既涵盖了与我国国民经济密切相关的经济物种(如水稻、家蚕、家鸡、鲤鱼、橡胶等),也包括了一些处在关键进化节点上的模式植物(如拟南芥、衣藻以及杨树)。GWH每年定期地搜集各个物种最新公布的转录组数据与蛋白质组数据,并通过统一的基因组注释流程对其进行基因组重注释,以期为生物学家提供更为可靠的基因注释信息。
4.3 基因表达数据库
GEN⑦http://bigd.big. ac.cn/gen的建设区别于NCBI、EMBLEBI对表达数据汇集和共享的模式,GEN主要以科学问题为导向,充分利用原始组学数据,汇总、挖掘、审编、整合出知识型的表达数据库,为科学研究人员提供数据、方法、信息与知识4个层面的内容。GEN将针对不同物种的特性,充分挖掘了解相应研究领域的共性科研问题和需求,将公共表达数据经过数据筛选、生物信息分析和数据审编等步骤,整理出物种、种属或特定类别群体的特异性表达库。
目前,GEN已经涵盖了基于二代测序的人、猪、小鼠、大鼠以及水稻的表达数据,基因在各种组织类型、环境状态与处理条件下的表达模式信息以及基于基本表达模式进一步分析与审编获得的知识性信息。随着对更多重要物种的表达数据的汇集与整理,同源基因在各物种中的表达进化等信息也会陆续被整合进来。未来,GEN系统将配合原始数据归档系统与基因组数据发布与展示系统,为科学研究人员提供更加多维全面的数据与信息源。
4.4 基因组变异数据库
GVM⑧http://bigd.big. ac.cn/gvm是一个以个体(物种)为单位收录其基因组中变异位点信息及其注释信息的共享平台,涉及的数据类型主要包括单核苷酸多态性(SNP)、小插入(small indel)或缺失片段。GVM提供多种数据利用功能,如用户在GVM平台上可以查看对应的物种信息、检索物种的变异数据、下载和提交变异数据;在变异检索页面中,通过同时设定多种检索条件,如位置信息、影响结果类型、关联基因信息、最小Allele频率,达到精确检索数据结果的目的。
GVM收录的数据主要来自于已公开发表的高粱、狼和狗以及水稻的SNP数据。其中,高粱的数据来源于48个个体,狼和狗的数据来源于78个个体,水稻的数据来源于5 152个个体。为了确保各个体变异数据标准的一致性,GVM 采用统一的标准注释流程对不同物种的变异数据进行处理和整理。
4.5 DNA甲基化数据库
MethBank⑨http://bigd.big. ac.cn/methbank是一个全基因组单碱基精度DNA甲基化的交互式数据库,允许用户检索和查询已有物种的全基因组单碱基甲基化分布、基因的甲基化水平分布、不同样本之间的差异甲基化区域、CpG岛、基因表达谱、特定基因或基因组区的遗传多态性等信息,并可以快速计算特定区间的甲基化水平。
目前,MethBank库整合了斑马鱼、小鼠、水稻、大豆、木薯、菜豆和番茄的高质量全基因组重亚硫酸氢盐测序甲基化图谱数据,提供了全基因组范围的甲基化水平概览,用户可以从数据库中直接获取所有搜集样本的基因的不同功能区域的不同序列模式的甲基化水平。未来,MethBank会持续升级并继续整合更多物种的高质量单碱基核苷酸甲基化组数据资源,为世界范围内的表观遗传和发育研究提供重要的资源储备。
5 结束语
组学大数据是国家重要的战略生物资源,科学家们产生的组学数据不仅仅是为了发表文章,更重要的是作为一种战略资源进行保护与再次利用,并充分发挥数据本身的价值。我国的组学数据产量较大,但数据存储量较少,导致我国的科学家使用数据时要跨过大西洋的海底线缆从美国下载,效率极低。要改变这种局面,不是一蹴而就的事情,也绝不是构建一套数据管理系统就能解决的问题。更重要的是获得更多国际期刊的认可并得到中国广大从事生命科学研究的科学家的认可,或者从某种程度上讲,也非常需要得到一些政府机构的认可与支持。因为只有这样,庞大的、耗资的数据库体系的开发与稳定运行才能持续,中国组学数据库建设的梦想才能实现。
致谢
在本文的撰写过程中,得到了北京大学罗静初教授的大力支持和帮助,在此表示真诚的感谢!同时感谢北京基因组研究所生命与健康大数据中心每一位成员的辛勤付出及对数据库系统建设做出的重要贡献!
[1] BENNETT S T, BARNES C, COX A, et al. Toward the $1000 human genome[J]. Future Medicine, 2005, 6(4): 373-382.
[2] HAYDEN C. Technology: the $1,000 genome[J]. Nature, 2014, 507(7492): 294-295.
[3] WHEELER D L, CHAPPEY C, LASH A E, et al. Database resources of the mational center for biotechnology information[J]. Nucleic Acids Research, 2015, 43(Database): D6- D17.
[4] STOESSER G, STERK P, TULI M, et al. The EMBL nucleotide sequence database[J]. Nucleic Acids Research, 1997, 25 (1): 7-13.
[5] COCHRANE G, AKHTAR R, ALDEBERT P, et al. Priorities for nucleotide trace, sequence and annotation data capture at the ensemble trace archive and the EMBL nucleotide sequence database[J]. Nucleic Acids Research, 2007, 36(Database): D5-D12.
[6] MASHIMA J, KODAMA Y, KOSUGE T, et al. DNA data bank of Japan (DDBJ) progress report [J]. Nucleic Acids Research, 2016, 44 (Database): D51-D57.
[7] TATENO Y, IMANISHI T, MIYAZAKI S, et al. DNA data bank of Japan (DDBJ) for genome scale research in life science[J]. Nucleic Acids Research, 2002, 30(1): 27-30.
[8] COCHRANE G, KARSCH-MIZRACHI I, TAKAGI T. The international nucleotide sequence database collaboration[J]. Nucleic Acids Research, 2016, 44(Database): D48-D50.
[9] WAN Y H, HE L M. Bioinformatics database resources on internet[J]. Journal of the China Society for Scientific and Technical Information, 2002, 21(4): 497-512.
Constructing the international database management system for omics big data
ZHAO Wenming, ZHANG Sisi, TANG Bixia, CHEN Tingting, HAO Lili, SANG Jian, LI Rujiao, XIAO Jingfa, ZHANG Zhang
Big Data Center in Beijing Institute of Genomics, Chinese Academy of Sciences, Beijing 100101, China
Omics data are the important elements of the biosciences, in recent years, with the rapid progress of the next generation sequencing (NGS) technology, the omics data show the explosive increasement. Drawing on the successful experiences from the international data centers, and considering the domestic requirements, lots of databases including genome sequencing archive, genome warehouse, gene expression nebulas, genome variation map, DNA methylation databank were constructed. These databases constitute the domestic omics data resources and provide the free service for all the scientists for the data storing, sharing and management.
omics data, big data, data sharing, bioinformatics, genome
Q-9
A
10.11959/j.issn.2096-0271.2016065

赵文明(1977-),男,中国科学院北京基因组研究所生命与健康大数据中心高级工程师,主要研究方向为生物组学大数据整合与挖掘、高性能计算。

张思思(1985-),女,博士,中国科学院北京基因组研究所生命与健康大数据中心工程师,主要研究方向为组学大数据整合与审编。

唐碧霞(1984-),女,中国科学院北京基因组研究所生命与健康大数据中心工程师,主要研究方向为三维基因组的可视化。

陈婷婷(1986-),女,中国科学院北京基因组研究所生命与健康大数据中心工程师,主要研究方向为组学大数据整合与审编。

郝丽丽(1983-),女,博士,中国科学院北京基因组研究所生命与健康大数据中心助理研究员,主要研究方向为转录组数据整合与挖掘分析。

桑健(1989-),男,中国科学院北京基因组研究所生命与健康大数据中心博士生,主要研究方向为组学大数据整合与挖掘。

李茹姣(1976-),女,博士,中国科学院北京基因组研究所生命与健康大数据中心高级工程师,主要研究方向为表观遗传学相关大数据整合和深度挖掘。

肖景发(1973-),男,博士,中国科学院北京基因组研究所生命与健康大数据中心研究员,主要研究方向为生命与健康相关组学大数据整合和深度挖掘。

章张(1980-),男,博士,中国科学院北京基因组研究所生命与健康大数据中心研究员,主要研究方向为分子进化建模和选择压力检测、序列组分建模分析。
2016-09-26
中国科学院先导基金资助项目(No.XDB13040500,No.XDA08020102);国家高技术研究发展计划(“863”计划)基金资助项目(No.2015AA020108);中国科学院关键技术人才基金资助项目
Foundation Items:Strategic Priority Research Program of the Chinese Academy of Sciences (No.XDB13040500, No.XDA08020102), The National High Technology Research and Development Program of China(863 Program) (No.2015AA020108), Key Technology Talent Program of the Chinese Academy of Sciences
