乌梁素海湖滨湿地硫酸盐还原菌种群分布
2016-03-24刘建丽武琳慧内蒙古大学环境与资源学院呼和浩特010021
刘建丽,赵 吉,武琳慧(内蒙古大学环境与资源学院,呼和浩特010021)
乌梁素海湖滨湿地硫酸盐还原菌种群分布
刘建丽,赵吉,武琳慧*
(内蒙古大学环境与资源学院,呼和浩特010021)
摘要:采用克隆文库构建和实时定量PCR技术,对乌梁素海湖滨湿地小河口陆向演化芦苇沉积物、湖滨沼泽水葱沉积物、湖滨碱蓬盐碱化草甸土壤、湖岸白刺荒漠化土壤等环境中硫酸盐还原菌(Sulfate reducting bacteria,SRB)多样性及其丰度进行了研究。结果显示:上述沉积物和土壤环境中硫酸盐还原菌功能基因apsA丰度存在显著差异,每克土壤中基因丰度(拷贝数)分别为1.34×107、8.07×105、5.27×106和2.37×104;系统发育树分析表明,硫酸盐还原菌属于变形杆菌门(Proteobacteria)中的五个科,分别为Acidithiobacillales、Desulfurella、Chromatiales、Desulfovibrionaceae和Desulfobacteriaceae,其中Chromatiales、Desulfovibrionaceae和Desulfobacteriaceae为优势菌群。沉积物和土壤环境中硫酸盐还原菌群落及apsA基因丰度存在显著差异,硫酸盐还原菌多样性与土壤总盐量、含水率及硫酸盐浓度之间存在显著相关(P<0.05)关系。
关键词:湖滨湿地;硫酸盐还原菌;群落结构;功能基因apsA
刘建丽,赵吉,武琳慧.乌梁素海湖滨湿地硫酸盐还原菌种群分布[J].农业环境科学学报, 2016, 35(2):358-363.
乌梁素海作为典型的浅草型湖泊湿地,在调节周围气候、维持生物多样性等方面起着至关重要的作用。近年来大量农灌退水和工业及生活污水的排入,导致水体呈现重度富营养化[1]。大型水生植物异常生长,湖泊周围土壤盐碱化现象严重,而且湖泊沼泽化趋势明显,水体生境受到破坏,水生态系统良性循环受阻[2]。面对乌梁素海湿地严重的水体富营养化及土壤盐碱化等问题,探究该环境中微生物群落结构变化及其多样性是目前研究热点之一。
硫酸盐还原菌(Sulfate reducting bacteria,SRB)的存在代表了厌氧环境的发生,其种群数量的多少一定程度上反映了厌氧程度的高低。水体富营养化及土壤盐碱化极易导致水体深层溶解氧降低和土壤结块,形成还原状态有机物无机化不完全,硫酸盐还原形成硫化氢气体,因此硫酸盐还原菌种群数量多少在一定程度上有指示作用[3]。随着生物学技术的不断发展,研究发现SRB形态各异、营养类型多样,在厌氧及微氧环境中都可以将硫化物作为电子受体进行新陈代谢[4-6]。但乌梁素海湖滨湿地环境中SRB功能菌群的分布和基因丰度目前知之甚少,硫酸盐还原菌功能基因apsA能够编码腺苷酰硫酸还原酶的α-亚基(αsubunit of adenosine -5' -phosphosulfate reductase reductase),该酶能将SO2-4还原为SO2-3[7],在硫循环过程中起主导作用。故本研究通过克隆文库构建和实时定量PCR技术分子手段,对乌梁素海湖滨湿地陆向分布梯度上SRB群落结构及功能基因apsA丰度进行研究,探究在沉积物和土壤环境中的SRB的多样性分布及丰度变化,旨在为研究乌梁素海湿地生态与环境提供理论基础,为湿地生态系统生物评估及生物修复提供数据基础。
1 材料与方法
1.1研究区概况及样品采集
研究区乌梁素海(108。43'~108。57'E,40。46'~41。03'N)是内蒙古高原干旱区最典型的浅草型湖泊,它的形成与黄河主流改道有关,是国际关注的湿地生态系统生物多样性的重点保护区之一。研究区属典型的温带大陆性季风气候,年均降雨量为215 mm,平均气温6.6℃,水生植物共6科6属11种,植被类型包括:挺水植物芦苇(Phragmites communis)、宽叶香蒲(Typhaphalatifolia);沉水植物菹草(Potamogetonerispus)、龙须眼子菜(Potamogeton pectinatus)和藻类;陆生植物以碱蓬和白刺为主[1]。小河口作为接纳来自总排干、通济渠和八排干等农灌退水和工业及生活污水的污水口,水体富营养化程度明显,且周边土壤盐碱化及荒漠化现象严重。因此,以乌梁素海湿地小河口为研究区域,按照该区域沉积物、盐碱化及荒漠化的地域分布特征设置一条陆向分布样带,选择具有代表性的4种类型植物土壤:湖中芦苇沉积物(108。54'649″E,40。46'367″N)、湖滨沼泽水葱(Scirpus validus Vahl)沉积物(108。56'692″E,40。456'861″N)、湖滨碱蓬(Suaeda salsa)盐碱化草甸土壤(108。56'682″E,40。56'863″N)和湖岸白刺(Nitraria tangutorum)荒漠化土壤(108。56'682″E,40。56'863″N),分别采集周围表层土样(0~20 cm),相应标记为A1、A2、A3、A4,每个样点按照“S”型分别设置三个平行样。样点分布示意图如图1所示。土壤理化性质的测定主要是根据《土壤农化分析实验教材》[8]和《土壤·水·植物理化分析教程》[9]。
样品内外做好标记后用封口袋密封,立即用冰袋保鲜,带回室内分别混匀并分装,分别于-20℃和-80℃保存。-20℃样品用于土壤理化性质的测定,-80℃样品用于分子生物学分析。
图1乌梁素海采样点示意图
Figure 1 Sampling locations in Wuliangsuhai
1.2样品DNA的提取及PCR扩增
1.3硫酸盐还原菌克隆文库的构建
本研究中选取APS7-F和APS8-R引物对硫酸盐还原菌功能基因apsA的保守片段进行扩增,采用菌落PCR方法鉴定阳性克隆子。将鉴定好的阳性克隆子接种于LB/AMP液体培养基中,37℃过夜培养。菌液保存,送华大基因测序,引物为M13R Forward Primer和M13F Reverse Prime(r
1.4实时定量分析
Illumina EcoTM实时荧光定量PCR仪用于定量分析,引物[11]RH1-aps-F:5'-CGCGAAGACCTKATCTTCGAC-3'和RH2-aps-R:5'-ATCATGATCTGCCAGCGGCCGGA-3'用于样品的定量扩增,片段大小为190 bp。SYBRRPremix Ex TaqTMⅡ(TRAN,BEIJING)试剂用于定量分析。经优化后的SYBRRGreenⅠ检测反应体系:SYBRRPremix Ex TaqTMⅡ(2×)10.0 μL,上下游引物(10 mmol·L-1)各0.8 μL,DNA模板0.5 μL,加ddH2O至20 μL。反应条件:预变性95℃30 s,35个循环PCR反应扩增(95℃15 s,63℃31 s)。溶解曲线分析条件为95℃15 s,63℃15 s,95℃15 s。
1.5数据处理及分析
本研究获得的序列均已通过Sequenin软件整理并提交,获取登录号为:KP337769-KP337883。
运用DOTUR软件对序列在相似性97%的基础上进行可分类单元操作(Operational taxonomic units,简称OTU),同时获得Rarefaction等指数。选取代表性的克隆序列,与NCBI在线数据库(http://www.ncbi. nlm.nih.gov/)进行比对,选择覆盖度和相似度都比较高的序列作为参考序列,通过Cluster W对齐序列,再用MEGA 5.2软件构建系统发育树。
2 结果与分析
2.1基质环境基本性质
四个样点土壤性质各不相同,数据表明(表1):有机碳、总氮和总磷的含量在A3样地含量最低;水溶性盐总量按照演化梯度总体上呈上升趋势;四个样点土壤pH值均大于8.0,偏碱性;硫酸根的含量介于3.80~7.49 g·kg-1之间;土壤含水率在陆向分布样带上呈下降趋势。

表1 土壤样品的基本理化性质Table 1 Physical and chemical properties of soil samples
2.2硫酸盐还原菌群落多样性
阳性克隆子测序后,共获得了121条有效序列。在给定的98%的相似水平上将所有获得的序列通过Dotur软件来进行OTU的分类,共分为34个OTUs。Rarefaction分析表明,本研究所获得SRB的生物信息在种水平上,还不能完全地代表真实环境中硫酸盐还原菌的多样性,说明这些样品中还包含着更多的有待于进一步挖掘的硫酸盐还原菌,增加取样量可能会得到更大的多样性,见图2。Shannon和Simpon多样性指数表明,克隆文库A3的多样性最高,而A4的多样性最低。

图2 apsA序列Rarefaction曲线Figure 2 Rarefaction curves of apsA gene sequence
本研究所得到的SRB apsA序列分布于5个科(图3):Chromatiaceae、Acidithiobacillales、Desulfovibrionaceae、Desulfobacteriaceae和Desulfurella,Desulfobacteriaceae科是主要的分布菌群。Acidithiobacillales属于Gammaproteobacteria(γ-变形菌纲),其他四个科都属于Deltaproteobacteria(δ-变形杆菌纲)。然而还有大部分序列没有高度相似的可培养SRB apsA序列作参考,推测各样点中可能存在新的硫酸盐还原菌种群。
2.3硫酸盐还原菌功能基因apsA丰度
采用实时定量PCR技术对四个样地中SRB功能基因apsA丰度进行检测(图4),结果显示四个样地(A1、A2、A3、A4)中硫酸盐还原菌功能基因apsA丰度存在显著差异,每克土样中apsA拷贝数分别为:1.34×107、8.07×105、5.27×106和2.37×104。因为apsA是硫酸盐还原酶α-亚基的编码基因,在每个细胞内仅有一个拷贝[12],所以本实验测定的aspA丰度能够确切地反映实际环境中硫酸盐还原菌的丰度。apsA丰度的最大值出现在A1,其后依次为A3、A2和A4。

图3 基于硫酸盐还原菌功能基因apsA的系统进化树Figure 3 Phylogenetic tree based on functional apsA gene of sulfate-reducing bacteria
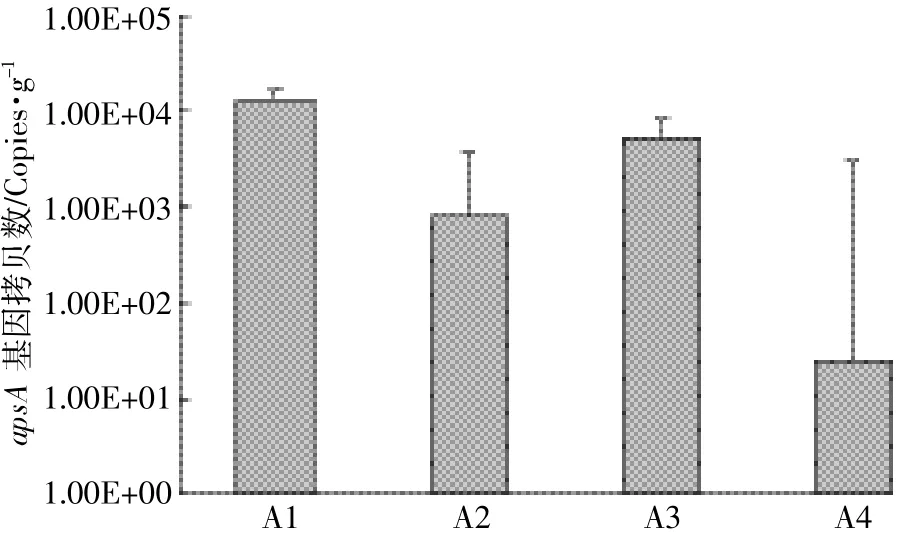
图4 定量PCR分析功能基因apsA拷贝数Figure 4 Copy number of functional gene apsA in four samples
2.4乌梁素海湿地土壤理化性质与SRB多样性的相关性分析
以SRB在优势菌群的相对丰度为参数,通过SPSS软件计算Pearson相关系数,分析土壤理化性质对SRB多样性的影响(表2)。发现Desulfobacteriaceae科的相对丰度与土壤总盐量及含水率之间存在显著相关(P<0.05),与其他理化性质相关性不显著;Desulfurella与硫酸盐浓度存在显著相关,与其他理化参数相关性不显著。
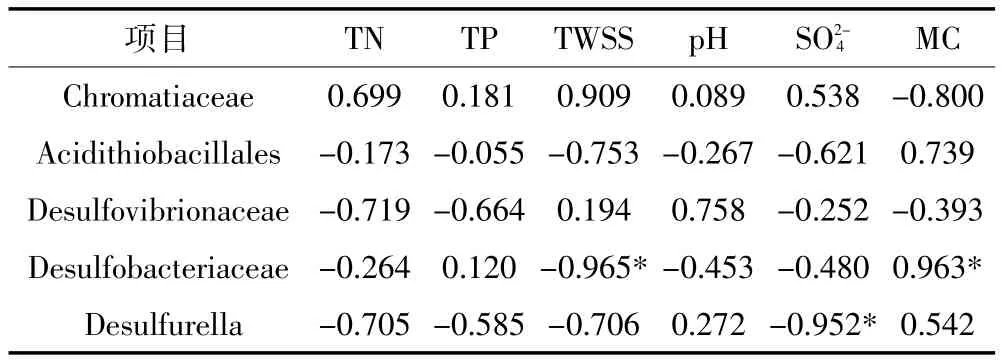
表2 乌梁素海湿地土壤理化性质与SRB多样性的相关性Table 2 Correlation coefficients between soil physic-chemical properties and SRB relative abundance in Wuliangsuhai wetland
3 讨论
硫酸盐还原菌在土壤、海洋沉积物、淹水稻田土壤、河流、湖泊及一些泥沼等自然环境中分布广泛,主要属于Proteobateria、Nitrospirae、Firmicutes、Thermodesulfobacteria和Euryarchaeota门。目前参与硫酸盐还原过程的微生物主要有革兰氏阳性/阴性硫酸盐还原细菌或古菌,还有部分能将含氮有机物分解为氨并将有机物脱硫后形成H2S。
功能基因apsA定量分析显示,乌梁素海湖滨湿地陆向分布带不同植被下,每克沉积物和土壤样品中apsA基因丰度为2.37×104~1.34×107个拷贝。根据图3的定量结果,A1功能基因apsA丰度最大(SO2-4浓度7.49 g·kg-1),A4功能基因apsA丰度最小(SO2-4浓度7.32 g·kg-1),可以看出硫酸盐浓度并不是唯一影响apsA基因丰度的因素。
本研究所获得的SRB属于变形杆菌门(Proteobac teria)中的五个科:Acidithiobacillales、Desulfurella、Chromatiales、Desulfovibrionaceae和Desulfob-acteriaceae,其中Chromatiales是变形菌门下的一类能够进行光合作用的细菌。研究发现硫酸盐还原菌可以与光营养型微生物共生,硫在这种共生关系中起着电子载体的作用[13]。Desulfovibrionaceae和Desulfoba-cteriaceae都属于革兰氏阴性嗜温不产芽孢型的变形菌门δ-变形菌纲,最早分离的SRB也属于这一类群,与ε-变形菌关系最近,主要分布于淡水、淡咸水或海水下的污泥或沉积物中,且多数菌株含氢化酶,部分菌株可以利用乙酸和CO2作碳源,通过氢的氧化和以SO2-4作末端电子受体来获取能量,少数菌株还有生物固氮能力[14]。湖区沉积物中的硫酸盐还原菌优势菌群为Desulfovibrionaceae和Desulfobacteriaceae,而在退水区及荒漠化样点中优势菌群为Chromatiales。初步推测,随着湿地的横向演化,使得湖心与湖岸生态系统产生差异,进而导致各自环境中硫酸盐还原菌类群的差异性,土壤总盐量、含水率及硫酸盐浓度可能是造成这一现象的主导因素(P<0.05)。研究发现在淹水稻田土壤环境中,Synthrophobacter、Desulforhabdus和Desulfobacterium sp.为优势物种,盐沼环境中由于Cl-和SO2-4的地球化学性质相似,常利用二者的摩尔比对硫酸盐还原菌进行定性及半定量推测,导致SRB的多样性较高[15-16]。硫酸盐还原菌对生态环境扰动所做出的快速响应,通过调查环境中SRB的群落结构,对湿地、矿山废水污染及油藏等环境的管理及评估,治理SRB引起的生物腐蚀都有一定的意义。
4 结论
乌梁素海湖滨湿地沉积物环境中硫酸盐还原菌群落多样性高于土壤环境中硫酸盐还原菌群落多样性,荒漠化土壤环境中群落多样性和apsA基因丰度都最低。土壤总盐量、含水率及硫酸盐浓度对SRB群落分布影响显著。
参考文献:
[1]李哲,王平.乌梁素海的环境问题及对策[J].环境与发展, 2014, 26(1):96-98. LI Zhe, WANG Ping. Environmental problems and solutions Wuliangsuhai[J]. Environment and Development, 2014, 26(1):96-98.
[2] Emadi M, Baghernejad M. Comparison of spatial interpolation techniques for mapping soil pH and salinity in agricultural coastal areas, Northern Iran[J]. Archives of Agronomy and Soil Science, 2014, 60(9):1315-1327.
[3] Xiao H. Studies on bacteriology of coastal water in Bohai Bay and the application of bacteria on assessment of the quality of coastal environment[D]. Qingdao:China Ocean University, 2005.
[4] Brioukhanov A, Pieulle L, Dolla A. Antioxidative defense systems of anaerobic sulfate-reducing microorganisms[J]. Current Research, Technology and Education Topics in Applied Microbiology and Microbial Biotechnology, 2010(1):148-159.
[5] Yu H, Chen C, Ma J, et al. Microbial community functional structure in response to micro-aerobic conditions in sulfate-reducing sulfur-producing bioreactor[J]. Journal of Environmental Sciences, 2014, 26(5):1099-1107.
[6] Pachiadaki M G, Lykousis V, Stefanou E G, et al. Prokaryotic community structure and diversity in the sediments of an active submarine mud volcano(Kazan mud volcano, East Mediterranean Sea)[J]. FEMS Microbiology Ecology, 2010, 72(3):429-444.
[7] Suzuki I. Oxidation of elemental sulfur by an enzyme system of Thiobacil-lus thiooxidans[J]. Biochimica et Biophysica Acta, 1965, 104(2):359-371.
[8]鲍士旦.土壤农化分析[M].北京:中国农业出版社, 2008. BAO Shi-dan. Soil agro-chemistrical analysis[M]. Beijing:China Agriculture Press, 2008.
[9]张韫.土壤·水·植物理化分析教程[M].北京:中国林业出版社, 2011. ZHANG Yun. Soil, water and plant physical and chemical analysis[M]. Beijing:China Forestry Publishing House, 2011.
[10] Hiibel S R, Pereyra L P, Breazeal M V R, et al. Effect of organic substrate on the microbial community structure in pilot-scale sulfate-reducing biochemical reactors treating mine drainage[J]. Environmental Engineering Science, 2011, 28(8):563-572.
[11] Xu X M, Fu S Y, Zhu Q, et al. Depth-related coupling relation between methane-oxidizing bacteria(MOBs)and sulfate-reducing bacteria (SRBs)in a marine sediment core from the Dongsha region, the South China Sea[J]. Applied Microbiology and Biotechnology, 2014, 98(24):10223-10230.
[12] Aktas D F, Lee J S, Little B J, et al. Effects of oxygen on biodegradation of fuels in a corroding environment[J]. International Biodeterioration and Biodegradation, 2013, 81:114-126.
[13]李永峰,刘晓烨,杨传平.硫酸盐还原菌细菌学[M].哈尔滨:东北林业大学出版社, 2013. LI Yong-feng, LIU Xiao-ye, YANG Chuan-ping. Sulfate reducing bacteria bacteriology[M]. Harbin:Northeast Forestry University Press, 2013.
[14]周德庆,徐士菊.微生物学词典[M].天津:天津科学技术出版社, 2005. ZHOU De-qing, XU Shi-ju. Microbiology dictionary[M]. Tianjin:Tianjin Science and Technology Press, 2005.
[15] Stubner S. Quantification of Gram-negative sulphate-reducing bacteria in rice field soil by 16S rRNA gene-targeted real-time PCR [J]. J Microbiol Methods, 2004, 57(2):219-30.
[16]陈庆强,唐媛,杨艳,等.长江口盐沼硫酸盐还原菌的分布特征与环境机制[J].沉积学报, 2012, 30(6):1088-1098. CHEN Qing-qiang, TANG Yuan, YANG Yan, et al. Distribution pattern of sulfate-reducing bacteria and its environmental mechanism in the saltmarsh of the Yangtze estuary[J]. Acta Sedimentologica Sinica, 2012, 30(6):1088-1098.
LIU Jian-li, ZHAO Ji, WU Lin-hui.Abundance and diversity of sulfate-reducing bacteria in littoral wetland of Wuliangsuhai[J]. Journal of Agro-Environment Science, 2016, 35(2):358-363.
Abundance and diversity of sulfate-reducing bacteria in littoral wetland of Wuliangsuhai
LIU Jian-li, ZHAO Ji, WU Lin-hui*
(College of Environment & Resources of Inner Mongolia University, Huhhot 010021, China)
Abstract:Abundance and diversity of sulfate-reducing bacteria(SRB)in sediments and soils from landward transect of Wuliangsuhai were investigated using clone library construction and real-time quantitative PCR(q-PCR)techniques. The abundance of apsA gene was significantly different among four studied samples. It was 1.34×107, 8.07×105, 5.27×106, and 1.34×107copies per gram of sample for landward evolved Phragmites australis sediments, lakeside Scirpus validus Vah sediments, lakeside Suaeda salsa salinized meadow soil, and lakeshore desertificated Nitraria tangutorum soil, respectively. Phylogenetic tree analysis showed that SRB communities in Wuliangsuhai consisted of five families. They were Acidithiobacillales, Desulfurella, Chromatiales, Desulfovibrionaceae and Desulfobacteriaceae in the Proteobacteria phylum. Among them, Chromatiales, Desulfovibrionaceae and Desulfobacteriaceae were dominant. The diversity of SRB was closely correlated with total water soluble salt, moisture content, and sulfate content(P<0.05).
Keywords:littoral wetland; sulfate-reducing bacteria(SRB); community structure; functional gene apsA
*通信作者:武琳慧E-mail:happyleis@sina.com
作者简介:刘建丽(1988—),女,山西吕梁人,硕士研究生,主要从事环境微生物学研究。E-mail:ljl2489@sina.com
基金项目:国家自然科学基金项目“蒙古高原沼泽化湿地甲烷及氨氧化菌的空间异质性与环境功能性研究”(31160129);内蒙古自然科学基金“蒙古高原湿地参与甲烷循环微生物群落结构与功能研究”(2012MS0610)
收稿日期:2015-09-18
中图分类号:X52
文献标志码:A
文章编号:1672-2043(2016)02-0358-06
doi:10.11654/jaes.2016.02.021
