典型生境中氨氧化古菌和厌氧氨氧化细菌的分布和丰度*
2016-03-13徐相龙陈祥春刘国华任争光王洪臣
徐相龙 陈祥春 刘国华# 范 强 王 皓 任争光 齐 鲁 王洪臣
(1.中国人民大学环境学院,北京 100872;2.济南热力有限公司,山东 济南 250100)
典型生境中氨氧化古菌和厌氧氨氧化细菌的分布和丰度*
徐相龙1陈祥春2刘国华1#范 强1王 皓1任争光1齐 鲁1王洪臣1
(1.中国人民大学环境学院,北京 100872;2.济南热力有限公司,山东 济南 250100)
运用分子生物学方法研究了某富营养化湖泊污泥(以下简称湖泊污泥)、某长期施肥的稻田土壤(以下简称稻田土壤)和某污水处理厂硝化污泥(以下简称硝化污泥)中氨氧化古菌(AOA)和厌氧氨氧化(ANAMMOX) 细菌的分布及丰度。结果表明,湖泊污泥、稻田土壤和硝化污泥中AOA的amoA基因Shannon指数分别为1.64、2.14、0.57,稻田土壤的生物多样性最丰富;但以上3种生境中AOA的amoA基因丰度分别为(1.45±0.13)×107、(7.24±0.25)×105、(3.46±0.22)×105拷贝数/g(以干质量计,下同),湖泊污泥中最大。湖泊污泥和稻田土壤中的AOA与水/沉积物中的古菌亲缘关系相近,既有来自水/沉积物分支,又有来自土壤/沉积物分支;硝化污泥中的AOA与土壤/沉积物中的古菌亲缘关系相近,全部来自土壤/沉积物分支。湖泊污泥、稻田土壤和硝化污泥中ANAMMOX细菌的16S rRNA基因Shannon指数都较小,生物多样性不丰富,但hzsB基因的丰度分别达到(1.32±0.17)×108、(2.88±0.28)×108、(7.76±0.25)×108拷贝数/g。湖泊污泥、稻田土壤中ANAMMOX细菌的优势种属于Brocadia属;硝化污泥中ANAMMOX细菌的Brocadia和Kuenenia几乎各占一半。
氨氧化古菌 厌氧氨氧化细菌 生物多样性 基因丰度

AOA和ANAMMOX细菌是环境科学领域新的重大发现[8],使人们重新认识全球氮循环过程。富营养化湖泊的污泥、长期施肥的土壤以及污水处理厂污泥中均含有高浓度的氮素,尤其是氨氮,是氮转化微生物的典型生境,了解这些典型生境中氮转化微生物类别及其特征对氮污染的治理具有重要的指导意义。本研究选择了某富营养化湖泊的岸边湿地带污泥、某含高浓度氮素的稻田土壤和某污水处理厂污泥3个典型生境,对其中的AOA和ANAMMOX细菌分布和丰度进行了研究,旨在为厌氧生物脱氮系统的构建提供理论基础。
1 材料与方法
1.1 实验材料
富营养化湖泊污泥(以下简称湖泊污泥)取自河北省安新县某富营养化湖泊的3个岛上,采样点为距离水陆交界线1 m处的岸边湿地带,3个采样点采集的污泥均匀混合后置于无菌袋中密封。长期施肥的稻田土壤(以下简称稻田土壤)取自上海市松江区某稻田,多点采集0~50 cm的表层土壤,均匀混合置于无菌袋中密封。污水处理厂硝化污泥(以下简称硝化污泥)取自北京市某污水处理厂的曝气池,采集后置于无菌袋中密封。上述样品在4 ℃条件下送至实验室,于-80 ℃冷冻保存直至DNA提取。
1.2 实验方法
1.2.1 DNA的提取与扩增
使用Qbiogene土壤DNA快速提取试剂盒对3种典型生境中的样品进行DNA提取。AOA采用聚合酶链式反应(PCR)进行扩增,引物为AOA特异性引物Arch-amoAF和Arch-amoAR[9]14684。AMMOXOX细菌采用巢式PCR进行扩增,第1轮采用浮霉菌通用引物PLA46f/630r[10-11],第2轮采用ANAMMOX细菌特异性引物Amx368f和Amx820r[12]。
1.2.2 系统发育树与生物多样性
扩增产物与pGEM©-T Easy载体进行连接,连接产物转化至Trans1-T1感受态细胞,用含有Amp、IPTG和X-Gal的LB平板于37 ℃下过夜培养,进行蓝白斑筛选。每个平板样品随机选取72个白色克隆,经阳性鉴定后取35个阳性克隆子进行测序,运用DOTUR软件按3%的差异度对amoA基因和16S rRNA基因去除载体序列,得到独立操作分类单元(OTU)。每个OTU中选取代表性序列通过美国国立生物技术信息中心(NCBI)数据库进行在线BLAST比对,并与AOA的amoA序列和ANAMMOX细菌的16S rRNA序列进行多重比对,采用MEGA 5.0软件的邻接法构建系统发育树[13]。用Shannon指数计算生物多样性。
1.2.3 实时荧光定量PCR
实时荧光定量PCR采用SYBY Green法,AOA定量PCR引物与1.2.1节相同,ANAMMOX细菌定量PCR引物使用hzsB基因引物AmxF和AmxR[14]。引物扩增体系为20 μL,其中SYBY Green mix 10 μL,引物(摩尔浓度为5 μmol/L)各0.4 μL,DNA模板1 μL,双蒸水补足至20 μL。AOA的反应条件为95 ℃预变性5 min,95 ℃变性15 s,55 ℃退火20 s,70 ℃延伸45 s,共进行40个循环。ANAMMOX细菌的反应条件为95 ℃预变性5 min,95 ℃变性15 s,55 ℃退火45 s,72 ℃延伸30 s,共进行40个循环。同时设置阴性对照和7个浓度梯度的标准品,每个样品(包括阴性对照和标准品)设置两次平行,以10倍基线荧光信号的标准偏差作为阈值,得到标准曲线与CT值。要求扩增斜率为-4.2~-3.5,扩增效率>95%,溶解曲线为单峰。
2 结果与讨论
2.1 不同生境中AOA的分布和丰度
2.1.1 不同生境中AOA的系统发育树分布
湖泊污泥、稻田土壤和硝化污泥中AOA的amoA基因分别得到7、14、2个OTU,分别以H-OTU、N-OTU、X-OTU标记并编号,系统发育树分布如图1所示。
由图1可见,3种生境中AOA的amoA基因序列在系统发育树中分成两大分支,分别归属于FRANCIS等[9]14685定义的土壤/沉积物分支和水/沉积物分支。其中,湖泊污泥有11个序列分布于土壤/沉积物分支,24个序列分布于水/沉积物分支;稻田土壤有13个序列分布于土壤/沉积物分支,22个序列分布于水/沉积物分支;硝化污泥的全部序列都位于土壤/沉积物分支。由此可见,湖泊污泥和稻田土壤中的AOA与水/沉积物中的古菌亲缘关系相近,而硝化污泥中的AOA与土壤/沉积物中的古菌亲缘关系相近。
通过对各OTU中的代表序列和已知菌种序列比对发现:土壤/沉积物分支中,N-OTU1#、H-OTU6#和N-OTU12#聚于一个小分支上,与来自土壤的CandidatusNitrososphaerasp. EN76(FR 773159)的相似度为89%~94%;水/沉积物分支中,H-OTU5#、N-OTU9#、N-OTU10#和N-OTU11#与来自硝化酸性沉积物的CandidatusNitrosotaleadevanaterraisolate Nd1.(JN 227489)的相似度为88%~95%,N-OTU7#、H-OTU3#、H-OTU4#和N-OTU14#与来自海洋的NitrosopumilusmaritimusSCM1(EU 239959)相似度为83%~91%。其余的OTU聚于土壤/沉积物分支中的另外两个小分支,推测这些簇群可能存在与已知菌种有区别的未被分离或培养的古菌。总的来说,硝化污泥的AOA全部来自土壤/沉积物分支,这可能与其单一的生存环境有关,湖泊污泥和稻田土壤的AOA既有来自水/沉积物分支,又有来自土壤/沉积物分支,可能是因为两者周期性的反复水陆交替,而使沉积物微生物兼顾两种特性。
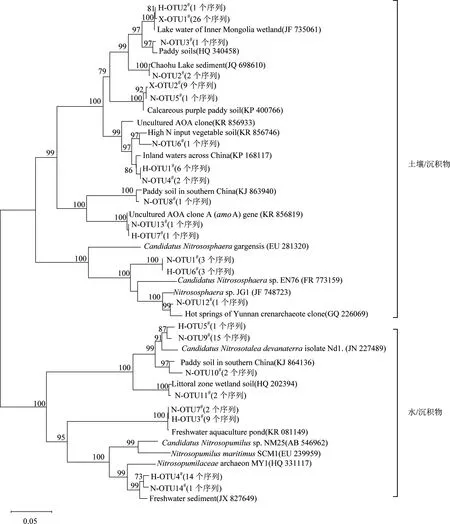
图1 3种生境中AOA的amoA基因系统发育树Fig.1 Phylogenetic tree for amoA gene of AOA in 3 habitats
2.1.2 不同生境中AOA的生物多样性
3种生境中AOA的amoA基因Shannon指数如表1所示。稻田土壤中的Shannon指数最大,为2.14,与WANG等[15]在高氮湿地沉积物中发现的AOA的amoA基因Shannon指数相近,这是因为南方稻田周期性地覆水和耕作导致了土壤生态环境的复杂性,造成了较高的AOA生物多样性。由于湖泊岸边湿地带也受到湖水涨落的交替影响,有着明显的边缘效应[16-17],因此其生物多样性也较丰富,Shannon指数为1.64。硝化污泥中AOA的amoA基因生物多样性最低,其Shannon指数仅为0.57,得到的OTU也只有2个,这是由于目前污水处理厂主要利用AOB进行硝化反应,曝气等条件不利于AOA的生长,而且污水处理厂的环境条件相对单一,对微生物起到一定的驯化作用,从而造成硝化污泥中AOA的amoA基因生物多样性偏低。

表1 3种生境中AOA的amoA基因Shannon指数
2.1.3 不同生境中AOA的丰度
运用实时荧光定量PCR得到湖泊污泥、稻田土壤和硝化污泥中AOA的amoA基因丰度分别为(1.45±0.13)×107、(7.24±0.25)×105、(3.46±0.22)×105拷贝数/g(以干质量计,下同)。虽然稻田土壤中AOA的amoA基因生物多样性高于湖泊污泥,但是丰度却只有湖泊污泥的0.050,这可能是由于南方稻田氨氮浓度较高促进了AOB生长而使AOA在环境中处于劣势。DI等[18]研究表明,环境中氨氮浓度的增加会促进AOB的丰度增加而对AOA的增长无明显作用。硝化污泥中AOA的amoA基因丰度是湖泊污泥的0.024,这主要是由于硝化污泥中氨氧化微生物以AOB为主。整体上看,在湖泊污泥中AOA的amoA基因生物多样性和丰度都较高。
目前,AOA在湖泊污泥、稻田土壤的氨氧化作用已经被证实[19-20]。近年来,在国内外多个污水处理厂中也发现有AOA的存在,并且有的污水处理厂中AOA甚至比AOB的数量还多[21-22]。
2.2 不同生境中ANAMMOX细菌的分布和丰度
2.2.1 不同生境中ANAMMOX细菌的系统发育树分布
湖泊污泥、稻田土壤和硝化污泥中ANAMMOX细菌的16S rRNA基因分别得到3、4、2个OTU,分别以H’-OTU、N’-OTU、X’-OTU标记并编号,系统发育树分布如图2所示。
目前,已报道的ANAMMOX细菌有Anammoxoglobus、Brocadia、Jettenia、Scalindua和Kuenenia5个属[23-25]。由图2可见,3种生境中ANAMMOX细菌的16S rRNA序列在系统发育树中可以聚成3个不同的分支。其中,湖泊污泥、稻田土壤和硝化污泥中分别有34、31、15个序列分布于Brocadia分支;稻田土壤和硝化污泥中分别有4、19个序列分布于Kuenenia分支;湖泊污泥中有1个序列介于Brocadia和Kuenenia之间。由此可见,湖泊污泥、稻田土壤中ANAMMOX细菌的优势种属于Brocadia属;硝化污泥中ANAMMOX细菌的Brocadia和Kuenenia几乎各占一半,Kuenenia略占优势,与徐亚慧等[26]研究的结果基本一致。由此可见,自然生态系统和人工水处理生态系统中均存在ANAMMOX细菌的Brocadia属类群,这为以自然环境污泥作为厌氧氨氧化启动的接种污泥培养富集菌种,并最终将其应用到人工水处理生态系统创造了可能性。
通过对各OTU中的代表序列和已知菌种序列比对发现:N’-OTU1#、N’-OTU3#、X’-OTU2#、H’-OTU1#和H’-OTU3#与CandidatusBrocadiafulgida(DQ 459989)的相似度为95%~99%;X’-OTU1#和N’-OTU4#与CandidatusKueneniastuttgartiensis(AF 375995)的相似度分别为96%、98%;而H’-OTU2#则介于Brocadia与Kuenenia之间。
2.2.2 不同生境中ANAMMOX细菌的生物多样性
3种生境中ANAMMOX细菌的16S rRNA基因Shannon指数如表2所示。3种生境中ANAMMOX细菌的生物多样性不是很丰富,虽然稻田土壤生物多样性最高,但其Shannon指数也只有0.92,说明这3种生境中ANAMMOX细菌都呈现较低的生物多样性。

表2 3种生境中ANAMMOX细菌的16S rRNA基因Shannon指数
2.2.3 不同生境中ANAMMOX细菌的丰度
运用实时荧光定量PCR得到湖泊污泥、稻田土壤和硝化污泥中ANAMMOX细菌的hzsB基因丰度分别为(1.32±0.17)×108、(2.88±0.28)×108、(7.76±0.25)×108拷贝数/g。3种生境中ANAMMOX细菌的hzsB基因丰度都较大且基本相当,原因可能是湖泊污泥和稻田土壤经常处于周期性的水陆交替状态,存在缺/厌氧界面,当有充足的ANAMMOX细菌生长的基质和适宜的生存条件时ANAMMOX细菌就能大量繁殖。硝化污泥中的ANAMMOX细菌可能来自厌氧池或缺氧池。
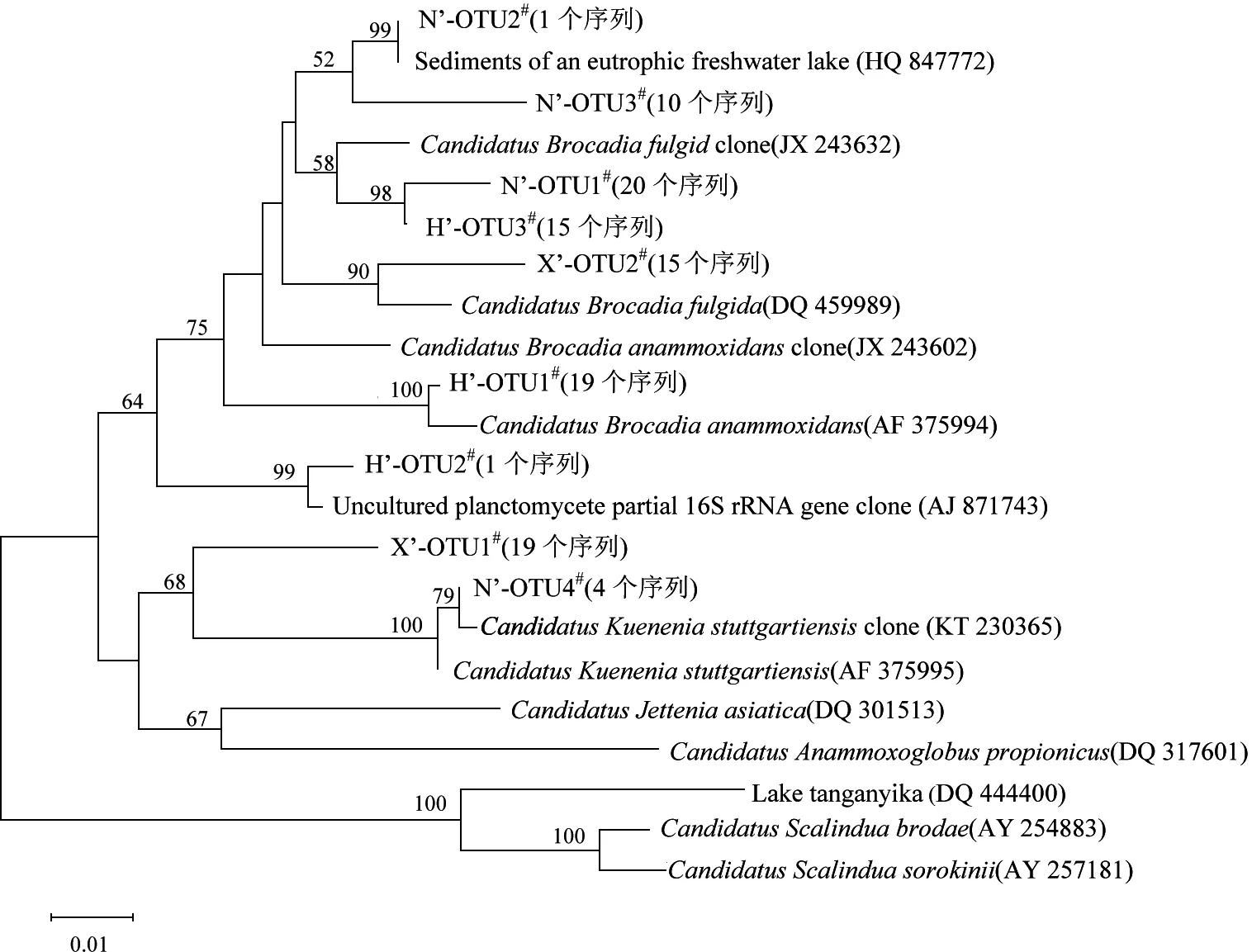
图2 3种生境中ANAMMOX细菌的16S rRNA基因系统发育树Fig.2 Phylogenetic tree for 16S rRNA gene of ANAMMOX bacteria in 3 habitats
3 结 论
(1) 湖泊污泥和稻田土壤中的AOA与水/沉积物中的古菌亲缘关系相近,既有来自水/沉积物分支,又有来自土壤/沉积物分支;硝化污泥中的AOA与土壤/沉积物中的古菌亲缘关系相近,全部来自土壤/沉积物分支。
(2) AOA的amoA基因Shannon指数在稻田土壤中最大,生物多样性最高,而在硝化污泥中最小,生物多样性最低。但是,湖泊污泥中amoA基因的丰度却远远高于稻田土壤和硝化污泥。
(3) 湖泊污泥、稻田土壤中ANAMMOX细菌的优势种属于Brocadia属;硝化污泥中ANAMMOX细菌的Brocadia和Kuenenia几乎各占一半。
(4) 3种生境中ANAMMOX细菌的16S rRNA基因生物多样性都不是很丰富,但hzsB基因的丰度却很高,甚至高于AOA的amoA基因。
4 展 望

[1] 贺纪正,张丽梅.氨氧化微生物生态学与氮循环研究进展[J].生态学报,2009,29(1):406-415.
[2] WUCHTER C,ABBAS B,COOLEN M J L,et al.Archaeal nitrification in the ocean[J].Proceedings of the National Academy of Sciences of the United States of America,2006,103(33):12317-12322.
[3] REIGSTAD L J,RICHTER A,DAIMS H,et al.Nitrification in terrestrial hot springs of Iceland and Kamchatka[J].FEMS Microbiology Ecology,2008,64(2):167-174.
[4] TREUSCH A H,LEININGER S,KLETZIN A,et al.Novel genes for nitrite reductase and Amo-related proteins indicate a role of uncultivated mesophilic crenarchaeota in nitrogen cycling[J].Environmental Microbiology,2005,7(12): 1985-1995.
[5] MARTENS H W,BERUBE P M,URAKAWA H,et al.Ammonia oxidation kinetics determine niche separation of nitrifying archaea and macteria[J].Nature,2009,461(7266):976-979.
[6] SCHLEPER C.Ammonia oxidation:different niches for bacteria and archaea?[J].The International Society for Microbial Ecology Journal,2010,4:1092-1094.
[7] JETTEN M S M,LOGEMANN S,MUYZER G,et al.Novel principles in the microbial conversion of nitrogen compounds[J].Antonie van Leeuwenhoek,1997,71(1):75-93.
[8] HORNER D M C,MARTINY A C.News about nitrogen[J].Science,2008,320(5877):757-758.
[9] FRANCIS C A,ROBERTS K J,BEMAN J M,et al.Ubiquity and diversity of ammonia-oxidizing archaea in water columns and sediments of the ocean[J].Proceedings of the National Academy of Sciences of the United States of America,2005,102(41).
[10] NEEF A,AMANN R,SCHLESNER H,et al.Monitoring a widespread bacterial group:insitudetection of planctomycetes with 16S rRNA-targeted probes[J].Microbiology,1998,144(12):3257-3266.
[11] PURKHOLD U,POMMERENING R A,JURETSCHKO S,et al.Phylogeny of all recognized species of ammonia oxidizers based on comparative 16S rRNA andamoA sequence analysis:implications for molecular diversity surveys[J].Applied and Environmental Microbiology,2000,66(12):5368-5382.
[12] AMANO T,YOSHINAGA I,OKADA K,et al.Detection of anammox activity and diversity of anammox bacteria-related 16S rRNA genes in coastal marine sediment in Japan[J].Microbes and Environments,2007,22(3):232-242.
[13] GARBEVA P,OVERBEEK L S,VUURDE J W L,et al.Analysis of endophytic bacterial communities of potato by plating and denaturing gradient gel electrophoresis (DGGE) of 16S rDNA based PCR fragments[J].Microbial Ecology,2001,41(4):369-383.
[14] 姜博,祝贵兵,周磊榴,等.低温高海拔湖泊岸边带厌氧氨氧化菌的存在、生物多样性及活性——以天山天池为例[J].环境科学学报,2015,35(7):2045-2051.
[15] WANG Shanyun,WANG Yu,FENG Xiaojuan,et al.Quantitative analyses of ammonia-oxidizing archaea and bacteria in the sediments of four nitrogen-rich wetlands in China[J].Applied Microbiology and Biotechnology,2011,90(2):779-787.
[16] 王为东,王大力,尹澄清,等.芦苇型湿地生态系统的潜水水质状态研究[J].生态学报,2001,21(6):919-925.
[17] WANG Weidong,WANG Dali,YIN Chengqing.A field study on the hydrochemistry land/inland water ecotones with reed domination[J].Acta Hydrochimica et Hydrobiologica,2002,30(2/3):117-127.
[18] DI H J,CAMERON K C,SHEN J P,et al.Nitrification driven by bacteria and not archaea in nitrogen-rich grassland soils[J].Nature Geoscience,2009,2:621-624.
[19] 叶磊,祝贵兵,王雨,等.白洋淀湖滨湿地岸边带氨氧化古菌与氨氧化细菌的分布特性[J].生态学报,2011,31(8):2209-2215.
[20] 王雨,祝贵兵,王朝旭,等.高含氮稻田深层土壤的氨氧化古菌和厌氧氨氧化细菌共存及对氮循环的影响[J].生态学报,2011,31(6):1487-1493.
[21] GAO Jingfeng,LUO Xin,WU Guixia,et al.Abundance and diversity based onamoA genes of ammonia-oxidizing archaea and bacteria in ten wastewater treatment systems[J].Applied Microbiology and Biotechnology,2014,98(7):3339-3354.
[22] LIMPIYAKORN T,FURHACKER M,HABERL R,et al.amoA-encoding archaea in wastewater treatment plants:a review[J].Applied Microbiology and Biotechnology,2013,97(4):1425-1439.
[23] SCHMID M C,MAAS B,DAPENA A,et al.Biomarkers for theinsitudetection of anaerobic ammonium oxidizing (anammox) bacteria[J].Applied and Environmental Microbiology,2005,71(4):1677-1684.
[24] KARTAL B,RATTRAY J,VAN NIFTRIK L A,et al.Candidatus“Anammoxoglobus propionicus” a new propionate oxidizing species of anaerobic ammonium oxidizing bacteria[J].Systematic and Applied Microbiology,2007,30(1):39-49.
[25] SCHMID M C,RISGAARD PETERSEN N,VAN DE VOSSENBERG J,et al.Anaerobic ammonium-oxidizing bacteria in marine environments:widespread occurrence but low diversity[J].Environmental Microbiology,2007,9(6):1476-1484.
[26] 徐亚慧,刘苗苗,张树军,等.反应器形式及污泥形态对厌氧氨氧化菌群落结构的影响[J].中国科学院大学学报,2014,31(1):67-73,84.
Distributionandabundanceofammonia-oxidizingarchaeaandanaerobicammonia-oxidizingbacteriaintypicalhabitats
XUXianglong1,CHENXiangchun2,LIUGuohua1,FANQiang1,WANGHao1,RENZhengguang1,QILu1,WANGHongchen1.
(1.SchoolofEnvironment&NaturalResources,RenminUniversityofChina,Beijing100872;2.JinanDistrictHeatingLimitedCompany,JinanShandong250100)
The distribution and abundance of ammonia-oxidizing archaea (AOA) and anaerobic ammonia-oxidizing (ANAMMOX) bacteria in typical habitats of lake sediment,paddy soil and nitrification sludge were investigated using molecular biology methods. Results showed thatamoA gene Shannon indexes of AOA in lake sediment,paddy soil and nitrification sludge were 1.64,2.14 and 0.57,respectively,indicating the richest biodiversity in paddy soil. However,amoA gene abundances of AOA in the 3 above habitats were (1.45±0.13)×107,(7.24±0.25)×105and (3.46±0.22)×105copies/g (dry weight),respectively,indicating the highest gene abundance in lake sediment. AOA in lake sediment and paddy soil was close to archaea of water/sediment branch,deriving from water/sediment branch or soil/sediment branch. AOA in nitrification sludge was close to archaea of soil/sediment branch,all deriving from soil/sediment branch. 16S rRNA gene Shannon indexes of ANAMMOX bacteria in the 3 above habitats were all low,indicating a poor biodiversity. However,hzsB gene abundance of ANAMMOX bacteria in the 3 above habitats reached (1.32±0.17)×108,(2.88±0.28)×108and (7.76±0.25)×108copies/g (dry weight),respectively. The dominant species of ANAMMOX bacteria in lake sediment and paddy soil belonged toBrocadiawhile that in nitrification sludge belonged toBrocadiaandKueneniaalmost equally.
AOA; ANAMMOX bacteria; biodiversity; gene abundance
徐相龙,男,1989年生,硕士研究生,主要从事污水的厌氧氨氧化脱氮技术研究。#
。
*中央高校基本科研业务费专项(No.15XNLD04)。
10.15985/j.cnki.1001-3865.2016.12.012
编辑:陈锡超 (
2016-06-13)
