杨树磷酸肌醇特异性磷脂酶C基因家族鉴定与分析
2016-02-13张杰伟丁莉萍陈亚娟王宏芝魏建华
张杰伟,丁莉萍,陈亚娟,王宏芝*,魏建华*
(1.北京市农林科学院北京农业生物技术研究中心,北京 100097;2.农业基因资源与生物技术北京市重点实验室,北京 100097)
杨树磷酸肌醇特异性磷脂酶C基因家族鉴定与分析
张杰伟1,2,丁莉萍1,2,陈亚娟1,2,王宏芝1,2*,魏建华1,2*
(1.北京市农林科学院北京农业生物技术研究中心,北京 100097;2.农业基因资源与生物技术北京市重点实验室,北京 100097)
为了对已测序杨树PopulustrichocarpaTorr.& Gray中磷酸肌醇特异性磷脂酶C (phosphoinositide-specific phospholipase C,PI-PLC)家族基因进行系统分析,利用杨树基因组数据库,通过生物信息学手段,鉴定杨树PI-PLC 家族基因的基因结构、染色体定位和编码蛋白,通过蛋白序列比对进行进化和分类分析。结果表明,杨树中含有7 个PI-PLC 家族基因,含有8~10个外显子,分布于杨树的4 条染色体上。MEME和Pfam保守结构域分析显示,杨树PI-PLC 蛋白均含有4个保守的EF、X、Y和C2结构域。蛋白质进化树分析结果表明PI-PLC 可分为2 个亚家族。
磷酸肌醇特异性磷脂酶C; 杨树; 进化分析; 基因家族
磷酸肌醇特异性磷脂酶C(phosphoinositide-specific phospholipase C,PI-PLC)是一类水解磷脂的酶类,其水解4,5-二磷酸磷脂酰肌醇(PIP2),产生1,4,5-三磷酸肌醇(IP3)和1,2-二酰甘油(DAG)[1]。植物细胞中,水溶性IP3进入细胞质后在一系列磷酸激酶的作用下可以迅速转化为IP6, 促进细胞内钙库中Ca2+内流,从而调节Ca2+浓度及Ca2+依赖的酶类或Ca2+依赖的通道[2];而脂溶性DAG停留在细胞膜上,在二酰甘油激酶的作用下生成磷脂酸(phosphatidic acid, PA),PA通过与一些蛋白激酶、脂激酶和NADPH氧化酶等蛋白相互作用, 从而调节植物细胞生长、非生物胁迫和细胞死亡等生理过程[3]。
高等植物中,拟南芥PI-PLC(AtPLC)的生物功能解析较为完整。拟南芥PI-PLC家族共有9个成员。其中AtPLC1(曾用名AtPLC1S)参与不依赖ABA的高渗胁迫信号转导[4];AtPLC2参与了内质网胁迫反应和生长素调节的生长发育[5-6];AtPLC3和AtPLC9在耐热性方面起着重要作用[7-8],过量表达AtPLC5引起拟南芥和烟草叶片早衰[9]。
2006 年杨树基因组草图测序完成[10],为后续通过生物信息学挖掘、鉴定和分析杨树基因组奠定了相关基础。本研究从杨树基因组数据库出发,重点分析杨树PI-PLC家族基因进化关系及编码蛋白保守结构域,为克隆杨树PI-PLC基因(PtPLC)提供有价值的信息,以期为进一步研究PtPLC基因的生物学功能奠定基础。
1 材料与方法
1.1 杨树PI-PLC家族基因全基因组、cDNA和蛋白序列的获得
拟南芥PI-PLC基因及蛋白序列下载自TAIR数据库(http://www.arabidopsis.org),杨树PI-PLC基因及蛋白序列均下载自JGI数据库(https://phytozome.jgi.doe.gov/pz/portal.html#!info?alias=Org_Ptrichocarpa)。
1.2 PI-PLC家族基因系统进化树的构建及编码蛋白保守结构
通过Clustal X(2.0)[11]软件对拟南芥和杨树的PI-PLC蛋白进行多序列比对分析,将序列比对结果使用MEGA 6.0[12]软件,采用Neighborjoining法(Bootstrap值设为1000)生成PI-PLC家族基因的系统进化树。应用在线软件MEME(http://meme.nbcr.net/meme/cgi-bin/meme.cgi)和Pfam 数据库(http://pfam.sanger.ac.uk/search)对拟南芥和杨树的PI-PLC蛋白保守结构进行分析[13-14]。
2 结果与分析
2.1 杨树PI-PLC 家族基因的鉴定和命名
根据拟南芥中已经鉴别出来的9个PI-PLC蛋白序列,分别利用phytozome 数据库(http://www.phytozome.net/search.php)提供的Blast 程序中进行BlastP,得到了21个杨树候选PI-PLC基因。利用保守域在线预测软件Pfam 和MEME进行验证EF、X、Y和C2保守域的存在,初步确定了7个杨树PI-PLC基因。其中杨树PI-PLC 基因分布在第1、8、9和10等4条染色体上,根据其在染色体上的定位分别命名为PtPLC1-7,每个成员的基因长度、氨基酸长度、染色体定位、外显子个数等详细信息见表1。杨树PtPLC基因均含有8~10个外显子(图1)。
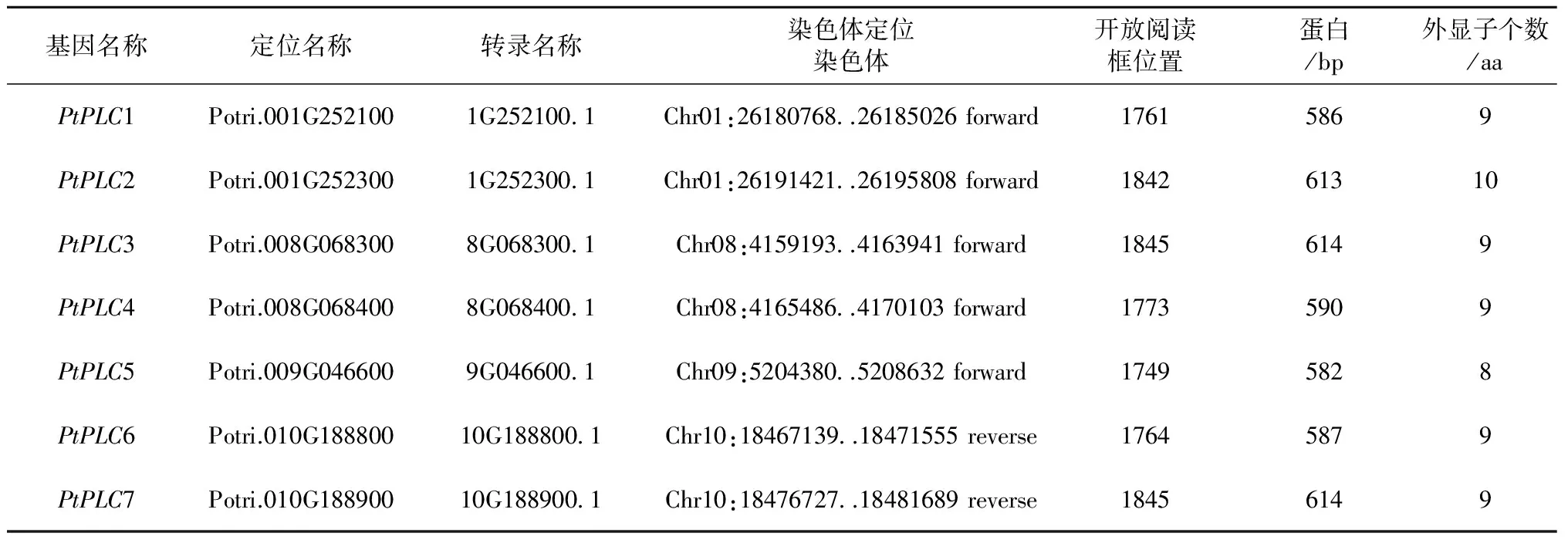
表1 杨树PI-PLC家族基因的基本特征Table 1 Characteristics of PI-PLC genes of poplar
2.2 杨树PI-PLC 家族蛋白相似性和系统进化分析
拟南芥AtPLC1~7含有561~584个氨基酸残基,预测分子量为64~67 kD,拟南芥AtPLC8和AtPLC9 均含有530个氨基酸残基,预测分子量均为61 kD,而杨树PtPLC1~7含有582~614个氨基酸残基,预测分子量为66~69 kD。利用进化树分析软件MEGA 6.0 对9个拟南芥和7个杨树PI-PLC蛋白序列进行进化分析。结果显示:这16个PI-PLC分为2个亚家族(按照催化结构域(X和Y结构域)的完成性),分别含有14和2个成员(图2)。第Ⅰ亚家族(具有完整催化结构域)由拟南芥AtPLC1~7和杨树PtPLC1~7组成,第Ⅱ亚家族(不具有完整催化结构域)由拟南芥AtPLC8和AtPLC9 组成。7个杨树PI-PLC蛋白的氨基酸序列相似性在51.67%~92.67%,其中PtPLC3 和PtPLC7氨基酸序列的相似性高达92.67%(表2)。

表2 杨树基因组中PI-PLC 蛋白氨基酸相似性分析Table 2 Amino acid seque nce similarity analysis of the OtPLC proteins
2.3 杨树PI-PLC 基因家族保守结构域分析
使用在线软件MEME和Pfam 对PtPLC 基因家族保守结构域进行分析,结果表明:拟南芥和杨树PI-PLC 基因家族均包含4个保守结构域。其中,元件1是EF结构域,2(X) 和3 (Y)组成PI-PLC催化结构域,元件4是C2结构域。进化树分析结果显示,杨树PtPLC1~7和拟南芥AtPLC1~7均聚类到第Ⅰ亚家族、拟南芥AtPLC8和AtPLC9聚类到第Ⅱ亚家族(图2)。他们均含有EF、X、Y、C2等4个结构域,这表明PI-PLC蛋白在进化过程中含有相似的保守结构域(图3)。

表3 杨树PtPLC1、4、6和7不同转录本的基本特征Table 3 Characteristics of PtPLC1, 4, 6, and 7 genes
3 讨论与结论
PI-PLC是磷酸肌醇信号途径中的一个关键酶,在许多植物中已经被证明参与非生物胁迫、调节骨架系统、C4作物的呼吸作用等多个生理过程[1,5-9,15-18]。随着分子生物学和生物信息学技术的发展,PI-PLC基因家族已经在拟南芥、水稻等多种高等植物中鉴定[19],但杨树PI-PLC基因家族尚未鉴定。本研究鉴定已测序的杨树PI-PLC基因家族含有7个成员,而已鉴定的拟南芥和水稻PI-PLC基因家族分别含有9个和4个成员,这说明在进化过程中,PI-PLC基因可能经历了不断发生谱系的特异扩张和拷贝丢失。杨树PI-PLC基因家族的7个成员含有8~10个外显子,均含有EF、X、Y和C2结构域,这表明PI-PLC基因家族的高度保守性。研究表明,虽然拟南芥AtPLC8和At PLC9 的Y 结构域缺失了一部分片段,但是拟南芥plc9 突变体较野生型植株对热胁迫更加敏感,同时AtPLC9过量表达植株表现出比野生型植株对热胁迫更加耐受[7]。推测这些酶所参与的磷脂信号在植物诸多生理过程中起着类似作用。而不同的物种中含有的成员数目不同,暗示不同物种的各成员在特定的组织响应不同的外界刺激从而精细调节各生理过程。
进化分析表明,已测序的杨树PI-PLC基因家族中不含有第Ⅱ亚家族的成员,PtPLC1-7与拟南芥AtPLC1-7聚类于第Ⅰ亚家族。到目前为止,拟南芥AtPLC1-5、AtPLC9已经被证明具有典型的PI-PLC生物学功能[7]。Hirayama等[20]从拟南芥中克隆到第一个PI-PLC基因AtPLC1,AtPLC1在正常条件下表达量很低,当在干旱、盐胁迫和低温胁迫时表达量急剧升高。后续的研究证实AtPLC1响应了ABA的次级信号转导[21]。AtPLC3(曾用名AtPLC1F)不仅在开花期诱导表达,而且参与了植株的耐热反应[8]。推测PtPLC2和PtPLC5可能响应干旱、高盐、低温等各种非生物胁迫以及参与到生长素调节的生长发育过程。AtPLC2参与了内质网胁迫反应和生长素调节的生长发育过程[5-6]。推测PtPLC4和PtPLC6可能在杨树中参与内质网胁迫和生长素调节的生长发育过程。截至目前,AtPLC6和AtPLC7的生物学功能还未见报道。推测PtPLC3、PtPLC4、PtPLC6和PtPLC7可能在特定时期木本植物中发挥重要功能。AtPLC5(曾用名AtPLC6)存在着典型的钙依赖的PI-PLC酶活性,Northern结果显示AtPLC5能够在冷胁迫下被强烈诱导[22]。最近的研究发现,AtPLC5超量表达后能都引起烟草和拟南芥的叶片早衰,同时在超量表达的AtPLC5转基因植株中能检测到PI-PLC酶活性[9]。与AtPLC5同源性最高的毛白杨PtoPLC1已经证明具有PI-PLC酶活性,并且其响应高盐、ABA等各种非生物胁迫[23]。表明聚类在一起的不同物种的同源基因可能具有不同的生物学功能,因此不能简单推测杨树PtPLC基因家族各成员的生物学功能,其各成员的生物学功能还需要从杨树中分离后逐一解析其功能。
杨树PtPLC家族不同基因有不同的转录本个数,其中:PtPLC2、PtPLC3和PtPLC5均只有一个预测的转录本(表1),PtPLC1、PtPLC4、PtPLC6和PtPLC7分别含有4、2、6和3个预测的转录本,每个预测的转录本的长度、氨基酸长度、染色体定位、外显子个数等详细信息见表3。因为植物PI-PLC基因家族含有EF、X、Y和C2等4个保守的结构域,故在转录本选择上均选取最大表达框。在未来的研究中要从杨树不同组织、不同发育阶段以及在不同逆境胁迫中克隆不同的转录本,验证是否存在其他预测PtPLC的转录本,并研究其生物学功能,以期完善对植物PI-PLC结构和生物学功能的认识。
[1]RUPWATE S D,RAJASEKHARAN R.Plant phosphoinositide-specific phospholipase C: an insight [J]. Plant Signa Behav, 2012, 7 (10):1281-1283.
[2]MUNNIK T, TESTERINK C. Plant phospholipid signaling:“in a nutshell” [J]. Lipid Res,2009, 50(S1): S260-S265.
[3]WANG X, DEVAIAH S P, ZHANG W, et al. Signaling functions of phosphatidic acid [J]. Prog Lipid Res, 2006, 45 (3): 250-278.
[4]TAKAHASHI S, KATAGIRI T, HIRAYAMA T, et al. Hyperosmotic stress induces a rapid and transient increase in inositol 1, 4, 5-trisphosphate independent of abscisic acid inArabidopsiscell culture [J]. Plant Cell Physiol, 2001,42(2): 214-222.
[5]KANEHARA K, YU C Y, CHO Y, et al.ArabidopsisAtPLC2 is a primary phosphoinositide-specific phospholipase C in phosphoinositide metabolism and the endoplasmic reticulum stress response [J]. PLoS Genet, 2015,11(9): e1005511.
[6]LI L, HE Y, WANG Y, et al.ArabidopsisPLC2 is involved in auxin-modulated reproductive development [J]. Plant J, 2015, 84(3): 504-515.
[7]ZHENG S Z, LIU Y L, LI B, et al. Phosphoinositide-specific phospholipase C9 is involved in the thermotolerance inArabidopsis[J]. Plant J, 2012, 69(4): 689-700.
[8]GAO K, LIU Y L, LI B, et al.Arabidopsisthalianaphosphoinositide-specific phospholipase C isoform 3 (AtPLC3) and AtPLC9 have an additive effect on thermotolerance [J]. Plant Cell Physiol, 2014, 55(11): 1873-1883.
[9]ZHANG J, XIA K, YANG Y, et al. Overexpression ofArabidopsisphosphoinositide-specific phospholipase C5 induces leaf senescence [J]. Plant Cell Tiss Organ Cult, 2015, 120(2): 585-595.
[10]TUSKAN G A, DIFAZIO S, JANSSON S, et al. The genome of black cottonwood,Populustrichocarpa(Torr. & Gray) [J]. Science, 2006,313(5793): 1596-1604.
[11]LARKIN M A, BLACKSHIELDS G, BROWN N P, et al. Clustal W and clustal X version 2.0 [J]. Bioinformatics, 2007, 23(21): 2947-2948.
[12]TAMURA K, STECHER G, PETERSON D, et al. MEGA6: molecular evolutionary genetics analysis version 6.0 [J]. Mol Biol Evol, 2013, 30(12): 2725-2729.
[13]BAILEY T L, BODEN M, BUSKE F A, et al. MEME SUITE: Tools for motif discovery and searching [J]. Nucleic Acids Res, 2009, 37(Web Server issue): W202-208.
[14]FINN R D, COGGILL P, EBERHARDT R Y, et al. The Pfam protein families database: towards a more sustainable future [J]. Nucleic Acids Res, 2016, 44(Database issue): D279-D285.
[15]TRIPATHY M K, TYAGI W, GOSWAMI M, et al. Characterization and functional validation of tobacco PLCδ for abiotic stress tolerance [J]. Plant Mol Biol Rep, 2012,30(2): 488-497.
[16]WANG C R, YANG A F, YUE G D, et al. Enhanced expression of phospholipase C 1 (ZmPLC1) improves drought tolerance in transgenic maize [J]. Planta, 2008, 227(5): 1127-1140.
[17]PAOLO G D, CAMILLI P D. Phosphoinositides in cell regulation and membrane dynamics [J]. Nature, 2006, 443(7112): 651-657.
[18]COURSOL S, GIGLIOLI-GUIVARC'H N, VIDAL J, et al. An increase in phosphoinositide-specific phospholipase C activity precedes induction of C4 phosphoenolpyruvate carboxylase phosphorylation in illuminated and NH4Cl-treated protoplasts fromDigitariasanguinalis[J]. Plant J, 2000, 23(4): 497-506.
[19]SINGH A, KANWAR P, PANDEY A,et al. Comprehensive genomic analysis and expression profiling of phospholipase C gene family during abiotic stresses and development in rice [J]. PLoS ONE, 2013, 8 (4): e62494.
[20]HIRAYAMA T, OHTO C, MIZOGUCHI T, et al. A gene encoding a phosphatidylinositol-specific phospholipase C is induced by dehydration and salt stress inArabidopsisthaliana[J]. Proc Natl Acad Sci USA, 1995, 92(1): 3903-3907.
[21]SANCHEZ J P, CHUA N H.ArabidopsisPLC1 is required for secondary responses to abscisic acid signals [J]. Plant Cell, 2001,13(5): 1143-1154.
[22]XU X, CAO Z, LIU G, et al. Cloning and expression of AtPLC6, a gene encoding a phosphatidylinositol-specificArabidopsisthaliana[J]. Chinese Sci Bull, 2004, 49(6): 567-573.
[23]ZHANG J, ZHANG Z, ZHU D, et al. Expression and initial characterization of a Phosphoinositide-specific phospholipase C fromPopulustomentosa[J]. J Plant Biochem Biotech, 2015, 24(3): 338-346.
(责任编辑:黄爱萍)
Genome-wide Analysis and Identification of Phosphoinositide-specific Phospholipase C Gene Family in Poplar (Populustrichocarpa)
ZHANG Jie-wei1,2, DING Li-ping1,2, CHEN Ya-juan1,2, WANG Hong-zhi1,2*, WEI Jian-hua1,2*
(1.BeijingAgro-BiotechnologyResearchCenter,BeijingAcademyofAgricultureandForestrySciences,Beijing100097,China;2.BeijingKeyLaboratoryofAgriculturalGeneticResourcesandBiotechnology,Beijing100097,China)
Phosphoinositide-specific phospholipase C (PI-PLC) is a conserved key enzyme in phosphoinositide signaling for animals and plants. It plays an important role in growth and development, as well as various abiotic and biotic stresses in the organisms. Based upon the existing database and bioinformatics methodologies, PI-PLC family gene structures and their positions on chromosome and duplication for poplar (PopulustrichocarpaTorr.&Gray) (Pt) were obtained. PI-PLC proteins were classified according to their phylogenetic relationship. The results showed that 7 PtPLC genes existed in the poplar genome on 4 chromosomes. Their multiple alignments and motif displays indicated that all of the PtPLC proteins contained 4 conserved domains (i.e., EF, X, Y, and C2). Phylogenetic analysis on the PtPLC family classified them into two groups. The information would be the basis for a study on the functions of PtPI-PLC gene family.
PI-PLC;Populustrichocarpa; phylogeny analysis;gene family
2016-07-07初稿,2016-10-24修改稿
张杰伟(1982-),男,博士,副研究员,研究方向:林木分子生物学(E-mail:jwzhang919@163.com) 共同第一作者:丁莉萍(1979-),女,助理研究员,研究方向:林木分子生物学(E-mail:dingluo2011@126.com); *通讯作者:王宏芝(1970-),副研究员,研究方向:林木分子生物学(E-mail:wanghongzhi@baafs.net.cn); 魏建华(1971-),研究员,研究方向:林木分子生物学(E-mail:weijianhua@baafs.net.cn)
国家高技术研究发展计划(863)项目(2013AA102702);国家自然科学基金(31270593);北京市自然科学基金(6163028);北京市农林科学院青年科研基金(QNJJ201416)
Q 945.78; S 601
:A
:1008-0384(2016)11-1181-06
张杰伟,丁莉萍,陈亚娟,等.杨树磷酸肌醇特异性磷脂酶C基因家族鉴定与分析[J].福建农业学报,2016,31(11):1181-1186.
ZHANG J-W,DING L-P,CHEN Y-J,et al.Genome-wide Analysis and Identification of Phosphoinositide-specific Phospholipase C Gene Family in Poplar (Populustrichocarpa)[J].FujianJournalofAgriculturalSciences,2016,31(11):1181-1186.
