布鲁氏菌非编码小RNA BSR1526突变株与过表达株的构建及毒力研究
2016-02-09徐晓阳欧红萍彭广能雷霜霜田一男曹雪峰王玉飞严光文钟志军
徐晓阳,欧红萍,彭广能,雷霜霜,田一男,曹雪峰,王玉飞,严光文,钟志军
布鲁氏菌非编码小RNA BSR1526突变株与过表达株的构建及毒力研究
徐晓阳1,欧红萍2,彭广能1,雷霜霜1,田一男1,曹雪峰1,王玉飞3,严光文4,钟志军1
目的 为探讨非编码小RNA BSR1526在布鲁氏菌胞内生存中的作用,构建BSR1526突变株与过表达株并分析它们在模拟胞内环境和小鼠体内的存活能力。方法首先采用Northern blot和RT-PCR验证布鲁氏菌16M在体外刺激条件中BSR1526的转录;然后采用融合PCR构建BSR1526缺失株;将BSR1526插入到质粒pBBR1-MCS4中,电转入16M中构建过表达株16M-BSR1526;最后分析BSR1526缺失株和过表达株在模拟胞内环境以及小鼠体内的存活力。结果BSR1526在布鲁氏菌16M中存在转录,且在不同刺激条件下转录水平不同。BSR1526缺失或过表达影响了布鲁氏菌16M在体外的生长能力,缺失后在热应激、氧化压力及酸性环境下的生存能力下降,在小鼠脾脏内的细菌数量显著下降,表明BSR1526影响了布鲁氏菌的毒力。结论非编码小RNA BSR1526影响着布鲁氏菌16M在胞内生存能力,缺失BSR1526后布鲁氏菌16M抵抗外界不良环境的能力下降,同时在小鼠体内的毒力下降。
布鲁氏菌;非编码小RNA;BSR1526;胞内生存
细菌非编码小RNA(Small non-coding RNA,sRNA)是近年来发现的一类RNA调控子,不编码蛋白质,长度在50~500个核苷酸之间[1]。过去它们被认为是“垃圾RNA”,但随着研究的深入,发现RNA的突变或异常表达与许多疾病的发生密切相关。sRNA广泛参与了生物生命活动的调控[2]。目前认为,细菌sRNAs 在应对环境变化的基因表达调控中发挥作用,是细菌适应环境压力、毒力以及代谢的重要调控子。sRNA广泛参与细菌多种生命活动的调控,如影响外膜平衡、新陈代谢、翻译以及群体感应和某些致病菌的毒力等[3-7]。
布鲁氏菌属于变形菌的α-2亚家族,无质粒,仅有2条染色体即Ⅰ号和Ⅱ号染色体[8-9]。布鲁氏菌为胞内寄生菌,主要寄生于宿主单核细胞内(主要为巨噬细胞),胞内生存繁殖是其致病的关键。适应胞内环境,如营养缺乏、酸性环境及氧化压力等,是布鲁氏菌实现胞内生存的前提。目前,布鲁氏菌中许多与胞内运输和增殖相关的基因已被鉴定,然而调控这些基因协调表达促使布鲁氏菌适应宿主体内恶劣环境的主要机制还不清楚。研究发现,细菌适应环境压力的重要调控子sRNAs 在细菌适应恶劣环境机制中起着重要作用。每种环境因素至少与一种sRNA 相关联,sRNA 将环境改变信号传导至细菌胞内有关基因,并引起一系列的级联效应[10]。因此,寻找布鲁氏菌中的sRNA,研究他们的功能和调控机制将有助于深入理解布鲁氏菌的胞内繁殖和致病机制。前期对羊种布鲁氏菌16M研究中,我们测序预测了1 321个sRNA,其中BSR1526就是其中之一[11]。本研究对BSR1526进行了Northern blot 及RT-PCR验证,确定其在16M中存在转录,并且可能参与布鲁氏菌胞内生存调控。因此,我们构建了BSR1526的突变株和过表达株,进一步研究BSR1526对布鲁氏菌胞内存活能力的影响。
1 材料与方法
1.1 材料 羊种布鲁氏菌(B.melitensis, 16M)为本室保存;大肠杆菌DH5α购自康为公司;pMD18-T Vector购自TaKaRa公司; pBBR1-MCS4(AmpR)为能在布鲁氏菌中复制的质粒,由Kenneth M. Peterson博士馈赠[12];布鲁氏菌培养的大豆胰蛋白胨琼脂(TSA)和大豆胰蛋白胨肉汤(TSB)购自生物梅里埃公司;LB用于大肠杆菌的培养;GEM(MgSO4.7H2O 0.2 g/L, Citric acid. H2O 2.0 g/L, K2HPO410.0g/L, NaNH4HPO4.4H2O 3.5 g/L, Glucose 20 g/L)培养基为营养缺乏培养基。
随机引物、dNTP、DNA分子量Marker、高保真酶购自Takara公司;快速小提质粒盒、Taq酶及XhoI和SpeI限制性核酸内切酶购自康为公司;琼脂糖凝胶DNA回收试剂盒、DNase I、RNasin@核糖核酸酶抑制剂和M-MLV逆转录酶购自Promega公司;MasterPure RNA Purification Kit购自Epicentre;MICROBExpress bacterial mRNA enrichment Kit购自Ambion;Trizol 购自Invitrogen;0.1% Triton X-100购自Sigma;DIG Northern Starter Kit购自Roche;紫外可见光分光光度计ND-1000;GeneAmp○R PCR System 9700;DYY-12型电脑三恒多用电泳仪。
SPF级6~8周龄BALB/c雌性小鼠,由军事医学科学院动物中心提供。
1.2 引物 GenBank中下载B.melitensis16M相关基因序列,Primer Premier 5 进行引物设计。引物由北京六合华大基因科技股份有限公司合成。
1.3 BSR1526的预测 16M于100 mL TSB培养基中37 ℃培养至对数生长中期,采用MasterPureTM RNA Purification Kit提取总RNA,DNase消化残存的DNA后用MICROBExpress Bacterial mRNA Enrichment Kit从总RNA中去除rRNA,纯化的mRNA通过Agilent生物分析仪进行质量检测。然后构建cDNA测序文库[13],采用IlluminaHiSeq2000测序仪对文库进行序列测定。对转录组数据分析并预测sRNA[14]。BSR1526为众多sRNA之一[11],本试验围绕该sRNA进行研究。
1.4 Northern blot验证BSR1526 参照Beckmann的方法,使用DIG Northern Starter Kit进行Northern blot[15]。
表1 引物序列
Tab.1 Primers used in this study

引物名称(Primers)5′—3′序列(5′—3′sequence)SI1526-p⁃FCGAAAAGGCACCCCAAGCATGSI1526⁃p⁃RTAATACGACTCACTAT⁃AGGGGGTGGTCGATTTCTGCAT⁃CAGSI1526⁃RT⁃FCACGAAGCCTTCAAATTGSI1526⁃RT⁃RGGTCGATTTCTGCATCAGSI1526⁃N⁃FGTCACATTCTGCAATTCATGSI1526⁃N⁃RGACATTCATCCCAGGTGGCAT⁃GCTTGGGGTGCCTTTTCGSI1526⁃C⁃FTCTGGGGTTCGAAATGACCGAT⁃CAATTGACACGGCTGATGSI1526⁃C⁃RTCAAGATGATGGTCGATGTCSI1526⁃I⁃FTCAACAGCCGGTCCATGTCCSI1526⁃I⁃RCTATGAGGGCGATGGCCATGSI1526⁃FCTCGAGCGAAAAGGCAC⁃CCCAAGCATGSI1526⁃RACTAGTGGTGGTCGATTTCTG⁃CATCAG
1.5 不同刺激条件下BSR1526的RT-PCR鉴定参照文献[11]进行。对不同刺激条件下(PH=7.0的TSB、pH=4.0的TSB、pH=7.0营养缺乏GEM培养基和1.5 mmol/L的H2O2培养基),BSR1526的转录水平进行验证。
1.6 缺失株和过表达株的构建 缺失株采用抗性基因替换和融合PCR构建突变株,具体参照文献[11]进行。构建正确后缺失突变株命名为16M-△BSR1526。过表达株构建时,以SI1526-F和SI1526-R为引物扩增目的片段。目的片段与pBBR1-MCS4质粒经XhoI和SpeI双酶切。然后用T4 DNA Ligase将两种酶切产物连接,转化,筛选抗性单克隆菌进行菌落PCR鉴定,鉴定正确后测序(测序结果略)。鉴定正确者即为阳性克隆,命名为16M-BSR1526。
1.7 BSR1526突变株和过表达株的RT-PCR验证 参照文献[11]进行。采用Trizol法提取总RNA,逆转录为cDNA。以cDNA为模板,SI1526-RT-F/SI1526-RT-R为引物,扩增BSR1526片段,以16S rRNA为内参进行PCR验证。
1.8 16M、16M-△BSR1526 和16M-BSR1526的生长曲线、体外应激实验和小鼠毒力实验 参照文献[11]进行。小鼠毒力实验的接种剂量为1×105CFU/只,接种后7 d、14 d、28 d、45 d后处死小鼠,计算小鼠脾脏内载菌数。
1.9 BSR1526靶基因的预测 利用TargetRNA2预测BSR1526的靶标基因。进行靶标预测时,对候选sRNA和mRNA序列进行计分,分数为极值分布,判断标准为P值,P值越小则相应mRNA越有可能是其靶标基因。TargetRNA2在线网址(http://cs.wellesley.edu/~btjaden/TargetRNA2/)。最后,查阅候选靶标mRNA分子的相关功能。
1.10 小鼠毒力实验 6~8周龄BALB/c雌性小鼠随机分为4组,腹腔注射分别接种16M、16M-△BSR1526、16M-BSR1526和空白对照组(PBS)。剂量为1×105CFU/只。接种后7 d、14 d、28 d、45 d,各组取3只小鼠处死,无菌条件下取小鼠脾脏,称重后放入1 mL 0.1% Triton X-100的生理盐水中匀浆,组织匀浆液进行倍比稀释,选取适当稀释度的匀浆液100 μL涂板计数。重复实验3次,计算平均值。
1.11 BSR1526靶基因的预测 利用TargetRNA2预测BSR1526的靶标基因。进行靶标预测时,对候选sRNA和mRNA序列进行计分,分数为极值分布,判断标准为P值,P值越小则相应mRNA越有可能是其靶标基因。TargetRNA2在线网址(http://cs.wellesley.edu/~btjaden/TargetRNA2/)。最后,查阅候选靶标mRNA分子的相关功能。
2 结 果
2.1 BSR1526的预测 经高通量测序我们预测了众多sRNA,其中BSR1526为候选sRNA之一。如图1,BSR1526位于布鲁氏菌Ⅰ号染色体BMEI1526和BMEI1533之间基因间区的正义链上,转录起点为1 577 556,终点为1 577 730,长175 nt。

图1 预测的BSR1526在染色体中的位置Fig.1 Location of BSR1526 on the B. melitensis 16M chromosome
2.2 Northern blot验证 为验证预测的BSR1526是否在16M中存在转录,提取标准培养条件下16M的总RNA,采用Northern blot进行验证。如图2所示, BSR1526在16M的总RNA中存在转录。

图2 Northern blot验证BSR1526Fig.2 Verification of candidate sRNA BSR1526 by Northern blot
2.3 不同刺激条件下BSR1526的RT-PCR鉴定 结果表明,BSR1526在总 RNA中存在转录,且在不同刺激条件下其转录水平存在差异(图3)。酸性条件下BSR1526的转录水平高于其他刺激条件。由于选用的条件为模拟的巨噬细胞内环境,BSR1526在这些条件下的转录存在差异,提示BSR1526参与了布鲁氏菌的胞内生存。
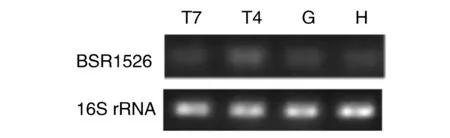
T7: TSB7.0; T4: TSB4.0; G: GEM7.0; H: H2O2.图3 BSR1526的RT-PCR验证Fig.3 Verification of candidate sRNA BSR1526 by RT-PCR
2.4 BSR1526缺失株和过表达株构建
2.4.1 缺失株构建 SI1526-N-F和SI1526-N-R为引物扩增出大小为530 bp的N端同源臂;以SI1526-C-F和SI1526-C-R为引物扩增出大小为638 bp的C端同源臂;以pUC19K为模板,Kana-F和Kana-R为引物,扩增出大小为1 093 bp的Kana基因;将3个片段融合成约2 000 bp片段;融合片段与pMD18-T Vector连接,转化DH5α态细胞,次日随机挑取单克隆菌进行菌落PCR,扩增出约2 000 bp片段的菌液进行测序,测序正确者提质粒并保菌。将阳性突变盒质粒电转入16M感受态细胞,挑取单克隆菌作为模板进行PCR鉴定,同时以野生株16M作为对照(图4)。结果显示,抗性基因与外侧引物组合鉴定,突变株能扩增出预期大小目的片段,而16M无扩增,表明构建成功,命名为 16M-ΔBSR1526。

1, 3: Identification of N-homologous arm; 2, 4: Identification of C-homologous arm; 1, 2: 16M-ΔBSR1526; 3, 4: 16M; 3, 4: 16M control; M: DL2000 marker.图4 BSR1526缺失突变株鉴定图Fig.4 Verification of BSR1526 mutant
2.4.2 过表达株构建 SI1526-F和SI1526-R为引物,扩增出过表达片段,回收的目的片段进行双酶切,将pBBR1-MCS4质粒进行双酶切。将双酶切产物纯化后,连接转化DH5α细胞,涂Amp抗性LB平板并进行菌落PCR,将鉴定正确的菌液送华大测序。Blast比对正确的即为阳性菌,提取阳性质粒MCS4-BSR1526,电转16M感受态细胞后涂抗性板,挑取单克隆菌以16M野生株为对照,同时16S为内参进行PCR(图5),结果表明,构建的过表达株扩增的条带比野生株条带亮,表明选取的单克隆菌模板DNA浓度高于野生株,过表达株构建成功。
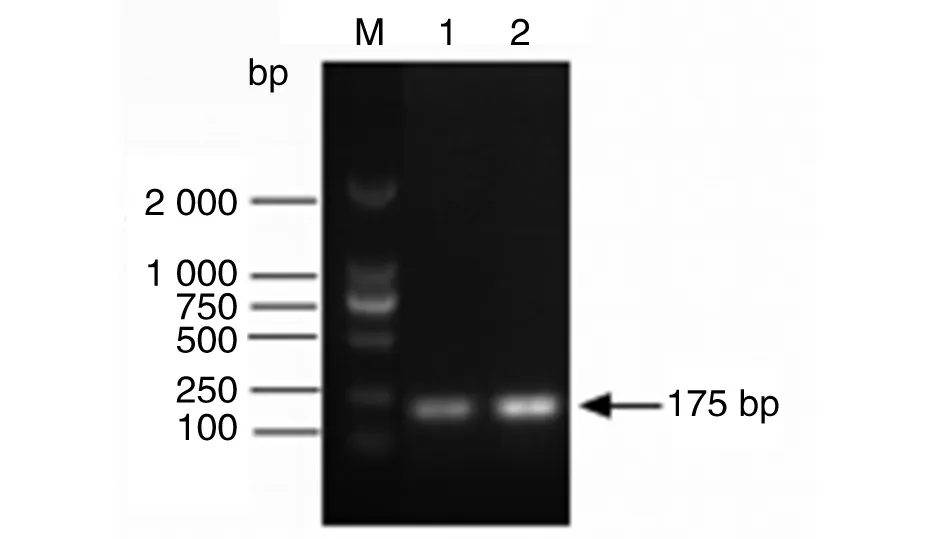
1: 16M; 2: 16M-BSR1526; M: DL2000 marker.图5 BSR1526过表达株构建鉴定图Fig.5 Verification of BSR1526 overexpression strain
2.5 16M-△BSR1526 和16M-BSR1526的RT-PCR验证 图6显示,16M-△BSR1526未扩增出BSR1955,16M-BSR1526扩增出亮度大于野生株16M的条带,表明16M-△BSR1526中缺失BSR1526转录,而16M-BSR1526中BSR1526转录水平增高,BSR1526过表达株构建成功。

图6 16M-△BSR1526 和16M-BSR1526的验证Fig.6 Verification of 16M-△BSR1526 and 16M-BSR1526 by RT-PCR
2.6 生长曲线 从图7可以看出,对数生长期16M-△BSR1526和16M-BSR1526与野生株16M的生长曲线类似,到达稳定期后突变株和过表达株的OD600值低于野生株。表明BSR1526的缺失或过表达影响了布鲁氏菌的生长。
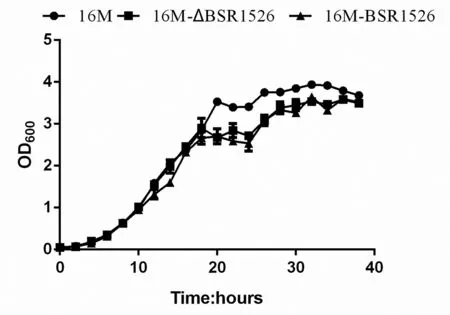
图7 野生株、突变株和过表达株的生长曲线Fig.7 Growth curve of 16M, 16M-△BSR1526 and 16M-BSR1526
2.7 从图8中可以看出,热应激和高渗透压条件下16M-△BSR1526和16M-BSR1526的存活率与野生株相比差异有统计学意义。在热应激、氧化压力及酸性条件下,16M-△BSR1526的存活率明显低于野生株16M。表明BSR1526缺失或过表达影响了布鲁氏菌适应胞内环境,BSR1526缺失或过表达后其抵抗热应激、氧化压力及酸性环境的能力下降。

图8 不同的刺激条件下16M、16M-△BSR1526和16M-BSR1526的存活率Fig.8 Intracellular survival of 16M, 16M-△BSR1526 and 16M-BSR1526 under stress conditions
2.8 小鼠毒力实验 从图9中可以看出,各个时间点脾脏中的过表达株的载菌量与野生株的载菌量无差异;而在第14 d,突变株小鼠脾脏中的载菌量明显低于野生株和过表达株,表明突变BSR1526后布鲁氏菌适应胞内环境的能力下降,被机体快速清除。提示BSR1526影响了布鲁氏菌的毒力。

图9 感染后小鼠脾脏载菌量Fig.9 Survival of 16M, 16M-△BSR1526 and 16M-BSR1526 in spleen of infected BALB/c mice
2.9 BSR1526靶基因的预测 BSR1526调控了45个靶标基因,其中25个位于布鲁氏菌I号染色体,20个位于布鲁氏菌II号染色体。这些靶基因编码的蛋白包括20个胞质蛋白、6个胞质膜蛋白、1个外膜蛋白、1个周质蛋白、17个蛋白定位尚不清楚。根据COG功能分类,这些靶基因主要包括碳水化合物的运输和代谢、无机离子转运和代谢、能量生产和转换、转录调控、功能预测、信号转导以及功能未知的蛋白等,它们可能参与布鲁氏菌胞内生存调控。
3 讨 论
作为一种胞内致病菌,适应胞内生存是布鲁氏菌致病的关键环节[16]。胞内存活和繁殖是布鲁氏菌主要的毒力特征,其自身拥有一套抵抗和适应巨噬细胞内杀菌或抑菌的能力,而这种适应主要通过调控基因的协调表达来实现[17]。最近研究表明,致病菌进入宿主后,感受环境改变并通过RNA调整自身毒力基因表达,有效地促进细菌的生长、繁殖,从而利于胞内菌在宿主胞内的侵染和存活[18]。迄今已发现超过200个sRNA,大部分集中于模式细菌如大肠杆菌、肠炎沙门氏菌等。随着研究的深入,越来越多的研究转向致病菌,如霍乱弧菌、产单核细胞李斯特菌、铜绿假单胞菌、结核分枝杆菌、沙门氏菌、肺炎链球菌和金黄色葡萄球菌等。这些病原菌中已证实sRNAs在转录后调控基因的表达,并在毒力调控中发挥作用[19-23]。目前关于布鲁氏菌sRNA的研究还处于起步阶段,了解sRNAs在布鲁氏菌胞内生存中的作用及其调控机制对理解布鲁氏菌致病机制至关重要。
研究表明,布鲁氏菌中存在sRNAs分子伴侣Hfq,且Hfq与布鲁氏菌对高氧、酸等胞内环境的适应和毒力有关[24]。Caswell CC等首次从B.abortus2308中鉴定出2个sRNAs(AbcR1 和 AbcR2),同时突变2个sRNAs导致布鲁氏菌的毒力下降,并且AbcR调控了大量靶基因,包括氨基酸代谢相关、多肽转运和代谢等,发现AbcR能加速靶标基因mRNA的降解[25]。国内Hao Dong等采用SIPHT 、NAPP软件对B.abortus2308在标准培养条件下的sRNA进行预测,新发现112种sRNA。对其中20个新发现的sRNA采用RT-PCR验证了7种sRNA的存在[26]。同时他们还发现一个调控铁代谢基因hemH的sRNA(BsrH),过表达BsrH影响了B.abortus2 308在应激条件下的生存,但对胞内以及小鼠体内的繁殖无影响[27]。本实验室预测了B.melitensis16M中21个sRNA,其中8个sRNA得到验证。同时我们还对其中的一个新的sRNA进行了初步研究。结果表明,BSR0602参与调控了布鲁氏菌的胞内生存和毒力[28]。Saadeh 等人通过Northern blot和RT-PCR对B.suis中发现的33个与Hfq相关的候选sRNA进行鉴定,验证出10个sRNA[29]。本实验室测序预测了羊种布鲁菌16M中1 321个sRNA,对其中一个涉及布鲁氏菌毒力相关的sRNA即BSR0441进行研究。结果表明,BSR0441参与调控布鲁氏菌在体内的生存和繁殖。同时发现,BSR0441的缺失和过表达影响了其靶基因的转录[11]。以上研究都表明,布鲁氏菌中存在大量sRNAs,并且这些sRNAs间接或直接参与调控布鲁氏菌胞内存活。
本研究中,经Northern blot和RT-PCR验证了布鲁氏菌中BSR1526的存在,BSR1526在酸性刺激条件下的转录水平高于其他刺激条件下的转录。本研究与郭英飞等人的研究结果相似,都表明sRNA在不同刺激条件下存在转录[30]。为进一步研究BSR1526在布鲁氏菌胞内生存中的作用,我们采用融合PCR构建BSR1526缺失株[31],通过RT-PCR验证缺失株中不存在BSR1526转录。将BSR1526基因插入到多拷贝质粒pBBR1-MCS4中,并将该质粒电转入16M中,构建过表达株16M-BSR1526。然后对16M-△BSR1526和16M-BSR1526的表型进行研究,结果显示,BSR1526影响了布鲁氏菌的生长能力。之前报道sRNA在细菌适应温度、氧化应激、pH等环境变化时起着重要的调控作用。如大肠杆菌的sRNA就是一种温度感应器,低温时DsrA 大量表达,诱导基因进行调控[32]。因此,我们对16M、16M-△BSR1526和16M-BSR1526在不同环境压力条件下的存活率进行进一步研究。结果表明,BSR1526缺失或过表达影响了布鲁氏菌适应胞内环境的能力,缺失后抵抗热应激、氧化压力及酸性环境的能力下降。小鼠毒力实验表明,缺失BSR1526后布鲁氏菌在第14 d在小鼠脾脏内的细菌量显著下降,表明BSR1526影响了布鲁氏菌的毒力。综合以上研究,表明 BSR1526在布鲁氏菌的胞内生存以及毒力中起着重要作用。
细菌sRNA主要通过不完全碱基配对与靶标mRNA结合,进而影响靶标mRNA的转录、翻译或稳定性发挥调控作用[4,28,33]。本实验对BSR1526的靶标基因进行了预测,发现BSR1526调控了45个靶标基因,这些靶基因在布鲁氏菌2条染色体中都有分布。BSR1526可能通过调控这些靶基因的表达来调控布鲁氏菌的胞内生存和毒力。45个靶基因中,BMEII0349,BMEII0703,BMEII0764与我们前期发现的BSR0441的靶基因一致,我们推测这3个靶基因可能同时受BSR0441和BSR1526调控[9]。后续我们将进一步验证BSR1526与靶基因之间的调控关系,分析BSR1526如何通过与靶标基因相互作用进而调控布鲁氏菌的胞内生存和毒力,更好的理解BSR1526在布鲁氏菌胞内生存以及毒力中扮演的角色。
[1] Altuvia S. Regulatory small RNAs: the key to coordinating global regulatory circuit[J]. J Bacteriol, 2004, 186 (20): 6679-6680. DOI: 10.1128/JB.186.20
[2] Xiang S, Fruehauf J, Li CJ. Short hairpin RNA-expressing bacteria elicit RNA interference in mammals[J]. Nat Biotechnol, 2006, 24(6): 697-702. DOI: 10.1038/nbt1211
[3] Van Puyvelde S, Steenackers HP, Vanderleyden J. Small RNAs regulating biofilm formation and outer membrane homeostasis[J]. RNA Biol, 2013, 10(2): 185-191. DOI: 10.4161/rna.23341
[4] Waters LS, Storz G. Regulatory RNAs in bacteria[J]. Cell, 2009, 136(4): 615-628. DOI: 10.1016/j.cell.2009.01.043
[5] Argaman L, Altuvia S. fhlA repression by OxyS RNA: kissing complex formation at two sites results in a stable antisense2target RNAcomplex[J]. J Mol Biol, 2000, 300(5): 1101-1112. DOI: 10.1006/jmbi.2000.3942
[6] Tu KC, Bassler BL. Multiple small RNAs act additively to integrate sensory information and control quorum sensing inVibrioharveyi[J]. Genes Dev, 2007, 21(2): 221-233. DOI: 10.1101/gad.1502407
[7] Storz G, Vogel J, Wassarman KM. Regulation by small RNAs in bacteria: expanding frontiers[J]. Mol Cell, 2011, 43(6): 880-891. DOI: 10.1016/j.molcel.2011.08.022
[8] Becker A, Overl?per A, Schlüter JP, et al. Riboregulation in plant-associated α-proteobacteria[J]. RNA Biol, 2014, 11(5): 550-562. DOI: 10.4161/rna.29625
[9] Zhong ZJ, Yu S, Xu J, et al. Research progress onBrucellacomparative genomics[J]. Chin J Zoonoses, 2011, 27(4): 346-350. (in Chinese)
钟志军,于爽,徐杰,等. 布鲁氏菌比较基因组学研究进展[J]. 中国人兽共患病学报, 2011, 27(4): 346-350.
[10] Li RL, Chen Y. The functions and mechanisms of small non-coding RNAs in bacteria: research progress[J]. Chin J Microecol, 2014, 11(26): 1359-1361. (in Chinese)
李瑞莲,陈杨. 细菌非编码小RNA功能及作用机制研究进展[J]. 中国微生态学杂志, 2014, 11(26) : 1359-1361.
[11] Zhong Z, Xu X, Li X, et al. Large-scale identification of small noncoding RNA with strand-specific deep sequencing and characterization of a novel virulence-related sRNA inBrucellamelitensis[J]. Sci Rep, 2016, 6: 25123. DOI: 10.1038/srep25123
[12] Elzer PH, Kovach ME, Phillips RW, et al.Invivoandinvitrostability of the broad-host-range cloning vector pBBR1MCS in sixBrucellaspecies[J]. Plasmid, 1995, 33(1): 51-57. DOI: 10.1006/plas.1995.1006
[13] Levin JZ, Yassour M, Adiconis X, et al. Comprehensive comparative analysis of strand-specific RNA sequencing methods[J]. Nat Methods, 2010, 7(9): 709-715. DOI: 10.1038/nmeth.1491
[14] Yoder Himes DR, Chain PS, Zhu Y, et al. Mapping the Burkholderia cenoce pacia niche response via high-throughput sequencing[J]. Proc Natl Acad Sci U S A, 2009, 106(10): 3976-3981. DOI: 10.1073/pnas.0813403106
[15] Beckmann BM, Grunweller A, Weber MH, et al. Northern blot detection of endogenous small RNAs (approximately 14 nt) in bacterial total RNA extracts[J]. Nucleic Acids Res, 2010, 38(14): e147. DOI: 10.1093/nar/gkq437
[16] Starr T, Child R, Wehrly TD, et al. Selective subversion of autophagy complexes facilitates completion of theBrucella[J]. Cell Host Microbe, 2012, 11(1): 33-45. DOI: 10.1016/j.chom.2011.12.002
[17] Dong BM, Wang JL, Tang N, et al. Research progress on pathogenic mechanism and cellular immunity ofBrucella[J]. Chin J Zoonoses, 2012, 28(6): 635-639. (in Chinese)
董炳梅,王金良,唐娜,等. 布鲁氏菌的致病机制与细胞免疫机制研究进展[J]. 中国人兽共患病学报, 2012, 28(6): 635-639.
[18] Gripenland J, Netterling S, Loh E, et al. RNAs: regulators of bacterial virulence[J]. Nat Rev Microbiol, 2010, 8(12): 857-866. DOI: 10.1038/nrmicro2457
[19] Arnvig KB, Young DB. Identification of small RNAs inMycobacteriumtuberculosis[J]. Mol Microbiol, 2009, 73(3): 397-408. DOI: 10.1111/j.1365-2958.2009.06777.x
[20] Tsui HC, Mukherjee D, Ray VA, et al. Identification and characterization of noncoding small RNAs inStreptococcuspneumoniaeSerotype2 Strain D39[J]. J Bacteriol, 2010, 192(1): 264-279. DOI: 10.1128/JB.01204-09
[21] Pfeiffer V, Papenfort K, Lucchini S, et al. Coding sequence targeting by MicC RNA reveals bacterial mRNA silencing downstream of translational initiation[J]. Nat Struct Mol Biol, 2009, 16(8): 840-846. DOI: 10.1038/nsmb.1631
[22] Mraheil MA, Billion A, Mohamed W, et al. The intracellular sRNA transcriptome of Listeria monocytogenes during growth in macrophages[J]. Nucleic Acids Res, 2011, 39(10): 4235-4248. DOI: 10.1093/nar/gkr033
[23] Oliver HF, Orsi RH, Ponnala L, et al. Deep RNA sequencing ofL.monocytogenesreveals overlapping and extensive stationary phase and sigma B-dependent transcriptomes, including multiple highly transcribed noncoding RNAs[J]. BMC Genomics, 2009, 10: 641. DOI: 10.1186/1471-2164-10-641
[24] Robertson GT, Roop RM Jr. TheBrucellaabortushost fact or I (HF-I) protein contributes to stress resistance during stationary phase and is a major determinant of virulence in mice[J]. Mol Microbiol 1999, 34(4): 690-700.
[25] Caswell CC, Gaines JM, Ciborowski P, et al. Identification of two small regulatory RNAs linked to virulence inBrucellaabortus2308[J]. Mol Microbiol, 2012, 85(2): 345-360. DOI: 10.1111/j.1365-2958.2012.08117.x
[26] Dong H, Peng X, Wang N, et al. Identification of novel sRNAs inBrucellaabortus2308[J]. FEMS Microbiol Lett, 2014, 354(2): 119-125. DOI: 10.1111/1574-6968.12433
[27] Peng X, Dong H, Wu Q. A new cis-encoded sRNA, BsrH, regulating the expression of hemH gene inBrucellaabortus2308[J]. FEMS Microbiol Lett, 2015, 362(2): 1-7. DOI: 10.1093/femsle/fnu017
[28] Wang Y, Ke Y, Xu J, et al. Identification of a novel small non-coding RNA modulating the intracellular survival ofBrucellamelitensis[J]. Front Microbiol, 2015, 6: 164.DOI: 10.3389/fmicb.2015.00164
[29] Saadeh B, Caswell CC, Chao Y, et al. Transcriptome-wide identification of Hfq-associated RNAs inBrucellasuisby deep sequencing[J]. J Bacteriol, 2015, 198(3): 427-435. DOI: 10.1128/JB.00711-15
[30] Guo YF, Wang YF, Gong CL, et al. Identification of novel transcripts and sRNA ofBrucellamelitensis by RNA-Seq[J]. Chin J Zoonoses, 2015, 31(3): 216-221. DOI: 1002-2694.2015.03.006 (in Chinese)
郭英飞,王玉飞,龚春丽,等. 基于RNA-Seq的羊种布鲁氏菌新转录本与非编码RNA鉴定[J]. 中国人兽共患病学报, 2015, 31(3): 216-221.
[31] Bai YX, Yang Y, Wang YF, et al. Construction ofBrucelladeletion mutant by using T cloning vector[J]. CN Biotechnol, 2010, 30(9): 62-67. (in Chinese)
白耀霞,杨 毅,王玉飞,等. 利用T载体克隆快速构建布鲁氏菌缺失突变株[J]. 中国生物工程杂志, 2010, 30(9): 62-67.
[32] Dudin O, Lacour S, Geiselmann J. Expression dynamics of RpoS/Crl-dependent genes inEscherichiacoli[J]. Res Microbiol, 2013, 164(8): 838-847. DOI: 10.1016/j.resmic.2013.07.002
[33] Arnvig K, Young D. Non-coding RNA and its potential role inMycobacteriumtuberculosis pathogenesis[J]. RNA Biol, 2012, 9(4): 427-436. DOI: 10.4161/rna.20105
Construction of mutant and overexpression strains with small noncoding RNA BSR1526 and study of its virulence inBrucella
XU Xiao-yang1, OU Hong-ping2, PENG Guang-neng1, LEI Shuang-shuang1,TIAN Yi-nan1, CAO Xue-feng1, WANG Yu-fei4,6,YAN Guang-wen6,ZHONG Zhi-jun1
(1.CollegeofVeterinaryMedicine,SichuanAgriculturalUniversity,Chengdu611130,China;2.ChengduVocationalCollegeofAgriculturalScienceandTechnology,Chengdu611130,China;3.DepartmentofLaboratoryMedicine,theGeneralHospitalofChinesePeople'sArmedPoliceForces,Beijing100039,China;4.CollegeofVeterinaryMedicine,NajingAgricalturalUniversity,Najing210095,China)
To evaluate the roles of small noncoding RNA BSR1526 inBrucellaintracellular survival, the mutant and overexpression strains of BSR1526 were constructed and their survival capabilities under stresses that simulate intracellular environment and that of in mice were analyzed. Firstly, BSR1526 was verified by Northern blot and its transcription under stress conditions was verified by RT-PCR. The mutant strains of BSR1526 were constructed by fusion PCR. BSR1526 was inserted into the plasmid pBBR1-MCS4, which transformed intoBrucellamelitensis16M to generate the overexpression strain16M-BSR1526. At last, the survival capabilities of 16M-△BSR1526 and 16M-BSR1526 in different stimuli resembling thatBrucellaprobably encounters during infection and mice were analyzed. Our results showed that BSR1526 was transcribed inBrucella,and with different transcription levels under the stress conditions. Deletion or overexpression of BSR1526 affected the growth ofBrucellainvitro. The 16M-△BSR1526 showed reduced survival under heat shock, oxidative stress and acidic conditions, and significant reduction in mice, indicating that BSR1526 influenced the virulence ofBrucella. In summary, small noncoding RNA BSR1526 affects intracellular survival ability ofBrucellamelitensis16M. The ability adapting to hostile environments and virulence in mice with mutant BSR1526 strain was decreased.
Brucella; non-coding small RNA; BSR1526; intracellular survival
Zhong Zhi-jun, Email: zhongzhijun488@126.com
10.3969/j.issn.1002-2694.2016.012.008
国家自然科学基金资助项目(No.31000548);成都大熊猫繁育研究基金会项目(No.CPF2015-4);北京市自然基金(No.6122030);北京市科技新星资助项目(No.Z131102000413062)联合资助。
钟志军,Email:zhongzhijun488@126.com; 王玉飞,Email:tosya@163.com
1.四川农业大学动物医学院,动物疫病与人类健康四川省重点实验室, 成都 611130; 2.成都农业科技职业学院,成都 611130; 3.武警总医院检验科,北京 100039; 4.南京农业大学动物医学院,南京 216095
R378
A
1002-2694(2016)12-1083-08
2016-05-09;
2016-07-25
Supported by the National Natural Science Foundation of China (No. 31000548), the Chengdu Giant Panda Breeding Research Foundation (No. CPF2015-4), the Natural Science Foundation of Beijing (No. 6122030), and the Beijing Novo Program (No. Z131102000413062).
