长穗偃麦草醇溶蛋白的遗传多样性分析
2016-02-06吴珊
吴 珊
(成都农业科技职业学院,四川 温江 611130)
长穗偃麦草醇溶蛋白的遗传多样性分析
吴 珊
(成都农业科技职业学院,四川 温江 611130)
为进一步发掘长穗偃麦草[Thinopyrumelongatum(Host) D.R.Dewey]优异种质,采用酸性聚丙烯酰胺凝胶电泳法(A-PAGE),对分别来自伊朗、土耳其、美国等7个国家的18个长穗偃麦草居群醇溶蛋白的遗传多样性进行了分析。这18个居群中共含有70种醇溶蛋白带型,平均多态性位点百分比(PPB)为75.40%(变幅41.43%~92.86%),平均Nei基因多样性指数(h)为0.241 1(变幅0.127 0~0.332 2),平均Shannon 信息指数(SI)为 0.361 1(变幅0.195 8~0.497 0)。其中17.35%的遗传变异来自长穗偃麦草居群间,82.65%的遗传变异来自居群内。此外,聚类结果显示,所选材料被分为5组,且聚类结果与材料的地理来源并无密切相关性。以上结果表明,长穗偃麦草的醇溶蛋白位点存在丰富遗传多样性,可用于改良小麦加工品质。
长穗偃麦草; 居群; 醇溶蛋白; 遗传多样性; 聚类分析
醇溶蛋白是小麦种子蛋白的主要成分之一,其组成与含量决定面团的黏着性[1-2],进而影响小麦加工品质[3]。一些学者对醇溶蛋白与小麦品质、抗病抗逆等性状之间的连锁进行分析发现,黑麦等醇溶蛋白组分与上述性状存在关联性[4-5]。醇溶蛋白等位基因间的差异主要体现在条带数量、相对迁移率、条带亮度和分子质量大小等方面。醇溶蛋白对加工品质也起重要作用,因此研究其在小麦近缘属种中的遗传多样性具有重要意义[6-8]。
长穗偃麦草(Thinopyrumelongatum)是小麦族偃麦草属的野生草本植物。长穗偃麦草有二倍体、四倍体和十倍体3种倍性类型。二倍体长穗偃麦草的染色体组构成为EE,是基本染色体组。长穗偃麦草具有高抗锈病、赤霉病和白粉病,还具有大穗、多花、抗低磷营养胁迫、抗寒、抗旱等优良性状[9-11]。现代小麦育种通常单株选种,导致栽培小麦的遗传基础趋向狭窄。因此,通过人工杂交引进外源优异基因具有重要的意义。二倍体长穗偃麦草作为普通小麦的一个重要野生近缘种,已经被广泛地应用于小麦品质及产量的改良[12]。本研究拟通过A-PAGE技术检测来自7个国家18个居群的二倍体长穗偃麦草居群间和居群内醇溶蛋白多样性,探讨长穗偃麦草种内不同居群间的遗传关系,为高效利用种质资源,改良普通小麦品质奠定基础。
1 材料和方法
1.1 供试材料
18个居群长穗偃麦草均由美国种质资源信息库惠赠。原产地分别为土耳其、伊朗、美国、中国、加拿大等国家(表1)。对照为普通小麦(T.aestivum)中国春。
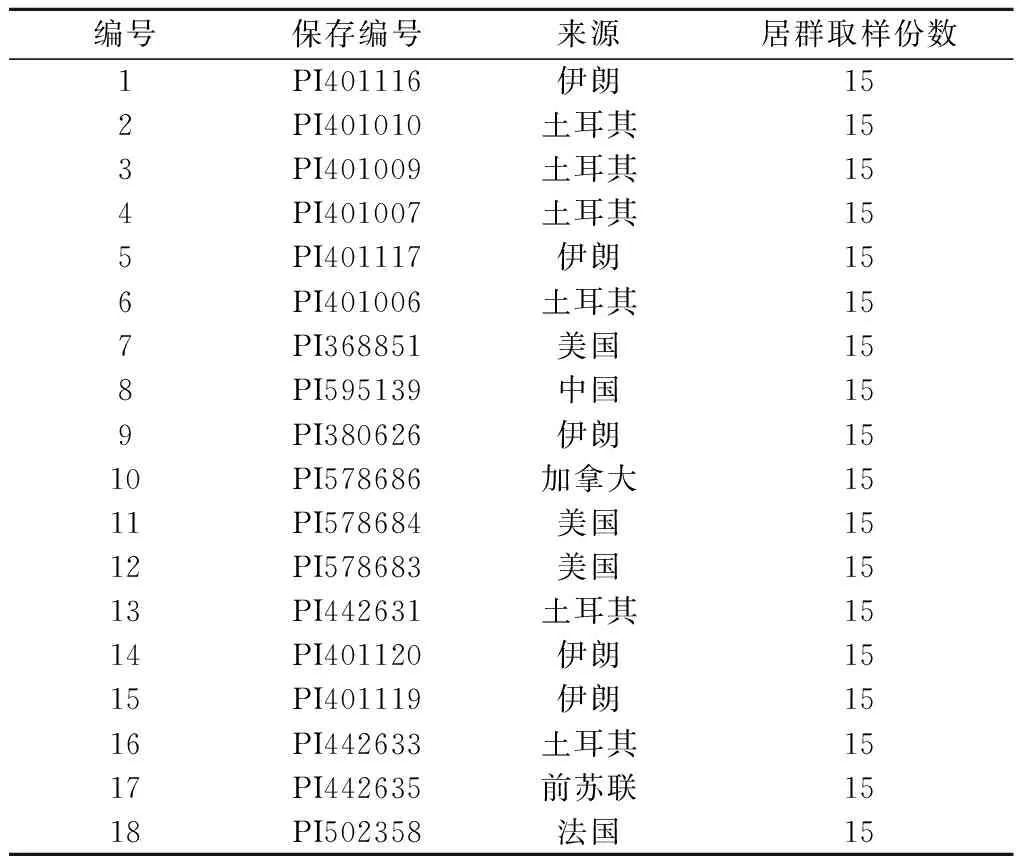
表1 供试材料
1.2 方法
1.2.1 醇溶蛋白提取和A-PAGE分析 种子磨粉后称100 mg,于1.5 mL 离心管按1 mg∶ 10 μL 比例加入样品提取液(25% α-氯乙醇,0.05% 甲基绿),振荡混匀,室温过夜浸提。电泳前3 000 r/min 离心2 min,取10 μL 上清液点样。采用SITA 于1986 年颁布的A-PAGE(pH值3.1)标准程序电泳分析醇溶蛋白。
1.2.2 醇溶蛋白指纹图谱数据转换 采用中国农业科学院作物品种资源研究所计算机室和浙江大学合作设计的分析软件GEL 2.0,对电泳图谱进行数据转换,生成0-1数据库。
1.2.3 统计分析 利用生成的0-1数据库进行各种多样性评价参数的统计分析。应用POPGENE version 1.31软件[13]计算各居群的多态性位点数(Npb)、多态性位点百分比(PPB)、Nei基因多样性指数(h),Shannon信息指数(SI),居群间的Nei遗传距离(D)、遗传一致度(GI)、遗传分化系数(Gst)和基因流(Nm)、总群体基因多样度(Ht)、各群体基因多样度(Hs)。根据Nei遗传距离,对7个国家18个居群的长穗偃麦草进行了UPGMA聚类分析。
2 结果与分析
2.1 长穗偃麦草居群内醇溶蛋白遗传多样性
A-PAGE检测结果表明,二倍体长穗偃麦草材料的醇溶蛋白有丰富的多样性(图1、表2)。在供试材料中,共检测到多态条带70条,多态条带比例为100%。
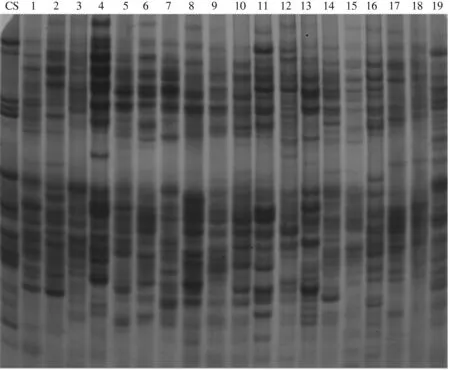
CS:中国春; 1—15:PI380626; 16—19:PI578686图1 部分供试长穗偃麦草的A-PAGE图谱
从Npb、PPB、及SI4个多样性参数来看,除来自法国编号为PI502358居群外的17个居群内变异都比较大,居群间的遗传差异比较小。居群PI368851、居群PI442635多态性位点数(Npb)较多,分别为65和63个,居群PI502358最少,仅为29个,平均为53个。居群PI368851的PPB最高(92.86%),居群PI502358最小(41.43%), PI401116、PI401006、PI595139、PI578683 四个居群Npb和PPB相同(80.00%),平均为75.40%。居群PI368851 ShannonSI最高(0.497 0),而居群PI502358最低(0.195 8),平均为0.361 1。居群PI368851h最大(0.332 2),居群PI502358最小(0.127 0),平均为0.241 1。按照各居群遗传变异由高向低的顺序,4个参数表现一致,可见不同参数得到的遗传变异的大小是一致的。

表2 长穗偃麦草居群内醇溶蛋白遗传多样性
2.2 长穗偃麦草居群间醇溶蛋白遗传分化
利用POPGENE version 1-31软件计算的总群体基因多样度、各群体基因多样度、遗传分化系数和基因流如表3所示。长穗偃麦草居群基因分化系数为 0.173 5,说明17.35%的遗传变异存在于居群之间,82.65%的变异存在于居群内。Wright[14]认为,居群间基因流大于1能发挥其均质化作用,反之小于1 则表明基因流成为遗传分化的主要原因。长穗偃麦草居群间基因流为2.382 4,大于1,说明基因流足以抵制居群由遗传漂变引起的遗传结果,维持遗传变异的多样性,防止近交衰退。

表3 长穗偃麦草居群间醇溶蛋白遗传多样性
2.3 长穗偃麦草遗传距离和聚类分析
居群间的遗传一致度与遗传距离列于表4。基于Nei’s遗传一致度和遗传距离可进一步分析居群间的遗传分化程度[15]。长穗偃麦草18个居群醇溶蛋白的平均遗传一致度和平均遗传距离分别为0.936 8和0.065 6。来自美国的PI578684居群和来自土耳其的PI401010居群的遗传一致度最大(0.985),其遗传距离也最近(0.015)。来自加拿大的PI578686居群和来自法国PI502358居群的遗传一致度最小,为0.853,其遗传距离也最远,为0.158。
根据长穗偃麦草居群间的遗传距离,采用UPG-MA法对18个居群进行聚类分析(图2),从图中可以看出,供试材料在遗传距离系数为2.67的水平上可以聚为5大类:来自伊朗的4个居群和来自土耳其的3个居群共同聚为类Ⅰ;类Ⅱ包括土耳其和美国各2个居群以及加拿大、中国和伊朗各1个居群;类Ⅲ为来自美国的PI368851和前苏联的PI442635居群;来自土耳其的PI401006居群和来自法国的PI502358居群分别单独聚为类Ⅳ和类Ⅴ。

表4 长穗偃麦草居群间遗传一致度与遗传距离

续表4 长穗偃麦草居群间遗传一致度与遗传距离
注:对角线上方为Nei遗传一致度,对角线下方为遗传距离。
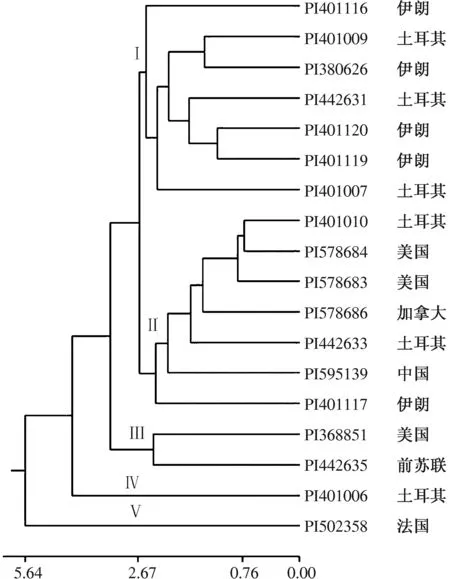
图2 18个长穗偃麦草居群间醇溶蛋白的遗传距离聚类分析
3 结论与讨论
醇溶蛋白条带多样性高且遗传稳定,其电泳条带的多样性可以反映材料之间的关系。陈玉清等[16]在40个四川小麦主栽品种中检测出38种醇溶蛋白带型,共分离出40条多态性带纹;兰秀锦等[17]对36份来自中国和中东的节节麦醇溶蛋白进行分析时共产生19种带型;杨瑞武等[18]对小麦族赖草属20种和1亚种共45份材料进行醇溶蛋白分析,共出现43种不同的带谱,从中分离出的38条带纹多态性高达100%;李桂江等[19]对133份野生二粒小麦的醇溶蛋白进行分析,得到122种谱带类型,电泳分离出77条不同的带纹;曾兴权等[20]对西藏小麦、杨瑞武等[21]对波兰小麦及其他小麦近缘属种的醇溶蛋白所做的研究均表明,利用A-PAGE法可以较好地研究小麦近缘属种材料间醇溶蛋白的遗传多态性。李培英等[22]研究表明,偃麦草也具有种质资源遗传多样性。本研究发现,长穗偃麦草醇溶蛋白带型遗传变异较大,18个居群共检测到70种带型。长穗偃麦草具有丰富的醇溶蛋白多样性,可以利用其与栽培品种杂交,丰富品种的遗传基础。刘华等[23]对我国小麦主要栽培品种、古老品种及国外引进品种共90份材料聚类分析发现,大多数系谱相同或相近的材料遗传相似系数很近,能聚在一起,反之亦然。许占友[24]研究表明,品种间醇溶蛋白遗传差异并不等于其地理来源的差异。李桂江等[19]对来自以色列16个地区的野生二粒小麦的醇溶蛋白分析发现,野生二粒小麦的醇溶蛋白与其来源地相关。张小英等[25]、徐黎黎等[26]、Wang等[27]和陈仕勇等[28]分别在研究密穗小麦、东方小麦、硬粒小麦和野生新麦草时得到了类似的结果。本研究表明,聚类结果与其地理来源并不完全一致,一般来源于同一国家居群间的遗传距离相对较小。绝大多数伊朗和土耳其的材料被聚在一起,但仍有部分材料存在较大差异,与其他材料聚在一起;其他来源地的材料在聚类中散布。长穗偃麦草的聚类结果并未严格地遵循材料来源地与聚类结果一致的规律,来自不同国家的材料聚在一起,表明基于醇溶蛋白的聚类结果受材料来源地的影响,但没有必然联系。同时注意到来自伊朗和土耳其的居群存在丰富的遗传多样性,推测其为长穗偃麦草的遗传多样性中心,这可能是由于长穗偃麦草起源于欧亚大陆,其他来源地的材料或多或少与其有血缘关系。
[1] Dachkevitch T,Redaelli R,Biancardi A M,etal. Gene-tics of gliadins coded by the group 1 chromosomes in the high-quality bread wheat cultivar Neepawa[J].Theor Appl Genet,1993,86(2/3):389-399.
[2] Metakovsky E V,Wrigley C W,Bekes F,etal.Gluten polypeptides as useful genetic markers of dough quality in Australian wheats[J]. Aust J Agric Res,1990,41(2):289-306.
[3] Metakovsky E V,Annicchiarico P,Boggini G,etal.Relationship between gliadin alleles and dough strength in Italian bread wheat cultivars[J].J Cereal Sci,1997,25(3):229-236.
[4] Payne P I.Genetics of wheat storage proteins and the effect of allelic variation on bread-making quality[J].Ann Rev Phyto,1987,38(1):141-153.
[5] Branlard G,Dardevet M,Saccomano R,etal.Genetic diversity of wheat storage proteins and bread wheat quality[M]//Wheat in a global environment. Netherlands:Springer 2001:157-169.
[6] 高艾英,张昊,湛平,等.小麦醇溶蛋白等位基因研究进展[J]. 山东农业科学,2007(1):25-28.
[7] Bietz J A. Genetic and biochemical studies of nonenzymatic endosperm proteinsv[J]. Wheat and Wheat Improv Agron Mono,1987,13(2):215-241.
[8] 张学勇,杨欣明,董玉琛. 醇溶蛋白电泳在小麦种质资源遗传分析中的应用[J].中国农业科学, 1995,28(4):25-32.
[9] 刘爱峰. 小偃麦种质系的鉴定及其抗病基因的染色体定位和SSR分子标记[D].泰安: 山东农业大学,2007.
[10] 方普,集临. 小偃麦细胞遗传学[M]. 北京: 中国农业科技出版社,1994:1-3.
[11] 王小华,陈春环,王长有,等. 普通小麦-长穗偃麦草抗白粉病异代换系的分子细胞学研究[J]. 麦类作物学报,2015,35(5): 4-8.
[12] 郑有良,周永红,魏育明. 小麦分子生物学应用研究[M]. 成都:四川科学技术出版社,2001.
[13] Yehf C, Boyle T J B. Population genetic analysis of co-dominant and dominant markers and quantitative traits [J].Belg J Bot,1997,129:157.
[14] Wright S. Evolution in Mendelian population [J].Genetics,1931,16(2):91-159.
[15] Nei M. Estimation of average heterozygosity and genetic distance from a small number of individuals [J]. Genetics,1978,89:583-590.
[16] 陈玉清,郑有良,魏育明. 四川小麦主栽品种醇溶蛋白遗传变异分析[J].四川农业大学学报,1999,17(3):254-260.
[17] 兰秀锦,魏育明,王志容. 中国节节麦与中东节节麦的醇溶蛋白遗传多样性比较研究[J].四川农业大学学报,1999,17(3):245-248.
[18] 杨瑞武,魏秀华,周永红,等. 赖草属植物醇溶蛋白的遗传多样性[J].云南植物研究,2004,26(1):103-110.
[19] 李桂江,伍碧华,侯永翠,等. 野生二粒小麦醇溶蛋白遗传多样性分析[J].麦类作物学报,2004,24(3):29-33.
[20] 曾兴权,韦泽秀. 西藏小麦地方品种醇溶蛋白的遗传多样性分析[J].西藏农业科技,2011,33(4):7-12.
[21] 杨瑞武,周永红,郑有良,等. 波兰小麦醇溶蛋白遗传差异及其与新疆稻麦的关系[J].麦类作物学报,2000,20(4):1-5.
[22] 李培英,孙宗玖,朱昊. 偃麦草种质资源遗传多样性的 RAPD 分析[J].中国草地学报,2012,34(3):12-20.
[23] 刘华,王宇生.小麦种质资源醇溶蛋白指纹图谱数据库的初步建立及应用[J].作物学报,1999,25(6):674-682.
[24] 许占友. 小麦分子遗传差异与杂种优势关系及其育性恢复基因分子标记的研究[D].北京:中国农业大学,1997.
[25] 张小英,李伟,魏育明,等.密穗小麦醇溶蛋白遗传多样性分析[J].西南农业学报,2005,18(6):680-683.
[26] 徐黎黎,李伟,魏育明,等.东方小麦醇溶蛋白遗传多样性分析[J]. 植物遗传资源学报,2005,6(2):195-199.
[27] Wang H Y, Wei Y M, Yan Z H,etal. Genetic variations of gliadin and HMW glutenins subunits in durum wheat [J].J Agr Biot,2006,14(5):721-727.
[28] 陈仕勇,周青平,张新全,等.野生新麦草种质醇溶蛋白遗传变异及地理分化研究[C]//中国草学会2013年学术年会论文集.天津: 中国草学会,2013.
Analysis of Genetic Diversity of Gliadins inThinopyrumelongatum
WU Shan
(Chengdu Vocational College of Agricultural Science and Technology, Wenjiang 611130, China)
In order to exploit the excellent germplasm resource ofThinopyrumelongatum(Host) D.R.Dewey, the gliadins of 18 populations, collected from Iran, Turkey, America and the other four countries, were analyzed and evaluated using A-PAGE methods. The results indicated that a total of 70 polymorphic bands were detected among the 18Thinopyrumelongatumpopulations, the average ofPPB,handSIwere 75.40% (varied range of 41.43%—92.86%), 0.241 1 (varied range of 0.127 0—0.332 2) and 0.361 1 (varied range of 0.195 8—0.497 0), respectively. 17.35% of molecular variation was among populations, and the great majority of genetic variation (82.65%) resided within populations. The cluster analysis indicated that all the accessions could be divided into five groups. There was no strong association between the clustering results and the geographical distribution. The above results showed that the gliadins ofThinopyrumelongatum(Host) D.R.Dewey had rich genetic diversity, and could be utilized to improve wheat processing quality.
Thinopyrumelongatum; population; gliadin; genetic diversity; cluster analysis
2015-09-21
国家自然科学基金项目(31171557)
吴 珊(1980 -),女,四川蒲江人,讲师,硕士,主要从事植物遗传育种及分子生物学研究。 E-mail:86015956@qq.com
S512.9
A
1004-3268(2016)03-0034-05
