7个草菇菌株的遗传多样性与生物学特性研究
2016-01-16,,,,,,
, , , , , ,
(1.广东省微生物研究所;省部共建华南应用微生物国家重点实验室;广东省菌种保藏与应用重点实验室;广东省微生物应用新技术公共实验室, 广州 510070;2.广东粤微食用菌技术有限公司, 广州 510070)
据文献记载,草菇栽培始于我国[1]。数十年的栽培技术与育种技术的发展,使得我国草菇品种众多,种源复杂。据全国食用菌品种认定委员会公告,经国家级认定的草菇品种至今尚只有1个(川草53)。可见现时国内流通的众多草菇品名尚处于未登记或未审核状态,菌种质量尚缺乏官方认证。要对各种草菇菌种进行准确的认证,首先要获得申请者所提供菌株的遗传多样性信息及生物学特性。
分子标记在食用菌栽培种或菌株之间的遗传多样性研究中得到广泛应用,但往往不同种类的分子标记对近缘菌株或栽培品种之间的差异有不同体现。例如22个商用白色双孢蘑菌株中,利用RAPD分析能直接展现出较好的多态性,而利用ITS分析则需要发掘各菌株序列的SNP才能体现较好的多态性[2]。在毛木耳和黑木耳研究中,应用ERIC-PCR能分辨出二者,但应用RAPD则不行[3]。在真姬菇的商业用和保藏菌株的研究中,基于ISSR的聚类和基于菌株本身农艺性状分类未能较好契合[4]。但某些生物学特性是能和分子标记的分类相契合的,例如在虫草无性分离物的研究中,其ERIC-PCR分析结果与各分离物体细胞不亲和性试验的结果具有较高的一致性[5]。结合不同的分子标记进行分析,可能会得到更精准的近缘分类结果。例如在20株榆黄蘑分别应用ISSR和SRAP时都展现较好的出多态性,但是结合两者后进行的聚类结果与菌株子实体形态分类及拮抗反应结果有更高的一致性[6]。
分子标记的多样性以及组合使用的多样性必然使遗传多样性信息更加丰富和复杂。目前我国公认有效的遗传多样性分析技术,部分被列为标准,如现行的国家标准“草菇菌种”(GB/T 23599—2009)中提及的ITS比对技术,“食用菌菌种真实性鉴定 酯酶同工酶电泳法”(NY/T 1097-2006)和“食用菌菌种区别性鉴定 拮抗反应”(NY/T 1845-2010)等。这些分类鉴定方法在区分属之间的菌株或品种时,有较好的分辨力。而在不同品种或不同菌株草菇之间,则尚未见到综合运用这些遗传多样性的研究方法进行分类。
在草菇的生物学特性和杂交育种研究中,同工酶分析、RAPD和SRAP分析都能较好地体现出菌株间[7]、亲子之间的差异性[8-9]。本研究从生理、蛋白质和基因3个水平上对7个馆藏草菇菌株进行了遗传多样性和生物学特性研究,并对其结果进行比较分析。
1 材料与方法
1.1 实验材料
本单位馆藏草菇菌株,分别记为V 1、V 2、V 3、……V 7,共7个菌株。
1.2 草菇菌丝形态观察
取各个菌株分别接种在平板培养基上,(32±1)℃下避光培养4 d后观察。
1.3 草菇菌丝体的扫描电镜观察
按1.2方法培养草菇菌丝体,培养天数为2~3 d,以靠近菌丝体边缘5 mm左右长度的环带区域为取材位置。取出的带琼脂的菌丝体块经脱水、叔丁醇置换、冷冻干燥、贴台、溅射Au后,使用扫描电镜(日立H-3000 N)进行观察并拍照。
1.4 草菇菌丝体拮抗试验
按三角形接种法把7个菌株的接种块按排列组合方式接种于平板培养基,在(32±1)℃下避光培养后观察是否存在拮抗现象。
1.5 草菇菌丝体蛋白提取
取液体培养基(改良马丁培养基)中培养6 d的新鲜草菇菌丝体,洗净吸干水分放入研钵中,液氮冻结,研磨成粉,加入适量含1%蛋白酶抑制剂混合物(生工,PL 026)的0.2 M磷酸钠缓冲液(pH=6.5)冰浴研磨至匀浆,4 ℃离心后所得上清液为草菇菌丝体蛋白提取液。
1.6 草菇菌丝体酯酶同工酶电泳分析
取1.5中得到的草菇菌丝体蛋白提取液为样品,参考标准“食用菌菌种真实性鉴定酯酶同工酶电泳法”(NY/T 1097-2006),稍作修正,进行电泳。凝胶显色后以凝胶成像系统拍照(英国UVItec)。使用软件UVIband识别并测量各条带的相对迁移率(Rf),列表纪录。泳道中各个迁移率位置上条带出现记为1,无则记为0。所得数据矩阵输入NTSYSpc(2.10 e),采用UPGMA法作树状聚类图。
1.7 草菇菌丝体基因组的ERIC-PCR试验
按1.5中描述取得新鲜菌丝体,60 ℃干燥。按Ezup柱式基因组DNA抽提试剂盒(生工生物,SK 8259)指示的步骤进行总DNA提取。所得DNA溶液作为PCR扩增模板。参照黄龙花等[10]的方法,以从华大购置的Oligo序列ERIC 2(5′-ATGTAAGCTCCTGGGGATTCAC-3)和ERICR(5′-AAGTAAGTGACTGGGGTGAGCG-3′)为引物,并采用Dream Taq Green PCR Master Mix混合液及其建议体系进行温度梯度PCR,筛选最佳退火温度,以最佳退火温度的反应体系对7个菌株模板进行PCR扩增,比较观察电泳后的产物条带。使用软件UVIband测量各条带的碱基对数,参照1.6对各菌株进行聚类分析。
1.8 草菇栽培
参照菌种国家标准(GB/T 23599—2009)相关条目制成草菇原种,取生长12 d原种下种。播种方式为混播,接种量为1包原种接入2筐栽培筐中。草菇栽培料为95%废棉、5%生石灰。栽培料堆制好后采用透气栽培筐栽培,每筐装入量控制在干料2.1 kg左右。栽培参数设置按廖世煌等[11]的方法加以调整进行。采菇时按每筐单独统计。
2 结果与分析
2.1 菌落外观特征与菌丝体扫描电镜观察
7个草菇菌株接种平板培养基,培养相同时间后,各菌株菌落呈现不同形态。从菌落类型划分,V 1和V 4基本呈匍匐型菌丝,其他则主要为气生型;从菌丝生长速度上看,V 4最慢,至结束培养时仍有大量空间未见菌丝覆盖。V 3和V 5稍慢,至结束培养时仍未长满培养基表面,其余菌株菌丝体均长满培养基。从菌丝形态来看,V 2和V 3菌丝整体浓密,呈短绒毛状。V 5、V 6和V 7粗壮菌丝明显,菌丝呈长丝状,其中V 5菌丝稀疏,V 6和V 7菌丝体较浓密。另外,V 1和V 2还呈现出明显的环状稀疏相间区域。
利用扫描电镜,在不同的放大倍数下观察了7个草菇菌株近菌落边缘区域部分的菌丝形态。视野内各个草菇菌株菌丝呈相互层叠、穿插或缠绕。各草菇菌株在50μm标尺视野(上排图片)内可见粗细不一的、有分支的菌丝。在10μm标尺视野(下排图片)内可见各菌株草菇菌丝顶端呈棒槌状隆起或钝圆状。
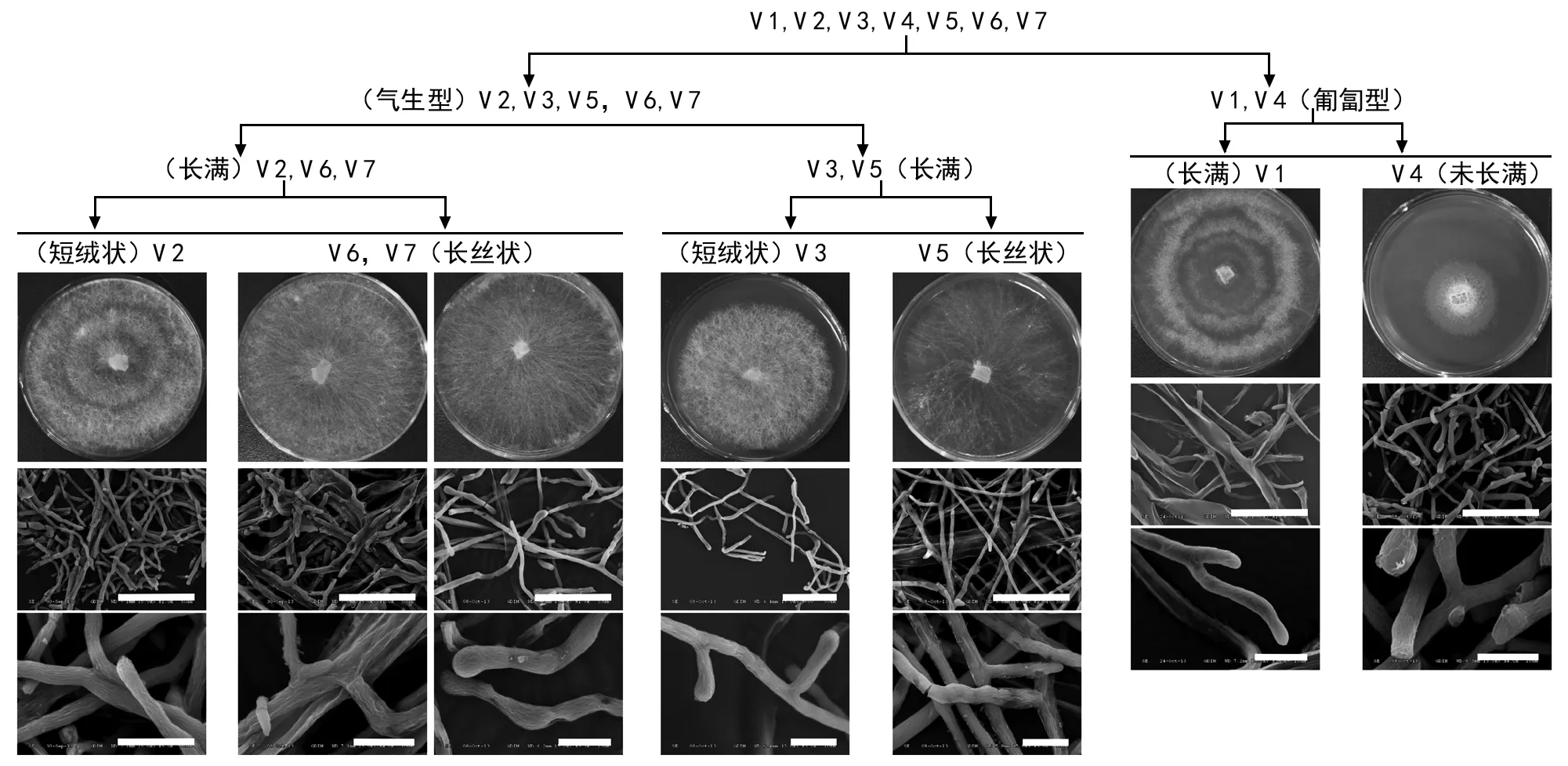
(白色标尺分别为上排50 μm,下排10 μm)。图1 从菌落外观特征对7个草菇菌株菌落进行分类,并图示各菌株菌丝体的扫描电镜照片
2.2 拮抗反应
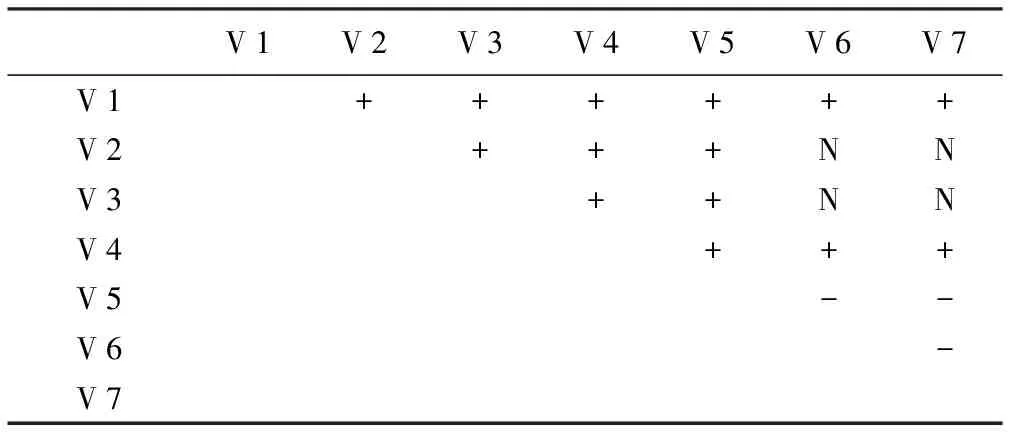
拮抗反应结果显示,V 1、V 4分别和其他6个菌株产生拮抗反应;V 2除了分别和V 6、V 7产生不明显的拮抗反应外(菌丝接触区域靠近V 2一边的形成较致密的菌丝环带),和其他4个菌株产生明显拮抗反应;V 3除了分别和V 6、V 7产生不明显的拮抗反应外(靠近V 3一端的菌丝较为浓密、靠近V 6、V 7一端较稀疏),和其他4个菌株产生明显拮抗反应;V 5分别和V 6、V 7无拮抗反应,和其他4个菌株产生拮抗反应;V 6和V 1、V 4产生拮抗反应,与V 2、V 3产生不明显拮抗反应,与V 7无拮抗反应;V 7情况与V 6相同。综上,除V 6和V 7两者无法区分外,其他5个菌株可以通过拮抗反应试验相互区分。
表1 7个草菇菌株菌丝体的两两拮抗反应结果判定
V1V2V3V4V5V6V7V1++++++V2+++NNV3++NNV4+++V5--V6-V7
注:“+”表示有拮抗反应,“-”表示无拮抗反应,“N”表示拮抗反应不明显。
2.3 菌丝体酯酶同工酶酶谱分析
7个草菇菌种的酯酶同工酶电泳图谱经软件进行条带分析后显示,酶条带出现在Rf值0.08~0.70之间。7个草菇菌种中共出现10条不同Rf值的酶带,其中9条为多态性酶带,多态性位点百分比为90%。其中Rf=0.08酶带只在V 5中出现、Rf=0.18酶带只在V 3中出现,而Rf=0.57酶带只在V 5中缺失。

图2 A:7个草菇菌株菌丝体酯酶同工酶酶谱;B:7个草菇菌株基于同工酶酶谱的聚类树状图
表2 7个草菇菌株的生物学特性与遗传多样性比较
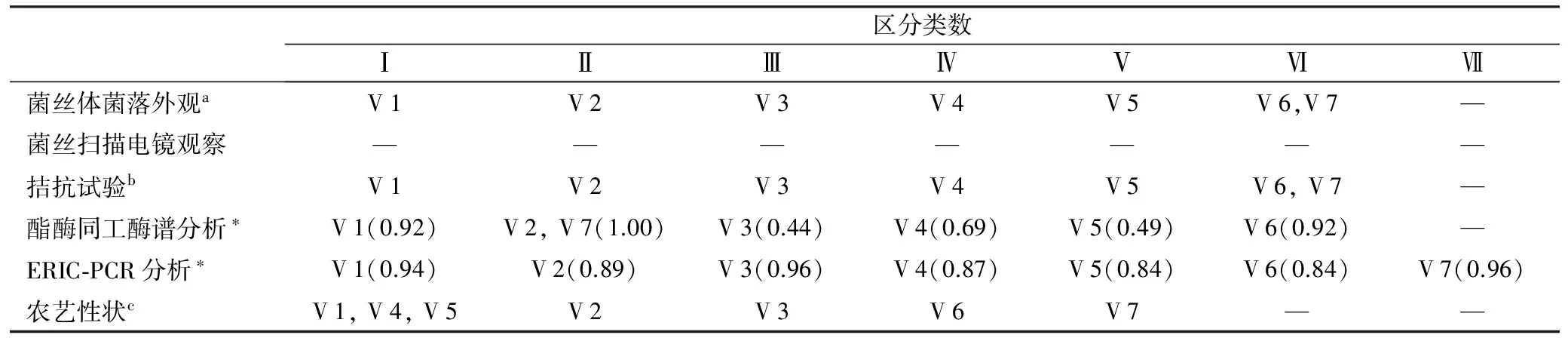
区分类数ⅠⅡⅢⅣⅤⅥⅦ菌丝体菌落外观aV1V2V3V4V5V6,V7—菌丝扫描电镜观察———————拮抗试验bV1V2V3V4V5V6,V7—酯酶同工酶谱分析∗V1(0.92)V2,V7(1.00)V3(0.44)V4(0.69)V5(0.49)V6(0.92)—ERIC⁃PCR分析∗V1(0.94)V2(0.89)V3(0.96)V4(0.87)V5(0.84)V6(0.84)V7(0.96)农艺性状cV1,V4,V5V2V3V6V7——
注:a分类依据见图1;b分类依据表1;c分类依据见图4;*括号给出该菌株在聚类分析中最近1个分枝点的相似性系数值。

注:泳道1~7分别是V 1~V 7,M为分子量标准物。图3 上图:2个适宜的退火温度下7个草菇菌株的ERIC-PCR产物图谱;下图:基于2个ERIC-PCR产物图谱多态性的菌株聚类树状图
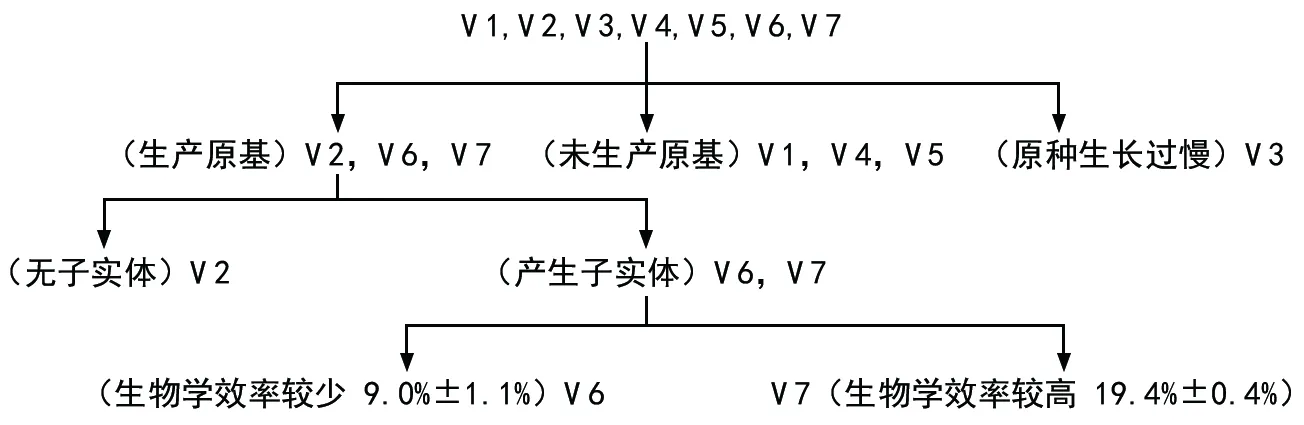
图4 从栽培特性对7个草菇菌株进行分类
根据同工酶图谱带型进行UPGMA法构建的聚类分析,7个草菇菌株同工酶带型的相似性系数在0.44~1.00之间。V 2和V 7最为相似,V 1和V 6较为相似,相似系数为0.92。V 4和前面四者在相似系数为0.69时归为一类,而V 5和V 3和其他菌株差异较大,在相似系数小于0.50时才被归为一类。
2.4 ERIC-PCR分析
结合2种退火温度下扩增的分子标记图谱,经软件识别,扩增得到不同分子量DNA片段条带一共14条,其中9条显示出多态性,多态性位点百分比为64%。其中V 3具有1条特异性条带(893 bp,退火温度47.1 ℃图谱),V 2缺失了其他菌株共有的2条条带(1.7 kb和1.4 kb,退火温度47.1 ℃图谱),V 4缺失了其他菌株共有的1条条带(171 bp,退火温度47.1 ℃图谱),V 5缺失了其他菌株共有的1条条带(1.7 kb,退火温度53.7 ℃图谱)。
根据2种退火温度下扩增的分子标记图谱进行聚类分析,结果显示7个菌株的相似系数范围在0.80~0.96之间。7个菌株在相似系数为0.80时分为2簇,其中1簇在相似系数为0.84时分为2支,分别是V 4和V 6。另外一簇也在相似系数为0.84时分为1支和1簇,包含V 1、V 2、V 3、V 5和V 7,其中V 5单独为1支;在相似系数为0.89时,V 2再单独分为1支;在相似系数为0.94时,V 1再单独分为1支;V 3与V 7最为相似。
2.5 不同草菇菌株的栽培状况比较
在相同的栽培条件下,7个草菇菌株中仅V 6和V 7能产生子实体,而且V 6和V 7所得产量差异显著(μ=0.05,LSD)。V 1、V 4和V 5无原基产生;V 2虽然有原基产生,但原基产生时间比V 6和V 7要迟,且原基没有发育成子实体;V 3在原种制作过程中生长十分缓慢,至其他菌株栽培结束时仍未长满菌种袋。
3 讨 论
所采用的7个草菇菌株通过多种分子标记的分析,体现出遗传多样性,而在生物学特性上,如菌落形态、拮抗性、栽培特性上也表现出差异之处。
7个受试菌株菌丝体,从菌丝菌落形态、菌丝浓密分布,菌丝长速等特征,凭肉眼几乎可逐一分辨。一般经验而言,匍匐性菌丝的草菇菌株不育。本实验中,V 1和V 4属于匍匐型菌株,在栽培试验中也未能产生原基(见图1和图4),符合这一判断,但气生型的菌株未必可育(如V 5),甚至不能完成原种的生长(如V 3)。有文献报道,草菇双核体菌丝的菌落形态与结实与否未见有直接关系[12]。另外,草菇菌丝体的生长速度与产量也没有严格的正相关[13-14]。以上结果在本研究中也有所体现。
黄敏敏等[15]通过扫描电镜观察了不同pH对金福菇菌丝粗细、锁状联合数量等的影响,以判断同一菌株菌丝体在不同培养环境下的变化。本研究尝试从菌丝微观结构角度来展现菌株间差异,显微视野中大部分菌丝的粗细与形状并无突出的差异,无法从中获取分辨7个菌株的可靠依据。
7个草菇菌株两两进行了拮抗试验,但有部分拮抗反应不太明显。影响草菇菌株间拮抗反应清晰度的原因很多,郑玲[16]的硕士学位论文中提及草菇菌龄、退化等因素会影响拮抗反应,而笔者认为,草菇气生菌丝旺盛,也可能是影响拮抗反应观察的原因之一。
同工酶图谱和ERIC-PCR图谱的聚类分析依据是泳道中条带的有无,因此对条带出现与否的判定很重要。本研究采用了具有条带识别功能的软件对所有参与聚类分析的图谱进行条带识别,人工判断为辅助,力求使每个泳道中条带存在与否的判定依据相一致。还采用了软件SPSS对上述3类标记的图谱进行聚类分析(文中未显示),但由于NTSYS在聚类过程的设置参数更符合由图谱转化成的1、0数据矩阵,所以采用后者进行聚类分析。SPSS聚类结果与本研究采用的聚类结果略有不同。
黄龙花等[10]成功通过一对ERIC-PCR引物区分出不同栽培品种的真姬菇;王永红[17]等曾利用ERIC-PCR检视不同保藏条件对同一草菇菌种遗传性状产生影响。通过比较7个草菇菌株的ERIC-PCR图谱发现,不同的反应体系会产生不同的区别效果。因此,选择联合2个图谱的多态性进行聚类分析。
通过生物学特性与遗传多样性可研究,可实现对7个草菇菌株进行分类。对各种实验方法的分类情况归纳为表2。从表2可见,利用ERIC-PCR作为分子标记可将7个草菇菌株完全分类。利用观察菌落外观、拮抗试验、同工酶图谱聚类分析可把7个菌株分为六类,但其中最相似的2个菌株的归类并不一致。利用栽培特性进行分类的结果稍逊于其他分类方法,同时它的分类判定也较为主观,在本研究中此方法可把7个菌株分为五类。而在2种可标出相似性系数的方法中,即使在同一区分类中出现相同的菌株,但它们的相似性系数并不一致,表明这2种方法体现出不同的遗传多样性样式。
综上所述,7个草菇菌株通过一系列的生物学特性与遗传多样性测试,可显示出它们相互之间的差异性。对于草菇菌种将来要进行审定、认证、保护等,有很好的参考作用。
[1]Shu-Ting Chang.The origin and early development of straw mushroom cultivation[J].Economic Botany,1977,31:374-376.
[2]MC Yadav,MP Challen,SK Singh,et al.DNA analysis reveals genomic homogeneity and single nucleotide polymorphism in 5.8 S ribosomal RNA gene spacer region among commercial cultivars of the button mushroom Agaricus bisporus in India[J].Current Science,2007,93(10):1 383-1 389.
[3]Ya-Li Wen,Hui Cao,Ying-Jie Pan.Assessment the genetic diversity of Auricularia strains by two PCR-based typing methods[J].Acta microbiologica Sinica,2004,44(6):805-810.
[4]Chengshu Qiu,Wenjuan Yan,Peng Li,et al.Evaluation of growth characteristics and genetic diversity of commercial and stored lines of Hypsizygus marmoreus[J].International Journal of Agriculture & Biology,2013,15(3):479-485.
[5]唐传红,唐庆九,杨焱,等.基于体细胞不亲和性及ERIC-PCR研究虫草无性分离物的亲缘关系[J].食用菌,2013(4):20-22,36.
[6]Qiu-sheng Zhang,Bing-lian Xu,Lin-de Liu.Analysis of genetic diversity among Chinese Pleurotus citrinopileatus Singer cultivars using two molecular marker systems (ISSRs and SRAPs) and morphological traits[J].World Journal of Microbiology & Biotechnology,2012,28(5):2 237-2 248.
[7]张萍,梁建光,杨立红,等.草菇不同菌株的生物学特性研究[J].中国食用菌,2005(2):17-19.
[8]谢宝贵,羿红,黄志龙,等.草菇杂交育种及同工酶分析[J].福建农业大学学报,2001,30(3):372- 376.
[9]傅俊生,朱坚,谢宝贵,等.草菇杂交菌株2628的鉴定与品比试验[J].中国农学通报,2010,26(14):48-53.
[10]黄龙花,夏凤娜,邵满超,等.2个不同来源真姬菇子实体同源性的分子鉴定[J].中国食用菌,2014,33(1):34-36.
[11]廖世煌,卢元祥.澳大利亚草菇室内栽培技术[J].食用菌,1995,17(5):34-36.
[12]薛承琴,周慧敏,汪虹,等.草菇子代单孢菌株的菌落类型与 A 因子分布规律研究[J].菌物学报,2013,32(1):89-95.
[13]王卓仁.八个草菇菌株的栽培比较试验[J].中国食用菌,2003,23(3):20-21.
[14]敖莉,杨维丽,李长田,等.10个草菇菌株比较研究[J].中国食用菌,2012,31(1):18-20.
[15]黄敏敏,江枝和,翁伯琦.不同pH 值条件下金福菇菌丝体形态的电镜观察[J].电子显微学报,2010,29(2):173-176.
[16]郑玲.草菇标准菌株库的建立.福建:福建农林大学,2011:21.
[17]王永红,郭俊,陈美标,等.海藻糖对草菇菌种的低温保护效应研究[J].徽生物李通报,2008,35(1):137-141.

图3 巯基乙醇·Tris法不同料液比提取乌拉尔甘草的蛋白质电泳结果比较
3.4 乌拉尔甘草与胀果甘草的蛋白质电泳结果比较
由图4可以看出,乌拉尔甘草种子和胀果甘草种子的蛋白质条带位置和数量均具有显著差异,胀果甘草种子蛋白质电泳的上部条带较乌拉尔甘草条带浅淡,但中部的2条带上方各出现1条新带,因此,可以依据这2条带来鉴定乌拉尔甘草和胀果甘草的种子。
4 结论与讨论
本研究通过分析比较8种蛋白质提取方法,发现巯基乙醇法和丙酮沉淀法在甘草种子蛋白质电泳试验中电泳条带多,背景清晰,成本低,适宜于进行甘草种子的SDS-PAGE电泳。1∶10和2∶10的料液比适宜于甘草种子蛋白质的提取。10%和12%的分离胶电泳效果差异不大,均适用于进行种质鉴定,乌拉尔甘草种子和胀果甘草种子的蛋白质条带位置和数量均具有显著差异,该研究结果为甘草种子纯度检测提供了有效的技术方法。

图4 巯基乙醇法鉴定乌拉尔甘草(左侧)与胀果甘草(右侧)的种子蛋白质电泳结果比较
参考文献:
[1]宋艳,梅李峰,周丽曼.瓜蒌种子蛋白质6种提取方法比较[J].中国实验方荆学杂志,2011,17(4):95-97.
[2]崔晓慧.黑芝麻中抗菌蛋白的分离纯化及其主要性质[D].中国农业科学院,2000.
[3]沙月霞.棉叶总蛋白提取及SDS-PAGE电泳的改良[J].棉花学报,2005,17(3):146-150.
[4]曾广娟,李春敏,张新忠,等.适于SDS-PAGE分析的苹果叶片蛋白质提取方法[J].华北农学报,2009,24(2):75-78.
[5]李莹莹,吴彩娥,杨剑婷,等.白果蛋白质提取及SDS-PAGE分析[J].食品科学,2010,31(22):36-40.
[6]于阳阳,包怡红.碱溶性东北山核桃蛋白的提取及SDS-PAGE分析[J].食品科学,2012,33(18):10-13.
