COⅠ条形码辅助分析雷州半岛红树林区鱼类的物种多样性*
2016-01-15郭昱嵩刘楚吾王中铎
张 顺 廖 健 柏 琴 陈 冲 郭昱嵩 刘楚吾 王中铎
(广东海洋大学 南海水产经济动物增养殖广东普通高校重点实验室 湛江 524025)
红树林生态系统(Mangrove ecosystem)是近海三大生态系统之一, 多分布于海陆交界的河口区, 受潮汐影响明显, 生态环境变化多样, 不仅物种多样性极其丰富(Field et al,1998), 还具有净化温室气体、抵抗水污染等重要的功能(Li et al, 2014), 国内外已有广泛的研究(张乔民等, 2001; Castellanos-Galindo et al,2013)。红树林因其独特的根部结构以及丰富的藻类等食物资源而成为鱼类理想的栖息地(Blaber, 2007;Gajdzik et al, 2014)和育幼场(Kimirei et al, 2013)。Nickerson(1999)研究表明, 美国、印度等地区60%以上的鱼类在其一生的发育过程中与红树林密切相关。我国雷州半岛拥有红树林及其附近滩涂面积约20279hm2, 是国家级红树林重点保护区之一。近些年来, 人口不断增加, 环境的恶化以及加上人为的过度开发, 致使原本生态环境比较脆弱的红树林生态系统逐渐被破坏, 近海渔业资源日渐枯竭。据“我国近海海洋综合调查与评价”显示, 近半个世纪以来, 我国红树林湿地丧失近 73%, 由 55万 hm2减至 15万hm2(谢庆裕, 2012)。通过相关学者对我国广东(Han et al, 2003)、广西(黄德练等, 2013)、台湾(Kuo et al, 1999)等地红树林的调查结果表明, 自红树林划入自然保护区后, 我国红树林湿地的物种多样性有所提升, 但相比鼎盛时期依然存在较大差距, 需要进一步研究和保护。
近年来, 世界鱼类数据库FishBase (http://fishbase.org/)已被广泛应用于鱼类形态学鉴定, 由于鱼类栖息环境变化大, 性状特征变异快, 及各区域研究水平不一, 单一的形态学分类法作为基本的分类法存在诸多的局限性。随之, DNA条形码(DNA barcodes)作为一个标准化和世界通用的物种识别系统被 Hebert等(2003)提出, 即通过使用短的、标准化的基因片段来进行物种鉴定, 成为了传统形态学分类的辅助工具(Hajibabaei et al, 2007)。Ward 等(2005)用 COⅠ基因(Cytochrome c oxidase subunit)Ⅰ中的655bp片段对澳大利亚207种海洋鱼类进行分析, 发现所有物种都能被有效地区分, 通过重建系统发生关系, 认为COⅠ作为海洋鱼类的 DNA条形码标准序列是可行的。王中铎等(2009)用COⅠ基因对南海硬骨鱼类40个物种89个样本进行了分析, 结果表明COⅠ序列广泛适用于硬骨鱼类的物种鉴别, 并可用于低级分类阶元的系统进化分析。DNA条形码技术应用于物种多样性的评价也有广泛的研究(Swartz et al, 2008;Triantafyllidis et al, 2011)。本文将形态学分类法与DNA条形码相结合应用于红树林鱼类物种的分类鉴定, 分析鱼类生态类群组成的特点, 以探讨红树林生态系统作为鱼类栖息地和育幼场的功能, 同时进一步完善该区域鱼类的DNA条形码数据库。
1 材料与方法
1.1 实验材料与形态学鉴定
2014年3月至2015 年7月于雷州半岛红树林海域 (廉 江 高 桥 (109°47′E, 21°36′N)、 雷 州 流 沙 湾(109°56′E, 20°26′N)、霞山东海岛(110°20′E, 20°6′N)、遂溪九龙山(110°17′E, 20°39′N))共采集成鱼和幼鱼样本1720尾, 每月采用围网、地笼、拖网以及 200目的手抄网进行随机采集。依据Fish Base数据库等鱼类形态分类系统(伍汉霖等, 2008)初步确认存在94个物种, 最终参考《拉汉世界鱼类系统名典》(伍汉霖等,2012)和综合分类学信息系统(Integrated Taxonomic Information System, ITIS)对物种的有效名以及分类地位进行确定。取背部肌肉组织提取DNA, 剩余样品用95%的乙醇固定, –40°C保存备用。
1.2 DNA的提取、PCR扩增及测序
取约 0.2g鱼的肌肉经 Protease K消化后, 参考《分子克隆实验指南》的酚/氯仿抽提法(萨姆布鲁克等,2008), 提取 DNA。COⅠ基因序列扩增引物(Ward et al, 2005): Fish F: 5′-TCAACC AACCACAAAGACAA TGGCAC-3′; Fish R: 5′-TAG ACTTCTGGGTGGCC AAAGAATCA-3′。PCR 反应总体积为 25μL, 包括2.5μL 10×Buffer, 2.0μL dNTP (2.5 mmol/L), 上下游引物各 1μL (5μmol/L), 0.2μL Taq DNA聚合酶(5U/μL),0.5—2.0μL模板 DNA, 超纯水补齐。PCR反应的循环参数为: 94°C 预变性 3min; 94°C 变性 30s、55°C 退火 30s、72°C延伸 50s, 共 30个循环; 最后 72°C延伸5min, 最后, 4°C下保存。扩增产物用1%琼脂糖凝胶电泳检测合格后, 委托生工生物工程(上海)股份有限公司进行测序。
1.3 物种鉴定与序列分析
利用 BioEdit软件对获得的碱基序列检测后, 导入NCBI (National Center for Biotechnology Information)数据库和BOLD (The Barcode of Life Data System)系统进行序列相似度比对, 并结合鱼类形态学资料进行辅助鉴定, 确保其准确性。通过 MEGA6.0软件分析 COⅠ基因序列特征及序列间的遗传距离。利用DNAsp5软件(Rozas et al, 1999)计算种间平均核苷酸差异数(K)及单倍型分析。选用 Kimura 2-parameter(K2P)计算遗传距离, 邻接法(Neighbor-Joining method, NJ)构建系统进化树, 经 1000次重复抽样(Bootstraps)检测分子系统树分枝的置信度。
1.4 鱼类生态类群的组成分析
依据Elliott等(2007)建立的鱼类生态类群分类方法, 并结合描述鱼类生活史特征的其它相关资料, 将红树林鱼类分为海洋偶见鱼类(marine stragglers,MS)、海洋洄游鱼类(marine migrants, MM)、红树林定居鱼类(estuarine species, ES)、溯河洄游鱼类(anadromous, AN)、降海洄游鱼类(catadromous, CA)、淡水洄游鱼类(freshwater migrants, FM)、淡水偶见鱼类(freshwater stragglers, FS)、两侧洄游鱼类(amphidromous, AM)八类。采用SPSS 17.0软件进行数据统计和分析。
2 结果与分析
2.1 COⅠ序列特征及序列间遗传距离分析
利用COⅠ条形码辅助形态分类法进行物种鉴定,获得115条有效长度约为560bp的碱基序列, 未存在插入和缺失。由表1可得知, A+T碱基含量(54.1%)高于 G+C含量(45.9%), 表现出 A+T碱基组成偏向性;第 1密码子位点的 G+C含量(55.1%)高于第 2和第3密码子位点(42.8%和 39.6%), 密码子组成也表现出一定的偏倚性。从总体来看, 含有295个多态位点, 约占52.7%, 简约信息位点数为 265个(约占47.3%), 转换/颠换之比为 1.56, 说明序列突变已达到饱和; 种间平均核苷酸差异数为49, 约占总核苷酸数的29.17%。
由各分类阶元K2P遗传距离(表2)可知, 种内不同个体的遗传距离平均为0.0032, 而同属不同物种间的遗传距离为0.0562, 约为种内遗传距离的17倍, 符合 Hebert等(2003)提出的“10×规则”。种间遗传距离在同科不同属间平均为 0.1432, 而同目不同科间为0.2115, 可见, K2P遗传距离值在科以上的高级阶元的增加明显变缓。此外, 虾虎鱼科与笛鲷科、鲻科和鲱科两两间平均遗传距离变化范围在0.2331—0.2534,科间的遗传距离变异较小。
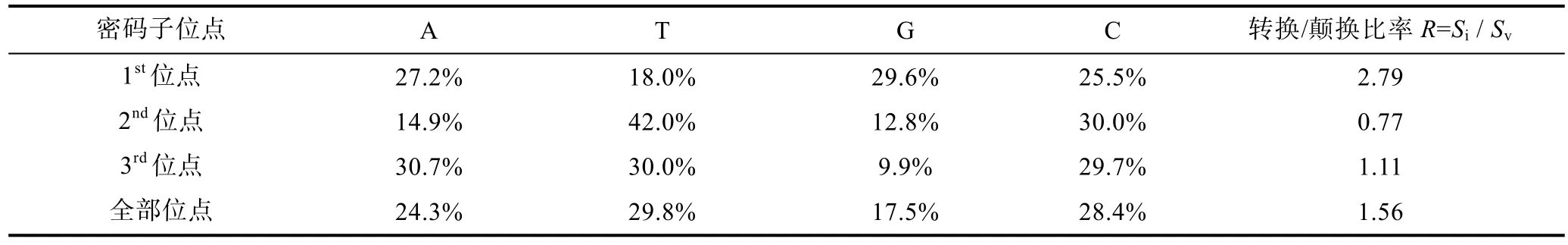
表1 94种鱼类的COⅠ基因序列碱基组成及变异情况Tab.1 Base composition and variation of all CO Ⅰ gene sequences of 94 fish species
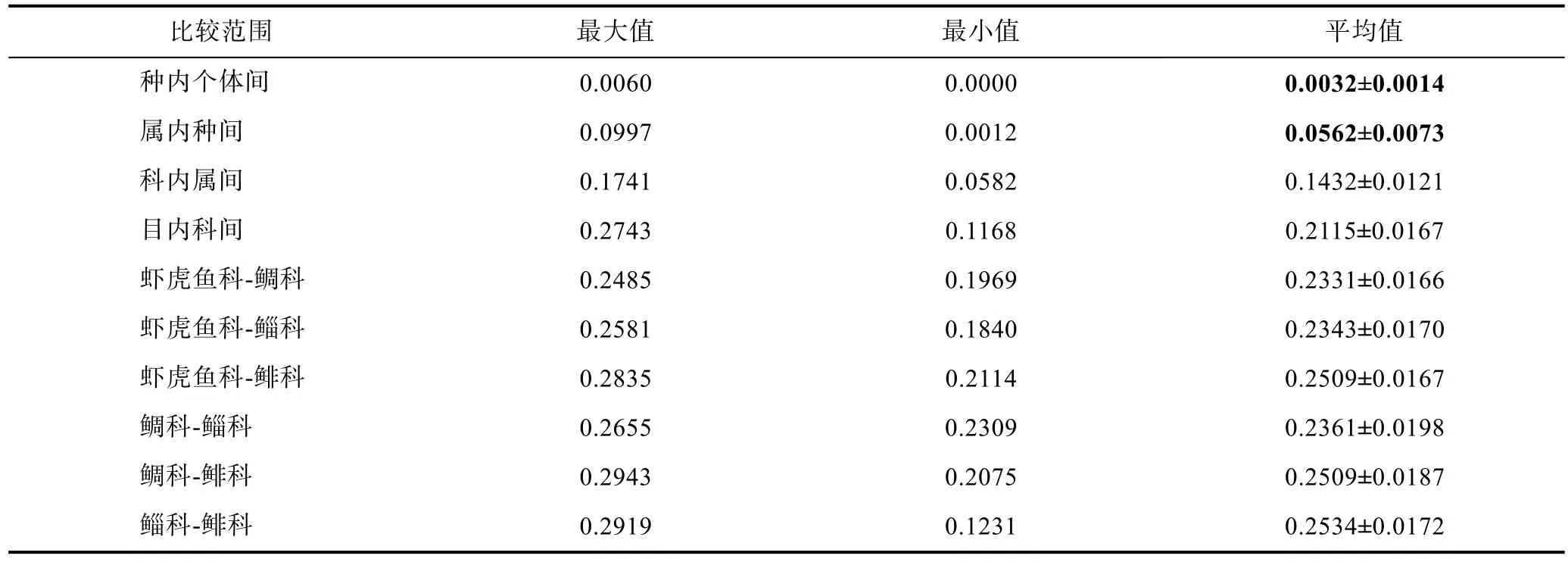
表2 各分类阶元遗传距离(K2P)统计表Tab.2 Genetic divergence (in K2P) within various taxonomic levels
2.2 物种的鉴定及有效种名的确定
经传统形态学鉴定, 94个物种分属于11目33科72属, 其最终的物种有效名、分类地位及生态类群见表 3。研究发现, 存在多种鱼类形态变异较小, 传统形态学难以鉴别, 最后通过形态学鉴定到属而未能确定其物种名的有 12 个物种(以“*”或“**”表示)。
由于形态学分类法存在一定的局限性, 再将所获取的COⅠ基因序列导入到NCBI的GenBank数据库和BOLD系统进行序列相似度比对, 其中59种鱼类形态学鉴定结果与两者比对结果相对应(Ident≥98%)。存在13种鱼类形态学鉴定结果仅与NCBI数据库相似度最高的比对结果相对应, 而与 BOLD系统未能高度匹配(“△”标注), 6种鱼类形态学鉴定结果仅与BOLD系统相似度最高的比对结果相对应(“”▽标注)。研究发现, 数据库中与形态鉴定为弹涂鱼(P.modestus)一致性最高的序列为尾斑舌塘鳢(Parioglossus dotui) (Ident≥99%), 但后者形态特征与分布情况同该鱼种差距甚大, 因此将该样本确定为弹涂鱼(“§”标注)。另外, 12个物种通过形态学可鉴定到种, 分子鉴定到科的有8种(以“*”表示), 分子鉴定到属的为 4种(以“**”表示), 说明数据库里的物种DNA条形码不够完善, 亟需补充。此外, 通过综合比较物种的有效命名和分类地位, 发现6个物种存在分歧, 本文最终以《拉汉世界鱼类名典》和综合分类学信息系统的信息为准(“”☆标注)。
2.3 鱼类群落组成
本次于雷州半岛红树林保护区共采集到鱼类 94种, 其中鲈形目鱼类有59种, 占总物种数的62.77%,其次是鲱形目和鲻形目分别为6种(占6.38%)和5种(占 5.32%), 而鲤形目的鱼类采集到 2种(占 2.13%),鲈形目中种类最多的科是虾虎鱼科, 29种(占 30.85%),其次是鲷科有6种(占6.38%)。可能是由于虾虎鱼类绝大多数体型细小, 更适宜于红树林湿地生态环境, 成为了代表性物种。
从生态类群组成来看(图 1), 红树林海域物种数占优势的是海洋洄游鱼类, 共有 31种, 占物种总数的32.98%, 其次是海洋偶见鱼类21种(占22.34%)和两侧洄游鱼类 16种(占 17.02%), 而红树林定居鱼类10种, 占物种总数的10.64%, 其次是淡水偶见鱼类6种和淡水洄游鱼类8种, 而采集到的降海洄游鱼类有1种, 为日本鳗鲡(A. japonica), 溯河洄游鱼类为1种,为暗纹多纪 鲀 (T. porphyreus)。研究发现, 四个采集点的各生态类群组成具有相似的特征, 以海洋洄游鱼类为优势种群, 以海洋偶见鱼类、红树林定居鱼类、两侧洄游鱼类居多。流沙湾和东海岛的海洋偶见鱼类物种数明显多于九龙山和高桥; 仅各1种的溯河洄游鱼类和降海洄游鱼类分别出现于流沙湾和东海岛; 东海岛的两侧洄游鱼类少于另外三个采样点。

表3 94种鱼类的物种名录、生态类群及相应DNA条码序列号Tab.3 The list of 94 fish species, ecological groups and the corresponding DNA GenBank accession numbers

续表

续表
2.4 聚类分析
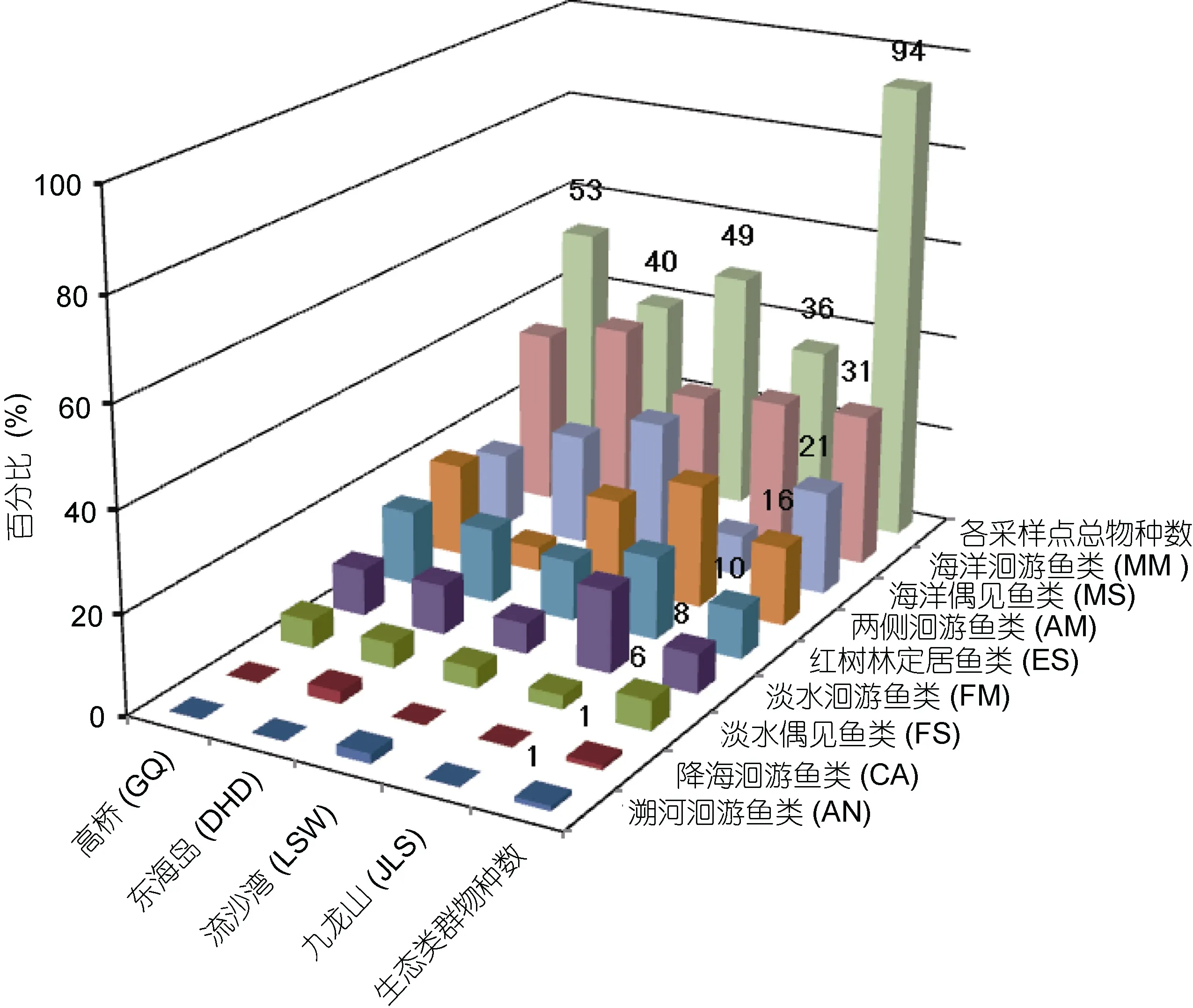
图1 雷州半岛各采样点的物种数及各生态类群的百分比组成Fig.1 Composition of fish species and ecological groups of each site in Leizhou Peninsula

图2 雷州半岛红树林94个物种的115条COⅠ序列构建的邻位连接树Fig.2 The neighbor-joining tree of 115 CO Ⅰ barcoding sequences of 94 species in mangrove waters of Leizhou Peninsula
基于115条COⅠ序列的101个单倍型构建系统关系树, 同种鱼类的不同个体间均可各自聚为独立的一支, 且属内不同物种间界限清晰(如图 2)。虾虎鱼科(Gobiidae)的弹涂鱼属(Periophthalmus)、青弹涂鱼属(Scartelaos)聚为一支, 与虾虎鱼科(Gobiidae)的背眼虾虎鱼属(Oxuderces)及其它的属亲缘关系很近(以◆表示), 从而可以进一步证实经数据库相似度比对高的物种并非尾斑舌塘鳢(Parioglossus dotui),而是弹涂鱼(P. modestus)。另外, 鲀形目(Tetraodontiformes)(以▲表示)及鲻科(Mugilidae)的鲻属(Chelon)(以○表示)也能各自聚为独立的一支, 且置信度高。其中, 庇 隆氏拟单角 鲀 (P. peroni)与同属于四齿 魨 科(Tetraodontidae)的其它鱼类聚类关系较远, 黑翼 文 鳐 鱼(H. rondeletii) 与 同 属 于 鹤 鱵 目(Beloniformes)(以□表示)的其它鱼类聚类关系较远,此外, 一些目内科属间的聚类关系出现一些异常, 即鲉形目(Scorpaeniformes)、鲽形目(Pleuronectiformes)(分别以■和◇表示)的物种与其它目的聚类关系比较混乱。
3 讨论
红树林同海草床和珊瑚礁海域存在相通性, 是鱼类重要的栖息地和育幼场, 研究其鱼类的种类对于保护物种多样性具有极其重要的作用(Honda et al,2013)。由于红树林中鱼类栖息环境变化大, 性状特征变异快及存在隐存种、新物种和杂交种(Zemlak et al,2009)等问题加大了传统形态学分类鉴定的难度。本研究将COⅠ条形码应用于雷州半岛沿岸红树林鱼类的分类鉴定, 提高物种鉴定的准确性及有利于推动DNA条形码数据库共享平台的建设, 同时也揭示出了一些问题。
3.1 应用COⅠ条形码进行鉴定发现的问题
应用 COⅠ条形码鉴别出红树林鱼类共计 94种,由此可见其作为通用引物的广泛适用性及其优势。但由于 DNA条形码数据库仍有欠缺, 本研究的鱼类样品中仍有一些物种未能进行分子鉴定, 以及由于时间久远和鉴定方法不够完善, 有的后期研究结果未及时更新至数据库。建议需加强标本资料的收集及进一步完善 DNA条形码数据库, 推动国际数据库共享平台的建设。另有研究指出单一的COⅠ基因并非适用于所有鱼类物种的鉴别(Krück et al, 2013)。水产养殖中出现了广泛培育杂交种及近海外来物种的侵袭等新的问题, 加大了研究难度, 新的研究工具 “多基因条形码”应加大研究和应用(Chesters et al, 2015),并及时补充低分化物种的其它相关数据(如线粒体DNA控制区等)进行更进一步的分析。
3.2 红树林对于不同生态类群鱼类的保育意义和生境危机
红树林区采集的鲤形目(Characiformes)鱼类有 2种(占 2.13%)与何秀玲等(2002)对雷州半岛红树林区的调查发现的鲤形目鱼类占总物种数的 3.90%近似,但远低于长江口潮沟的优势类群鲤形目(占物种总数14.94%)(金斌松, 2010), 这可能是由于长江口的径流量较雷州半岛红树林河口的径流量大, 更适宜于鲤形目鱼类的生存。由生态类群分析来看, 红树林海域的物种数占优势的是海洋洄游鱼类、海洋偶见鱼类和两侧洄游鱼类, 其特征同金斌松(2010)研究的长江口盐沼潮沟鱼类的生态类群组成相似, 表明红树林生态系统是许多海水、淡水和洄游性鱼类在特定生活阶段的重要栖息地和育幼场所, 且与已有的红树林调查一致(Elliott et al, 2007; Franco et al, 2008; 黄德练等, 2013)。红树林采集到的10种定居鱼类, 占物种总数的 10.64%, 主要是虾虎鱼类, 其依靠特化的生理构造, 比其它类型鱼类更能适应盐度波动、潮汐涨落等复杂多变的红树林生态环境, 成为了代表性物种。
3.3 COⅠ条形码在系统进化分析中的适用性和局限性
系统关系树中的同种鱼类的不同个体间均可各自聚为独立的一支, 且属内不同物种间界限清晰, 但对于科以上的聚类关系分析不是很清晰, 如鲉形目(Scorpaeniformes)、鲽形目(Pleuronectiformes)的物种与其它目的聚类关系比较混乱, 表明 COⅠ条形码具备一定的低阶元系统进化分析能力, 但对于高级阶元的系统进化分析准确性明显降低。究其原因, 可能是 COⅠ条形码序列长度仅约 560bp, 信息位点数目有限, 科间变异较大, 且在高级阶元时碱基置换趋于饱和, 这与COⅠ条码序列间K2P遗传距离值在种以上的高级阶元的增加明显变缓的特征相符合。
为了获得较为准确的物种系统发生关系, 构建比较客观的进化树, 可以选择 mtDNA不同区域的多组序列的组合比较的途径; 或选择核 DNA序列和mtDNA不同区域相结合的方法; 或选择整个mtDNA全序列来进行研究的方法。近年来, 这些方法也得到过验证(Rocco et al, 2007; Keskin et al, 2009; Catanese et al, 2010)。此外, 研究发现, 庇 隆氏拟单角 鲀 (P.peroni)与同属于四齿魨科(Tetraodontidae)的其它鱼类聚类关系较远, 黑翼文鳐鱼(H. rondeletii)与同属于鹤 鱵 目(Beloniformes)的其它鱼类聚类关系较远, 在一定程度上说明了这两种鱼类目前的分类地位有待进一步的商议。
综上所述, 本文利用 COⅠ条形码技术可有效快捷地鉴定出红树林海域的鱼类并发现物种多样性极其丰富, 红树林生态系统更是许多淡水、海水和洄游性鱼类的重要栖息地和育幼场所。虽然COⅠ条形码在属内的同物种间界限清晰, 但对于科以上的聚类关系分析不是很清晰, 说明其具备一定的低阶元系统进化分析能力, 但对于高级阶元的系统进化分析准确性有所降低, 其原因与序列长度较短有关, 对于高阶元系统关系分析, 应该加强对“多基因条形码技术”的研究, 更加准确的确定物种的系统进化关系和分类地位。由于采样方法的限制以及近海生态环境的多变, 对组成生态类群的鱼类样本采集不够全面, 并且加上人类的过度捕捞, 导致近海渔业资源的枯竭,给研究工作带来了一定的困扰。支持 Thomsen等(2012)提出的可以尝试利用 DNA条形码来检测水体中的体表细胞或分泌物来研究鱼类的种群分布和活动范围, 使条形码技术成为鱼类迁徙、洄游等相关研究的便利工具。
致谢 感谢湛江市红树林保护区林广旋工程师和我校海洋资源与环境监测中心张瑜斌老师等在样本采集方面给予的无私帮助, 谨致谢忱。王中铎, 郭昱嵩, 陈荣玲等, 2009. 南海常见硬骨鱼类COⅠ条码序列. 海洋与湖沼, 40(5): 608—614
伍汉霖, 邵广昭, 赖春福等, 2012. 拉汉世界鱼类系统名典.台湾: 水产出版社, 10—550
伍汉霖, 钟俊生, 2008. 中国动物志(硬骨鱼纲): 鲈形目 5(虾虎鱼亚目). 北京: 科学出版社, 164—750
何秀玲, 叶 宁, 宣立强, 2003. 雷州半岛红树林海区的鱼类种类调查. 湛江海洋大学学报, 23(3): 3—10
张乔民, 隋淑珍, 2001. 中国红树林湿地资源及其保护. 自然资源学报,16(1): 28—34
金斌松, 2010. 长江口盐沼潮沟鱼类多样性时空分布格局. 上海: 复旦大学博士论文, 12
黄德练, 吴志强, 黄亮亮等, 2013. 广西钦州港红树林区鱼类
物种多样性分析. 海洋湖沼通报,(4):135—142
谢庆裕, 2012. 我国红树林半世纪来丧失73%. 南方日报, A20萨姆布鲁克J, 拉塞尔D W编著, 黄培堂译, 2008. 分子克隆实
验指南(上、下二册). 3版. 北京: 化学工业出版社, 693 Blaber S J M, 2007. Mangroves and fishes: issues of diversity,dependence, and dogma. Bulletin of Marine Science, 80(3):457—472
Castellanos-Galindo G A, Krumme U, Rubio E ASpatial variability of mangrove fish assemblage composition in the tropical eastern Pacific OceanReviews in Fish Biology and Fisheries, 23: 69—86
Catanese G, Manchado M, Infante C, 2010. Evolutionary relatedness of mackerels of the genus Scomber based on complete mitochondrial genomes: strong support to the recognition of Atlantic Scomber colias and Pacific Scomber japonicus as distinct species. Gene, 452(1): 35—43
Chesters D, Zheng W M, Zhu C D, 2015. A DNA Barcoding system integrating multigene sequence data. Methods in Ecology and Evolution, 6(8): 930—937
Elliott M, Whitfield A K, Potter I C et al, 2007. The guild approach to categorizing estuarine fish assemblages: a global review. Fish and Fisheries, 8(3): 241—268
Field C B, Osborn J G, Hoffman L L et al, 1998. Mangrove biodiversity and ecosystem function. Global Ecology and Biogeography Letters, 7(1): 3—14
Franco A M, Elliott M, Franzoi P et al, 2008. Life strategies of fishes in European estuaries: the functional guild approach.Marine Ecology Progress Series, 354: 219—228
Gajdzik L, Vanreusel A, Koedam N et al, 2014. The mangrove forests as nursery habitats for the ichthyofauna of Mida Creek (Kenya, East Africa). Journal of the Marine Biological Association of the United Kingdom, 94(5):865—877
Hajibabaei M, Singer G A C, Hebert P D N et al, 2007. DNA barcoding: how it complements taxonomy, molecular phylogenetics and population genetics. Trends in Genetics,23(4): 167—172
Han W D, Lui J K, He X L et al, 2003. Shellfish and fish biodiversity of mangrove ecosystems in Leizhou Peninsula,China. Journal of Coastal Development, 7(1): 21—29
Hebert P D N, Cywinska A, Ball S L et al, 2003. Biological identifications through DNA barcodes. Proceedings of the Royal Society B: Biological Sciences, 270(1512): 313—321
Honda K, Nakamura Y, Nakaoka M et al, 2013. Habitat use by fishes in coral reefs, seagrass beds and mangrove habitats in the Philippines. PLoS One, 8(8): e65735
Keskin E, Can A, 2009. Phylogenetic relationships among four species and a sub-species of Mullidae (Actinopterygii;Perciformes) based on mitochondrial cytochrome B, 12S rRNA and cytochrome oxidase II genes. Biochemical Systematics and Ecology, 37(5): 653—661
Kimirei I A, Nagelkerken I, Mgaya Y D et al, 2013. The mangrove nursery paradigm revisited: otolith stable isotopes support nursery-to-reef movements by indo-Pacific fishes.PLoS One, 8(6): e66320
Krück N C, Tibbetts I R, Ward R D et al, 2013. Multi-gene barcoding to discriminate sibling species within a morphologically difficult fish genus (Sillago). Fisheries Research, 143: 39—46
Kuo S R, Lin H J, Shao K T, 1999. Fish assemblages in the mangrove creeks of northern and southern estuaries Taiwan.Estuaries and Coasts, 22(4): 1004—1015
Li N, Chen P M, Qiao P P et al, 2014. Research advances on marine ecological effect and repairing techniques of coastal mangrove wetland. Asian Agricultural Research,6(8): 60—64
Nickerson D J, 1999. Trade-offs of mangrove area development in the Philippines. Ecological Economics, 28(2): 279—298
Rocco L, Liguori I, Costagliola D et al, 2007. Molecular and karyological aspects of Batoidea (Chondrichthyes,Elasmobranchi) phylogeny. Gene, 389(1): 80—86
Rozas J, Rozas R, 1999. DnaSP version 3: An integrated program for molecular population genetics and molecular evolution analysis. Bioinformatics, 15(2): 174—175
Swartz E R, Mwale M, Hanner R, 2008. A role for barcoding in the study of African fish diversity and conservation. South African Journal of Science, 104(7—8): 293—298
Thomsen P F, Kielgast J, Iversen L L et al, 2012. Detection of a diverse marine fish fauna using environmental DNA from seawater samples. PLoS One, 7(8): e41732
Triantafyllidis A, Bobori D, Koliamitra C et al, 2011. DNA barcoding analysis of fish species diversity in four north Greek lakes. Mitochondrial DNA: The Journal of DNA Mapping, Sequencing, and Analysis, 22(S1): 37—42
Ward R D, Zemlak T S, Innes B H et al, 2005. DNA barcoding Australia’s fish species. Philosophical Transactions of the Royal Society B: Biological Sciences, 360(1462):1847—1857
Zemlak T S, Ward R D, Connell A D et al, 2009. DNA barcoding reveals overlooked marine fishes. Molecular Ecology Resources, 9(S1): 237—242
猜你喜欢
杂志排行
海洋与湖沼的其它文章
- CPA-核酸试纸条快速检测副溶血性弧菌(Vibrio parahaemolyticus)方法的建立及其在海产品检测中的应用*
- 日本囊对虾(Marsupenaeus japonicus)秋繁同生群仔虾不同耐干露性能群体间的形态表型差异*
- 小黄鱼(Pseudosciaena polyactis)形态性状与体质量的相关性及通径分析*
- 基于微卫星标记的洞庭青鲫与三个鲫品系群体遗传多样性分析*
- 豆油替代鱼油对赤点石斑鱼(Epinephelus akaara)生长、体组成及体脂肪酸组成的影响*
- 青鱼(Mylopharyngodon piceus)新发病病原类志贺邻单胞菌(Plesiomonas shigelloides)的分离鉴定*
