基于海洋细菌基础数据库的软件系统设计
2015-12-20李艳君颜焱锋崔玉军钱扬会赵强元
李艳君,颜焱锋,崔玉军,钱扬会,赵强元
基于海洋细菌基础数据库的软件系统设计
李艳君,颜焱锋,崔玉军,钱扬会,赵强元
目的:建立海洋细菌基础数据库软件系统,以实现对医院收集保存的大量海洋细菌的规范化、系统化管理。方法:完善菌株背景资料和部分16S rRNA序列信息,编写软件系统,进行调试运行。结果:建立了基于海洋细菌基础数据库的软件系统,具备菌种信息管理、菌种鉴定和菌种保藏功能,能够实现菌株资料的查询、基于16S rRNA序列的菌种鉴定和菌株保藏信息的管理。结论:数据库软件系统的建立方便了大量海洋菌株的管理,同时为菌种鉴定提供了有效工具。
海洋细菌;数据库;软件系统
0 引言
海洋细菌在海洋环境中分布广泛,容易引发海上作业人员的各类感染性疾病[1],值得深入研究。本科室在前期工作中分离保存了大量海洋细菌,获得一系列研究数据[2-4],加强了菌株收集、保存和数据的系统化管理,将为后续研究提供宝贵资源。然而,由于数据库的局限性,传统生化方法不足以实现对海洋细菌的鉴定,16S rRNA序列是实现细菌分类鉴定的金靶标之一,16S rRNA核酸指纹数据库和相关软件系统将为海洋细菌鉴定提供有力工具。因此,本研究综合应用微生物学、分子生物学和生物信息学手段,建立了大规模的海洋细菌管理查询系统和16S rRNA核酸指纹数据库,从而实现对我院大量海洋菌株的规范化、信息化管理以及对未知海洋细菌进行基于16S rRNA的分类鉴定。
1 菌株信息的完善和实验数据的积累

对我院保存的816株海洋细菌背景资料、鉴定结果进行完善和补充,保证数据信息的可靠。挑选432株代表性菌株进行16S rRNA测序,同时登录美国国家生物技术信息中心(NationalCenterforBiotechnology Information,NCBI)网站收录8 000余条已知种属、背景信息清晰的16S rRNA序列。
2 数据库软件系统的整体结构及其主要功能
2.1 数据库软件系统的整体结构
本研究设计开发的软件系统以海洋细菌基础数据库为核心,包含菌种信息管理查询、菌种鉴定和菌种保藏3个功能模块,其整体结构如图1所示。基础数据库为3个模块提供所需要的数据,同时可以通过人机交互界面直接调用、维护数据库中的内容。用户通过人机交互界面进行数据输入、参数选择等操作。根据运算结果的数据特点,得到xls、txt和jpg格式的输出文件。单机版数据库软件可在Win9x/me/ xp/7等微软操作系统下运行,主界面如图2所示。
2.2 菌种信息管理模块
2.2.1 菌株检索
软件系统可实现单独或者组合查询功能,用户可以根据菌株的某一类特征对全库进行查询,获得所需信息。点击工具栏中的“菌种信息”按钮,将启动菌种信息管理模块。进入“检索”选项卡,用户可以自行选择时间范围及其他各类背景参数,通过“与、或、非”逻辑运算符进行细菌背景资料的组合查询(如图3所示)。
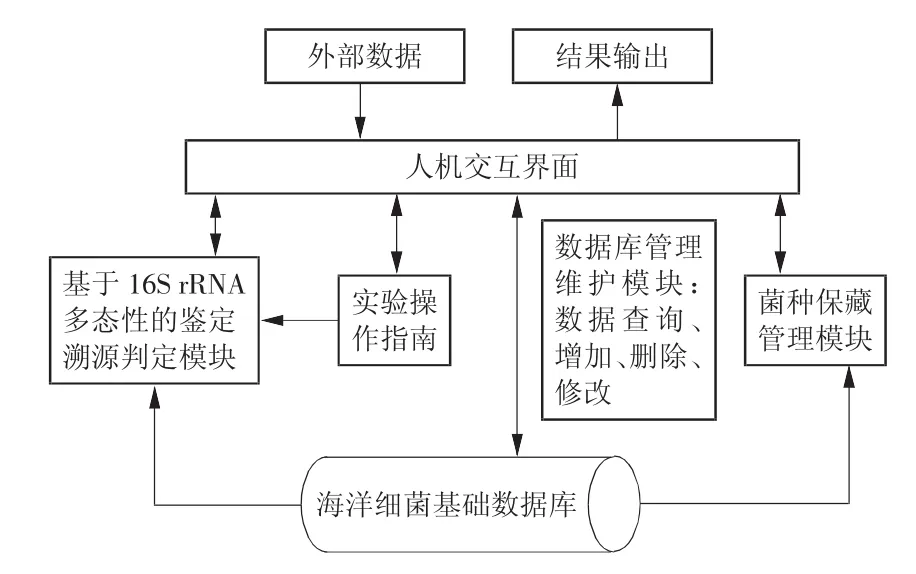
图1 软件系统整体结构示意图
图2 基础数据库的主界面
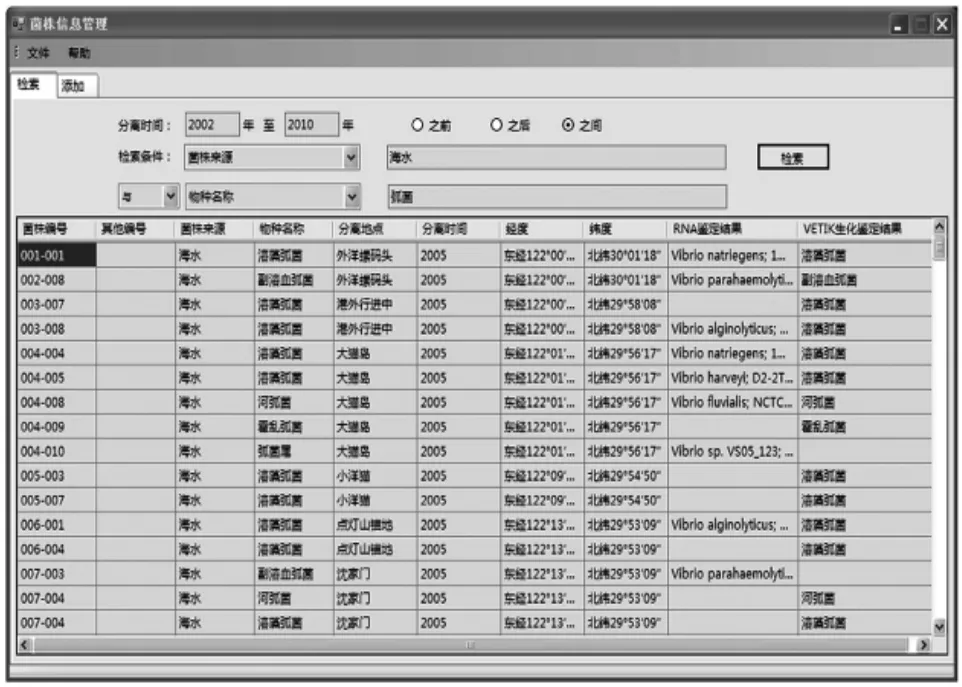
图3 菌株信息管理界面
2.2.2 菌株添加
在菌株添加功能中,为保证数据库资料的准确性和保密性,系统禁止一般用户进行修改,只有得到授权后才能对数据库中的信息进行增减和修改。点击进入“添加”选项卡,用户可添加新录入的菌株信息(如图4所示)。其中,菌株编号、物种名称、分离时间等用红色“*”标注的字段为必填字段。为保证数据库录入菌株的唯一性,添加菌株前需先检索并确认此菌株编号在数据库中不存在。
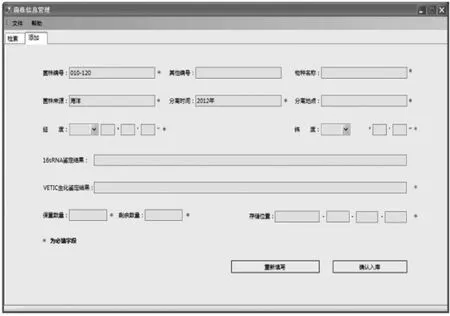
图4 菌株信息添加界面
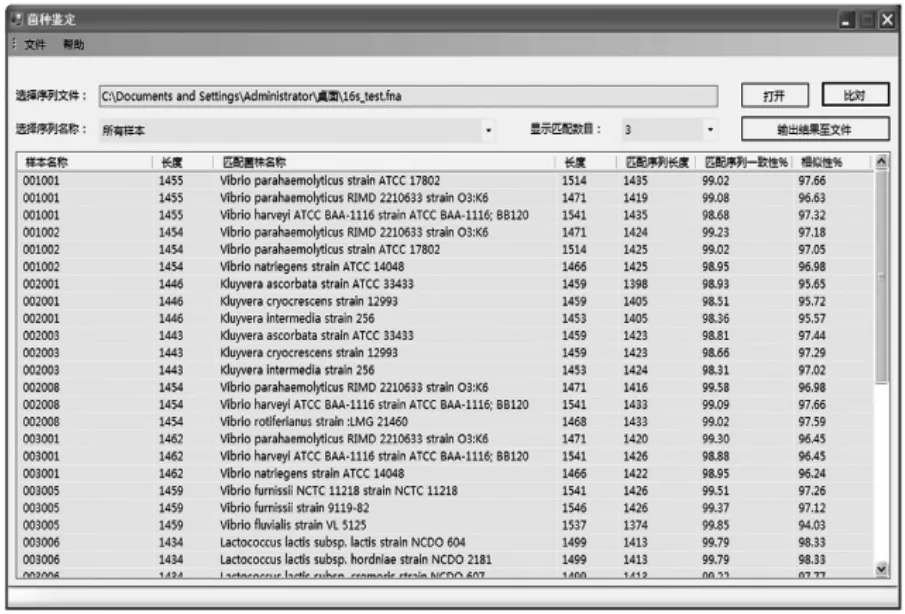
图5 16S rRNA序列文件导入界面
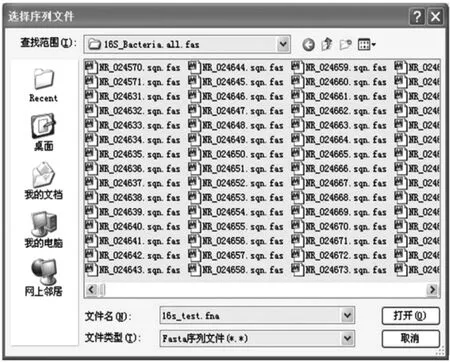
图6 基于16S rRNA序列比对的菌株鉴定界面
2.3 菌种鉴定模块
菌种鉴定模块以16S rRNA序列比对为基础,除了本研究中新获得的432条16S rRNA序列信息外,我们在数据库中还收录了8 000多条已知种属16S rRNA序列,并将本地化比对程序与数据库系统进行了整合和优化。点击工具栏中的“菌种鉴定”按钮,将启动菌种鉴定模块。点击“打开”按钮,选择待鉴定菌株的16S rRNA的Fasta序列文件(如图5所示)。点击“比对”按钮,若干分钟后,界面下方将出现待鉴定菌株与本地化16S rRNA数据库的比对结果,系统通过序列的一致性参数及覆盖比例计算相似性指数(similar index,SI),按照从高到低的顺序显示1~5条(用户可选)序列在列表中(如图6所示)。点击“输出结果至文件”按钮,可以将鉴定结果输出至本地Excel文件。
2.4 菌种保藏模块
菌种保存模块以海洋细菌基础数据库为核心,记录菌种的保藏位置、保种数量、剩余数量等资料,并可根据菌种的使用情况进行实时更新;同时菌种的保藏资料与菌种的背景资料库进行了关联,可方便调用其分离时间、地点、鉴定结果等信息,方便临床及科研人员对海洋细菌分离株进行深入研究。点击工具栏中的“菌种保藏”按钮,将启动菌种保藏模块,用户在“菌株编号”一栏中输入菌株编号后,点击“检索”按钮,菌种的背景信息及保藏信息将显示在对应的文本栏中。此时除“菌株编号”不能编辑外,保种数量、剩余数量及存储位置等信息均可任意修改,编辑完成后点击“修改”按钮即可保存入库。如果此菌株在库中已不存在,用户可点击“删除”按钮对其进行删除操作(如图7所示)。
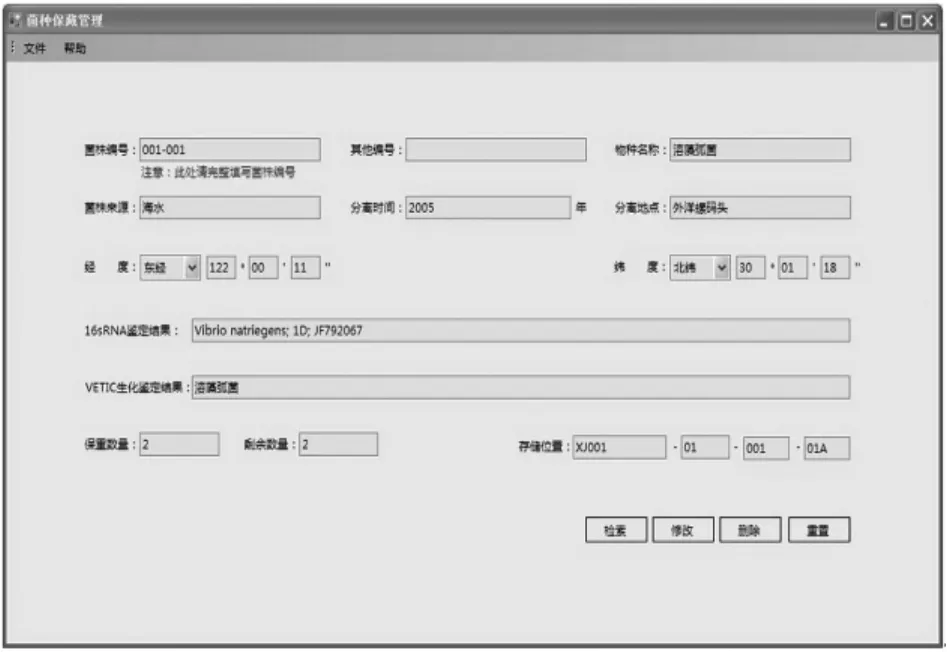
图7 菌种保藏管理界面
3 数据库与软件系统的设计与实现
3.1 数据库的构建
我们采用Microsoft Access 2007构建底层的关系数据库[5],数据库主要由海洋细菌背景信息和NCBI 16S rRNA序列信息2个部分组成,在Access中分别以对应名称的二维表格实现。
海洋背景信息表为本底数据库,是软件系统管理和保藏模块的直接操作对象,包含菌株编号、菌株来源、分离时间、分离地点、物种名称、经度、纬度、VETIK生化鉴定结果、16S rRNA鉴定结果、保种数量、存储位置和剩余数量等12个特征属性,分别以文本或数字形式存储,其中“菌株编号”被设为主键。
NCBI 16S rRNA序列信息表的作用主要是为软件系统的菌株鉴定功能提供16S rRNA序列及已知菌株的物种分类信息,是对NCBI数据库中的16S rRNA序列和背景信息整合而成的规范化表格,包含菌株ID、物种分类和DNA序列3个属性,以文本形式存储,其中“菌株ID”被设为主键。
3.2 软件系统的实现
我们选择Visual Studio 2010设计前台的应用程序,所开发的人机交互界面采用Windows视窗模式,由标题栏、菜单栏、工作窗口构成,用以处理数据录入、浏览、查询、更新等操作。系统以ADO技术来连接并访问在后台运行的Access数据库,用SQL语言对后台数据库进行检索、添加、删除和编辑。
为实现系统的菌株鉴定功能,我们设计了基于序列相似性的物种判断模块。用户提交未知菌株的16S rRNA序列后,系统后台调用NCBI开发的短序列比对工具Blast[6],对所提交的序列与数据库中已知菌株的16S rRNA进行相似性比对。比对完成后,后台对Blast输出的结果进行处理,计算数据库中已知序列与未知菌株序列的相似性指数,此参数由Blast输出结果中序列的一致性和匹配序列的覆盖度(匹配序列的长度除以未知菌株序列的长度)相乘所得[7-9]。系统按照相似性高低,挑选匹配上的10条已知菌株的物种信息输出至图形用户界面。
为方便用户对数据库进行查询和维护,我们在设计菌株管理模块和保藏模块时,所编写的SQL语言支持用户对数据库的模糊查询和不同检索条件的任意组合。
4 展望
数据库在使用过程中可根据需求进行优化,比如增加16S rRNA构建发育树的功能,可以更加直观地表现不同海洋细菌菌属之间的进化关系,从而进行菌株分型的研究。临床菌株的分离和保存也是开展后续研究不可或缺的宝贵资源,加强临床菌株的保存和规范化管理对于后续科学研究至关重要,将该数据库进行改造,实现与检验信息系统的对接,将能够实现对临床菌株更好的保存和管理。
5 结语
综上所述,本研究构建的海洋细菌基础数据库相关软件系统能够实现对大量海洋细菌的规范化、信息化管理以及对未知菌种的快速分类鉴定,为我军海战伤员救治的战备工作提供了良好支撑,并为后续更深入研究海洋细菌的致病性、耐药性及海洋细菌活性物质的开发利用奠定基础。
[1]Vignier N,Barreau M,Olive C,et al.Human infection with Shewanella putrefaciens and S.algae:report of 16 cases in Martinique and review of the literature[J].Am J Trop Med Hyg,2013,89(1):151-156.
(▶▶▶▶)(◀◀◀◀)
[2]马聪,郝秀红,马学斌,等.中国海域海洋细菌抗生素敏感性分析[J].中国抗生素杂志,2013,38(1):53-57.
[3]马聪,陈昌国,蒋学兵,等.中国海域海洋细菌分布特征分析[J].解放军医学杂志,2012,37(9):909-913.
[4]马骢,郝秀红,付凯飞,等.海洋中分离的河流弧菌致病能力的研究[J].中华医院感染学杂志,2009,19(7):734-737.
[5]SilberschatzA,KorthHF,SudarshanS.Databasesystemconcepts[M]. New York:McGraw-Hill,2002.
[6]Tatusova T A,Madden T L.BLAST 2 Sequences,a new tool for comparing protein and nucleotide sequences[J].FEMS Microbiology Letters,1999,174(2):247-250.
[7]Shine J,Dalgarno L.Determinant of cistron specificity in bacterial ribosomes[J].Nature,1975,254(5 495):34-38.
[8]Brosius J,Palmer M L,Kennedy P J,et al.Complete nucleotide sequenceofa16SribosomalRNAgenefromEscherichiacoli[J].ProceedingsoftheNationalAcademyofSciences,1978,75(10):4801-4805.
[9]Kim O S,Cho Y J,Lee K,et al.Introducing EzTaxon-e:a prokaryotic 16S rRNA gene sequence database with phylotypes that represent uncultured species[J].International journal of systematic and evolutionary microbiology,2012,62(Pt 3):716-721.
(收稿:2013-12-22 修回:2014-03-25)
Design of software system based on marine bacteria basic database
LI Yan-jun1,YAN Yan-feng2,CUI Yu-jun2,QIAN Yang-hui1,ZHAO Qiang-yuan1
(1.Department of Laboratory,Navy General Hospital,Beijing 100048,China; 2.Institute of Microbiology and Epidemiology,Academy of Military Medical Sciences,Beijing 100071,China)
ObjectiveTo establish the software system of the marine bacteria database in order to regularly and systematically manage the marine bacteria in our hospital.MethodsThe background information of bacteria and part of 16S rRNA sequences were complemented,and the software were developed and debugged.ResultsThe software based on the database was established and possessed the bacteria management,identification,and conserve modules and could realize the functions of querying bacteria information,identifying species based on 16S rRNA sequence and managing the conserved bacteria.ConclusionThe software system of the database provided useful tools for conveniently managing and identifying the marine bacteria.[Chinese Medical Equipment Journal,2015,36(1):58-60,65]
marine bacteria;database;software system
R318;TP311.13
A
1003-8868(2015)01-0058-04
10.7687/J.ISSN1003-8868.2015.01.058
国家自然科学基金(31170008);海军总医院创新培育基金(CX201003)
李艳君(1976—),女,博士,副主任医师,主要从事微生物检验、海洋细菌鉴定及基因组多态性方面的研究工作,E-mail:liyanjun1230@163.com。
100048北京,海军总医院检验科(李艳君,钱扬会,赵强元);100071北京,军事医学科学院微生物流行病研究所分析微生物实验室(颜焱峰,崔玉军)
赵强元,E-mail:zhaoqiangyuan001@163.com
