银杏叶绿素a/b结合蛋白基因(GbLhcb4)及其启动子克隆
2015-12-20王欢利刘新亮郁万文李广平曹福亮
王欢利,刘新亮,郁万文,李广平,曹福亮
(南京林业大学 林学院 南方现代林业协同创新中心, 江苏 南京 210037)
银杏叶绿素a/b结合蛋白基因(GbLhcb4)及其启动子克隆
王欢利,刘新亮,郁万文,李广平,曹福亮
(南京林业大学 林学院 南方现代林业协同创新中心, 江苏 南京 210037)
为了探究银杏中叶绿素a/b结合蛋白基因(GbLhcb4)的结构及其相关的转录调控元件,基于Roche 454高通量测序获得的GbLhcb4基因片段,采用RACE技术分离得到该基因的cDNA序列,并通过染色体步移技术获得该基因启动子序列,结合生物信息学技术分析基因序列的特征、系统进化地位及启动子区域的转录调控元件。序列分析表明,GbLhcb4基因全长1 200 bp,含有一个834 bp开放阅读框,编码277个氨基酸,相对分子量为30.65 kD,等电点为5.17。同源分析表明,该基因编码蛋白与北美云杉PsLhcb4蛋白(GenBank登录号:ABK21281.1)相似性最高,为74.7%。启动子顺式作用元件预测表明,启动子区域包含多个光响应元件及逆境响应元件,同时还存在激素响应、组织特异性表达、细胞周期等相关调控元件。此研究分离得到银杏GbLhcb4基因及启动子序列,并进行了相关的生物信息学分析,为探究银杏GbLhcb4基因应答环境信号的转录调控机制提供了分子基础。
银杏;叶绿素a/b结合蛋白基因;启动子分离;RACE技术
光合作用产物是人类赖以生存和发展的物质基础,因此植物光合作用的研究一直是生物学的研究热点。在高等植物中,捕光叶绿素a/b结合 蛋 白(Light-harvesting chlorophyll a/b-binding proteins,LhcPs)是光合作用中的重要功能蛋白与叶绿素a/b 形成复合体,将光能迅速传到光系统I(PS I)和光系统II(PS II)的反应中心,使光能转化为化学能,促进光合反应的进行[1]。LhcPs在类囊体膜上的含量最为丰富,其结合的叶绿素约占类囊体膜上色素量的50%。研究发现编码LhcPs的同源基因定位于细胞核,分属于10个基因家族。PS I包含4种捕光色素蛋白,分别由Lhca1、Lhca2、Lhca3和Lhca4编码;PS II包含3种主要捕光色素蛋白(major Lhc II),分别由Lhcb1、Lhcb2和Lhcb3编码,以及3种次要捕光色素蛋白(minor Lhc II),分别由Lhcb4(CP29)、Lhcb5(CP24)和Lhcb6(CP26)编码[2]。主要捕光色素蛋白具有高度的同源性,形成同源或异源三聚体;次要捕光色素蛋白大多以单体的形式存在,包括PSII内部天线叶绿素a/b结合复合体CP29、CP26和CP24,较主要捕光色素蛋白更靠近PS II反应中心[3]。主要和次要捕光色素蛋白复合体构成PS II外周捕光天线,其功能主要有以下四个方面:传递和捕获光能;分配和平衡 PS II和 PS I 中的能量;光保护和过剩能量耗散;维持类囊体膜的结构[4-8]。
自20世纪末期至今已有少数植物的Lhcb基因被相继克隆,如拟南芥[9-10]、小麦、水稻[11]、毛竹[12]、长白松[13]、银杏[14-16]、大麦[3]、葡萄[17]、花椰菜[18]、萝卜、羽衣甘蓝、盐生杜氏藻[19]等。此外有研究表明在低温、高盐、干旱及病害的条件下Lhc基因表达下调[3]。目前NCBI公布的银杏Lhc基因为GbLhcb1,而另几种Lhc基因还未见报道[17]。本研究基于银杏叶片高通量测序的基础上,根据基因注释结果为Lhcb4的基因片段设计5′ 及3′ 引物,利用RACE技术扩增GbLhcb4基因全长序列,并进行生物信息学分析;同时应用染色体步移技术扩增GbLhcb4基因的启动子区域,进而分析有关的调控元件,目的在于丰富Lhcb基因家族成员,以便进一步了解Lhcb家族基因在不同植物中的结构、功能及其表达情况。
1 材料与方法
1.1 材 料
银杏叶片于2012年4月下旬采自南京林业大学银杏园,叶片经酒精擦拭后,液氮速冻,-80 ℃保存。
SmarterTMRACE cDNA Amplification Kit、Advantage 2 Polymerase Mix和Genome WalkerTMUniversal Kit购自clontech公司。各种限制性内切酶、T4 DNA 连接酶、ExTaq DNA聚合酶、PMD-18T 载体、DNA凝胶纯化回收试剂盒均购自大连宝生物工程公司。其他试剂均为国产分析纯。试验中所选用的引物序列及反应程序参考表1和表2。引物序列交由上海捷瑞生物工程有限公司合成。基因测序由上海美吉生物工程有限公司完成。
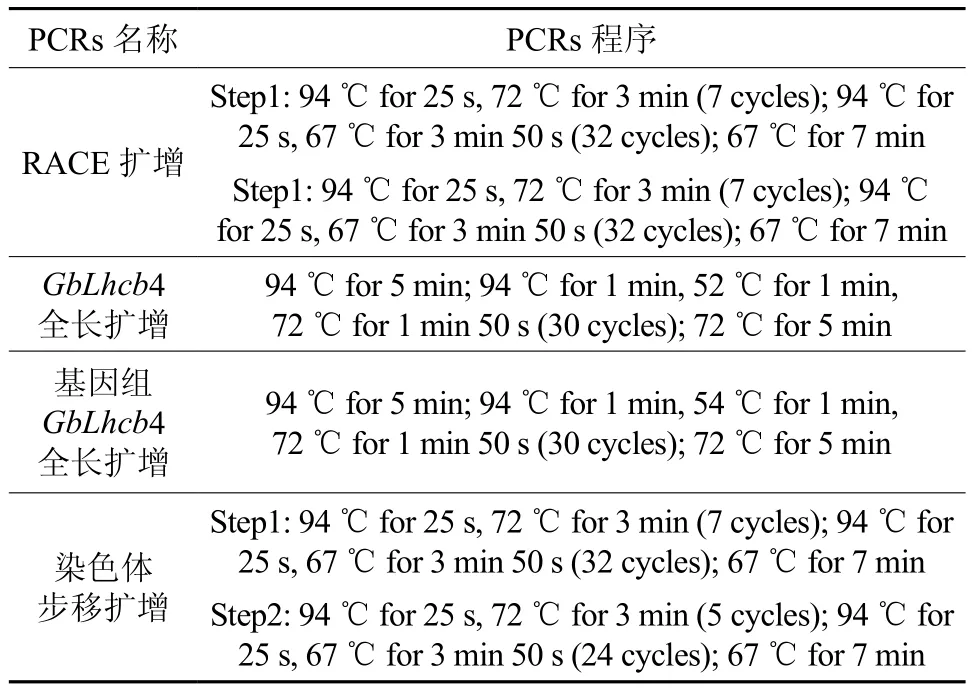
表2 PCR扩增程序Table 2 Amplification program of PCR
1.2 方 法
1.2.1 RNA及DNA的提取
叶片总RNA的提取采用CTAB法[20],DNA的提取参考魏春红等[21]的改良CTAB法。采用1%琼脂糖凝胶电泳检测核酸的完整性并用紫外分光光度计(UV2102,UNICO)检测核酸的浓度。
1.2.2 cDNA的合成
反转录cDNA合成采用Takara公司的Prime ScriptTM第一链cDNA合成试剂盒5' RACE和3'RACE反转录cDNA模板合成参考Clontech公司的SmarterTMRACE cDNA Ampli fication Kit说明书操作。
1.2.3 RACE反应及基因全长的获得
RACE PCR 使用巢式PCR 扩增,扩增程序参见Clontech公司的SmarterTMRACE cDNA Ampli fication Kit说明书。反应过程采用clontech公司的Advantage 2 Polymerase Mix基因。PCR产物经1%琼脂糖凝胶电泳分离,采用Agarose Gel DNA puri fieation Kit回收目的片段,亚克隆至PMD-18 T Vector中,热激法转入大肠杆菌后测序。
1.2.4 GbLhcb4基因全长及基因组序列的扩增
扩增选用Takara公司的ExTaq DNA聚合酶。引物序列及反应程序参见表1和表2。
1.2.5 启动子的克隆及测序
基因启动子参考Clontech公司的Genome WalkerTMUniversal Kit 说明书。基因组酶切选用平末端限制酶DraI、EcoRV、PvuII和StuI酶切12 h,分别纯化回收;回收产物与Genome WalkerTMUniversal Kit 试剂盒提供的接头连接,16 ℃条件下连接过夜,连接产物稀释10 倍,置于-20 ℃保存,作为步移PCR 扩增模板。步移中使用到两组巢式引物AP1、Lhcb4 gsp1及AP2、Lhcb4 gsp1(见表1)。扩增序列连接到pMD18-T 载体并测序。
1.2.6 生物信息学分析
用生物信息学序列比对软件Vector NTI Suite 11.5进行不同物种间同源基因的核苷酸序列比对、测序片段的拼接。采用Primer Premier 5.0和Oligo 6生物学软件设计引物。利用BlastP(http://www.ncbi.nlm.nih.gov/BLAST/) 进 行 同源 性 搜 索。ProtParam (http://au.expasy.org/tools/protparam.html)计算蛋白的分子量及等电点。Signal P4. 1 Server (http:∥ www. cbs. dtu. dk /services / SignalP)预测信号肽及剪切位点。蛋白质的疏水性和跨膜结构的预测分别采用ProtScale Server(http://www.expasy.org/cgi-bin/protscale.pl) 和 TMHMM Server Version 2.0(http://www.cbs.dtu.dk/services/TMHMM)。使用植物调控元件 分 析 软 件 PLACE(http://www.dna.affrc.go.jp/PLACE/) 及 PlantCare(http://bioinformatics.psb.ugent.be/webtools/plantcare/html)对克隆的启动子序列进行转录调控元件预测。用SMART(http://smart.embl-heidelberg.de/)和 MOTIFSCAN(http://prosite.expasy.org/)等软件对GbLhcb4蛋白的功能结构域进行分析。依据NCBI数据库提供的已发布Lhcb氨基酸序列,结合银杏GbLhcb序列,用MEGA5.03提供的近邻相接法(Neighbor-Joining Tree)绘制系统发育树。进化树各节点处数字表示bootstrap值,重复1 000次。
2 结果与分析
2.1 GbLhcb4 cDNA 的克隆及序列分析
测序结果拼接后得到1 200 bp全长序列(GeneBank登录号:KC818609),包含一个834 bp 最大读码框(ORF);blast分析结果显示该基因与植物Lhcb4基因序列有较高的同源性,因此,将银杏中该基因定义为GbLhcb4。序列分析表明,GbLhcb4含有保守的植物基因翻译起序列AACAATGG,与Lutcke等[22]统计的植物基因翻译起始序列一致。GbLhcb4编码1条278个氨基酸残基的多肽(见图1),预测分子质量为30.65 kD,等电点为5.17。SignalP预测显示GbLhcb4蛋白质不存在信号肽序列。蛋白疏水性和跨膜结构显示,GbLhcb4可能存在3个跨膜结构域,分位于142~164、179~201和246~263氨基酸段朝向类囊体膜内,羧基端朝向基质一侧(见图2)。
用SMART和MOTIFSCAN在线分析软件对GbLhcb4基因编码的氨基酸进行分析,显示第64~261位包括一个典型的捕光叶绿素a/b结合蛋白功能域(chlorophyll a/b binding domain);第 24~ 26,53~ 55,263~ 265和 276~ 278位为蛋白激酶C的磷酸化位点(Protein kinase C phosphorylation site),蛋白激酶C 是G 蛋白偶联受体系统中的效应物,在非活性状态下是水溶性的,游离存在于胞质溶胶中,激活后成为膜结合的酶;第70~75,107~112和150~155位为N-肉豆蔻酰位点(N-myristoylation site);第22~25,23~26和187~190位为N-糖基化位点(N-glycosylation site)(见图1)。
GbLhcb4氨基酸序列与北美云杉Picea sitchensis(ABK21281.1) 的 同 源 性 最 高,为74.7%;而与伞藻Acetabularia acetabulum(DAA05897.1 )的同源性最低,为46.3%;与蓖 麻Ricinus communis(XP_002528105.1)、 陆地棉Gossypium hirsutum(ACO51068.1)、毛果杨Populus trichocarpa(XP_002323575.1)、 绿豆Vigna radiata(AAD27878.1)、玉米Zea mays(NP_001105502.1)、拟南芥Arabidopsis thaliana(NP_181539.1)和小立碗藓Physcomitrellapatenssubsp. Patens(XP_001785452.1)相似性为64.7%~69.9%(见图3)。

图1 GbLhcb4基因核酸和编码氨基酸序列及启动子序列Fig. 1 Gene nucleic acid, encoded amino acid sequences and promoter sequence of GbLhcb4
2.2 GbLhcb4 进化树分析
采用近邻相接法(Mega5.03)对不同植物Lhcb的氨基酸序列进行进化树分析,结果发现,Lhcb4蛋白的进化独立其他5类Lhcb蛋白。其中植物Lhcb4氨基酸根据所选物种分为5组(见图3):藻类植物、苔藓类植物、裸子植物、单子叶植物和双子叶植物。其中单子叶植物玉米、水稻和毛竹等为一组,分化时间最迟,集中在0.01~0.03;双子叶植物龙眼、蓖麻、陆地棉和拟南芥等为一组,银杏和北美云杉作为裸子植物一组,集中在0.02~0.14;小立碗藓作为较原始的苔藓类植物单独为一组,分歧时间大约为0.2;伞藻作为更原始的藻类植物单独为一组,分歧时间最早。
2.3 GbLhcb4 内含子序列分析
克隆的基因片段测序结果显示,GbLhcb4基因DNA序列长度为1 192 bp,与GbLhcb4基因cDNA序列相比,该基因包含2个外显子和1个内含子(见图1)。
2.4 启动子顺式作用元件预测和序列分析
经过2轮PCR,从银杏基因组DNA的PvuII酶切体系中扩增一条位于750~1 000 bp之间的目的条带。将目的片段回收亚克隆后直接测序,结果表明,目的片段实际长度为766 bp。利用Vector NTI Suite 11.5软件对测序结果进行序列分析发现,该序列中包含有15 bp的GbLhcb4基因编码区、89 bp的5'-UTR区和662 bp的基因启动子区(见图1)。

图2 Lhcb4 氨基酸序列多重对比分析Fig.2 Multiple comparative analyses of Lhcb4 amino acid sequences
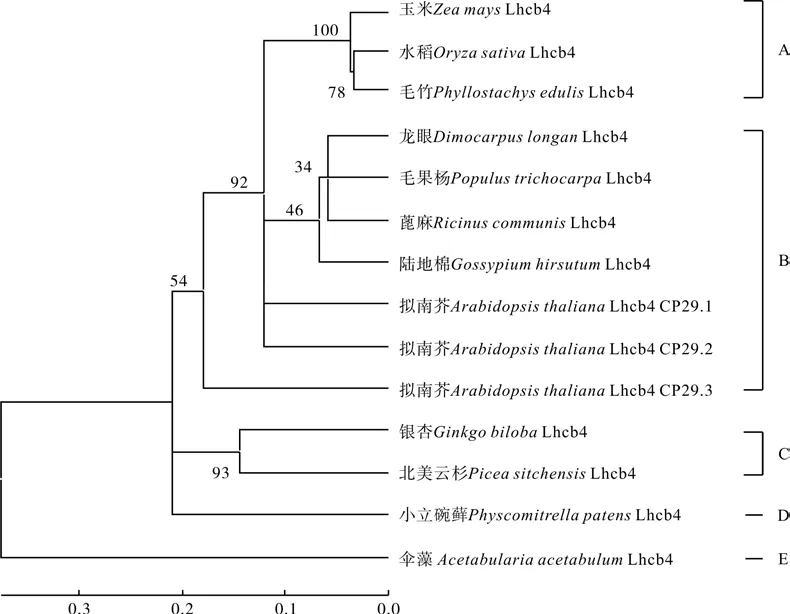
图3 采用MEGA5.03构建植物Lhcb4蛋白系统进化树Fig. 3 Phylogenetic tree of Lhcb4 protein system constructed by MEGA5.03
利用在线软件PLACE和PlantCARE对GbLhcb4基因启动子序列中可能含有的顺式作用元件进行分析,结果发现该段序列含有3个拷贝的CAATBOX和4拷贝的TATABOX。除此之外该启动子区域还具有22个光响应元件(ACE,CGACGDSAMY3,GATABOX,GBOX,GT1CONSENSUS,IBOX,REALPHALGLhcb21,SORLIP1AT,SORLIP2AT)、26个 逆 境 胁 迫响 应 元 件(ACGTATERD,CCAATBOX1,C U R E C O R E C R,D O F C O R E Z M,GT1GMSCAM4,MYB1AT,MYBPZM,M Y B 2 C O N S E N S U S,P RE AT P R O D H,SURECOREATSULTR11,UPRMOTIFIAT,WRKY71O,WUNMOTIF)、6个激素响应顺式作用元件(ARFAT,ARR1AT,CGTCAMOTIF,MYB1AT,MYB2CONSENSUS,WRKY71O)(见表3);3个组织特异性表达元件包括1个叶肉组织特异性表达元件(CACTFTPPCA1)、1个K离子通道调节组织特异表达元件(TAAAGSTKST1)和1个MYB识别位点(MYBPZM),2个调节细胞周期相关的元件(E2FCONSENSUS,HEXMOTIFTAH3H4),1个叶绿体核糖体蛋白基因负性调节元件(S1FBOXSORPS1L21)(见表3)。
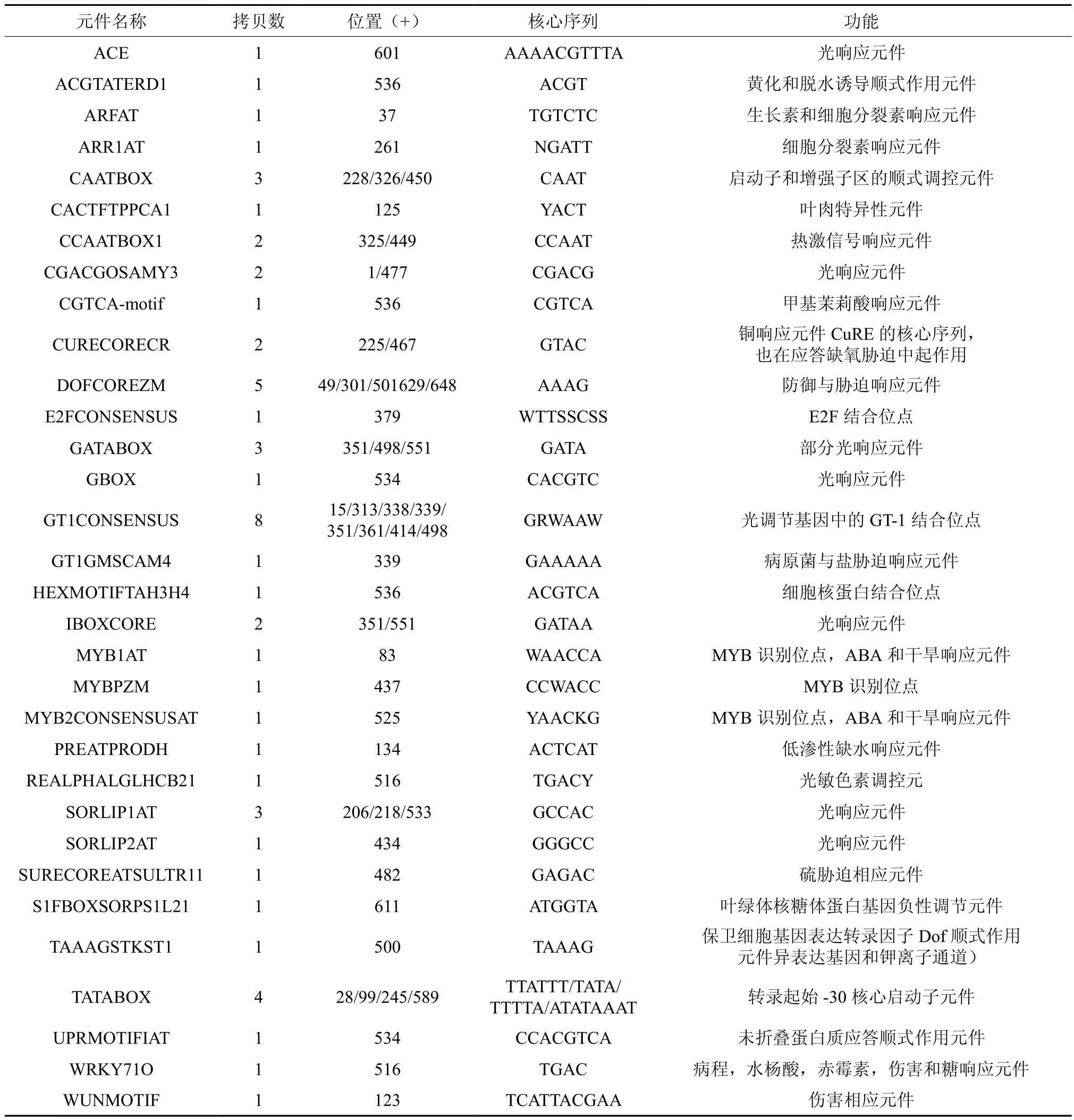
表3 GbLhcb4启动子区域主要顺式作用元件分析Table 3 Some important cis-acting regulatory elements in promoter sequence of GbLhcb4
3 讨 论
本研究从古老的裸子植物银杏中分离得到捕光叶绿素 a/b 结合蛋白基因GbLhcb4,序列分析结果表明GbLhcb4基因与高等植物Lhcb4基因具较高的相似性,显示了Lhcb4进化过程中的保守性。系统进化树分析表明,GbLhcb4在进化上分化较早,仅次于伞藻和小立碗藓,而与裸子植物北美云杉具有相近的分化时间,这与物种由简单到复杂的进化理论相一致。蛋白序列分析结果显示:此蛋白含有3个跨膜区域和1个叶绿素a/b结合位点,此研究表明Lhcb4可能是一类膜蛋白,执行与色素分子结合的功能[21];4个蛋白激酶C的磷酸化位点可能与调节Lhc蛋白的光合磷酸化过程相关,完成PS I和PS II之间的转换,从而使两个系统的光能利用达到平衡[24-25];3个N-豆蔻酰基化位点和3个N-糖基化位点这可能与蛋白质翻译后修饰有关。
此外,启动子的克隆和顺式作用元件分析是进行基因转录调控机制研究和基因功能分析的前提。目前许多学者对Lhc基因进行了研究,研究结果表明Lhc基因的表达不仅受光[26]和昼夜节律[27]的调控, 而且还受到植物不同的发育阶段[27-28]与激素水平[10,24,29]及外界环境因子[30-31]的影响。然而,不同Lhc基因的诱导条件存在一定差异,但是控制Lhc基因特异性表达的机制还未得到系统的揭示。银杏GbLhcb4启动子序列分析发现,其存在多种光响应、逆境响应、激素响应、组织特异性表达、细胞周期和叶绿体核糖体蛋白基因负性调节元件。本研究分离到的银杏GbLhcb4基因及其启动子序列,为开展银杏GbLhcb4基因功能及转录调控机理的研究,揭示Lhcb4基因响应环境信号的分子机制提供了遗传基础。
[1]Raghvendra A S. Photosynthesis: a comprehensive treatise[M].Cambridge: Cambridge University Press, 1998: 644-653.
[2]Jansson S, Pichersky E A. A nomenclature for genes encoding the chlorophyll a/b binding proteins of higher plants [J]. Plant Mol.Biol. Rep., 1992,10(3): 242-253.
[3]Xia Y, Ning Z, Bai G,et al.Allelic variations of a light harvesting chlorophyll A/B binding protein gene (Lhcb1) associated with agronomic traits in barley[J]. PLoS ONE, 2012,7(5): e37573.
[4]Klimmek F, Sjodin A, Noutsos C. Abundantly and rarely expressed Lhc protein genes exhibit distinct regulation patterns in plants[J]. Plant Physiol., 2006,140(3): 793-804.
[5]Teramoto H, Ono T, Minagawa J. Identification ofLhcbgene family encoding the light-harvesting chlorophyll-a/b proteins of photosystem II inChlamydomonas reinhardtii[J]. Plant Cell Physiol., 2001,42(8): 849-856.
[6]Liu C, Zhang Y, Cao D,et al.Structural and functional analysis of the antiparallel strands in the lumenal loop of the major lightharvesting chlorophyll a/b complex of photosystem II (LhcIIb)by site-directed mutagenesis[J]. J. Biol. Chem., 2008,283(1):487-495.
[7]Kolanus W, Scharnhorst C, Kühne U,et al.The structure and light-dependent transient expression of a nuclear-encoded chloroplast protein gene from pea (Pisum sativumL.)[J]. Mol.Gen. Genet., 1987,209(2): 234-239.
[8]Luciński R, Jackowski G. The structure, functions and degradation of pigment binding proteins of photosystem Ⅱ[J].Acta Biochim. Pol., 2006,53(4): 693-708.
[9]McGrath J M, Terzaghi W B, Sridhar P,et al.Sequence of the fourth anf fifth Photosystem II type I chlorophyll a/b binding protein genes ofArabidopsis thalianaand evidence for the presence of a full complement of the extended CAB gene family[J]. Plant Mol. Biol., 1992,19(5): 725-733.
[10]Xu Y H, Liu R, Yan L,et al.Light-harvesting chlorophyll a/b-binding proteins are required for stomatal response to abscisic acid in Arabidopsis[J]. J. Exp. Bot., 2012,63(3): 1095-1106.
[11]Pavan U. Genome-wide analysis of the family of light-harvesting chlorophyll a/b-binding proteins in Arabidopsis and rice.[J]. Plant Signal Behav., 2010,5(12): 1537-1542.
[12]高志民, 刘 成, 刘颖丽, 等. 毛竹捕光叶绿素a/b结合蛋白基因cab-PhE1的克隆与表达分析[J]. 林业科学, 2009,45(3):145-149.
[13]Jansson S, Gustafsson P. Type and type-genes for cholophyll a/b binding proteins in the gymnospermPinus sylvestris(Soots pine):cDNA cloning and sequence analysis[J]. Plant Mol. Bio.,1990,14: 287-296.
[14]王义强, 谭晓风, 邢伟一, 等. 银杏叶绿素结合蛋白基因表达、全长cDNA克隆及分析[C]// 曹福亮. 全国第十九次银杏学术研讨会论文集.北京: 中国林业出版社, 2012: 63-72.
[15]王义强, 何 方, 谭晓风, 等. 基于EST银杏光合作用相关基因表达分析[J]. 中南林业科技大学学报, 2011, 31(3): 54-59.
[16]王义强,谭晓风,陈介南,等.银杏雌树成熟叶cDNA文库的构建[J].中南林业科技大学学报,2009, 29 (1): 6-9.
[17]Petit A N, Wojnarowiez G, Panon M L,et al.Botryticides affect grapevine leaf photosynthesis without inducing defense mechanisms[J]. Planta, 2009,229(3): 497-506.
[18]Damarajua S, Schledeb S, Eckhardta U,et al.Functions of the water soluble chlorophyll-binding protein in plants[J]. J. Plant Physiol., 2011,168(12): 1444-1451.
[19]魏 亮. 盐生杜氏藻光系统Ⅱ主要捕光叶绿素a/b结合蛋白基因的克隆与表达研究[D]. 成都: 四川大学, 2006.
[20]程水源, 陈昆松, 杜何为, 等. 银杏RNA的提取[J]. 果树学报 , 2005,22(4): 428-429.
[21]魏春红, 李 毅. 现代分子生物学实验[M]. 北京: 高等教育出版社 , 2006: 135-137.
[22]Lutcke H A, Chow K C, Mickel F S,et al.Selection of AUG initiation codons differs in plants and animals[J]. EMBO J.,1987,6(1): 43-48.
[23]Chinn E, Silverthorme J. Light-dependent choloplast development and expression of a light -harvesting chlorophyll a/b-binding protein gene in the gymnospermGinkgo biloba[J].Plant Physiol., 1993,103: 727-732.
[24]未 丽, 徐秉良, 雷江丽, 等. 胡杨叶绿素a/b结合蛋白基因的克隆及序列特性分析[J]. 中国农业科技导报, 2008,10(4):63-69.
[25]Kargul J, Barber J. Photosynthetic acclimation: Structural reorganisation of light harvesting antenna-role of redoxdependent phosphorylation of major and minor chlorophyll a/b binding proteins[J]. FEBS J., 2008,275(6): 1056-1068.
[26]Thompson W F. Physiological and molecular studies of lightregulated nuclear genes in high plants[J]. Annu. Rev. Plant Physiol. Plant Mol. Biol., 1991,42: 423-466.
[27]Aoki S, Kato S, Ichikawa K,et al. Circadian expression of thePpLhcb2 gene encoding a major light-harvesting chlorophyll a/b-binding protein in the mossPhyscomitrella patens[J]. Plant Cell Physiol., 2004, 45(1): 68-76.
[28]Chang Y C, Walling L L. Spatial and temporal expression of Cab mRNAs in cotyledons of the developing soybean seedling[J].Planta, 1992,186(2): 262-272.
[29]石兰馨, 张晓平, 梁厚果. 捕光叶绿素a/b结合蛋白和cab基因[J]. 植物生理学通讯, 1995,31(6): 470-476.
[30]乌云塔娜,王 淋,叶生晶.杜仲甲羟戊酸激酶(EuMK)基因鉴定及生物信息学分析[J].经济林研究,2014,32(1):6-12.
[31]梁楠松,周 姗,李蕾蕾,等.小黑杨bHLH转录因子基因和启动子的克隆与分析[J].经济林研究, 2013,31(4):58-66.
Cloning of light-harvesting cholorophyll a/b-binding protein coding gene(GbLhcb4) and promoter sequence fromGinkgo biloba
WANG Huan-li, LIU Xin-liang, YU Wan-wen, LI Guang-ping, CAO Fu-liang
(Collaborative Innovation Center of Southern Modern Forestry, College of Forestry, Nanjing Forestry University, Nanjing 210037,Jiangsu, China)
In order to explore structure and cis-regulatory elements of the gene of light-harvesting cholorophyll a/b-binding proteins in ginkgo (GbLhcb4), full-length cDNA sequence and promoter ofGbLhcb4 were cloned from ginkgo by using method of rapid ampli fication of cDNA ends and genome walking methods respectively, which based on the fragment ofGbLhcb4 acquired by Roche 454 high-throughput sequencing. The sequence analysis indicated that the length ofGbLhcb4 (GenBank number: KC818609) was 1200 bp, which contained a 834 bp open reading frame and encoded a 277 aa protein, with a predicted molecular weight of 30.65 kD and the isoelectric point of 5.17. Multiple alignment of deduced amino acid sequence showed that it had the highest sequence similarity,approximately 74.7% similarity with other Lhcb4 inPicea sitchensis. Cis-regulatory elements analysis revealed that light responsiveness elements, stress responsiveness, phytohormone responsiveness and tissue-speci fic cis-regulatory elements was in the promoter region ofGbLhcb4. The full-length sequence and promoter ofGbLhcb4 were obtained in this research and the structure andcis-elements were analyzed by a series of bioinformatics soft-wares. It will be helpful to understand the transcriptional regulatory mechanism ofGbLhcb4,when ginkgo responded to environment signals.
Ginkgo biloba; chlorophyll a/b binding protein gene (GbLhcb4); promoter isolation; RACE technologies separation
S792.95;Q786
A
1673-923X(2015)05-0114-08
10.14067/j.cnki.1673-923x.2015.05.020
2014-02-10
国家自然科学基金项目(31170627);江苏省自然基金项目(BK2010019);江苏省普通高校研究生科研创新计划项目(CXZZ12_0546);南京林业大学优秀博士学位论文创新基金项目
王欢利,博士生
曹福亮,教授;E-mail:samcaoster@gmail.com
王欢利,刘新亮,郁万文,等. 银杏叶绿素a/b结合蛋白基因(GbLhcb4)及其启动子克隆[J].中南林业科技大学学报, 2015,35(5): 114-121.
[本文编校:谢荣秀]
