我国草鱼野生群体D-Loop序列遗传变异分析
2015-11-29傅建军王荣泉沈玉帮宣云峰徐晓雁刘承初李家乐
傅建军 王荣泉 沈玉帮 宣云峰 徐晓雁 刘承初 李家乐
我国草鱼野生群体D-Loop序列遗传变异分析
傅建军1, 2王荣泉3沈玉帮1宣云峰3徐晓雁1刘承初2李家乐1
(1. 上海海洋大学省部共建水产种质资源发掘与利用教育部重点实验室, 上海 201306; 2. 上海海洋大学食品科学与工程博士后流动站, 上海 201306; 3. 苏州市吴江水产养殖有限公司, 农业部大宗淡水鱼类繁育与健康养殖技术重点实验室, 苏州 215221)
利用线粒体DNA的D-Loop区序列, 对来自长江水系(邗江、吴江、九江、石首、木洞和万州)、珠江水系(肇庆)和黑龙江水系(嫩江)的8个草鱼野生群体开展了遗传变异分析。在424尾鱼中检测到34个变异位点, 34个单倍型, 单倍型多样性介于0.474—0.708。群体间Kimura双参数遗传距离介于0.0020—0.0049。长江下游3个群体间遗传距离最近, 遗传分化不显著(>0.05); 肇庆群体与长江上游3个群体遗传距离较近, 与九江群体遗传分化不显著(>0.05); 嫩江群体与长江上游2个群体遗传距离较近, 与万州群体遗传分化不显著(>0.05)。遗传距离与地理距离存在极显著正相关(=0.61,<0.01)。分子方差分析显示, 不同流域间遗传变异占总变异26.24%, 差异极显著(<0.01)。34个单倍型分为2个分支, 分化极显著(ST=0.644,<0.01), 推测分化时间为第四纪更新世纪晚期。
草鱼; 野生群体; D-Loop序列; 遗传变异
草鱼()为我国土著鱼类, 自然分布于长江、珠江及黑龙江水系等流域中[1]。草鱼在我国具有悠久的养殖历史, 其养殖产量在淡水鱼类中位居世界第一[2]。近几十年来, 由于受环境破坏及不当利用等原因[3], 草鱼野生资源锐减并存在种质退化风险。当前, 开展良种培育是减少对草鱼野生资源过度开发和破坏, 并实现草鱼资源可持续利用的有效途径。
分子标记作为鱼类种质资源研究的有效手段[4], 可为制定合理的育种方案提供数据参考。其中, 线粒体DNA(mtDNA)变异可用于鱼类亲缘关系[5]、种属分类[6]、遗传分化[7]和遗传多样性[8]等研究。mtDNA标记被广泛用于草鱼群体遗传研究中。李思发等[9]和张四明等[10]用mtDNA限制性片段长度多态性(RFLP)方法分别对长江中下游和中游水系草鱼群体开展了遗传变异研究; Zhao等[11]基于3个线粒体编码基因(ND5、ND6和Cyt)和控制区(D-Loop)序列对长江流域草鱼群体进行了遗传结构及演化进行了分析; 宋晓等[12]通过线粒体D-Loop区和Ⅱ基因序列对我国土著群体和国外移居群体进行了遗传变异分析; 李树华等[13]基于线粒体D-Loop和Cyt基因序列对长江中游草鱼亲本增殖放流的遗传效果进行了评估。此外, 诸如RAPD[14]、TRAP[15]、ISSR[16]和SSR[17—19]等其他分子标记被广泛用于草鱼群体遗传研究。然而, 众多研究在样本采集、研究方法及侧重点等方面存在差异, 因此具有进一步开展草鱼群体遗传结构研究的必要。
本研究对长江、珠江和黑龙江水系的8个草鱼野生群体的mtDNA D-Loop序列进行PCR扩增和测序, 开展了群体间遗传变异和系统分化等研究, 旨在补充完善我国草鱼野生群体的种质资源研究, 为后期遗传育种提供数据参考。
1 材料与方法
1.1 实验材料与DNA提取
草鱼8个野生群体收集于长江、珠江及黑龙江水系(表1), 在前期研究[19]的基础上补充了一些个体(2013年5月)。实验样本为剪取的鳍条组织, 以无水乙醇固定。基因组DNA采用苯酚/氯仿法提取, 通过1% 琼脂糖胶检测其完整性, 经NanoDrop分光光度仪检测其纯度及浓度。DNA样品被稀释成20 ng/µL的终浓度, 保存于–20℃备用。
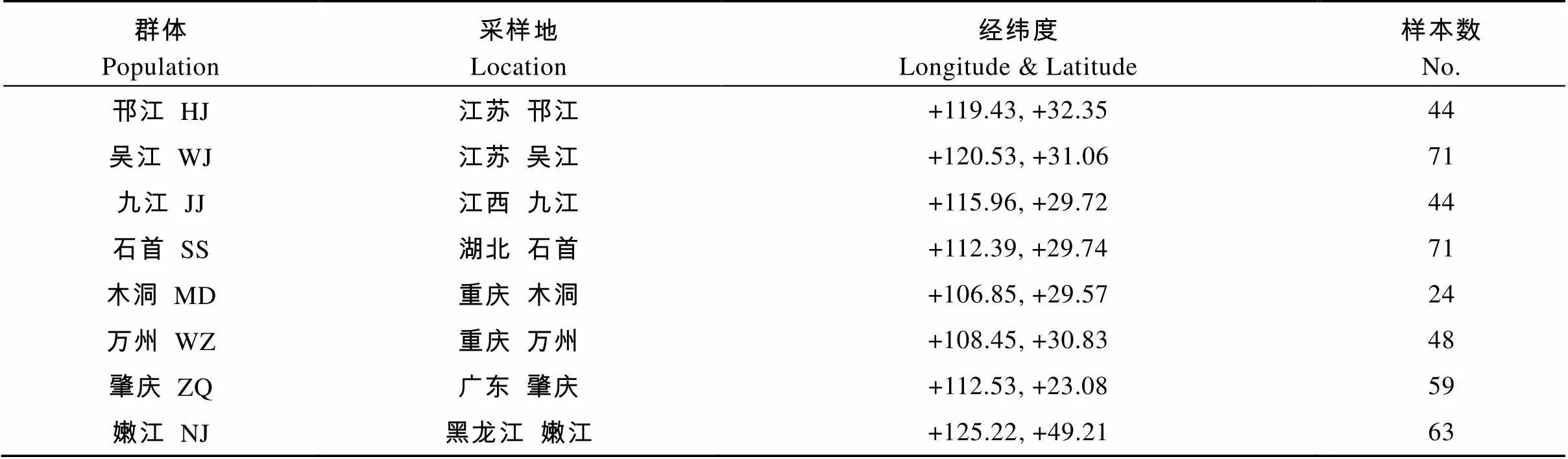
表1 草鱼样本采集信息
1.2 PCR扩增与测序
根据草鱼mtDNA全序列[20](GenBank序列号: NC_010288)设计D-Loop区的引物, 引物由上海生工生物工程有限公司合成。上游引物和下游引物序列分别为: DLF: 5¢-CCTAGCGCCCAGAAAAGGGAGAT T-3¢; DLR: 5¢-GCGGGGGATTGAGGGCATACTC-3¢。
PCR扩增体系为50 μL, 包括10× PCR Buffer 5 μL、MgCl2(25 mmol/L)、3 μL、dNTPs (2.5 mmol/L) 4 μL、DNA聚合酶(2.5 U/μL) 1 μL、上游及下游引物(10 mmol/L)各1 μL、基因组DNA (20 ng/μL) 2 μL, 补充无菌水33 μL。所需试剂购自天根生化科技(北京)有限公司。PCR扩增程序为: 94℃预变性3min; 94℃变性30s, 50℃复性30s, 72℃延伸2min, 扩增35个循环; 72℃延伸10min; 4℃保存。扩增反应在Eppendorf梯度PCR仪上完成。反应产物经1%琼脂糖凝胶电泳检测, 由上海迈浦生物科技有限公司通过ABI3730XL测序仪进行双向测序。
1.3 序列整理与数据分析
使用BioEdit 7.0软件[21]进行序列编辑, 并用Clustal X 1.81软件[22]进行同源比对和长度确定。采用DnaSP 5.0软件[23]统计多态性位点数()、单倍型个数()、单倍型多样性(d)、核苷酸多样性()、平均核苷酸差异数()和Tajima’s值等遗传多样性参数。
利用Arlequin 3.5软件[24]进行核苷酸组成统计、分子方差分析(AMOVA)和遗传分化指数(-statistics,ST)计算。利用MEGA 5.1软件[25]计算群体间的Kimura双参数模型(Kimura 2 Parameter, K2P)遗传距离, 并构建邻接(Neighbor-Joining, NJ)进化树。根据各采样点间的地理距离[26], 利用SPSS 16.0软件[27]分析与遗传距离间的相关性。
利用MEGA 5.1软件对单倍型构建最大简约(Maximum Parsimony, MP)进化树, 并计算单倍型分支间的K2P遗传距离, 利用Network 4.6软件[28]构建单倍型的简约中介(Reduced-Median, MJ)网络图, 并推算单倍型分化时间。
2 结果
2.1 草鱼D-Loop序列的变异位点与单倍型分布
共测序获得424尾草鱼的D-Loop区(898 bp)序列。核苷酸组成显示A+T含量(66.71%)明显高于G+C含量(33.29%)。在D-Loop区共发现34个变异位点。共检测到34个单倍型, 其中Hap1、Hap6、Hap20和Hap21为优势单倍型, 分别占个体数的32.31%、10.14%、28.54%和13.21%; 20个单倍型为除肇庆群体外的各群体所特有(表2)。
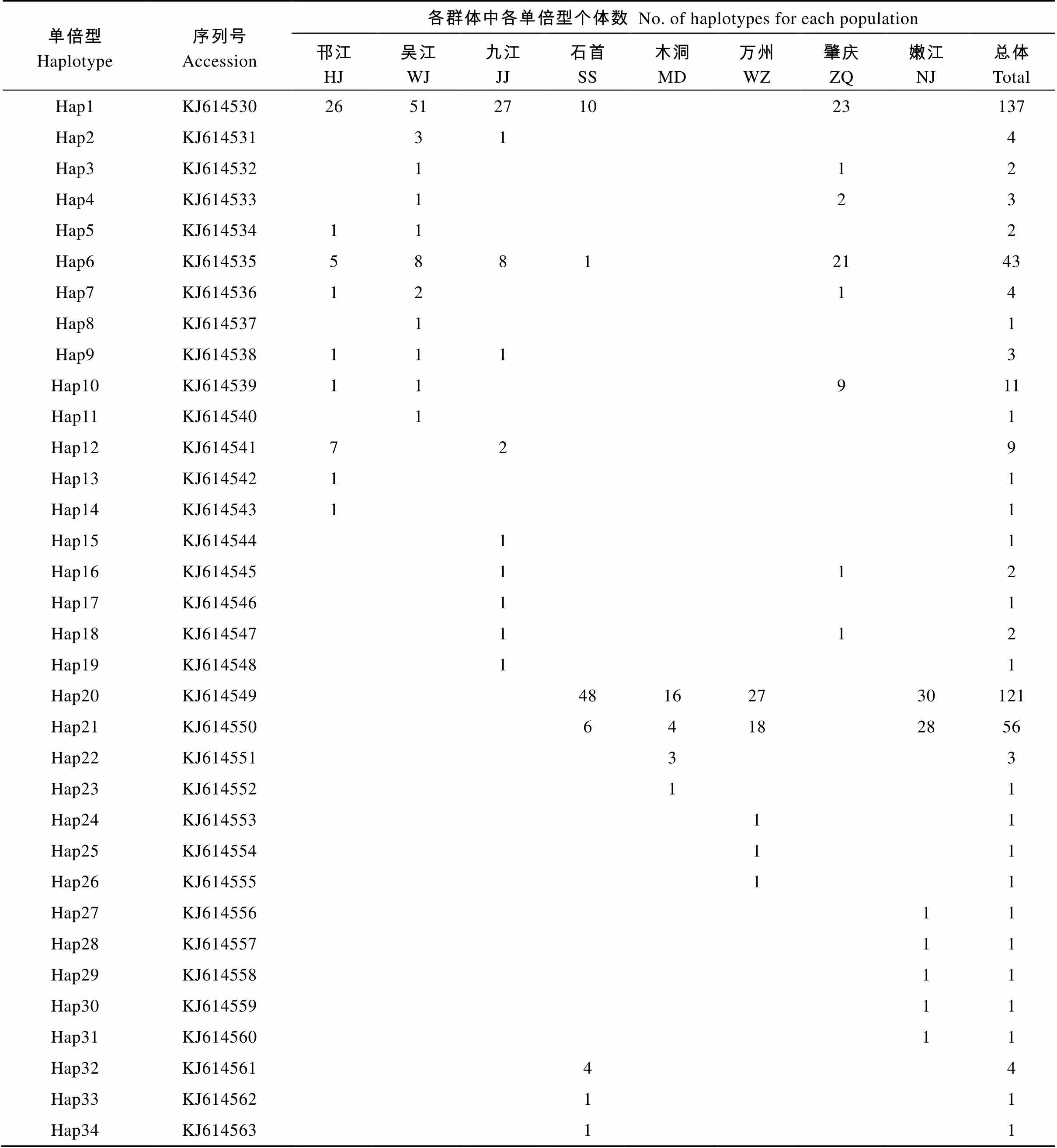
表2 草鱼群体中的单倍型分布情况
2.2 草鱼野生群体遗传多样性与群体间遗传距离
我国草鱼野生群体整体单倍型多样性为0.787(表3)。各群体的单倍型多样性介于0.474— 0.708, 核苷酸多样性介于0.0018—0.0037。其中, 吴江群体的遗传多样性水平最低(d= 0.474,=0.0018), 肇庆群体的单倍型多样性最高(d=0.708), 嫩江群体的核苷酸多样性最高(=0.0037)。群体中性检验显示, 邗江、吴江、九江和石首群体的Tajima’s值为负值; 其余群体为正值, 其中肇庆群体显著偏离中性(<0.05)。
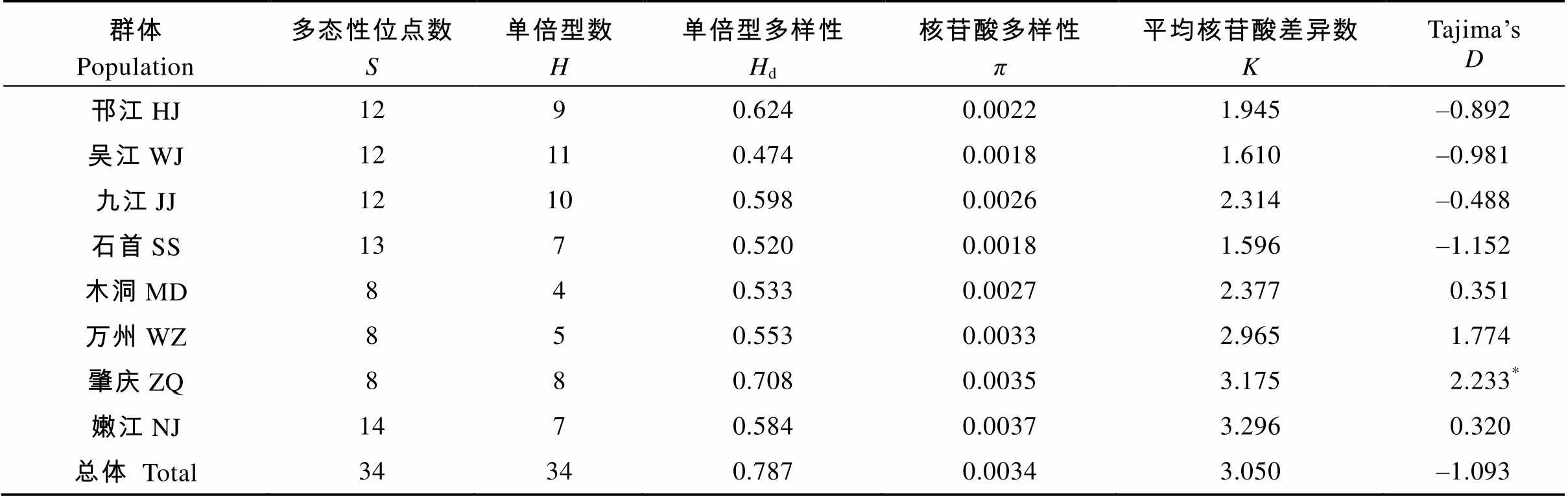
表3 草鱼群体D-Loop区序列遗传多样性参数
注:* 表示达到显著性水平(<0.05)
Note:* means of statistical significant (<0.05)

表4 草鱼群体间的K2P遗传距离(左下角)和遗传分化指数FST(右上角)
注: * 表示遗传分化显著(<0.05); **表示遗传分化极显著(<0.01)
Note: * and ** means of significant and extreme significant of genetic differentiation, respectively
我国草鱼野生群体间K2P遗传距离介于0.0020— 0.0049, 如表4(左下角), 其中邗江与吴江群体间遗传距离最近, 嫩江与邗江群体的遗传距离最远。遗传距离与地理距离具极显著正相关(=0.61,<0.01, 图1)。基于群体间K2P遗传距离构建NJ树显示, 8个群体聚为2支(图2)。其中一支由长江下游的邗江群体(HJ)、吴江群体(WJ)和九江群体(JJ)首先聚类, 再与肇庆群体(ZQ)聚类; 另一支由石首群体(SS)先与长江上游的木洞群体(MD)和万州群体(WZ)聚类, 然后与嫩江群体(NJ)聚类; 最后, 以上2个分支聚到一起。
2.3 草鱼野生群体间遗传分化
我国草鱼野生群体间的遗传分化指数如表4(右上角)所示。其中长江下游3个群体(HJ、WJ和JJ)之间及其他4对群体间(JJ与ZQ、SS与MD、MD与WZ、及WZ与NJ)遗传分化不显著(>0.05); 其余群体间遗传分化均达显著水平(<0.05)或极显著水平(<0.05)。
基于遗传分化分析的结果, 将8个群体分成5组, 长江水系分成上游(MD和WZ)、中游(SS)和下游(HJ、WJ和JJ)流域, 及珠江流域(ZQ)和黑龙江流域(NJ), 进行分子方差分析(AMOVA), 结果如表5所示, 不同流域间的方差组分占26.24%, 遗传分化指数ST为0.262, 达极显著水平(<0.01), 说明不同水系间及长江水系不同江段间存在显著遗传分化。

表5 草鱼群体D-Loop序列变异的分子方差分析(AMOVA)
注: **表示差异极显著(<0.01)
Note: ** means of statistical extreme significant
2.4 草鱼单倍型的发生关系
基于草鱼单倍型构建MP进化树(图3), 34个单倍型被分为2个主分支(LGA和LGB)。其中LGA包含26个单倍型, 有308尾鱼, 为优势分支; LGB包含8个单倍型, 有116尾鱼。分支间平均K2P遗传距离为0.0087, 分化水平极显著(ST=0.644,<0.01)。
利用单倍型构建的MJ网络图与MP进化树相吻合(图4)。2个分支各有2个优势单倍型, 并具有一定的地域性; 其中2个为长江中下游4个群体和肇庆群体所有, 另外2个为长江中上游3个群体和嫩江群体所有。估算4个优势单倍型间的分化时钟, 不同分支的优势单倍型间分化时间介于距今51077—28924年前, 分支内优势单倍型间分化时间介于距今11414—9464年前, 依此推测2个单倍型分支的分化大约发生在第四纪更新世晚期。
3 讨论
3.1 草鱼D-Loop区的变异特征
本研究对草鱼D-Loop区序列分析表明, A+T含量达到66.71%, 明显高于G+C含量, 这与Zhao等[11]在草鱼中研究发现66.8%的A+T含量相符合。研究发现草鱼D-Loop区34个变异位点, 34个单倍型, 高于Zhao等[11]利用4个mtDNA序列的研究结果, 而与宋晓等[12]的研究结果相似。分析造成本研究中草鱼D-Loop区多态性较高的原因, 与所用群体数及样本数较多有关。对比Zhao等[11]的研究, 其样本数尽管不多, 但其单倍型多样性水平与本文差异不大, 分析采用更多序列片段易于检测到较多突变位点和单倍型。因此, 在样本数充足的情况下, 可只用D-Loop区等序列开展遗传变异分析, 以降低实验成本; 而当样本量不太充足时, 建议增加mtDNA序列片段来开展群体遗传研究。
3.2 我国草鱼野生群体遗传多样性
在本研究中我国草鱼野生群体, 呈现较高的单倍型多样性, 说明我国草鱼野生资源具有较高遗传多样性。各群体的单倍型多样性水平与Zhao等[11]和李树华等[13]对长江水系的草鱼群体及宋晓等[12]对中国土著群体的研究结果相类似, 明显高于宋晓等[12]研究中日本群体(Tong)和美国群体(MSB)的单倍型多样性, 这与旅居群体的原始亲本较少有关。比较基于线粒体多态性的草鱼群体遗传研究, 发现结果不尽相同; 李思发等[9]和张四明等[10]利用基于片段长度多态性的RFLP技术检测到的单倍型数目明显少于Zhao等[11]和宋晓等[12]利用基于序列突变位点而确定的单倍型数, 可能与检测技术和研究群体等的差异有关。
本次研究表明吴江群体单倍型多样性最低, 推测可能与太湖流域悠久的圩田开发历史[29]有关, 原有水生资源的不断破坏, 导致遗传多样性降低, 可能也是廖小林等[30]认为草鱼群体存在“隐蔽瓶颈效应”现象[31]的原因。长江水系中上游群体的遗传多样性水平较低, 推测与其特殊的水文条件和水利设施有关[32]。长江流域部分野生群体呈现相对较低的单倍型多样性水平, 可能还受各江段原种场运营管理模式及常年增殖放流等因素影响; 尽管李树华等[13]在相关研究中未发现放流模式对野生群体的遗传多样性和遗传结构产生明显的影响, 但是近年来长江流域的增殖放流对草鱼野生种质资源的影响应该受到重视。在研究中肇庆群体的单倍型多样性最高, 与宋晓等[12]研究发现中国土著群体中珠江群体单倍型多样性最高相符, 与我们前期研究中发现肇庆群体等位基因数最多相吻合[19], 这可能与珠江流域温暖适宜的水域环境有关。长江下游2个群体(HJ和JJ)分布于长江干流, 拥有广阔的水域环境及适宜的水文条件, 可能导致其单倍型多样性略高于嫩江群体和长江上游群体, 与宋晓等[12]的部分研究结果相似。
3.3 我国草鱼系统演化和群体间遗传分化
本研究4个优势单倍型在石首群体中均有分布, 可为草鱼起源于第三纪后期的上新世的长江中游[1]提供分子证据, 而长江于早更新世晚期才从三峡东西贯通[33], 草鱼从而由中游扩散分布于整个长江水系。4个优势单倍型分为2支并具一定地域性分布, 推测是在地貌变化和种群演化两方面因素影响下形成。2个分支的分化时间推算发生于第四纪更新世晚期, 可能是冰期和间冰期交替导致地理隔离与融合造成[34]单倍型分化。2个分支内表现地域性差异, 其分化时间与中国主要水系形成时期(1.2—1.0百万年前)[33]相吻合, 这可能是物种在扩散中的选择和保留; 其中一个分支由长江中游经长江下游往珠江水系方向演化, 而另一个分支则由长江中游向长江上游和经过江河平原向古嫩辽河扩散演化, 这符合对珠江水系和黑龙江水系草鱼种群的起源推测[1]。
长江、珠江和黑龙江水系的草鱼在经历长期的演化过程后发生了显著遗传分化。在长江水系内部, 下游3个群体间遗传距离最近, 遗传分化不显著, 与李思发等[9]、张四明等[10]及Zhao等[11]的研究结果相符合; 中游的石首群体与下游和上游2个群体遗传距离接近, 大部分群体间存在显著遗传分化, 与Zhao等[11]研究发现长江水系不同江段无显著遗传分化不尽相同, 群体间遗传距离与地理距离呈正相关, 与我们基于微卫星标记的研究结果相符[19]。肇庆群体与长江下游3个群体间的遗传距离较近, 与除九江群体外的其他群体呈极显著的遗传分化。一方面支持珠江水系草鱼起源于长江中下游草鱼种群扩散的说法[1], 也可能与灵渠沟通湘江(长江支流, 有草鱼产卵场分布[9])和漓江(珠江支流)有关; 另一方面说明珠江水系草鱼种群在长期的适应过程中累积了特异的遗传资源。相对其他群体, 嫩江群体与长江上游2个群体的遗传距离最近, 在优势单倍型上也具有明显的共享现象; 根据相关文献[35, 36], 两者的地貌及河流坡降等方面具有相似之处, 而Zhang等[37]发现水文情势与草鱼的繁殖行为密切相关, 是否相近的环境导致群体间的遗传相似还有待进一步研究。
木洞、万州、肇庆及嫩江4个群体呈现相对较高的核苷酸多样性及平均核苷酸差异数, 这可能是在种群进化中平衡选择[38]的结果, 通过保留更多遗传多样性, 维持种群较高的适合度。以上4个群体的中性检验[39]显示可能经历不同程度遗传瓶颈效应, 与我们利用微卫星的研究结果相符[19], 推测在草鱼种群演化过程中, 地貌变化促进草鱼物种扩散并造成单倍型分化的同时, 气温变化、环境恶劣也抑制了草鱼种群过度繁衍。
[1] Shen Y B, Zhang J B, Li J L. Advances in studies on genetic resources of grass carp [J]., 2011, 27(7): 369—373 [沈玉帮, 张俊彬, 李家乐. 草鱼种质资源研究进展. 中国农学通报, 2011, 27(7): 369—373]
[2] FAO. The State of World Fisheries and Aquaculture 2012 [M]. Rome, 2012, 209
[3] Liu S P, Duan X B, Chen D Q,. Studies on status of fishery resources in the middle reach of the Yangtze River [J]., 2005, 29(6): 708—711 [刘绍平, 段辛斌, 陈大庆, 等. 长江中游渔业资源现状研究. 水生生物学报, 2005, 29(6): 708—711]
[4] Liu Z J, Cordes J F. DNA marker technologies and their applications in aquaculture genetics [J]., 2004, 238(1—4): 1—37
[5] Lu X, Wang H, Liu B,. An effective method for parentage determination of the clam () based on SSR and COI markers [J]., 2011, 318(1—2): 223—228
[6] Mishina T, Takada M, Takeshima H,. Molecular identification of species and ploidy offishes in Lake Biwa, using mtDNA and microsatellite multiplex PCRs [J]., 2014, 61(2): 169—175
[7] Mabuchi K, Miya M, Senou H,. Complete mitochondrial DNA sequence of the Lake Biwa wild strain of common carp (L.): further evidence for an ancient origin [J]., 2006, 257(1—4): 67—77
[8] Hu J, Hou X Y, Yin S W,. Genetic diversity and divergence ofof different geographic populations of the south China sea revealed byⅠ and Cytgene analyses [J]., 2014, 38(6): 1008—1016 [胡静, 侯新远, 尹绍武, 等. 基于mtDNAⅠ和Cyt基因序列对南中国海不同海域波纹唇鱼群体遗传多样性的研究. 水生生物学报, 2014, 38(6): 1008—1016]
[9] Li S F, Lü G Q, Bernatchez L. Diversity of mitochondrial DNA in the populations of silver carp, bighead carp, grass carp and black carp in the middle- and lower reaches of the Yangtze River [J]., 1998, 44(1): 82—93 [李思发, 吕国庆, 贝纳切兹 L. 长江中下游鲢鳙草青四大家鱼线粒体DNA多样性分析. 动物学报, 1998, 44(1): 82—93]
[10] Zhang S M, Wang D Q, Deng H,. Mitochondrial DNA variations of silver carp and grass carp in populations of the middle reaches of the Yangtze River revealed by using RFLP-PCR [J]., 2002, 26(2): 142—147 [张四明, 汪登强, 邓怀, 等. 长江中游水系鲢和草鱼群体mtDNA遗传变异的研究. 水生生物学报, 2002, 26(2): 142—147]
[11] Zhao J, Cao Y, Li S,. Population genetic structure and evolutionary history of grass carpin the Yangtze River, China [J]., 2011, 90(1): 85—93
[12] Song X, Li S F, Wang C H,. Grass carp () genetic structure analysis among native populations in China and introduced populations in USA, Europe and Japan based on mitochondrial sequence [J]., 2009, 33(4): 709—716 [宋晓, 李思发, 王成辉, 等. 草鱼中国土著群体与欧美日移居群体遗传差异的线粒体序列分析. 水生生物学报, 2009, 33(4): 709—716]
[13] Li S H, Chen D Q, Duan X B,. Genetic effects of released matureon natural populations based on the mitochondrial DNA markers in the middle reaches of the Yangtze River [J]., 2014, 44(3): 45—50 [李树华, 陈大庆, 段辛斌, 等. 基于线粒体DNA标记的长江中游草鱼亲本增殖放流的遗传效果评估. 淡水渔业, 2014, 44(3): 45—50]
[14] Xue G, Liu J, Liu J. RAPD analysis of grass carp population in three-river waters [J]., 1998, 5(1): 1—5 [薛国雄, 刘棘, 刘洁. 三江水系草鱼种群RAPD分析. 中国水产科学, 1998, 5(1): 1—5]
[15] Zhang Z, Cao Z, Zhou J,. Genetic structure analyses of different populations of grass carp () using the TRAP technique [J]., 2007, 4(1): 27—32
[16] Chen Q, Wang C, Lu G,. Analysis of genetic variation in grass carp () from native and colonized regions using ISSR markers [J]., 2009, 37(5): 549—555
[17] Liu F, Xia J, Bai Z,. High genetic diversity and substantial population differentiation in grass carp () revealed by microsatellite analysis [J]., 2009, 297(1—4): 51—56
[18] Chen Q, Wang C, Lu G,. Microsatellite genetic diversity and differentiation of native and introduced grass carp populations in three continents [J]., 2012, 140(4—6): 115—123
[19] Fu J J, Li J L, Shen Y B,. Genetic variation analysis of wild populations of grass carp () using microsatellite markers [J]., 2013, 35(2): 192—201 [傅建军, 李家乐, 沈玉帮, 等. 草鱼野生群体遗传变异的微卫星分析. 遗传, 2013, 35(2): 192—201]
[20] Wang C, Chen Q, Lu G,. Complete mitochondrial genome of the grass carp (Teleostei): insight into its phylogenic position within Cyprinidae [J]., 2008, 424(1—2): 96—101
[21] Hall T A. BioEdit: a user-friendly biological sequence alignment editor and analysis program for windows 95/98/ NT [J]., 1998, 41: 95—98
[22] Thompson J D, Gibson T J, Plewniak F,. The Clustal X windows interface: flexible strategies for multiple sequence alignment aided by quality analysis tools [J]., 1997, 25(24): 4876—4882
[23] Rozas J, Sá nchez-DelBarrio J C, Messenguer X,. DnaSP, DNA polymorphism analyses by the coalescent and other methods [J]., 2003, 19(18): 2496—2497
[24] Excoffier L, Lischer H E. Arlequin suite ver3.5: a new series of programs to perform population genetics analyses under Linux and Windows [J]., 2010, 10(3): 564—567
[25] Tamura K, Peterson D, Peterson N,. MEGA5: molecular evolutionary genetics analysis using maximum likelihood, evolutionary distance, and maximum parsimony methods [J]., 2011, 28(10): 2731—2739
[26] Karney C F F. Algorithms for geodesics [J]., 2013, 87(1): 43—55
[27] Cleophas T J, Zwinderman A H. SPSS for Starters [M]. Springer, Dordrecht New York Heidelberg London. 2009, 15—19
[28] Network 4.6.1.1. User Guide. www.fluxs-engineering.com
[29] Zhuang H F. Cultivation of diked paddy-fields in ancient south-east China and their effects on ecological environment [J]., 2005, 20(3): 87—94 [庄华峰. 古代江南地区圩田开发极其对生态环境的影响. 中国历史地理论丛, 2005, 20(3): 87—94]
[30] Liao X L, Yu X M, Tan D Q,. Microsatellite DNA analysis of genetic diversity of grass carp in Yangtze River system [J]., 2005, 29(2): 113—119 [廖小林, 俞小牧, 谭德清, 等. 长江水系草鱼遗传多样性的微卫星DNA分析. 水生生物学报, 2005, 29(2): 113—119]
[31] Luikart G, Sherwin W B, Steele B M,. Usefulness of molecular markers for detecting population bottlenecks via monitoring genetic change [J]., 1998, 7(8): 963—974
[32] Wang S Y, Liao W G, Chen D Q,. Analysis of eco-hydrological characteristics of the four Chinese farmed carps’ spawning grounds in the middle reach of the Yangtze River [J]., 2008, 17(6): 892—897 [王尚玉, 廖文根, 陈大庆, 等. 长江中游四大家鱼产卵场的生态水文特性分析. 长江流域资源与环境, 2008, 17(6): 892—897]
[33] Li C A, Zhang Y F. Geoscientific factors analyses on the through cutting of main drainages and the formation of flood damage in China [J]., 1997, 16(59): 61—65 [李长安, 张玉芬. 中国主要水系贯通和洪灾形成的地学因素分析. 大自然探索, 1997, 16(59): 61—65]
[34] Shi Y. Characteristics of late Quaternary monsoonal glaciation on the Tibetan Plateau and in East Asia [J]., 2002, (97—98): 79—91
[35] Xu D X, Zhang G X, Yin X R. Runoff variation and its impacting factor in Nenjiang River during 1956—2006 [J]., 2009, 20(3): 416—421 [徐东霞, 章光新, 尹雄锐. 近50年嫩江流域径流变化及影响因素分析. 水科学进展, 2009, 20(3): 416—421]
[36] Huang S. Upper Yangtze River annual runoff analysis and its forecast [D]. Thesis for Doctor of Science. Sichuan University, Chengdu. 2006 [黄胜. 长江上游干流区径流变化规律及预测研究. 博士学位论文, 四川大学, 成都. 2006]
[37] Zhang G, Chang J, Shu G. Applications of factor-criteria system reconstruction analysis in the reproduction research on grass carp, black carp, silver carp and bighead in the Yangtze River [J]., 2000, 29(3): 419—428
[38] Biswas S, Akey J M. Genomic insights into positive selection [J]., 2006, 22(8): 437—446
[39] Tajima F. Statistical method for testing the neutral mutation hypothesis by DNA polymorphism [J]., 1989, 123(3): 585—595
GENETIC VARIATION ANALYSIS BASED ON D-LOOP SEQUENCES OF WILD POPULATIONS OF GRASS CARP () IN CHINA
FU Jian-Jun1, 2, WANG Rong-Quan3, Shen Yu-Bang1, XUAN Yun-Feng3, XU Xiao-Yan1, LIU Cheng-Chu2and LI Jia-Le1
(1. Key Laboratory of Exploration and Utilization of Aquatic Genetic Resources, Ministry of Education, Shanghai Ocean University, Shanghai 201306, China; 2. Postdoctoral Research Station of Food Science and Technology, Shanghai Ocean University, Shanghai 201306, China; 3. Key Laboratory of Conventional Freshwater Fish Breeding and Health Culture Technology Germplasm Resources, Ministry of Agriculture, Suzhou Wujiang Area Aquaculture Limited Company, Suzhou 215221, China)
In this study we analyzed the genetic variations in the mtDNA D-Loop sequences of eight wild populations of grass carp () including six populations (Hanjiang, Wujiang, Jiujiang, Shishou, Mudong, and Wanzhou) from the Yangtze River, the Zhaoqing population from the Pearl River, and the Nenjiang population from Heilongjiang River. A total of 34 variable sites and 34 haplotypes were detected in 424 individuals, and the haplotype diversity ranged from 0.474 to 0.708 among the eight populations. The pairwise population K2P genetic distances were between 0.0020 and 0.0049. The genetic distance between the three populations from the lower reach of the Yangtze River was the closest and their genetic differences were insignificant (>0.05). The Zhaoqing population also showed a close genetic distance with the three populations from the lower reach of the Yangtze River; and it did not have significant genetic differences compared to the Jiujiang population (>0.05). The Nenjiang population displayed a close genetic distance with the two populations from the upper reach of the Yangtze River. The genetic difference between the Nenjiang population and the Wanzhou population (>0.05) was insignificant. Furthermore we observed an extreme significant correlation between the genetic distance and the geographic distance (<0.01). The AMOVA analysis suggested that there were highly significant differences between populations (<0.01), and that the genetic variation among different watersheds in China accounted for 26.24% of the total variation. The haplotypes divided into two major branches which exhibited remarkable evolutionary differences (ST=0.644,<0.01), and this branching might originate in the late Pleistocene.
; Wild population; D-Loop sequence; Genetic variation
10.7541/2015.46
Q346+.1
A
1000-3207(2015)02-0349-09
2014-04-21;
2014-09-11
现代农业产业技术体系建设项目(编号: CARS-46-04); 国家科技支撑计划项目-大宗淡水主养鱼类新品种选育(编号: 2012BAD26B02); 上海市重点学科建设项目(编号: Y1101)资助
傅建军(1986—), 男; 浙江金华人; 博士; 研究方向为水产动物种质资源与遗传育种。E-mail: jjfu@shou.edu.cn
李家乐, 教授, 博士生导师; 研究方向为水产动物种质资源与遗传育种。E-mail: jlli2009@126.com
猜你喜欢
杂志排行
水生生物学报的其它文章
- 达氏鲟生长激素基因cDNA克隆、表达及免疫荧光定位研究
- 杂交鲟对六种蛋白原料的表观消化率
- SUPPRESSION OF LIGASE4 OR XRCC6 ACTIVITIES ENHANCES THE DNA HOMOLOGOUS RECOMBINATION EFFICIENCY IN ZEBRAFISH PRIMORDIAL GERM CELLS
- 大兴安岭硅藻的中国新记录属、种
- 鲢快速逃逸游泳行为研究
- EFFECT OF FIBROUS ROOT OF COPTIS CHINENSIS FRANCH AND BERBERINE ON THE NON-SPECIFIC IMMUNITY AND RESISTANCE AGAINST AEROMONAS HYDROPHILAINFECTION IN GRASS CARP (CTENOPHARYNGODON IDELLA)
