DNA条形码技术在部分菱属植物分子鉴定中的应用
2015-11-28董晶莱高广春
董晶莱,高广春,黄 嬛,李 白,李 军*
(1.嘉兴学院 医学院,浙江 嘉兴 314001;2.嘉兴市农业科学研究院,浙江 嘉兴 314016)
DNA条形码技术在部分菱属植物分子鉴定中的应用
董晶莱1,高广春1,黄 嬛1,李 白2,李 军2*
(1.嘉兴学院 医学院,浙江 嘉兴 314001;2.嘉兴市农业科学研究院,浙江 嘉兴 314016)
为了弥补形态学鉴定方法的不足,本研究利用DNA条形码技术,选取标准基因序列对部分菱属植物进行初步的分子鉴定。通过对浙江、江苏2省南湖菱、两角菱、四角菱的ITS,matK和rbcL序列扩增及多重序列比对,探寻菱属植物的分子鉴定方法。克隆了菱属植物692 bp的ITS序列、878 bp的matK序列及685 bp的rbcL序列,其中matK和rbcL序列不存在变异位点,不能用于鉴定菱属植物;ITS序列存在20个变异位点,包括6处插入/缺失和14处碱基置换,在部分菱属植物的分子鉴定中具有应用价值。
菱属;分子鉴定;DNA条形码
菱科(Trapaceae)菱属(Trapa L.)植物为一年生水生草本植物,在中国南方尤其以长江下游太湖地区和珠江三角洲栽培最多。菱果肉含有丰富的蛋白质、不饱和脂肪酸及多种维生素和微量元素,菱壳具有抗食道癌、乳腺癌、子宫颈癌等药用价值。菱属植物包含多个栽培种和野生种,栽培种包括南湖菱(Trapa acornis Nakano)、四角菱(Trapa quadrispinosa Roxb)、二角菱 (Trapa bispinosa Roxb.)及乌菱(Trapa bicornis Osbeck)等;野生种包括野菱(Trapa incise var.Sieb.)、耳菱(Trapa potanini V.Vassil.)、冠菱(Trapa litwinowii V.Vassil.)、格菱(Trapa pseudoincisa Nakai.)及细果野菱(Trapa maχimowiczii Korsh)等[1-2]。菱属植物的分类学研究多集中在常规的形态分类学、数量分类学、细胞分类学以及花粉形态学等方面[3-8],不同分类方法对菱属植物的聚类分析存在一定差异。由于菱属植株的形态及果肉等营养形态基本相同,传统的形态分类学方法在菱属植物的鉴别上有局限性。近年来已有应用分子生物学方法研究菱属植物亲缘关系及分子鉴别的研究报道[1-2,9-12],大多是利用 DNA指纹标记对菱属植物进行分类及系统进化研究。鉴于菱在农业及医药领域的重要作用,及其种质混乱、鉴别方法不统一的现状,对单一品种菱建立一套可靠的分子鉴定方法势在必行。
DNA条形码技术是利用标准的、具有足够变异的、易扩增且相对较短的DNA片段,基于物种内的特异性和物种间的多样性而创建的一种新的生物身份识别系统,可实现对物种的快速自动鉴定[13],该技术已被成功应用于生物物种的分类和鉴定等领域[2,14-16]。常用的 DNA序列有 matK,trnH-psbA,rbcL和ITS等,通过单一片段或多片段组合的方式构建不同植物的 DNA条形码标准[17]。
matK基因位于 trnK基因的内含子中,约1 500 bp,编码一种成熟酶(matuease)。matK序列的变异较均一,变异中转换和颠换以及密码子3个位置的变异频率没有严重的偏离,增强了分子系统树的可靠性[15]。rbcL基因编码核酮糖-1,5-二磷酸羧化/加氧酶的大亚基,rbcL序列的变异主要存在于种以上水平,物种水平上通常变异不大,其变异位点较均匀地分布于整个基因上[17]。核糖体DNA ITS(18S-5.8S-26S)片段广泛分布于真核生物和真菌中,是系统学研究中最常用的片段之一,常用作种水平区分的片段[2]。因此,本研究以浙江省和江苏省的栽培种南湖菱、四角菱及二角菱为研究材料,选取matK,rbcL和ITS序列对菱属植物进行分子鉴定,为菱属植物的鉴定提供理论依据。
1 材料与方法
1.1 材料和试剂
试验所用材料包括浙江嘉兴的南湖菱 (T. acornis Nakano),果皮绿白色,试验编号为T1-1;浙江温州和江苏南京的二角菱(T.bispinosa Roxb),果皮暗紫色,试验编号为T2-1和 T2-2;浙江嘉兴和江苏南京的四角菱(T.quadrispinosa Roxb),果皮分别为水红色和绿色,试验编号分别为T3-1和T3-2。菱角种类的鉴定参照中国菱属植物分类方法[18-19]。
DNA聚合酶和dNTPs等购于宝生物工程 (大连)有限公司,引物和PCR产物测序委托英潍捷基 (上海)贸易有限公司完成。
1.2 总DNA提取和PCR扩增
总DNA提取方法:用清水洗净菱角果实,解剖刀快速取出菱胚,每个测试样品分别取5个菱胚进行混合,用 CTAB法提取总DNA[20],保存于-20℃备用。
用于PCR反应的ITS,matK和rbcL序列引物分别:ITS-F为5'-CGTAACAAGGTTTCCGTAGG-3',ITS-R为5'-TCCTCCGCTTATTGATATGC-3';matK-F为5'-CGATCTATTCATTCAATATTTC-3',matK-R为5'-TCTAGCACACGAAAGTCGAAGT-3';rbcL-F为5'-ATGTCACCACAAACAGAAAC-3',rbcL-R为5'-TCGCATGTACCTGCAGTAGC-3'。
克隆ITS序列的PCR反应体系为20μL,2× GC buffer I 10μL、dNTPs(2.5 mmol·m L-1)0.5μL、上下游引物 (10μmol·m L-1)各0.5μL、DNA模板1μL、Taq DNA聚合酶0.2μL、ddH2O 7.3μL。反应条件为94℃预变性3 min;然后94℃变性30 s,56℃退火30 s,72℃延伸1 m in,共36个循环;最后72℃延伸5 m in,4℃保存。matK和rbcL序列的PCR反应体系为20μL,10× PCR buffer 2μL、dNTPs(2.5 mmol·m L-1)0.5μL、上下游引物(10μmol·m L-1)各0.5μL、DNA模板1μL、Taq DNA聚合酶0.2μL、ddH2O 15.3μL。反应条件为94℃预变性3 min;然后94℃变性30 s,50℃退火30 s,72℃延伸1 min,共36个循环;最后72℃延伸5min,4℃保存。PCR产物进行琼脂糖凝胶电泳检测,然后割胶回收目的条带,送英潍捷基 (上海)贸易有限公司进行回收、纯化及测序。
1.3 序列分析及多重比对
分别将ITS、matK和rbcL序列在NCBI进行Blast分析,用DNAMAN软件进行多重序列比对,然后用MEGA 4.1软件构建系统发育树。
2 结果与分析
2.1 ITS序列扩增及比对
通过PCR扩增,5个测试样品均获得了692 bp的ITS序列 (图1),序列分析表明ITS序列包含262 bp的ITS1序列、166 bp的5.8S rDNA序列及264 bp的ITS2序列。序列比对表明,5个测试样品的ITS序列相似度达95%以上,(G+C)含量偏高。南湖菱ITS序列 (G+C)含量为58.96%,(A+T)含量为41.04%。

图1 菱属植物ITS序列引物PCR结果
通过GenBank数据库检索,获得9个菱属植物的ITS序列:江苏苏州四角菱 (AY315464)、江苏泰州四角菱 (AY315466)、江苏南通二角菱(AY315465)、安徽芜湖二角菱 (AY315468)、江苏南通乌菱 (AY315463)、安徽芜湖乌菱(AY315470)、江苏南京野菱 (AY315462)、湖北梁子湖细果野菱 (AY035757)及欧菱 (FM887019)的ITS序列。多重序列比对表明,ITS序列存在20个变异位点 (图2),其中6处插入/缺失和10处碱基置换位于ITS1序列,4处碱基置换位于ITS2序列,5.8S rDNA序列中不存在变异位点。6处插入/缺失分别位于第17,18,71,107,130和207 bp处;14处碱基置换分别为12 bp(A↔G),15 bp(A↔G),36 bp(A↔G),63 bp(C↔T),482 bp(T↔C),508 bp(A↔G),606 bp(A↔G)的转换和9 bp(T↔G)、10 bp(C↔G)、11 bp(A↔C)、67 bp(T↔G)、134 bp(C↔G)、201 bp(T↔G)、448 bp(T↔G)的颠换。本试验的5个测试样品仅在第36 bp及67 bp处有差异 (图2),浙江温州二角菱在第36 bp为 “G”,其余4种菱均为 “A”;浙江温州二角菱和浙江嘉兴四角红菱在第67 bp为 “G”,其余3种菱角为 “T”。
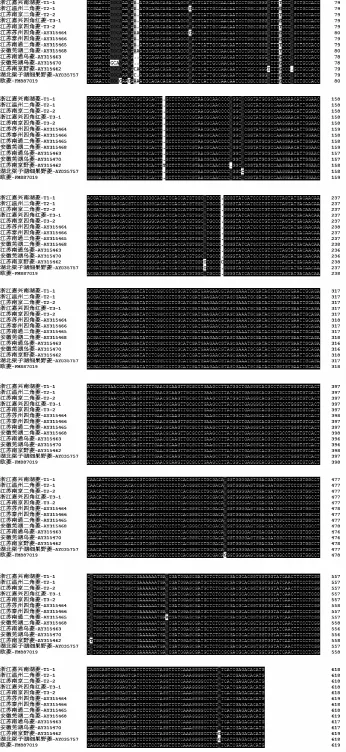
图2 不同种菱属植物的ITS序列比对
2.2 matK基因扩增及序列比对PCR扩增及测序结果表明,5个测试样品都获得了878 bp的matK序列 (图3),样品间序列相似度为100%。统计分析表明,菱属植物的matK序列 (G+C)含量偏低,为32.80%, (A+T)含量为67.20%。GeneBank中尚未有菱属植物的matK基因登录,通过Blast检索到海桑科、千屈菜科等科属植物的matK基因序列与本试验克隆的matK序列相似度达95%以上。用MEGA 4.1软件构建matK序列系统发育树,结果表明,在matK序列涉及到的植物中,菱属植物与海桑科植物亲缘关系最近,与千屈菜科植物次之,与桃金娘科植物亲缘关系最远 (图4)。

图3 菱属植物matK及rbcL序列PCR结果
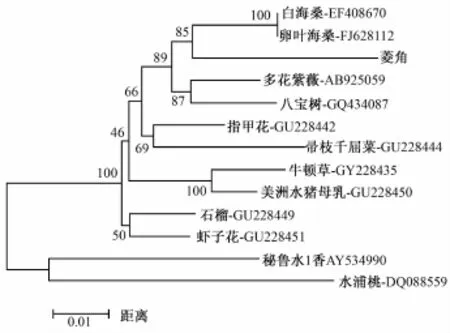
图4 菱属植物与其他物种的matK序列系统进化树
2.3 rbcL基因扩增及序列比对
5个测试样品都获得了685 bp的rbcL序列片段 (图3),样品间序列相似度为100%,(G+C)含量为41.17%,(A+T)含量为58.83%。将克隆的rbcL序列与GeneBank数据库中欧菱和细果野菱的序列进行序列比对发现 (图5),rbcL序列存在5个变异位点,包括1处插入/缺失和4处碱基置换,1处插入/缺失位于10 bp处,4处碱基置换包括第109 bp和110 bp处的2处碱基颠换,4 bp和111 bp处的2处碱基转换。

图5 菱角与欧菱和细果野菱的rbcL序列比对
3 小结和讨论
自加拿大动物学家Paul Hebert[21]于2003年首次提出DNA条形码的概念以来,该研究已成为生物分类学研究的热点和前沿。该技术利用基因组中一段通用的标准短序列进行物种鉴定,现已提出10多条植物候选DNA条形码序列,其中以ITS,matK,rbcL及trnH-psbA的单一片段或几个片段的组合应用较多[22]。 现已在胡椒属[16]、 锦葵科[15]、忍冬科[23]、 石斛属[24]、 重楼属[14]等植物的系统进化及分类鉴定中得到广泛应用,同时也在药用植物藏药雪莲[25]、 女贞子[26]、 杜仲[27]等基原植物的分子鉴定中得到应用。
本研究以南湖菱、二角菱和四角菱为研究材料,克隆ITS、matK及rbcL序列,通过多重序列比对,找寻菱属植物序列变异位点。在测试的3个DNA条形码序列中,ITS序列变异位点较多,有20个,均位于ITS1和ITS2序列,包括6处插入/缺失和14处碱基置换,5.8S rDNA序列中不存在变异位点,这与保曙琳等[2]的研究报道一致。由于rDNA比cpDNA具有更快的进化速率,而且不存在cpDNA的母系单向遗传问题,所以rDNA ITS序列是近年来用于探讨植物种内变异和种间、近缘属间分子系统关系的重要分子标记之一,已被广泛应用于胡椒属、锦葵科、石斛属及重楼属等科属植物的分子鉴定研究中[2]。据保曙琳等[2]的研究报道,南湖菱与二角菱在ITS序列中的鉴别位点在508号碱基上,南湖菱为 “G”,二角菱为 “A”。本研究结果表明,在第508号碱基上,除来源于江苏南通的二角菱 (GenBank登录号AY315465)为“A”外,包括南湖菱在内的其余菱均为 “G”,因此,ITS序列第508号碱基是否能作为鉴别南湖菱与二角菱的位点还有待进一步扩大采样地点及样本数量来分析。测试样品中matK和rbcL序列不存在变异位点,不能用来鉴定菱属植物。
本试验的5个研究材料涉及南湖菱、四角菱及二角菱3个种,均为栽培种,各种之间的差异为种内差异。ITS序列变异位点分析表明,3个种之间具有较高的遗传稳定性,ITS序列在菱属植物种内分子鉴定及系统进化分析中具有重要的应用价值。
[1] 姜维梅,丁炳扬.国产菱属植物亲缘关系的RAPD分析[J].浙江大学学报:农业与生命科学版,2004,30(2):191-196.
[2] 保曙琳,丁小余,常俊,等.长江中下游地区菱属植物的DNA分子鉴别 [J].中草药,2004,35(8):926-930.
[3] Kadono Y.A preliminary study on the variation of Trapa in Japan[J].Acta Phytoax Geobot,1987,38:199-210.
[4] 于丹.中国东北菱属植物的研究 [J].植物研究,1994,14(1):40-47.
[5] 胡仁勇,丁炳扬,黄涛,等.国产菱属植物数量分类学研究 [J].浙江大学学报:农业与生命科学版,2001,27(4):419-423.
[6] 陈家宽,周进.湖北斧头湖浮叶水生植物群落学研究:I.菱群落的结构 [J].水生生物学报,1995,19 (1):40-48.
[7] 丁炳扬,黄涛,姜维梅,等.菱属植物的幼苗形态及其系统学意义 [J].浙江大学学报:自然科学版,1999,26(3):92-98.
[8] Shalabh B,Akash J,Jasm ine C.Trapa natans(Water Chestnut):an overview[J].International Research Journal of Pharmacy,2012,3(6):31-33.
[9] Hoque A,Anai T,Arima S.Analysis of molecular diversity in water chestnut based on RAPD markers[J].Biotechnology,2005,4(2):144-148.
[10] Huang Y L,Shi SH.Phylogenetics of Lythraceae sensu lato:a preliminary analysis based on chloroplast rbcL gene,psaA-ycf3 spacer,and nuclear rDNA in ternal transcribed spacer(ITS)sequences[J].International Journal of Plant Sciences,2002,163(2):215-225.
[11] Graham S A,Hall J,Sytsma K,et al.Phylogenetic analysis of the Lythraceae based on four gene regions and morphology[J]. International Jou rnal of Plant Sciences,2005,166(6):995-1017.
[12] Kim C,Na H R,Choi H K.Molecular genotyping of Trapa bispinosa and T.japonica(Trapaceae)based on nuclear AP2and chloroplast DNA trnL-F region[J].American Journal of Botany,2010,97(12):e149-e152.
[13] 任保青,陈之瑞.植物DNA条形码技术 [J].植物学报,2010,45(1):1-12.
[14] 朱英杰,陈士林,姚辉,等.重楼属药用植物DNA条形码鉴定研究 [J].药学学报,2010,45(3):376-382.
[15] 王柯,陈科力,刘震,等.锦葵科植物DNA条形码通用序列的筛选 [J].植物学报,2011,46(3):276-284.
[16] 郝朝运,邬华松,范睿,等.胡椒属植物 DNA条形码初步研究 [J].热带作物学报,2013,34(5):870-874.
[17] 高连明,刘杰,蔡杰,等.关于植物 DNA条形码研究技术规范 [J].植物分类与资源学报,2013,34(6):592-606.
[18] 颜素珠.中国水生高等植物图说 [M].北京:科学出版社,1983,121-130.
[19] 万文豪.中国菱属植物分类研究 [J].江西大学学报:自然科学版,1984(2):71-78.
[20] Doyle J J.A rap id DNA isolation procedure for small quantities of fresh leaf tissue[J].Phytochem ical Bulletin,1987,19:11-15.
[21] Hebert P D N,Ratnasingham S,de Waard J R.Barcoding animal life:cytochrome c oxidase subunit1 divergences among closely related species[J].Proceedings of the Royal Society of London B:Biological Sciences,2003,270(Suppl 1):S96-S99.
[22] 宁淑萍,颜海飞,郝刚,等.植物DNA条形码研究进展[J].生物多样性,2008,16(5):417-425.
[23] 刘震,陈科力,罗焜,等.忍冬科药用植物 DNA条形码通用序列的筛选 [J].中国中药杂志,2010,35(19):2527-2532.
[24] 黄海,李劲松,符岸军,等.石斛属植物DNA条形码序列的筛选 [J].热带作物学报,2010,31(10):1769-1777.
[25] 刘建全,陈之瑞,路安民.藏药雪莲原植物水母雪莲及其混淆种类的 ITS序列比较和分子鉴定 [J].中草药,2001,32(5):443-445.
[26] 李美妮,韩蕊莲,韩建萍,等.基于ITS2序列的女贞子原植物及其混伪品的分子鉴定 [J].世界科学技术:中医药现代化,2011,13(4):644-649.
[27] 章群.中药杜仲原植物的分子鉴定 [J].生态科学,2004,23(2):141-143.
(责任编辑:侯春晓)
S567.21+9
A
0528-9017(2015)04-0530-04
10.16178/j.issn.0528-9017.20150427
2014-11-24
嘉兴市科技计划项目 (2013AY21047);十二五浙江省高校重点学科 (药理学);2011年嘉兴市重点科技创新团队——天然药物与健康食品研发技术;国家级大学生创新创业训练计划项目 (201410354021)
董晶莱(1992-),女,本科生。E-mail:gaogcjx@163.com。
李 军。E-mail:lijun jx1@163.com。
文献著录格式:董晶莱,高广春,黄嬛,等.DNA条形码技术在部分菱属植物分子鉴定中的应用 [J].浙江农业科学,2015,56(4):530-533,557.
