碳同位素标记法鉴别芳烃降解菌研究
2015-11-22胡恒宇赵东风刘春爽
胡恒宇,赵东风,刘春爽
(中国石油大学化学工程学院,山东青岛266580)
碳同位素标记法鉴别芳烃降解菌研究
胡恒宇,赵东风,刘春爽
(中国石油大学化学工程学院,山东青岛266580)
从油田采出水中富集培养石油烃降解菌,利用13C标记的甲苯作为碳源和变性梯度凝胶电泳技术,从中鉴定和考察能够降解甲苯的细菌群落。结果表明:从采出水中富集培养的复合菌群经过500 d的厌氧降解,对石油烃降解率达到36.4%,甲烷产量为201.2 μmol;以13C标记的甲苯为碳源,经过60 d的培养,复合菌群对其降解率达到67%,12C标记的甲苯降解率为69%,两种碳源标记甲苯的降解率差异不显著;将离心中的重带13C-DNA片段回收测序,鉴别出3种芳烃降解菌。
碳同位素;菌株鉴定;CsCl密度梯度;DGGE分析;芳烃
油藏环境中的有机物质(特别是残余原油)降解过程中会产生大量副产物和气体,参与石油降解的菌群形成互营共生关系,最终使有机质一步步地降解[1]。油藏环境中的微生物群体是由微好氧菌和兼性厌氧型细菌组成的,油藏中的脂肪烃提供了它们存活的基础。油藏中的菌群有发酵菌、硫酸盐/硝酸盐还原菌、铁还原菌、厌氧产甲烷菌等[2-4],分属于不同的菌属,在自然状态下可以降解和利用石油烃,但是单纯利用内源微生物,在自然状态下的石油烃降解效率低[5]。现有的细菌群落分析技术,如通过聚合酶链式反应(PCR)扩增和变性梯度凝胶电泳(DGGE)技术,虽然可以很好地分析石油烃降解微生物的群落结构,但不能准确分离降解芳烃的菌[6]。用稳定性同位素探针(SIP)和DGGE技术结合可以准确分析和鉴定石油烃降解菌群的结构[7-8]。笔者利用13C标记的和未标记的芳烃分别作为碳源,使菌群利用碳源合成自身的核酸(DNA),带有13C的DNA片段会在氯化铯(CsCl)密度梯度中和其他DNA片段分离,从而鉴别分离出高效芳烃类降解菌。
1 试验设计与方法
1.1 药品与仪器
13C标记的甲苯标准品(国家标准物质信息中心购买,美国)、12C标记的甲苯标准品(色谱纯)、氯化铯CsCl、己烷(分析纯)、二硫化碳、厌氧工作站(Plas Labs,Lansing,MI,USA)、OIL-510型红外分光测油仪、贝克曼库尔特高速离心机Optima L-XP(美国)、日本爱宕手持折射仪R-5000、吉尔森Minipuls 3蠕动泵。
1.2 石油烃降解菌群的初级培养
菌溶液为复合石油烃降解菌群,经过实验室长期培养和保存。具体培养方法为:采集来自油田的采出水,从采油井泵取保存于塑料桶(约10 L)中,立即送到实验室并富集培养。取样地油井的油层温度为50~65℃,油层压力为9~10 MPa,原油密度(0.929±0.04)g/cm3,原油黏度(168.4 ±3.0)mPa·s,总矿化度9.563 g/L,饱和分(33.9±0.7)%,芳香分(32.7±0.7)%,胶质(28.6±0.6)%,沥青质(4.9±0.1)%。驱油类型为聚丙烯酰胺驱。
在厌氧操作箱中,首先进行初级培养,用红外测油仪测定采出水中石油烃含量为0.05 g/mL。后取50 mL的采出水和5 g原油、50 mL灭菌无机盐培养基充入到150 mL无菌瓶中,进行50℃的富集培养,以灭菌作为对照,多组培养。经过实验室500 d的培养,筛选出高效降解石油烃的复合菌群,表1是其石油降解率、CH4产量随着天数的变化情况,每次测定重复5次。

表1 复合石油烃降解菌的降解参数Table 1 Characteristics of complex hydrocarbon degradation bacteria
对降解前后的石油烃萃取后进行四组分分离,再通过气相色谱质谱连用得知其降解前后的石油中饱和烃变化情况,结果见图1和图2,可以确定这是高效石油烃降解菌群,可以在后续试验时,从中鉴别出哪些是芳烃类降解菌。

图1 降解前饱和烃的气相色谱-质谱总离子图Fig.1 Saturated hydrocarbon GC-MS TIC map before degradation
所用无机盐培养基:KH2PO45.0 g;K2HPO45.0 g;NH4Cl 5.0 g;NaCl 1.0 g;MgCl22.0 g;CaCl20.1 g;酵母粉1.0 g;L-半胱氨酸盐酸盐0.5 g;定容到1 L;pH值7.0~7.2。

图2 降解后饱和烃的气相色谱-质谱总离子图Fig.2 Saturated hydrocarbon GC-MS TIC map after degradation
1.3 芳烃降解培养
通过《化学化工物性数据手册》查得,在本实验的50℃下,1 L水中最多可以溶解0.588 g甲苯,所以进行本实验时应该控制甲苯的使用质量浓度为小于0.5 g/L。在厌氧操作箱中,先取上述经过初培养的复合菌液,取清液并且过滤,滤掉油和杂质,利用气相色谱测定培养液中和培养瓶上层空气中的甲苯的初始含量,由于清液过滤也可能含有极少量的石油烃,在后期培养时,测定的甲苯降解率只是相对值。再分别取0.5 g12C和13C标记的甲苯标准品,分别加入到1 L培养基中,再分装为50 mL同时转入到150 mL无菌培养瓶中,再加入50 mL的复合菌液,以加菌液灭菌和不加菌液为对照,分别做多组重复,每次测定取样重复5次,培养60 d。所用选择无机盐培养基:KH2PO45.0 g;K2HPO45.0 g;NH4Cl 5.0 g;NaCl 1.0 g;MgCl22.0 g;CaCl20.1 g;L-半胱氨酸盐酸盐0.5 g;硫酸亚铁铵0.5 g;定容到1 L;pH值7.0~7.2。
1.4 SIP和DGGE分析方法
取12C和13C标记的甲苯处理培养液离心收集菌体(12 000 r/min,15 min),除去杂质,用DNA提取试剂盒提取菌液DNA(Tiangen Biotech公司)[9]。再用等密度梯度离心法使DNA在氯化铯溶液中高速离心,达到分离的目的[10]。离心时氯化铯的初始密度为1.692 g/mL,并用手持折射仪矫正[11]。上述提取的DNA 2 mg和1.5 mL溴化乙啶(4 mg/mL,对DNA进行染色以便观察)、200 mL CsCl TE缓冲溶液充分混合。然后再取0.2 mL混合液和4.8 mL的氯化铯TE缓冲溶液在5.1 mL的快封离心管中准备离心。离心参数为48 000 r/min,离心时间25 h,温度4℃。离心结束后在离心管底部用针刺穿,然后以一定的流速通过蠕动泵在离心管顶部加入水,在顶部形成恒定的压力,这样底部的氯化铯溶液以恒定的速率流出,收集不同密度的氯化铯溶液,分装。再用聚乙二醇除去氯化铯,回收DNA并用70%的乙醇冲洗,保存在4℃条件下备用[11]。上述离心得到的DNA经16SrDNA基因的扩增,采用引物341F和534R,使用的PCR仪器为9700PCR[12],所用程序为94℃5 min,55℃60 s,72℃1 min,一共进行35个循环,温度为4℃。将扩增得到的16SrDNA进行DGGE分离,仪器为DCode System,所用变性剂质量分数为30%~60%,在电压20 V下电泳30 min,然后150 V下电泳8 h,温度为60℃。然后切胶回收测序[13],得到序列后在数据库中比对,以确定相似菌种[14]。
1.5 石油烃的测定方法
采用红外分光光度测油仪时,参照GB/T16488 -1996,首先将培养液转移到分液漏斗中,然后酸化到pH≤2,用CCl4洗涤3次,加入NaCl振荡,静置分层后,取下层萃取液加入含有Na2SO4的玻璃砂心漏斗中,并用真空泵抽滤;与此同时,对上层溶液在重复刚才的操作。然后将漏斗中的滤液转移到容量瓶中,CCl4定容。稀释一定倍数以后,用红外分光测油仪确定石油烃含量[15-16]。
1.6 甲苯的测定方法
每次取样时对培养瓶中的溶液进行萃取,培养液加入CS2,然后常温下进行水浴振荡2 h,4℃保存过夜。用梨形漏斗收集下层有机相于三角瓶中,对上层水相继续加入CS2振荡10 min,重复3次,将三角瓶中的有机相溶液用无水硫酸钠过滤并且进行减压蒸发,用CS2定容。而后进行甲苯回收率的检测,配成甲苯的0.5 g/L的溶液,振荡溶解,然后稀释若干浓度梯度,50℃过夜,按照上述萃取方法进行萃取,最后通过色谱检测甲苯,计算回收率。用岛津GC-2010气相色谱仪测定甲苯含量。色谱柱为弹性石英毛细柱(50 m×0.2 mm×0.5 μm),检测器FID温度180℃、进样口温度150℃。先用甲苯标准溶液逐级稀释,用色谱检测,然后计算甲苯含量[17]。
1.7 气相色谱和质谱连用的测定方法
GC-MS分析。采用Agilent 6890N气相色谱/ 5975i质谱联用仪。弹性石英毛细柱(60 m×0.25 mm ×0.25 μm)。进样口温度300℃,程序升温初温50℃进行1 min,以20℃/min的速度提升温度到120℃,再以4℃/min的速度升温到250℃,最后以3℃/min的速度升温到310℃,维持30 min。质谱EI源,70 eV,照射灯丝电流100 μA,进行全扫描[17-18]。
1.8 数据分析
采用Origin8.0画图软件,数据分析采用SPSS统计软件,采用标准差(SD)和最低显著性检验(LSD)比较和分析各组数据之间的差异。
2 结果分析
2.1 细菌降解石油烃的分析
通过油藏中的采出水富集培养石油烃降解菌,从表1可以看出,在培养初期50~200 d,石油烃降解率很低,到200 d时,达到4.1%。从250 d开始,石油烃降解率逐渐升高,并在各个测量时间点的时候差异显著,最终在500 d时达到36.4%。直到400 d时开始有甲烷产生,为130.7 μmol;到500 d时,甲烷产量为201.2 μmol。
有研究[19]表明,甲烷是石油烃降解的终端产物。近年来有学者从油藏采出水中分离得到降解石油的菌群[20]。这些菌群中包含有专门降解芳香烃的菌群[21-22],可以为降解石油提供丰富的菌源。从图1和图2(0~90 min)可以看出,饱和烃分布完整,且图2中从左到右大部分的饱和烃都被降解,表明这种降解菌的降解效果很理想。
2.2 细菌降解甲苯的分析
图3为12C和13C标记甲苯的降解情况。可以看出甲苯的降解率随着培养天数的增加而显著增加。其中13C标记的甲苯处理的甲苯降解率在60 d达到67%,而12C标记甲苯处理的甲苯降解率,在60 d达到69%,两种标记处理的甲苯降解率差异不显著。在培养瓶顶部空气中也未检出甲苯,说明甲苯没有挥发,完好地溶解于培养液中。对照处理的甲苯降解率为0,说明甲苯的降解与培养基溶液无关。
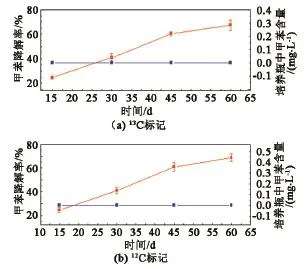
图3 12C和13C标记甲苯的降解Fig.3 Degradation situation of12C-toluene and13C-toluene
2.3 降解12C和13C标记甲苯的细菌群落分析
由于不同轻重同位素组成的化合物具有相同的物理化学性质,所以细菌可以代谢它们来合成自身的物质(如DNA核酸)。用13C标记的石油烃经过微生物菌群的降解合成了自身的13C-DNA,通过在氯化铯溶液中高速离心,就可以把13C-DNA和12C-DNA分开[10]。重同位素取代轻同位素将导致其相对分子质量增加,所以在离心的时候就呈现出分离,12C-DNA在上层,密度小,13C-DNA在下层,密度大[11]。本实验经过60 d的降解后提取菌液DNA,在离心后收集到1.64~1.73 g/mL DNA的氯化铯溶液,然后进行DGGE分析,结果见图4和图5。由图4看出,细菌降解12C标记的甲苯时,在低密度中含有丰富的细菌群落,回收条带多,而在高密度中没有细菌群落,这与用普通的12C标记甲苯进行培养相符。这是因为经过离心,13CDNA会在高密度中,12C-DNA会在低密度中。细菌降解13C标记的甲苯时,在高密度溶液中回收了大量条带,而在低密度溶液中回收的条带偏少。这是因为细菌降解了13C标记的甲苯,形成13C DNA在高密度溶液。所以在图5中,从高密度梯度中切胶(如红色箭头标明)回收13C-DNA,然后进行测序用于后期分析鉴定降解甲苯的细菌。有研究表明,通过碳源标记的方法,可以鉴定出降解特定化合物的细菌群落[11]。

图4 细菌降解12C和13C甲苯处理的DGGE电泳回收条带数Fig.4 Bacterial degradation DGGE recovery bands of12C-toluene and13C-toluene treatments

图5 细菌降解13C标记的甲苯处理的DGGE分析Fig.5 Bacterial degradation DGGE analysis of13C-toluene treatment
2.4 芳烃降解菌的鉴定分析
将高密度的13C DNA条带回收送测序公司克隆测序,测出3株(a、b、c)该芳烃降解菌的序列,见表2。通过NCBI数据库进行比对,从中得知相似度92%是1株降解氯苯的细菌和1株柴油降解菌,相似度90%的是1株高效石油烃降解菌。从中得知,本实验室培养的是高效石油烃降解菌,其中13C标记甲苯培养时,从重密度溶液中分离得到芳烃降解菌条带,从而鉴别出了此芳烃降解菌的相似序列,可以在以后应用此菌群进行石油降解和生物修复。

表2 芳烃降解菌的分析和鉴定Table 2 Analysis and identification of aromatic hydrocarbon degrading bacteria
3 结 论
(1)从油田采出水中富集培养复合的石油烃降解菌群,在500 d对石油烃降解率达到36.4%,并且在400 d开始产气为130.7 μmol。
(2)以13C标记的甲苯为碳源和以12C标记的甲苯培养菌群,经过60 d的培养,测得甲苯降解率分别为67%和69%。
(3)通过回收高密度的13C-DNA,进行测序发现有3株相似序列,其中有2种石油烃降解菌,还有1种为氯苯降解菌。
[1] GIEG L M,DUNCAN K E,SUFLITA J M.Bioenergy production via microbial conversion of residual oil to natural gas[J].Appl Environ Microbiol,2008,74(10):3022-3029.
[2] PHAM V D,HNATOW L L,ZHANG S,et al.Characterizing microbial diversity in production water from an A-laskan mesothermic petroleum reservoir with two independent molecular methods[J].Environ Microbiol,2009,11(1):176-187.
[3] GRAY N D,SHERRY A,LARTER S R,et al.Biogenic methane production in formation waters from a large gas field in the North Sea[J].Extremophiles,2009,13(3):511-519.
[4] DUNCAN K E,GIEG L M,PARISI V A,et al.Biocorrosive thermophilic microbial communities in Alaskan worth slope oil facilities[J].Environ Sci Technol,2009,43(20):7977-7984.
[5] JONES D M,HEAD I A,GRAY N D,et al.Crude oil biodegradation via methanogenesis in subsurface petroleum reservoirs[J].Nature,2008,451(7175):176-180.
[6] BAMFORTH S M,SINGLETON I.Bioremediation of polycyclic aromatic hydrocarbons:current knowledge and future directions[J].Journal Chemical Technology Biotechnology,2005,80(7):723-736.
[7] CHEN Y,MURRELL J C.When metagenomics meets stableisotope probing:progress and perspectives[J]. Trends in Microbiology,2010,18(4):157-163.
[8] XIA W,ZHANG C,ZENG X,et al.Autotrophic growth of nitrifying community in an agricultural soil[J].The ISME Journal,2011,5(7):1226-1236.
[9] GU G Zh,LI Z,ZHAO D F,et al.Isolation and characterization of a thermophilic oil-degrading bacterial consortium[J].China Petroleum Processing and Petrochemical Technology,2013,15(2):82-90.
[10] FREITAG T E,CHANG L,PROSSER J I.Changes in the community structure and activity of betaproteobacterial ammonia-oxidizing sediment bacteria along a freshwater marine gradient[J].Environ Microbiol,2006,8(4):684-696.
[11] ZHANG L M,OFFREA P R,HE J Zh,et al.Autotrophic ammonia oxidation by soil thaumarchaea[J]. PNAS,2010,107(40):17240-17245.
[12] KE S Z,ZHANG M,SHI Z,et al.Phenol degradation and microbial characteristics in up flow anaerobic sludge blanket reactors at ambient and mesophilic temperatures[J].International Journal of Environment and Pollution,2008,32(1):68-77.
[13] WATANABE K,KODAMA Y,HARAYAMA S.Design and evaluation of PCR primers to amplify bacterial 16S bosomal DNA fragments used for community fingerprinting[J].Journal of Microbiological Methods,2001,44(3):253-262.
[14] 李政,赵朝成,张云波,等.16种EPA-PAHs复合污染土壤的菌群修复[J].中国石油大学学报(自然科学版),2012,36(1):175-181. LI Zheng,ZHAO Chaocheng,ZHANG Yunbo,et al. Bioremediation of 16 EPA-PAHs combined contaminated-soil with microbial consortium[J].Journal of China University of Petroleum(Edition of Natural Science),2012,36(1):175-181.
[15] MISHRA S,JYOT J,KUHAD R C,et al.Evaluation of inoculum addition to stimulate in situ bioremediation of oily-sludge-contaminated soil[J].Applied Environmental Microbiology,2001,67(4):1675-1681.
[16] 刘春爽,赵东风,吴文华,等.芦苇修复新疆石油污染土壤效果[J].中国石油大学学报(自然科学版),2012,36(2):186-190. LIU Chunshuang,ZHAO Dongfeng,WU Wenhua,et al.Phytoremediation of petroleum contaminated soil by reed in Xinjiang[J].Journal of China University of Petroleum(Edition of Natural Science),2012,36(2):186-190.
[17] 吴伟林.石油烃厌氧降解菌的筛选及其降解特性研究[D].青岛:中国石油大学,2011. WU Weilin.Study on the screening and degrading characteristics of anaerobic hydrocarbon degrading bacteria[D].Qingdao:China University of Petroleum,2011.
[18] MCFARLIN K M,PRINCE R C,PERKINS R,et al. Biodegradation of dispersed oil in arctic seawater at-1℃[J].Plos One,2014,9:e84297.
[19] GROSSI V,CRAVO L C,GUYONEAUD R,et al.Metabolism of n-alkanes and n-alkenes by anaerobic bacteria:a summary[J].Organic Geochemistry,2008,39(8):1197-1203.
[20] AITKEN C M,JONES D M,LARTER S R.Anaerobic hydrocarbon biodegradation in deep subsurface oil reservoirs[J].Nature,2004,431(7006):291-294.
[21] SAYARA T,POGNANI M,SARR M,et al.Anaerobic degradation of PAHs in soil:impacts of concentration and amendment stability on the PAHs degradation and biogas production[J].International Biodeterioration and Biodegradation,2010,64(4):286-292.
[22] DOU J,LIU X,HU Z,et al.Anaerobic BTEX biodegradation linked to nitrate and sulfate reduction[J]. Journal of Hazardous Materials,2008,151(2/3):720-729.
(编辑 刘为清)
Identification of arene degradation bacteria using carbon isotope labeling method
HU Hengyu,ZHAO Dongfeng,LIU Chunshuang
(College of Chemical Engineering in China University of Petroleum,Qingdao 266580,China)
The degradation bacteria of petroleum hydrocarbon were enriched and cultured from the oilfield water.Using13C-toluene as carbon source and denaturing gradient gel electrophoresis(DGGE)technology to identify and investigate the bacterial community which could degrade the toluene.The results show that after 500 days anaerobic degradation the petroleum hydrocarbon degradation rate is 36.4%by complex bacterium,and the methane yield is 201.2 μmol.It is also found that after 60 days culture the carbon source13C-toluene degradation rate is 67%by the complex bacterium.Meanwhile,after 60 days culture the12C-toluene degradation rate is 69%,and two kinds of toluene degradation rate are not significantly different.13C-DNA is recovered and identified for three types of aromatic hydrocarbon degrading bacteria.
carbon isotope;bacteria identification;CsCl density gradient;DGGE analysis;arene
T 992.4
A
胡恒宇,赵东风,刘春爽.碳同位素标记法鉴别芳烃降解菌研究[J].中国石油大学学报(自然科学版),2015,39(6):57-62.
HU Hengyu,ZHAO Dongfeng,LIU Chunshuang.Identification of arene degradation bacteria using carbon isotope labeling method[J].Journal of China University of Petroleum(Edition of Natural Science),2015,39(6):57-62.
1673-5005(2015)06-0057-06
10.3969/j.issn.1673-5005.2015.06.007
2015-05-09
中国石油天然气集团公司科学研究与技术开发项目(2008D-4704-2)
胡恒宇(1983-),男,博士研究生,研究方向为原油降解及资源化利用。E-mail:hhyu01@163.com。
