利用双荧光素酶报告系统鉴定靶向作用猪CD163基因的miRNA
2015-10-28吴俊静彭先文乔木等
吴俊静 彭先文 乔木等
摘要:为了进一步筛选获得抑制猪繁殖与呼吸综合征病毒(PRRSV)增殖的候选miRNA,为猪繁殖与呼吸综合征(PRRS)防治及猪的抗病育种提供新策略,通过3′RACE(Rapid-amplification of cDNA ends)测序获得了猪CD163基因3′UTR的完整序列,生物信息学分析发现其具有miR-181c、miR-181d、miR-23b、miR-4262等miRNA的作用靶点。利用双荧光素酶报告系统检测发现,与阴性对照组相比,miR-181c、miR-181d、miR-23b和miR-4262均可极显著(P<0.01)抑制荧光素酶的相对活性,其中miR-181d抑制效果最佳。
关键词:双荧光素酶报告系统;miRNA;CD163基因;猪
中图分类号:R318; Q78 文献标识码:A 文章编号:0439-8114(2015)19-4854-05
DOI:10.14088/j.cnki.issn0439-8114.2015.19.052
Abstract: CD163 gene is one of the most important PRRSV entry mediators, which determines the cell susceptibility for PRRSV. If we can identify the miRNAs inhibiting CD163 gene expression, the anti-PRRSV miRNAs can be further acquired. In the present study, we got the full porcine CD163 3′UTR sequence by 3′RACE (Rapid-amplification of cDNA ends) sequencing, and found that it existed targeting sequences of miRNA,including miR-181c,miR-181d,miR-23b and miR-4262,by bioinformatics analysis. The results of dual luciferase reporter assay showed that miR-181c,miR-181d,miR-23b and miR-4262 could significantly reduced the related luciferase activity of reporter construct compared with the NC control group (P<0.01), especially for miR-181d. These results indicated that the 4 miRNAs could directly target to porcine CD163 gene,which layed a foundation for the further screening of anti-PRRSV miRNAs in the future.
Key words: dual luciferase reporter system; miRNA; CD163 gene; pig
猪繁殖与呼吸综合征(Porcine reproductive and respiratory syndrome,PRRS)是由猪繁殖与呼吸综合征病毒(PRRSV)感染引起的一种危害极大的传染性疾病,因发病猪耳部发绀呈紫红色斑块状,又称蓝耳病,主要导致母猪繁殖障碍(流产、死胎、弱胎、木乃伊胎等)、各年龄阶段猪的呼吸道症状、哺乳仔猪的高死亡率、免疫抑制和持续性感染等[1],造成巨大的经济损失。PRRSV侵入宿主细胞首先通过与细胞膜表面上的特异性受体结合,利用细胞内吞作用感染易感细胞[2],因此这些特异性受体存在与否决定了细胞对PRRSV的易感性。CD163是PRRSV感染宿主细胞的关键受体之一,参与介导病毒的内吞和增殖。
CD163是一种清道夫受体(Scavenger receptor),在PRRSV感染宿主细胞中起重要作用,主要参与病毒脱壳和基因组RNA的释放过程。在PRRSV非易感细胞系(BHK-21、PK-15、NLFK)中转染CD163真核表达质粒可以使这些细胞系感染 PRRSV 并在细胞内产生子代病毒粒子[3,4]。CD163的表达量高低与PRRSV复制效率呈正相关,上调CD163表达会增加病毒增殖,而下调CD163表达可降低病毒增殖[5]。猪肺泡巨噬细胞(PAM)是PRRSV的天然靶细胞,而采用CD163的抗体可以有效阻止PRRSV感染PAM[3]。永生化的PAM细胞系因无法正常表达CD163而变为PRRSV非易感细胞[6]。CD163是PRRSV感染过程中的关键受体,在PRRSV感染PAM或者是Marc-145细胞的过程中都必须要有CD163的表达。
MicroRNA(miRNA)是一类长约22 bp、高度保守的内源性非编码RNA分子,其表达具有明显的组织和时间特异性,能够互补或部分互补的与靶mRNA结合,使mRNA降解或介导其翻译抑制。成熟的miRNA可同时封闭多个靶基因的翻译,干预调节miRNA可改变多个靶基因的表达水平,且干预调节效率很高[7]。miRNA在病毒感染和宿主天然免疫中起关键作用,参与病毒与宿主的互作,它有可能是细胞最原始的免疫方式。在小鼠中发现miR-126可通过靶向作用于VEGFR2调控病源引起的天然免疫反应[8]。在人巨噬细胞中,miR-155可靶向作用于SOCSS1,刺激干扰素信号通路下游基因表达,参与抗病毒天然免疫过程[9]。
猪CD163基因在PRRSV感染宿主猪细胞的过程中起关键作用,同时miRNA介导的基因调控对调节机体免疫功能具有重要作用,广泛参与机体抗病毒反应。miRNA多数是通过与靶基因的3′UTR区序列特异性识别来实现对基因表达的调控作用,因此本研究构建了猪CD163基因3′UTR双荧光素酶报告载体,筛选鉴定了靶向调控猪CD163基因的miRNA,可为筛选抑制PRRSV感染增殖的miRNA奠定基础。endprint
1 材料与方法
1.1 主要试剂与仪器
Marc-145细胞系由华中农业大学保存,用含10%胎牛血清的DMEM培养基对Marc-145细胞进行培养。psi-CHECK2载体、双荧光素酶检测试剂盒、T4 DNA连接酶和感受态大肠杆菌DH5α购自Promega公司;去内毒素质粒小量抽提试剂盒购自Omega公司;Opti-MEM无血清培养基、胎牛血清购自Gibco公司;DMEM培养基、磷酸盐缓冲液(PBS)购自HyClone公司;Trizol试剂和脂质体(LipofectaminTM 2000)购自Invitrogen公司;3′RACE (Rapid-amplification of cDNA ends)试剂盒、限制性内切酶XhoⅠ和NotⅠ、Taq DNA聚合酶、dNTP均购自Takara公司。引物由生工生物工程(上海)股份有限公司合成;miRNA由上海吉玛制药技术有限公司人工合成。
1.2 猪CD163基因的3′RACE测序
提取湖北白猪(湖北省农业科学院畜牧兽医研究所实验猪场)肝脏组织总RNA,使用Takara的3′RACE Adaptor引物进行反转录反应,合成cDNA第一链。反应体系为10 μL,反应程序为:42 ℃ 60 min;70 ℃ 15 min。
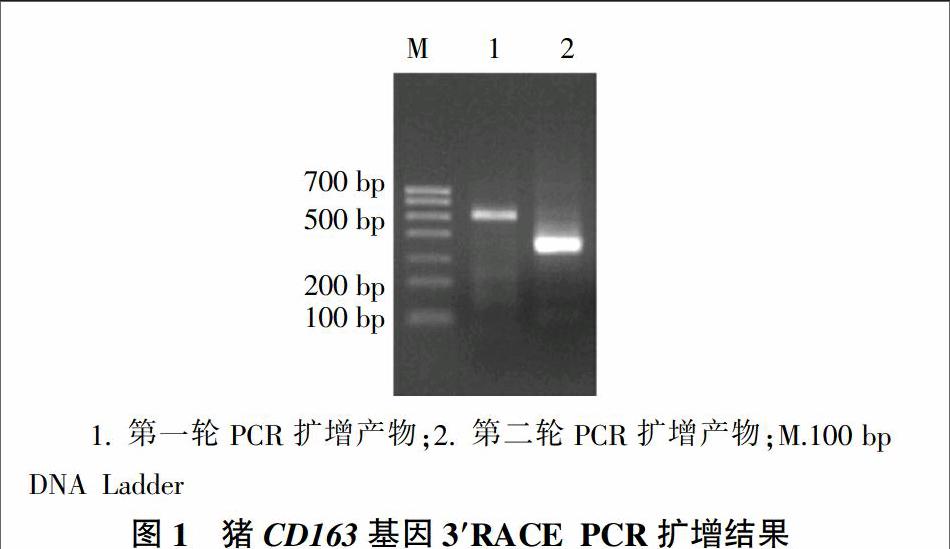
参考猪CD163基因GenBank的cDNA序列(登录号:NM_213976.1)设计外、内两个上游特异性引物(外引物:5′-CAGGTCGCTCATCTTTTGTTGC-3′和内引物:5′-TACCGTCGTTCCACTAGTGATTT -3′),结合Takara 3′RACE试剂盒的外、内两个下游锚定引物(外引物:5′-TACCGTCGTTCCACTAGTGATTT-3′和内引物:5′-CGCGGATCCTCCACTAGTGATTTCACTATAGG-3′),利用槽式降落PCR扩增3′UTR区片段。第一轮外引物PCR扩增反应体系为50 μL,其中反转录cDNA 2 μL,反应程序为:95 ℃预热4 min;95 ℃变性30 s,62~52 ℃退火30 s(每个循环降低0.5 ℃),72 ℃延伸90 s,共20个循环;95 ℃变性30 s,52 ℃退火30 s,72 ℃延伸90 s,共15个循环;72 ℃延伸10 min;4 ℃保存。第二轮内引物PCR扩增反应体系为50 μL,其中第1轮PCR产物1 μL,反应程序为:95 ℃预热4 min;95 ℃变性30 s,64~54 ℃退火30 s(每个循环降低0.5 ℃),72 ℃延伸90 s,共20个循环;95 ℃变性30 s,54 ℃退火30 s,72℃延伸90 s,共15个循环;72℃延伸10 min;4 ℃保存。PCR反应产物用1.2%琼脂糖凝胶电泳检测,从琼脂糖回收目的DNA,送北京奥科鼎盛生物科技有限公司测序。
1.3 靶向猪CD163基因3′UTR的miRNA预测
利用生物信息学工具TargenScan,参考人CD163基因3′UTR序列,预测调控CD163基因表达的物种间保守miRNA(miR-181a、miR-181b、miR-181c、miR-181d、miR-23a、miR-23b、miR-23c等)及其作用靶位点序列(TGAATGT和AATGTGA),详见http://www.targetscan.org/cgi-bin/targetscan/vert_61/view
_gene.cgi?taxid=9606&rs=NM_203416&members=&showcnc=0&shownc=0&showncf=。对比新扩增获得的猪CD163基因3′UTR区序列,发现也包含这些保守miRNAs的靶位点序列(TGAATGT和AATGTGA)。人工合成miR-181c、miR-181d、miR-23a、miR-4262及阴性对照(NC)miRNA模拟物。
1.4 猪CD163基因3′UTR双荧光素酶报告载体的构建
结合猪CD163基因CDS序列和以上3′RACE测序结果,设计包含猪CD163基因3′UTR区的1对引物,PCR扩增猪CD163基因3′UTR区的 469 bp片段。所用引物:上游引物:5′-CCGCTCGAGTCGCT
CATCTTTTGTTGC-3′,含有XhoⅠ酶切识别位点(CTCGAG)和保护碱基(CCG);下游引物:5′-ATTTGCGGCCGCTTTTATTTAATGCCCTTG-3′,含有NotⅠ酶切识别位点(GCGGCCGC)和保护碱基(ATTT)。PCR扩增反应体系为50 μL,反应程序为:95 ℃预热4 min;95 ℃变性30 s,55 ℃退火30 s,72 ℃延伸90 s,5个循环;95 ℃变性30 s,60 ℃退火30 s,72 ℃延伸90 s,共25个循环;72 ℃延伸10 min;4 ℃保存。PCR反应产物用1.2% 琼脂糖凝胶电泳检测,采用过柱离心的方法从琼脂糖回收纯化PCR产物,并对其进行测序验证。
将切胶纯化回收后的PCR产物及psiCHECK-2载体分别用限制性内切酶XhoⅠ和NotⅠ双酶切,然后采用T4 DNA 连接酶将线性化psiCHECK-2载体与CD163基因3′UTR 片段相连,连接体系为10 μL:psi-CHECK2载体2 μL,CD163基因3′UTR扩增片段6 μL,T4连接酶1 μL,10× T4 DNA连接酶缓冲液1 μL。混合体系于4 ℃连接过夜。取5 μL的连接产物连接反应产物转化50 μL感受态大肠杆菌DH5α,采用菌液PCR筛选阳性克隆,并将用XhoⅠ和NotⅠ双酶切正确的重组质粒进行测序鉴定,测序鉴定正确的重组质粒命名为psi-CHECK2-CD163-3UTR。
1.5 荧光素酶活性的检测endprint
在转染前1 d,将5×104个细胞接种在24孔板中,加入无抗生素的培养基培养细胞。转染当天,更换细胞新鲜培养液,用Opti-MEM无血清培养基稀释质粒和脂质体,每孔加入200 ng psi-CHECK2-CD163-3UTR质粒、80 nmol/L各miRNA、2 μL LipofectaminTM 2000脂质体。将细胞培养板放入无菌的5% CO2培养箱中,培养5 h后更换为新鲜完全培养基,24 h后裂解细胞检测荧光素酶活性。上述试验每组设3个平行孔,以不进行转染的细胞作为空白对照,每组试验重复3次。
用冷PBS清洗细胞2次,加入120 μL细胞裂解液1×PLB,室温摇晃15 min充分裂解细胞,然后将裂解液收集至1.5 mL离心管中,按照Promega 公司的双荧光素酶检测试剂盒(Dual-Luciferase Reporter Assay)说明书提供的方法,采用PerkinElmer公司的VICTORTM X2 Multilabel Plate Reader仪器检测双荧光素酶活性。
1.6 数据分析
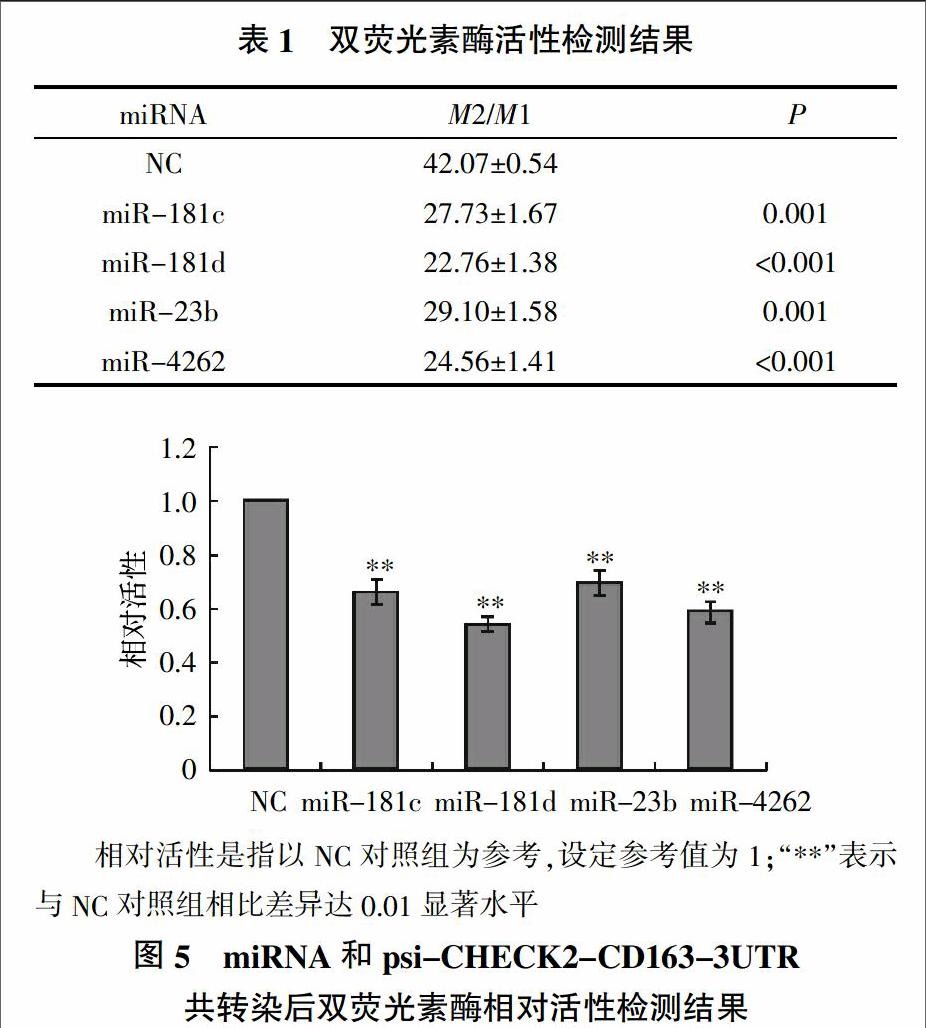
萤火虫荧光值(M1)为内参荧光值,海肾荧光值(M2)为报告基因检测值,结果以M2与M1的比值(M2/M1)计算,利用SPSS 17.0对检测结果进行统计分析。数据以平均数±标准差表示,检验标准以P<0.05为有统计学差异。
2 结果与分析
2.1 猪CD163基因的3′RACE测序及分析
miRNA多数是通过与靶基因的3′UTR区序列特异性识别来实现对基因表达的调控作用,而目前GenBank数据库中猪CD163基因的3′UTR序列并不完整,无法进行调控猪CD163基因表达miRNAs的筛选鉴定研究。为了获得猪CD163基因3′UTR的全序列,采用3′RACE结合槽式降落PCR技术,成功扩增获得了猪CD163基因的特异性片段(图1),片段测序结果与GenBank数据库中已知的猪CD163 mRNA序列(NM_213976.1)进行比对分析,获得猪CD163 CDS下游(3′)新的181 bp核苷酸序列,该序列包含PolyA特征的3′UTR核苷酸序列。
利用TargetScan生物学软件预测可调控CD163基因3′UTR区的物种间保守miRNA(miR-181a、miR-181b、miR-181c、miR-181d、miR-23a、miR-23b、miR-23c等)及作用靶位点序列(TGAATGT和AATGTGA),发现新扩增获得的猪3′UTR区也包含这些保守miRNA的作用靶位点序列。
2.2 猪CD163基因3′UTR双荧光素酶报告载体的构建与鉴定
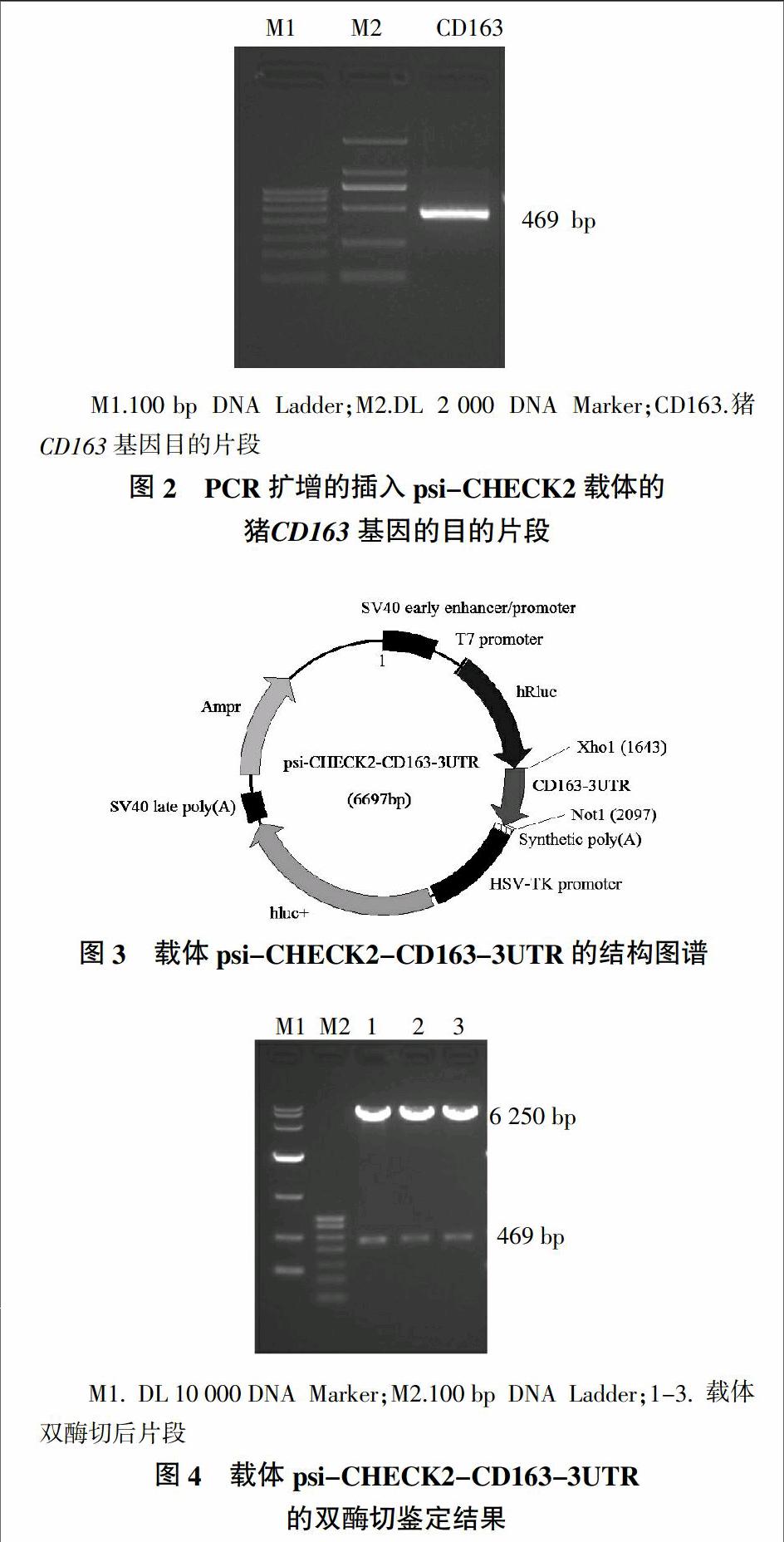
根据获得的猪CD163基因的CDS和3′RACR测序结果,设计包含3′UTR区的1对引物,在5′端和3′端分别引入XhoⅠ和NotⅠ酶切识别位点及保护碱基,利用PCR方法扩增获得了猪CD163基因的469 bp长的目的片段(图2),将该片段插入双荧光素酶报告载体psi-CHECK2的XhoⅠ和NotⅠ酶切位点之间,构建猪CD163基因3′UTR双荧光素酶报告基因载体psi-CHECK2-CD163-3UTR,构建的载体结构如图3所示。采用菌液PCR鉴定阳性克隆,并用XhoⅠ和NotⅠ限制性内切酶进行双酶切鉴定,证实酶切后得到的469 bp的猪CD163基因3′UTR片段与预期大小相符(图4)。抽提的重组质粒经测序再次验证插入序列的正确性。
2.3 miRNA对猪CD163基因3′UTR的调控
为了检测以上预测的miRNA是否能靶向调控猪CD163基因3′UTR,将psi-CHECK2-CD163-3UTR和相应miRNA模拟物分别共转染Marc-145细胞,采用双荧光素酶报告系统检测荧光素酶活性,结果如表1和图5所示。与NC对照组相比,转染miR-181c、miR-181d、miR-23b和miR-4262可以极显著(P<0.01)降低双荧光素酶报告载体psi-CHECK2-CD163-3UTR的荧光素酶活性,其中miR-181d抑制效果最佳(降低46%)。说明miR-181c、miR-181d、miR-23b和miR-4262均可靶向作用于猪CD163基因3′UTR,对猪CD163基因具有一定的调控作用。
3 小结与讨论
CD163是PRRSV感染宿主细胞的一个关键受体,主要参与病毒脱壳和基因组RNA的释放过程。它由单核细胞-巨噬细胞特异表达,结构包含信号肽、9个串联的SRCR末端重复序列、一个跨膜区以及胞质尾区。2005年Welch等[10]首次发现CD163与PRRSV感染有关,PRRSV非易感系的猪肾细胞表达CD163后,可以被PRRSV感染并产生子代病毒。Calvert等[3]将从各种细胞系中分离得到CDl63分子转染到PRRSV不敏感的细胞系后,这些细胞内都产生子代病毒粒子。并且CD163表达量的高低与PRRSV复制效率呈正相关,上调CD163表达会增加病毒增殖,而下调CD163表达可降低病毒增殖[5]。Ren等[11]发现猪CD163基因CDS区域序列变异与猪PRRSV易感性显著相关。
近几年有文献报道miRNA参与调控PRRSV感染。在人工miRNA方面,Xiao等[12]和Xia等[13]针对PRRSV病毒基因组特点,设计人工miRNA,利用内源性miRNA代谢途径加工成熟后靶向降解PRRSV RNA,从而达到抑制病毒感染的效果。这表明人工miRNA的应用可能成为有效抗PRRSV的新策略。在内源性抗PRRSV增殖的miRNA研究方面,Wang等[14]发现与天然免疫相关的miR-125b能显著抑制PRRSV的RNA复制和病毒增殖,miR-125b并不是直接作用于PRRSV的基因组,而是作用于宿主细胞中NF-κB的抑制因子κB-Ras2,通过调控NF-κB信号通路抑制PRRSV的增殖。endprint
CD163在PRRSV感染宿主细胞过程中具有重要作用,PRRSV必须依赖CD163的表达才能在宿主细胞中繁殖。因此,如果能鉴定获得抑制CD163基因表达的miRNA,就可以进一步筛选获得抑制PRRSV增殖的候选miRNA,为PRRS防治及猪的抗病育种提供新策略。本研究扩增获得了猪CD163基因3′UTR的完整序列,生物信息学分析发现其具有miR-181c、miR-181d、miR-23b、miR-4262等miRNA的作用靶点。利用双荧光素酶报告系统鉴定发现miR-181c、miR-181d、miR-23b和miR-4262均可靶向作用猪CD163基因的3′UTR,其中miR-181d抑制效果最佳。这4个miRNA也可能是潜在的能抑制PRRSV增殖的miRNA。然而,本结果还需要借助体外细胞学实验进一步验证。
Guo等[15]发现miR-181能与PRRSV RNA非编码序列结合,抑制PRRSV感染,且体内实验证实鼻滴miR-181模拟物可抑制PRRSV在仔猪体内增殖,缓解攻毒后仔猪发病症状。研究还表明宿主miR-181在PAM细胞表达量较低,而在非易感细胞或组织中高表达。在此基础上,Gao等[16]进一步研究发现miR-181能靶向抑制CD163基因表达,从而抑制PRRSV感染,与本研究结果一致。Zhang等[17]研究结果显示,上调表达miR-23能抑制PRRSV感染,而干扰掉内源性miR-23表达会增强PRRSV增殖,表明miR-23具有一定的抗PRRSV增殖效应。他们进一步研究指出miR-23能通过激活IRF3/IRF7诱导产生Ⅰ型干扰素,从而抑制PRRSV感染。然而并未研究miR-23与CD163基因的靶向调控关系,很可能miR-23能通过多种途径(包括抑制受体基因CD163的表达)达到抑制病毒的效果。有关miR-4262的相关研究报道比较少,其功能还不十分明确。
本研究利用双荧光素酶报告系统明确了miR-181c、miR-181d、miR-23b和miR-4262能靶向作用猪CD163基因的3′UTR,对猪CD163基因具有负向调控作用,可为进一步筛选抗PRRSV增殖miRNA奠定基础。
参考文献:
[1] OPRIESSING T, BAKER R B, HALBUR P G. Use of an experimental model to test the efficacy of planned exposure to live porcine reproductive and respiratory syndrome virus[J]. Clin Vaccine Immunol, 2007, 14(12):1572-1577.
[2] DELPUTTE P L, COSTERS S, NAUWYNCK H J. Analysis of porcine reproductive and respiratory syndrome virus attachment and internalization: Distinctive roles for heparan sulphate and sialoadhesin[J]. J Gen Virol, 2005, 86(5): 1441-1445.
[3] CALVERT J G, SLADE D E, SHIELDS S L, et al. CD163 expression confers susceptibility to porcine reproductive and respiratory syndrome viruses[J]. J Virol, 2007, 81(14): 7371-7379.
[4] WELCH S K, CALVERT J G. A brief review of CD163 and its role in PRRSV infection[J]. Virus Res, 2010, 154(1-2): 98-103.
[5] PATTON J B, ROWLAND R R, YOO D, et al. Modulation of CD163 receptor expression and replication of porcine reproductive and respiratory syndrome virus in porcine macrophages[J]. Virus Res, 2009, 140(1-2): 161-171.
[6] LEE Y J, PARK C K, NAM E, et al. Generation of a porcine alveolar macrophage cell line for the growth of porcine reproductive and respiratory syndrome virus[J]. J Virol Methods, 2010, 163(2): 410-415.
[7] CARTHEW R W, SONTHEIMER E J. Origins and mechanisms of miRNAs and siRNAs[J]. Cell,2009,136(4):642-655.
[8] AGUDO J, RUZO A, TUNG N, et al. The miR-126-VEGFR2 axis controls the innate response to pathogen-associated nucleic acids[J]. Nat Immunol, 2014, 15(1): 54-62.
[9] WANG P, HOU J, LIN L, et al. Inducible microRNA-155 feedback promotes type I IFN signaling in antiviral innate immunity by targeting suppressor of cytokine signaling 1[J]. J Immunol, 2010, 185(10): 6226-6233.endprint
[10] WELCH S K W, SLADE D E, SHIELDS S L, et al. Identification and characterization of a cellular gene essential for PRRS virus infection, 2005 international PRRS symposium [J]. St Louis Missoud, 2005, 2:3-22.
[11] REN Y W, ZHANG Y Y, AFFARA N A, et al. The polymorphism analysis of CD169 and CD163 related with the risk of porcine reproductive and respiratory syndrome virus (PRRSV) infection[J]. Mol Biol Rep, 2012, 39(11):9903-9909.
[12] XIAO S, WANG Q, GAO J, et al. Inhibition of highly pathogenic PRRSV replication in MARC-145 cells by artificial microRNAs[J]. Virol J, 2011, 8:491.
[13] XIA B, SONG H, CHEN Y, et al. Efficient inhibition of porcine reproductive and respiratory syndrome virus replication by artificial microRNAs targeting the untranslated regions[J]. Arch Virol, 2013, 158(1): 55-61.
[14] WANG D, CAO L, XU Z, et al. MiR-125b reduces porcine reproductive and respiratory syndrome virus replication by negatively regulating the NF-κB pathway[J]. PLoS One, 2013, 8(2):e55838.
[15] GUO X K, ZHANG Q, GAO L, et al. Increasing expression of microRNA 181 inhibits porcine reproductive and respiratory syndrome virus replication and has implications for controlling virus infection[J]. J Virol, 2013, 87(2):1159-1171.
[16] GAO L, GUO X K, WANG L, et al. MicroRNA 181 suppresses porcine reproductive and respiratory syndrome virus (PRRSV) infection by targeting PRRSV receptor CD163[J]. J Virol, 2013, 87(15): 8808-8812.
[17] ZHANG Q, GUO X K, GAO L, et al. MicroRNA-23 inhibits PRRSV replication by directly targeting PRRSV RNA and possibly by upregulating type I interferons[J]. Virology, 2014, 450-451:182-195.endprint
