绿色杜氏藻DHDDS基因生物信息学分析及表达研究
2015-10-27潘益芳龚一富俞凯朱帅旗王何瑜
潘益芳 龚一富 俞凯 朱帅旗 王何瑜
(宁波大学海洋学院,宁波 315211)
绿色杜氏藻DHDDS基因生物信息学分析及表达研究
潘益芳 龚一富 俞凯 朱帅旗 王何瑜
(宁波大学海洋学院,宁波 315211)
绿色杜氏藻是一种能产生重要次生代谢产物类胡萝卜素的单细胞绿藻,脱氢多萜醇焦磷酸合成酶(dehydrodolichyl diphosphate synthase,DHDDS)是其在类胡萝卜素合成途径中的相关酶。旨在研究DHDDS基因表达与类胡萝卜素含量之间的关系。提取绿色杜氏藻总RNA,通过转录组测序获得DHDDS基因全长,对该基因进行生物信息学分析;并用不同浓度甲基茉莉酸(MeJA)处理绿色杜氏藻,采用实时定量PCR研究该基因转录差异。结果显示,绿色杜氏藻DHDDS基因全长2 211 bp,含有一个1 740 bp长的开放阅读框(ORF),编码579个氨基酸序列。DHDDS蛋白质理论等电点为7.63,相对分子质量为62 472.7 Da。预测结果表明,DHDDS蛋白不含信号肽,也不存在跨膜区域,该蛋白定位于细胞质基质。氨基酸序列比对结果显示,绿色杜氏藻DHDDS蛋白与小球藻的同源性最高(59%)。实时定量PCR结果表明,经100 μmol/L MeJA处理的绿色杜氏藻DHDDS表达水平最高,具有极显著差异;在该浓度下,类胡萝卜素含量均高于其他浓度组。且DHDDS基因的表达与类胡萝卜素含量呈一定的相关性。绿色杜氏藻DHDDS是一种定位于细胞质基质中的酶,其与绿色杜氏藻类胡萝卜素合成途径有关。在一定浓度范围的MeJA诱导下,低浓度的MeJA对其表达起促进作用,高浓度MeJA对其表达有抑制作用,且其表达与类胡萝卜素含量呈一定的相关性。
绿色杜氏藻;脱氢多萜醇焦磷酸合成酶;类胡萝卜素;甲基茉莉酸
极端嗜盐绿色杜氏藻(Dunaliella viridis)又称绿色杜氏藻,是一种单细胞真核绿藻,较其它藻类更能忍受高盐度,是迄今为止发现的最耐盐的生物之一[1]。由于其结构简单,无细胞壁,因此绿色杜氏藻成为研究耐盐机制理想的模式生物[2]。绿色杜氏藻细胞中含有丰富的类胡萝卜素、甘油、多糖等能提高免疫力、清除自由基、预防心血管病的物质[3],可以作为天然的保健品,具有一定的营养价值;目前,对绿色杜氏藻的研究主要集中在耐盐机制[4]和类胡萝素积累机制[5]这两个方面。
类胡萝卜素通常是指含有40个碳原子的萜类色素,主要通过萜类途径合成,即甲羟戊酸途径(MVA)和2-C-甲基-D-赤藓糖醇-4-磷酸途径(MEP)。脱氢多萜醇焦磷酸合成酶(dehydrodolichyl diphosphate synthase,DHDDS)存在于萜类合成的第二阶段,将异戊烯基焦磷酸(isopentenyl diphosphate,IPP)与法呢基焦磷酸(farnesyl-phyophosphate,FPP)或焦磷酸双牛龙牛儿基丙酮(geranylgeranyl diphosphat,GGPP)通过不断缩合,直至形成焦磷酸多萜醇(polypeno-pyrophosphat,Pol-PP)[6],Pol-PP经脱磷酸和还原后得到多萜醇(dolichol,Dol)。DHDDS在许多糖蛋白的N-糖基化中起着非常重要的作用,是多萜醇合成途径中的关键酶,也是类胡萝卜素合成途径的相关酶。
甲基茉莉酸(Methyl Jasmonate,MeJA)是一种广泛存在于植物体内的生长调节物质。有报道认为外源性茉莉酸类化合物能有效刺激植物次生代谢物的生物合成[7,8],能诱导植物产生抗毒素。在细胞工程中,MeJA 是促进类胡萝卜素等次生代谢产物积累的诱导因子[9]。植物中次生代谢产物的合成通过诱导因子调节次生代谢途径中关键酶的表达来调节[10]。马君兰等[11]研究表明,在大豆生长过程中通过喷洒MeJA 能改变苯丙氨酸解氨酶(PAL)的酶活性,从而影响其次级代谢,使大豆中异黄酮的含量增加。廖巧[12]研究表明,在青蒿生长过程中用 MeJA 喷洒的植株α-氨基己二酸还原酶(AAR)、乙醛脱氢酶1(ALDH1)基因的表达水平显著提高,其青蒿素含量亦显著提高。桂连友[13]研究表明,外源 MeJA 能够提高茶树叶片脂氧合酶(LOX)的活性。施江[14]研究表明,外源 MeJA能诱导茶树鲜叶中不同的次生代谢途径,能降低非挥发性次生产物含量,增加挥发性产物含量,提高茶树鲜叶的品质。但是MeJA诱导藻类次生代谢产物积累方面的报道较少,王鑫威等[15]研究表明,一定浓度的MeJA可以促进单位细胞雨生红球藻次生代谢产物虾青素的合成。章丽等[16]研究表明,一定浓度的外源MeJA能诱导盐生杜氏藻β-胡萝卜素的积累。因此本研究使用MeJA作为诱导子,试图探究其在绿色杜氏藻DHDDS合成代谢途径中的影响。
如今,DHDDS基因已经在拟南芥[17]、玉米[18]、斑马鱼[19]等动植物中被克隆出来,但尚未有绿色杜氏藻中的DHDDS基因的相关报道。本实验通过绿色杜氏藻转录组测序,序列拼接和分析得到DHDDS基因序列,对其进行生物信息学分析,同时用诱导子甲基茉莉酸处理绿色杜氏藻,研究DHDDS的转录差异,旨在为进一步阐明DHDDS在类胡萝卜素合成代谢途径及其调控机制奠定基础。
1 材料与方法
1.1 材料
1.1.1 藻种 绿色杜氏藻藻种(D. viridis)由宁波大学海洋生物工程重点实验室微藻室提供。绿色杜氏藻培养液为PKS培养基,25±0.5℃,4 000 lx光强,光暗周期比12 h∶12 h,每天摇瓶2-3次。
1.1.2 引物设计与合成 利用Primer Premier5.0软件设计引物,由生工生物工程(上海)有限公司合成。
1.2 方法
1.2.1 绿色杜氏藻总RNA的提取 取纯化培养的处于对数生长期的绿色杜氏藻,离心收集,按照TaKaRa RNAiso plus kit(TaKaRa,大连)操作说明书提取总RNA。经1.2%琼脂糖凝胶电泳检测 RNA质量。按照反转录试剂盒(TaKaRa,大连)的操作说明,反转录获得cDNA,保存于-20℃备用。
1.2.2 转录组测序获得基因序列 绿色杜氏藻DHDDS基因全长序列由本实验室绿色杜氏藻转录组测序获得的。
1.2.3 DHDDS的生物信息学分析 将测序获得的 序 列 在NCBI(National Center for Biotechnology Information,http://www.ncbi.nlm.nih.gov/)网站查找其开放阅读框(Open Reading Frame,ORF)并进行核酸、氨基酸比对[20];通过CBS(http://www. cbs.dtu.dk/)网站预测信号肽、转运肽及其跨膜结构域[21-23];使用ExPASy(http://cn.expasy.org)中文镜像网站预测蛋白的疏水性/亲水性、分子量、等电点及二、三级结构[24,25]。利用DNAMAN 4.0 软件推导DHDDS基因的氨基酸序列;用Vector NTI 7.1 软件对测序获得的绿色杜氏藻DHDDS基因进行多序列比对;MEGA 3.0 软件的邻接归并法(NJ法)构建系统进化树[26];用SOPMA[27](https://sopma. expasy.org/)预测绿色杜氏藻DHDDS氨基酸序列的二级结构。
1.2.4 绿色杜氏藻DHDDS表达特征分析 将绿色杜氏藻细胞以1×105cells/mL接种到PKS培养液中,培养2 d后,向培养瓶中分别添加不同体积的MeJA母液,分别将培养瓶中MeJA终浓度调整为0(对照组)、50、100、200和500 μmol/L,每个外源MeJA诱导组设3个重复组。处理24 h,提取不同处理组绿色杜氏藻的总RNA。用NanoDrop 2000超微量核酸定量光谱仪(Thermo,美国)测定RNA样品的浓度和纯度。荧光定量PCR 目的基因引物:DHDDS-1:5'-CCTGGAAACGCTTGAATAAAT-3'和DHDDS-2:5'-CAGTTGCTCGTATCCGTATCG-3',β-actin内参基因引物:BACTIN-1:5'-CGTGACTTGACGGACTACCTG-3'和 BACTIN-2:5'-TAGTTTTTGTCCAGAGCCGAG-3'。 荧光定量PCR反应在Realplex4型定量PCR仪(Eppendorf,德国)上进行,反应体系为:SYBR®Premix Ex TaqTM(2×)10 μL,上下游引物(10 μmol/L)各0.8 μL,cDNA模板2 μL,ddH2O 6.4 μL。PCR反应程序为:94℃ 3 min;94℃30 s,58℃ 30 s,72℃ 20 s,40个循环。反应结束后确认扩增曲线和融解曲线。计算绿色杜氏藻DHDDS基因相对表达量,每个样品重复3次。采用SPSS软件进行数据统计分析。
1.2.5 绿色杜氏藻类胡萝卜素含量的提取和测定用0、50、100、200和500 μmol/L的MeJA处理绿色杜氏藻,培养10 d,吸取20 mL藻液置于50 mL离心管中,4 000 r/min离心8 min,用双蒸水冲洗沉淀2 次,加16 mL纯丙酮,冰浴超声5 min,-20℃保存24 h后,8 000 r/min离心10 min,重复提取直到藻体呈白色。以1∶10的量加入60% KOH于色素提取液中,振荡后静置分层,以除去叶绿素和中性脂肪的影响。全部提取液用 0.22 μm 的注射式膜过滤,定容到100 mL,在722型分光光度计上测定450 nm波长下提取液的最大吸光度A,以胡萝卜素分子平均吸收系数250为依据,按下式计算:
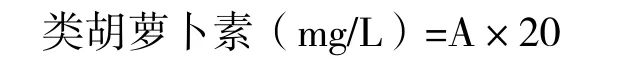
式中,A为测定的最大吸光度;20为换算系数。以上操作都在遮光条件下进行。
2 结果
2.1 绿色杜氏藻DHDDS生物信息学分析
2.1.1 绿色杜氏藻DHDDS的基本理化性质分析绿色杜氏藻转录组测序获得的DHDDS基因序列全长为2 211 bp,其中5'非翻译区(Untranslated Regions,UTR)为204 bp,3' UTR为270 bp,开放阅读框(ORF)1 740 bp(核苷酸序列位置269-2 008),编码579 个氨基酸的蛋白质(图1),该蛋白的分子式为C2718H4262N822O810S33,原子个数8 645,推导的蛋白质理论等电点(pI)为7.63,相对分子质量为62 472.7 Da。正电荷残基(Arg+Lys)总数为54,负电荷残基(Asp+Glu)总数为53,不稳定系数为60.79,该蛋白分类为不稳定蛋白。脂肪系数为74.54,其疏水性平均值为-0.317,表现为亲水性,无明显的疏水区域。具有3个主要保守区,保守序列为AFIMDGNRR,YAFSI(D/E)NFKR和IRTSG(E/A)(T/I/A)RLS(N/D)F(L/M)LWQ,到目前为止,这是多萜醇磷酸合成酶的共同特征[28],属于顺式-IPPS蛋白家族。同源性比对发现绿色杜氏藻与小球藻(Chlorella variabilis)DHDDS基因的氨基酸序列相似性最高,达到了59%,与虎刺梅(Callorhinchus milii)、侏儒鸟(Manacus vitellinus)、斑马鱼(Danio rerio)、小家鼠(Mus musculus)和小头虫(Capitella teleta)的同源性分别为46%、46%、43%、43%和40%。
2.1.2 绿色杜氏藻DHDDS亚细胞定位的预测 通过CBS网站在线预测,结果显示DHDDS不含信号肽,也不存在跨膜区域,其定位于细胞质基质。

图1 绿色杜氏藻DHDDS基因全长及其推导氨基酸序列
2.1.3 绿色杜氏藻DHDDS二级结构和三级结构的预测 用SOPMA预测绿色杜氏藻DHDDS氨基酸序列的二级结构,有34.72%的α-螺旋,15.54%的延长链,7.08%的β-转角和42.66%的无规则卷曲。DHDDS 三级结构的预测结果如图 2 所示。该蛋白是典型的T字型结构,其酶的活性位点在其T字交叉处,其中Lys40靠近该蛋白质的活性位点,是在多个物种中高度保守的残基,表示强烈的进化约束。带正电的Arg235在酶的活性位点的组织中起重要作用,可能与活性位点与FPP的结合有关。

图2 绿色杜氏藻DHDDS蛋白三级结构预测图
2.1.4 绿色杜氏藻DHDDS的生物进化分析 通过NCBI数据库中Blastp对DHDDS氨基酸序列进行相似性搜索和比对,选取同源性较高的11个物种构建进化树,序列如下:麦芽糖假丝酵母(Candida maltosa,EMG47928.1);伞状毛霉菌(Lichtheimia corymbifera,CDH54945.1);短花药野生稻(Oryza brachyantha,XP_006655828.1);小米(Setaria italica,XP_004964740.1);玉米(Zea mays,ACG35019.1);马铃薯(Solanum tuberosum,XP_006353865.1);甜橙(Citrus sinensis,XP_006480091.1);葡 萄(Vitis vinifera,XP_002263977.1); 草 莓(Fragaria vesca subsp. Vesca,XP_004289930.1);小球藻(Chlorella variabilis,XP_005848062.1);新型微藻(Auxenochlorella protothecoides,KFM24359.1)。结果(图3)显示,12个不同物种的DHDDS基因大体分成4支,小球藻(Chlorella variabilis)和新型微藻(Auxenochlorella protothecoides)聚成一支,真菌麦芽糖假丝酵母(Candida maltosa) 和 伞 状 毛 霉 菌(Lichtheimia corymbifera)聚成一支,双子叶植物甜橙(Citrus sinensis),草莓(Fragaria vesca subsp. Vesca),葡萄(Vitis vinifera)和马铃薯(Solanum tuberosum)聚成一支,而绿色杜氏藻(Dunaliella viridis)则与单子叶植物玉米(Zea mays)、小米(Setaria italica)和短花野生稻(Oryza brachyantha)聚成一支。可以看出绿色杜氏藻与单子叶植物的亲缘关系较近,与其他植物亲缘关系稍远。

图3 绿色杜氏藻与其他物种DHDDS氨基酸序列的系统进化树
2.2 绿色杜氏藻在MeJA诱导下类胡萝卜素含量和DHDDS表达分析
实时荧光定量结果(图4)表明,经过不同浓度的甲基茉莉酸(MeJA)处理绿色杜氏藻,DHDDS基因相对表达水平具有一定差异性,呈现先增加后减少再增加的趋势。浓度为100 μmol/L MeJA处理绿色杜氏藻,DHDDS转录水平最高,经500 μmol/L MeJA处理绿色杜氏藻,其DHDDS转录水平最低。经50 μmol/L MeJA处理的绿色杜氏藻DHDDS表达水平不具有显著差异(F50=1.454,P>0.005)。经100 μmol/L和500 μmol/L MeJA处理的绿色杜氏藻DHDDS表达水平具有极显著差异(F100=3.630,P<0.001;F500=0.157 127,P<0.001),经200 μmol/L MeJA处理的绿色杜氏藻DHDDS表达水平具有显著差异(F200=0.379,P<0.05)。不同浓度MeJA处理对绿色杜氏藻类胡萝卜素含量的影响,结果(图5)表明,随着MeJA浓度的升高,绿色杜氏藻类胡萝卜素含量呈现先上升后下降的趋势,MeJA浓度在100 μmol/L时,绿色杜氏藻类胡萝卜素含量最高。与对照相比,具有显著差异(C100=9.386 667,P<0.05)。MeJA浓度在1 000 μmol/L时,未检测到类胡萝卜素含量。
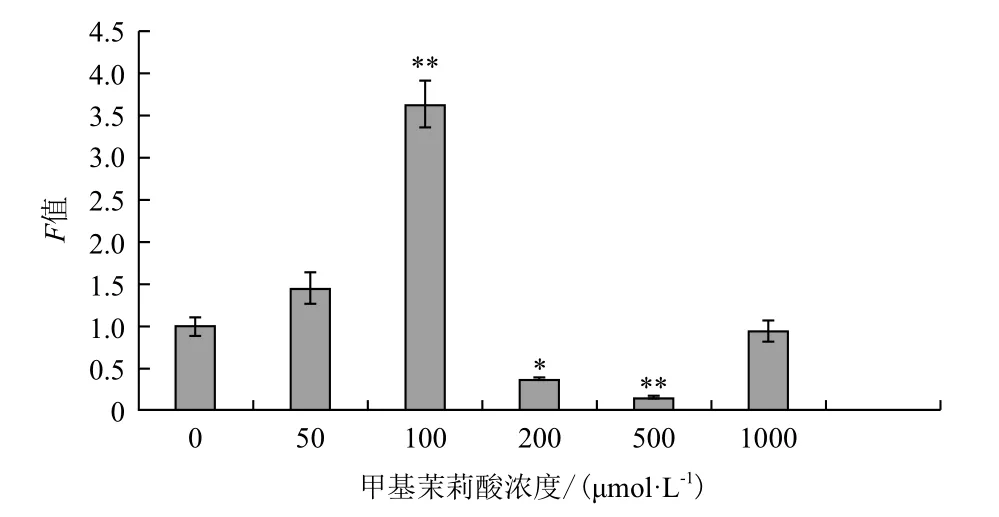
图4 甲基茉莉酸浓度对绿色杜氏藻DHDDS转录的影响
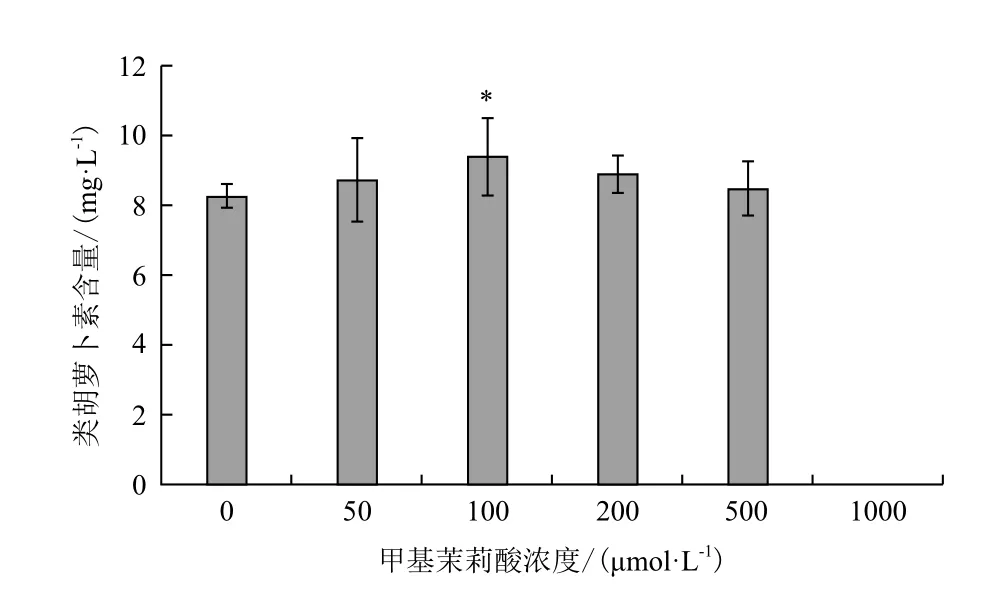
图5 甲基茉莉酸浓度对绿色杜氏藻类胡萝卜素含量的影响
3 讨论
本研究从绿色杜氏藻转录组测序结果中获得DHDDS基因序列全长,对DHDDS核苷酸序列及氨基酸序列进行比对。结果显示绿色杜氏藻DHDDS基因核酸序列未能在其他物种中找到相似序列,而氨基酸序列比对结果显示与之相似的物种有很多。这一结果证明不同物种之间的氨基酸序列比对相较于核苷酸序列比对,其相似性更高、更广泛[29]。生物信息学方法预测,DHDDS的开放阅读框为 1 740 bp,编码579个氨基酸,序列已上传至GenBank(登录号 KP125328)。
绿色杜氏藻DHDDS含有3个保守位点,分别位于 28-36 aa,76-83 aa,228-243 aa,保守结构域分析结果表明,DHDDS蛋白属于顺式-IPPS蛋白家族。其中分析保守区域发现,28-36 aa序列同目前不同物种已获得的DHDDS活性中心相同[28],基本可确定该区域是绿色杜氏藻DHDDS的活性中心。绿色杜氏藻DHDDS蛋白等电点为7.63,相对分子质量为62 472.7 Da,二级结构中无规则卷曲含量最多,其次为α-螺旋,延长链和β-转角。不含有转运肽、信号肽、跨膜结构域等结构,定位于细胞质,该结论同Núria Cunillera等[17]分析的拟南芥DHDDS定位于细胞质粗面内质网上相近。根据氨基酸序列比对制作系统进化树,发现绿色杜氏藻DHDDS与单子叶植物的亲缘关系较近,与其他植物的亲缘关系稍远。该结论虽与形态分类学[30]的结果有一定的差异,但对判断不同植物之间的亲缘关系也具有一定的借鉴意义。
MeJA是与植物代谢及防御相关的内源激素[31]。实时定量PCR结果显示,绿色杜氏藻DHDDS的表达水平受到MeJA浓度的影响,其中100 μmol/L的MeJA处理的绿色杜氏藻,其DHDDS的表达较对照组明显提高。同时,该浓度下,绿色杜氏藻细胞内类胡萝卜素含量也高于其他浓度组。而经500 μmol/L的MeJA处理后,绿色杜氏藻DHDDS的表达则受到抑制,绿色杜氏藻细胞内类胡萝卜素含量也低于其他浓度组。该结果也与前人的研究结果相符。Yukimune等[32]分别在曼地亚红豆杉和欧洲红豆杉细胞悬浮培养液中加入100 μmol/L的MeJA,发现能明显提高曼地亚红豆杉紫杉醇的含量,而欧洲红豆杉则表现出最高的浆果赤霉素III的含量。余龙江等[9]在红豆杉胚性细胞培养过程中加入不同浓度的MeJA,结果表明在浓度100 μmol/L时紫杉醇产量出现最大值。朱颖等[33]研究茉莉酸甲酯对杜氏盐藻β-胡萝卜素含量的影响发现,MeJA 能诱导盐藻β-胡萝卜素的积累,其中100 μmol/L的MeJA处理效果最佳,比对照增加了33.85%。而Choi等[34]报道高浓度的MeJA能抑制甾体类甘油生物碱的积累。这可能是高浓度的MeJA对绿色杜氏藻DHDDS的表达也有类似的作用。
绿色杜氏藻DHDDS氨基酸序列较长,是玉米(Z. mays)的1.91倍,是甜瓜(C. smelo)的1.97倍,是葡萄(V. vinifera)的2倍。本实验首次获得了绿色杜氏藻DHDDS基因,并全面地对DHDDS进行了生物信息学分析,探讨了DHDDS的表达与类胡萝卜素含量之间的关系,为今后深入研究DHDDS结构和功能提供了可靠依据。目前对绿色杜氏藻DHDDS的研究尚不完全,对于其在类胡萝卜素合成机制中的具体作用仍不是非常清楚,还需要进一步研究探索。
4 结论
通过绿色杜氏藻转录组测序,得到DHDDS基因全长序列,该基因全长为2 211 bp,ORF为1 740 bp。生物信息学分析表明,该蛋白是一种亲水性不稳定蛋白;二级结构主要组成元件为α-螺旋和无规则卷曲;3D模型成功构建,具有3个主要保守区域,属于顺式-IPPS蛋白家族。定位于细胞质基质,与绿色杜氏藻类胡萝卜素合成途径有关。不同浓度的诱导子甲基茉莉酸对绿色杜氏藻DHDDS基因的表达有影响,一定范围内,低浓度的MeJA对其表达起促进作用,高浓度MeJA对其表达有抑制作用,且其表达与类胡萝卜素含量呈一定的相关性。
[1]姬翔. 杜氏盐藻(Dunaliella salina)14-3-3蛋白cDNA全长的克隆与序列分析[D]. 郑州:郑州大学, 2006.
[2]张翅. 杜氏盐藻(Dunaliella salina)cDNA文库构建及表达序列标签分析[D]. 武汉:华中科技大学, 2009.
[3] 伍先绍, 贺稚非, 龚霄. 杜氏盐藻及其在功能食品中的应用[J].中国食品添加剂, 2008(2):127-130.
[4]刘红涛, 冯书营, 陈涛, 等. 杜氏盐藻分子生物学最新进展及展望[J]. 中国生物工程杂志, 2007, 27(10):113-118.
[5]冯书营, 刘红涛, 李杰, 等. 杜氏盐藻基因工程研究现状及应用前景[J]. 中国生物工程杂志, 2007, 27(2):108-112.
[6]Sakaihara T, Honda A, Tateyama S, et al. Subcellular fractionation of polyprenyl diphosphate synthase activities responsible for the syntheses of polyprenols and dolichols in spinach leaves[J]. J Biachem, 2000, 128(6):1073-1078.
[7] Mueller MJ, Brodschelm W, Spannagl E, et al. Signaling in the elicitation process is mediated through the octadecanoid pathway leading to jasmonic acid[J]. Proc Natl Acad Sci USA, 1993, 90:7490.
[8]Tamogami S, Rakwal R, Kodama O. Phytoalexin production elicited by exogenously applied jasmonic acid in rice leaves(Oryza sativa L.)is under the control of cytokinins and ascorbic acid[J]. FEBS Lett, 1997, 412:61.
[9]余龙江, 朱敏, 刘幸福, 等. 茉莉酸甲酯对中国红豆杉胚性细胞紫杉醇生物合成的影响[J]. 武汉植物学研究, 1999, 17(4):371-374.
[10]Qi FH, Zhan YG, Jing TZ. A review on elicitors and their regulation on secondary metabolites in plant cell culture[J]. Nat Prod Res Dev, 2008, 20:568-573.
[11]马君兰, 赵越. 外源茉莉酸甲酯(MeJA)对大豆异黄酮合成途径的影响[J]. 东北农业大学学报, 2011, 42(5):14-18.
[12]廖巧. 甲基茉莉酸诱导青蒿素积累的机理研究[D]. 重庆:西南大学, 2012.
[13]桂连友. 外源茉莉酸甲酯对茶树抗虫作用的诱导及其机理研究[D]. 杭州:浙江大学, 2004.
[14]施江. 外源茉莉酸甲酯诱导对茶树鲜叶次生代谢产物的影响[D]. 北京:中国农业科学院, 2014.
[15]王鑫威, 王丽丽, 龚一富, 等. 茉莉酸甲酯对雨生红球藻虾青素含量和dxs基因表达的影响[J]. 水产学报, 2011, 12:1822-1828.
[16]章丽, 龚一富, 刘晓丹, 等. 外源MeJA胁迫对盐生杜氏藻生理生化特性的影响[J]. 生物学杂志, 2013, 3:38-42, 47.
[17]Núria C, Montserrat A, Oriol F, et al. Characterization of dehydrodolichyl diphosphate synthase of Arabidopsis thaliana, a key enzyme in dolichol biosynthesis[J]. FEBS Letters, 2000(477):170-174.
[18]Alexandrov NN, Brover VV, Freidin S, et al. Insights into corn genes derived from large-scale cDNA sequencing[J]. Plant Mol Biol, 2009, 69(1-2):179-194.
[19]Hu MC, Gong HY, Lin GH, et al. XBP-1, a key regulator of unfolded protein response, activates transcription of IGF1 and Akt phosphorylation in zebrafish embryonic cell line[J]. Biochem Biophys Res Commun, 2007, 359(3):778-783.
[20]吕军, 张颖, 冯立芹, 等. 生物信息学工具BLAST的使用简介[J]. 内蒙古大学学报:自然科学版, 2003, 34(2):179-187.
[21]Emanuelesson O, Nielsen H, Brunak S, et al. Predicting subcellular localization of proteins based on their N-terminaal amino acid sequence[J]. J Mol Biol, 2000, 300(4):1005-1016.
[22]Emanuelesson O, Nielsen H, von Heijne G. Chloro P, a neural network-based method for predicting chloroplast transit peptides and their cleavage sites[J]. Protein Science, 1999, 8(5):978-984.
[23]Bendsten JD, Nielsen H, Heijne GV, et al. Prediction of signal peptides:SignalP3. 0[J]. J Mol Biol, 2004, 340:783-795.
[24]Gasteiger E, Hoogland C, Gattiker A, et al. Protein identification and analysis tools on the ExPAsy Aerver. In:John M, Walker ed. The Proteomics Protocols Handbook[M]. New Jersey:Humana Press, 2005:571-607.
[25]Higgins D, Thompson J, Gibson T, et al. CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gapenalties and weight matrix choice[J]. Nucleic Acides Res, 1999, 22:4673-4680.
[26]Guex N, Peitsch MC. SWISS-MODEL and the Swiss-PdbViewer: An environment for comparative protein modeling[J]. Electrophoresis, 1997, 18:2714-2723.
[27]Geourjon C, Deleage G. SOPMA:Significant improvement in protein secondary structure prediction by consensus prediction from multiple alinments[J]. Comput Appl Biosci, 1995, 11(6):681-684.
[28]Stephan Z, Julia D, Rong W, et al. Whole-exome sequencing links a variantin DHDDS to retinitis pigmentosa[J]. The American Journal of Human Genetics, 2011, 88:201-206.
[29]李嵘, 王喆之. 植物萜类合成酶3-羟基-3-甲基戊二酰辅酶A还原酶的生物信息学分析[J]. 广西植物, 2006, 26(5):464-473.
[30]崔大方. 植物分类学[M]. 第3版. 北京:中国农业出版社,2010:85-255.
[31]Schroeder JI, Allen GJ, Hugouvieux V, et al. Guard cell signal transduction[J]. Annual Review of Plant Biology, 2001, 52(1):627-658.
[32]Yukimune Y, Tabata H, Higashi Y. Methyl jasmonate-induced overproduction of paclitaxel and baccatin III in taxus cell suspension cultures[J]. Nat Biotechnol, 1996, 14(9):1129-1132.
[33]朱颖, 王丽丽, 柴芸彬, 龚一富. 茉莉酸甲酯对杜氏盐藻β-胡萝卜素含量的影响[J]. 宁波大学学报:理工版, 2010, 1:13-17.
[34]Choi D, Bostock RM, Avdiushko S, et al. Lipid-derived signals that discriminate wound and pathogen responsive isopernoid pathways in plants:Methyl jasmonate and the fungal elicitor arachidonic acid induce different 3-hydroxy-3-methylglutaryl-coenzyme A reductase genes and antimicrobial isoprenoids in Solanum tuberosum L. [J]. Proc Natl Acad Sci, 1994, 91(6):2329-2333.
(责任编辑 马鑫)
Bioinformatics Analysis and Expression Study of DHDDS of Dunaliella viridis
Pan Yifang Gong Yifu Yu Kai Zhu Shuaiqi Wang Heyu
(School of Marine Science,Ningbo University,Ningbo 315211)
Dunaliella viridis is a unicellular green alga producing valuable secondary metabolite carotenoids, and dehydrodolichyl diphosphate synthase(DHDDS)is an enzyme in the synthesis pathway of carotenoids. It was to investigate the correlation between expression of DHDDS gene in D. viridis treated with different concentrations of MeJA and the content of carotenoids. Total RNA was extracted from D. viridis and the full length of DHDDS was acquired by de-novo transcriptome sequencing, then the sequence was analyzed by bioinformatics website and software to predict the protein structure. Transcriptional levels of D. viridis treated with different concentrations of MeJA were examined by Realtime quantitative PCR(RT-qPCR). The total length of gene DHDDS was 2 211 bp, containing a 1 740 bp open reading frame(ORF)that encoded 579 amino acids. Theoretical isoelectric point of DHDDS protein was 7.63. Relative molecular mass was 62 472.7 Da. Bioinformatics prediction revealed that there was no signal peptide and transmembrane domain in DHDDS protein, and it was located in cytoplasmic matrix. Sequence alignment showed that DHDDS of D. viridis shared the highest homology of 59% with that of Chlorella variabilis. The RT-qPCR showed that the expression of DHDDS gene in D. viridis treated by 100 mol/L of MeJA was the highest with significant difference:at this concentration,the content of carotenoids was higher than other groups. There was certain correlation between the expression of DHDDS gene and the content of carotenoids. Conclusively, the DHDDS of D. viridis is an enzyme located in cytoplasmic matrix, relating to synthesis pathway of carotenoids. Within certain concentration, MeJA in lower concentration shows promoting effect on the expression, and inhibiting in higher concentration, there is a correlation between the expression and the content of carotenoids.
Dunaliella viridis;DHDDS;carotenoids contents;MeJA
10.13560/j.cnki.biotech.bull.1985.2015.07.015
2014-12-02
浙江省科技厅重点科技创新团队项目(2012R10029-07,2010R50029),宁波市科技项目(2013C10018)
潘益芳,女,硕士研究生,研究方向:植物分子生物学;E-mail:pannyfang@126.com
龚一富,男,副教授,研究方向:植物分子生物学;E-mail:gongyifu@163.com
