巴西蕉诱导的香蕉枯萎病菌1号和4号小种致病相关基因表达分析
2015-10-21肖义炜李春强李文彬孙建波曾会才彭明
肖义炜 李春强 李文彬 孙建波 曾会才 彭明

摘 要 目前对于香蕉枯萎病菌的致病分子机制尚不十分清楚。为了阐明枯萎病菌的致病分子机制,从香蕉枯萎病菌1号(Foc1)和4号小种(Foc4)的比较蛋白质组学分析发现的表达量差异较大的蛋白中,选取了9个致病相关蛋白或潜在的致病基因,利用荧光定量PCR方法进行基因表达谱分析。结果显示:与对照相比,巴西蕉处理的Foc4中,上调表达的基因有:酰胺转移酶基因(amidotransferase)、线粒体过氧化物还原酶基因(mitochondrial peroxiredoxin)、几丁质酶基因(chitinnase 1)、羧肽酶基因(carboxypeptidase cpds),差异表达不明显的有腺苷激酶基因(adenosine kinase)、NADP-依赖型甘油脱氢酶基因(NADP-dependent glycerol dehydrogenase)、磷酸甘油酸激酶基因(phosphoglycerate kinase)、谷胱甘肽还原酶基因(glutathione reductase)、酰胺酶基因(amidase family protein)。Foc1中,与对照相比,上调表达的基因有:腺苷激酶基因、NADP-依赖型甘油脱氢酶基因,下调表达的基因有:酰胺转移酶基因、羧肽酶基因。综合分析显示,酰胺转移酶、过氧化物酶、几丁质酶、羧肽酶基因可能在Foc4的致病作用中起作用。
关键词 香蕉枯萎病;尖孢镰刀菌古巴专化型;基因表达谱;致病相关基因
中图分类号 S668.1 文献识别码 A
Abstract The understanding of Fusarium oxysporum f. sp. cubense molecular pathogenic mechanism is still not clear. Nine pathogenesis related protein genes or potential virulence genes were selected from lots of differences expression proteins discovered in comparative proteomic analysis of F. oxysporum f. sp. cubense race 1 and race 4 and to analyze the gene expression profile by real time PCR. The results indicated that amidotransferase,mithchondrial peroxiredoxin,chitinnase 1,carboxypeptidase cpds were up-regulated compared with the controls in Foc4 group induced by Musa AAA Cavendish cv Brazil. Adenosine,NADP-dependent glycerol dehydrogenase,phosphoglycerate kinase,glutathione reductase and amidase family protein expressions were barely noticeable difference in two races. In Foc1,adenosine and NADP-dependent glycerol dehydrogenase were up-regulated in induced group compared with the control group. Amidotransferase and carboxypeptidase cpds were down-regulated. Those suggested that amidotransferase,mithchondrial peroxiredoxin,chitinnase 1 and carboxypeptidase cpds might play an important role in the pathogenesis of Foc4.
Key words Banana wilt; Fusarium oxysporum f. sp. cubense; Gene expression profile; Pathogenicity-related gene
doi 10.3969/j.issn.1000-2561.2015.11.009
香蕉(Musa spp.)生長在热带、亚热带,是全世界最主要的水果之一;也是发展中国家最重要的四大作物之一[1]。尖孢镰刀菌古巴专化型(Fusarium oxysporum f. sp. cubense,Foc)引起的香蕉枯萎病,也称巴拿马病,是目前防治难度大、蔓延快的土传维管束病害,严重影响世界各香蕉生产国香蕉产业的发展。香蕉枯萎病病原菌有4个生理小种,其中1号和4号小种分布最广,危害性最大[2]。20世纪40年代至60年代,1号小种引起的香蕉枯萎病的爆发,导致香蕉在世界范围毁灭性破坏。后来发现起源于中国南部的“Cavendish”亚群(如巴西蕉)抗1号小种,于是取代“Gros Michel”成为栽培最广的品种[3]。然而,该亚群对4号小种是易感的。由于缺乏高抗4号小种且品质优良的香蕉栽培品种,所以4号小种引起的香蕉枯萎病迅速蔓延到各香蕉生产区,并在澳洲和许多亚洲国家造成重大损失,已成为香蕉产业最严重的问题。
香蕉枯萎病是土传维管束病害,由于病原菌是在维管束中侵染为害,香蕉假茎粗大,化学药剂很难在维管束中达到有效的抑制浓度,因此,至今没有十分有效的化学药剂用于该病的防治。由于具有生产价值的香蕉品种都是三倍体,杂交育种困难,香蕉抗病育种大都依靠体细胞变异和田间芽变选育,除培育出一些耐病品种,如台湾的宝岛蕉(AAA)、农科一号(AAA)等外,至今尚未培育出对Foc4高抗的品种,也未找到十分有效的防治措施,只能在栽培上采取一些综合防控措施来预防和降低发病率,香蕉枯萎病的防控仍然是一个难题。
目前对香蕉枯萎病病原菌生物学特性比较清楚[4],检测体系基本建立[5],遗传变异方面也有可喜的进展[4,6]。在香蕉抗病方面,香蕉根系中的通气组织[7]、维管束结构、细胞大小及排列的紧密、导管的大小与多少等因素与品种的抗病性相关[8]。还发现有些香蕉被枯萎病菌侵染后,会被诱导产生凝胶[9]或某些化合物[10],从而阻止病原菌的进一步扩展。但迄今,对病原菌致病和香蕉抗/感病的分子机制尚不清楚。在香蕉中克隆到的抗病相关基因还很少,只分离到一些香蕉RGA(Resistance gene analog sequence)片段[11-12]和SSH克隆[13],主要是一些细胞壁强化基因,但在抗枯萎病中的作用尚不清楚。在病原菌致病机制方面,目前报道的有:与胞外酶和过氧化氢酶活性有关的Foatf1基因,该基因缺失导致毒力降低[14];pgc2基因,其融合表达的PGC2蛋白能使香蕉组织变软和坏死[15];4号小种中克隆到的foABC1基因[16-17],对植保素或抗毒素类物质具有忍耐性。而在香蕉与枯萎病菌互作方面则尚未见有报道。
为了阐明枯萎病菌的致病分子机制,本项研究前期试验中已对巴西蕉处理的Foc1和Foc4进行了比较蛋白质组学分析,共鉴定出99个差异蛋白;在Foc4中鉴定出46个高丰度或特异表达的蛋白(尚未发表)。本文从这些差异蛋白中选取了9个基因,对这些基因进行基因表达谱分析,为进一步揭示香蕉枯萎病菌的致病机制奠定基础。
1 材料与方法
1.1 材料
由于Foc1与Foc4存在小种致病性差异,Foc1不侵染巴西蕉(Musa AAA Cavendish subgroup cv. Brazil)而Foc4侵染,因此本研究采用巴西蕉分别处理Foc1与Foc4。巴西蕉种植在中国热带农业科学院生物技术所试验大棚,取吸芽进行组织培养获得幼苗(袋装苗),转接到大培养瓶培养50 d后用于试验。香蕉枯萎病菌1号小种(Fusarium oxysporum f. sp. cubense race 1,Foc1)N2菌株和4号小种(Fusarium oxysporum f. sp. cubense race 4,Foc4)B2菌株由黄俊生研究员惠赠,均分离自海南岛,并进行了基因组测序[18]。
1.2 方法
1.2.1 香蕉处理及采样 将香蕉枯萎病菌1号小种(Foc1)和4号小种(Foc4)分别接种到三角瓶中,同时放入28 ℃摇床,180 r/min培养48 h。此时菌液浓度约106 cells/mL,用PDB培養液调节菌液浓度使Foc1和Foc4浓度一致。而后分别用1 000 mL三角瓶分装,每瓶500 mL,分装时摇匀使每瓶浓度一致。取苗龄相同、长势一致(4~5片叶,约15 cm高)、健康无染病的巴西蕉组培苗,用无菌水将根部清洗干净;然后将香蕉根部浸入分装好的菌液中进行处理,每瓶放1株香蕉苗,对照不用香蕉处理,3个重复。处理8 h后将香蕉苗取出,然后放入28 ℃培养箱继续培养。采样:每个样品收集菌液100 mL,每24 h取样1次,连续4 d,共0、24、48、72 h和96 h 5个时间点。
1.2.2 RNA提取 将采集到的同一批次相同处理的三个重复的样品混在一起,用CTAB法分别提取Foc1和Foc4的总RNA,具体提取方法见文献[19]。
1.2.3 RT合成第一条链cDNA 按照TIANGEN公司反转录试剂盒操作步骤合成cDNA第一链。
1.2.4 荧光定量PCR检测 以蛋白质学数据库所得序列设计荧光定量PCR基因引物(表1),产物大小为150 bp左右,设计好的引物由上海生工生物技术有限公司合成。仪器为AB公司的StepOnePlus Real-Time PCR系统,操作步骤按照Takara公司荧光定量PCR试剂盒进行。
2 结果与分析
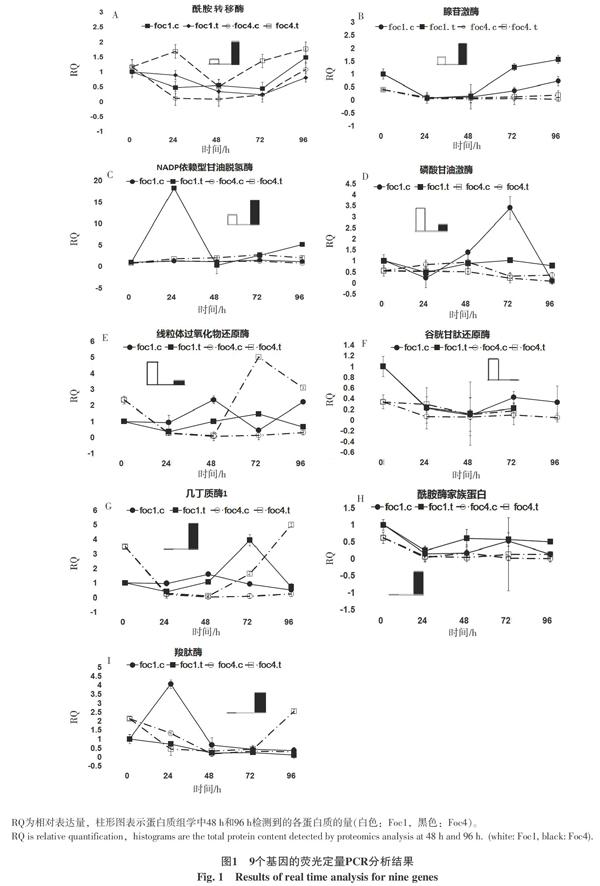
图1是9个基因的荧光定量PCR分析结果。从图1-A可以看出,香蕉处理后的Foc4中,各时间点酰胺转移酶的表达量较对照高很多,而在Foc1中该基因的表达量与对照相比差别不大。而且,香蕉处理组中,各时间点Foc4中酰胺转移酶的表达量比Foc1中均高出许多,在前期蛋白质组学分析中检测到的蛋白质的量,Foc4也比Foc1高(图1-A柱状图)。
在巴西蕉处理及未处理的Foc4中,腺苷激酶基因表达水平在整个试验阶段一直处于低水平状态,且在处理与对照中其表达量基本相同;在对巴西蕉非致病的Foc1中,试验前2 d处理与对照的腺苷激酶基因表达量基本一致;72 h后,腺苷激酶基因的表达水平有所增加,但处理较对照增加幅度更大(图1-B)。48 h后Foc4中的基因表达量比Foc1低许多,而在蛋白质组学分析中检测到的蛋白质的量,Foc4是Foc1的2倍,蛋白质与基因表达之间存在差异,其原因可能是基因转录的mRNA水平并不能完全代表其相应蛋白质的丰度,因为蛋白质水平的表达不仅取决于基因转录的速率,也与mRNA的出核、细胞浆定位、转录物的稳定性、mRNA翻译调节及蛋白质的降解密切相关。对蛋白质合成的调控不仅局限于转录过程,在蛋白质合成的其他阶段也存在调控机制[20-21]。研究表明,mRNA和蛋白质无论在单个基因还是在整个细胞系中相关系数一般小于0.5[21],在酵母中某些基因mRNA的丰度和相应翻译的蛋白质水平可相差30倍之多,而且在低丰度蛋白质中更加明显[22]。
在香蕉处理的Foc1中,NADP依赖型甘油脱氢酶在24 h表达量快速升高近20倍,而后又迅速下降到几乎为0,72 h后又重新开始上升,而对照一直处于低水平。与对照相比,香蕉处理的Foc4中,该基因的表达水平在各时间点均有所升高(图1-C)。表明在1号和4号小种中,巴西蕉的诱导使NADP依赖型甘油脱氢酶基因的表达量增高。在48 h和96 h时间点该基因的表达总量Foc4与Foc1相当,蛋白质组学分析中检测到的蛋白质量,Foc4较Foc1多。
Foc4中,香蕉处理组与对照组的磷酸甘油激酶基因表达无明显差异且各时间点的变化不大。Foc1对照组表达量波动较大,处理组该基因的表达量基本不变(图1-D)。该基因表达量和蛋白质组学中检测到的蛋白质量均为Foc1较Foc4高。
从图1-E可以看出,在Foc4中,48 h前,线粒体过氧化物还原酶基因处理组和对照组表达量几乎相同但极低;72 h后,处理组表达量升高3~5倍,而对照组却几乎为0或表达量极低。在Foc1中,处理与对照组相比较并未见明显改变且各时间点的表达水平变化不大。蛋白质组学中48 h和96 h检测到的蛋白质量Foc1大于Foc4,而基因表达量48 h时 Foc1大于Foc4,96 h 时Foc4大于Foc1;可能的原因是蛋白质翻译较基因表达滞后,96 h时表达基因还没有完全被翻译成过氧化物還原酶。
在Foc1和Foc4中,处理与对照的谷胱甘肽还原酶基因和酰胺酶家族蛋白基因表达量差别不大,且前后几天的变化也不明显(图1-F、H)。24 h后,Foc1与Foc4中的表达量差异不明显,虽然蛋白质组学中检测到的蛋白质量差异较大,推测这2个基因均未参与到病原菌的致病过程之中。
与对照相比,48 h后香蕉处理的Foc1和 Foc4中,几丁质酶基因表达量都有升高。但最后一天(96 h),Foc1中该基因表达量回复到原点,而在Foc4中其表达量却继续升高至5倍以上,Foc4对照中表达量接近0(图1-G)。前期蛋白质组学分析中,Foc4的几丁质酶蛋白量也较Foc1中高很多。推测几丁质酶在Foc4侵染过程中起作用。
从图1-I可以看出,24 h时,Foc1中羧肽酶基因在对照组中的表达量高于处理组,之后处理组与对照组该基因均低水平表达并基本无差别。而在Foc4中,72 h后羧肽酶基因在处理组中的表达量远高于对照组。48 h后羧肽酶基因在Foc4中的表达量较Foc4中高,蛋白质组学分析中检测到的蛋白量Foc4中很高,而Foc1中几乎检测不到。
3 讨论与结论
酰胺转移酶是生物体内嘌呤产物合成途径的关键酶[23],在对数生长期的细胞中稳定表达,在对数生长末期合成终止[24]。RIG-I(retinoic acid-induced gene I,维甲酸诱导基因I)对先天性免疫至关重要,但是某些病原菌,如疱疹病毒(herpesvirus),能够通过酰胺转移酶介导的脱酰氨基作用劫持活化的 RIG-I 避免抗病毒细胞因子产生[25]。本研究中,酰胺转移酶基因在香蕉处理后期的Foc4中有明显增长,其原因可能是Foc4受到植物寄主巴西蕉的诱导后该酶合成增加,以抵御寄主免疫产生的抗病因子;而在Foc1中处理与对照的表达量差别不大,可能是巴西蕉抑制了Foc1中该基因的表达,而Foc4则克服了这种抑制;这些结果显示该酶很可能参与了 Foc4对巴西蕉的致病过程,但具体机制有待进一步研究。
腺苷激酶是一个嘌呤补救途径中存在的一种酶,催化嘌呤的磷酸化作用,也在甲基化作用中扮演重要角色[26]。虽然有报道称该酶与黄单胞菌(Xanthomonas campestris pv. campestris)致病性相关[27],但本研究中对巴西蕉致病的Foc4中腺苷激酶的表达量处理与对照之间却无明显变化。因此推测腺苷激酶与香蕉枯萎病菌的致病性不相关。
NADPH主要参与合成代谢,在甘油代谢过程主要参与细胞的高渗透压生理调节和胞内氧化还原平衡调节[28]。营养状况也影响真菌侵染,基因突变后,变成营养缺陷型从而失去致病能力。Sweigard等[29]发现灰梨孢菌咪唑甘油磷酸脱氢酶基因pth3突变后,使组氨酸无法合成而成为非致病性突变株。本研究中NADP依赖型甘油脱氢酶在香蕉处理后期Foc4和Foc1中表达量有增加趋势,表明枯萎病菌在侵染时的营养需求增大。
磷酸甘油酸激酶主要参与糖降解途径,目前尚未发现致病相关性的报道,本实验结果也未体现其与致病性有关。研究表明,过氧化物还原酶在过氧化代谢中起着至关重要的作用[30]。过氧化物还原酶能清除自由基活性氧(ROS),在细胞毒性过氧化物的解毒作用中起催化作用,还能够作为分子伴侣在面临氧化应激时参与细胞的损伤修复[31]。本研究中线粒体过氧化物还原酶在处理后期的Foc4中表达量明显升高,可能的原因是受到侵染的巴西蕉生成了较多的自由基,以及病原菌体内的新陈代谢增加导致游离自由基较多,使病原菌产生相关清除自由基的应激反应,降解这些氧自由基,因此,推测该基因可能与Foc4的致病性有相关。但具体情况有待进一步验证。
几丁质是甲壳动物、昆虫、线虫、真菌等物种外壳的主要成分,起营养和保护等作用[32]。几丁质酶主要用于降解寄主细胞壁,或者在致病过程中降解病原菌自身的几丁质从而利于侵染,研究表明寡孢根霉菌(Rhizopus oligosporus)的chi3几丁质酶基因在菌丝的生长过程中表达,有利于菌丝顶端细胞松弛形成膨压并使末端伸长[33]。缺失几丁质酶的病毒对寄主的致死时间明显推迟[34]。本实验中,处理后期的Foc4中,几丁质酶表达量快速上升,可能是由于香蕉处理后菌群内的几丁质升高导致菌体本身需要几丁质酶的降解,或者因为香蕉的诱导使几丁质酶表达增加使菌丝顶端细胞松弛有利于侵染。但具体情况还有待对该基因进行敲除、互补和功能验证等进一步研究。
谷胱甘肽还原酶虽然是细胞的重要抗氧化剂,但鲜有致病相关性的报道,本实验中也未发现其与致病性相关。酰胺酶是一种在生物体中无所不在的酶,这些酶通常涉及多种代谢[35],主要用于水解冬酰胺,并未发现在病原菌致病方面报道,本实验也未发现其与枯萎病菌的致病性相关。
羧肽酶是催化水解多肽链含羧基末端氨基酸的酶。病原真菌能够分泌羧肽酶等分泌蛋白,作为潜在的致病因子对某些病原菌的毒力很重要,可为病原真菌专一性提供碳源[36]。研究表明,L,D-羧肽酶对单增李斯特菌(Listeria monocytogenes)的毒力起作用[37]。此外,在米曲霉(Aspergillus oryzae)和黑曲霉(Aspergillus niger)中也分离到几个羧肽酶[38]。本研究中,72 h后Foc4中处理组羧肽酶基因的表达量远远高于对照组,而且在Foc4中蛋白质组学检测到的羧肽酶蛋白量也很高,而在Foc1中几乎检测不到。推测其可能作为致病因子参与了Foc4的致病过程,或者在降解植物抗病反应所释放的小分子肽类中起作用,具体情况有待进一步研究。
综上所述,本研究在对Foc1 和Foc4的比较蛋白质组学分析的基础上,选取9个差异蛋白进行基因表达分析。结果显示,所讨论的9个基因中,酰胺转移酶、过氧化物酶、几丁质酶、羧肽酶基因可能在Foc4的致病作用中起作用。
参考文献
[1] Heslop-Harrison J S, Schwarzacher T. Domestication, genomics and the future for banana[J]. Ann Bot, 2007, 100(5): 1 073-1 084.
[2] Hwang S C, Ko W H. Cavendish banana cultivars resistant to fusarium wilt acquired through somaclonal variation in Taiwan[J]. Plant Dis, 2004, 88(6): 580-588.
[3] Price N S. The origin and development of banana and plantain cultivation. In Bananas and Plantains. Edited by Gowen S[C]. London: Chapman and Hall, 1995: 1-13.
[4] Koenig R L, Ploetz R C, Kistler H C. Fusarium oxysporum f.sp. cubense consists of a small number of divergent and globally distributed clonal lineages[J]. Phytopthology, 1997, 87: 915- 923.
[5] 刘景梅, 陈 霞, 王壁生, 等. 香蕉枯萎病菌生理小种鉴定及其SCAR标记[J]. 植物病理学报, 2006, 36(1): 28-34.
[6] Groenewald S. Application of high-throughput AFLP analysis in assessing genetic diversity of Fusarium oxysporum f. sp. cubense[M]. CLAUDINE Pieq, ANNE Vezina, Eds. 2th Intenlational symposium on Fusarium wilt on banana. Brasilia: Salvador de Bahla Brazil, 2003.
[7] Aguilar E A, Turner D W, SivasithamParam K. Proposed mechanisms on how Cavendish bananas are predisposed to Fusarium wilt during hypoxia[J]. Infomusa, 2000, 9(2): 9-13.
[8] 胡玉林, 謝江辉, 江新华, 等. 威廉斯香蕉8818及其抗枯萎病突变体的细胞学与组织学研究[J]. 果树学报, 2008, 25(6): 877-880.
[9] Beckman C H, Mace M E, Halmos S, et al. Physical barrers associated with resistance in Fusarium oxysporum f. sp. cubense wilt of bananas[J]. Phytopathology, 1961, 51: 507-515.
[10] Kuc J. Increasing crop productivity and value by increasing disease resistance through non-genetic techniques[C]. Proceedings of a Symposium on Forest Potentials: Productivity and Value(August 20-24, 1985), Weyerhaeuser Co, Tacoma, Washington, 1985: 147-190p.
[11] 孟祥春, 徐金刚, 黄秉智, 等. 香蕉抗病基因类似序列(RGA)的克隆与分析[J]. 分子植物育种, 2007, 5(6S): 57-60.
[12] 徐金刚, 向 旭, 蔡礼鸿, 等. 孟祥春.香蕉抗病种质RLK一RGAS的克隆及序列差异分析[J]. 农业科技通讯, 2008(8): 80-84.
[13] Van den Berg N, Berger D, Hein I, et al. Tolerance in banana to Fusarium wilt is associated with early up-regulation of cell wall-strengthening genes in the roots[J]. Molecular Plant Pathology, 2007, 8: 333-341.
[14] Xingzhu Qi, Lijia Guo, Laying Yanga, et al. Foatf1, a bZIP transcription factor of Fusarium oxysporum f. sp. cubense, is involved in pathogenesis by regulating the oxidative stress responses of Cavendish banana(Musa spp.)[J]. Physiological & Molecular Plant Pathology, 2013, 84(5): 76-85..
[15] Dong Z Y, Wang Z H. Isolation and characterization of anexopolygalacturonase from Fusarium oxysporum f. sp. cubenserace race 1 and race 4[J]. BMC Biochemistry, 2011, 12(51): 1-9.
[16] Fravel D R, Moravec B C, Jones R W, et al. Characterization of two ABC transporters from biocontrol and phytopathogenic Fusarium oxysporum[J]. Physiological and Molecular Plant Patnology, 2008, 73: 2-8.
[17]李敏慧, 庄楚雄, 姜子德. 香蕉枯萎病菌4号小种致病相关基因foABC1的分离[J]. 菌物研究, 2006, 4(3): 94-95.
[18] Guo L J, Han L J, Yang L Y, et al. Genome and Transcriptome Analysis of the Fungal Pathogen Fusarium oxysporum f. sp. cubense Causing Banana Vascular Wilt Disease[J]. PLoS One, 2014, 9(4): e95543.
[19] Li C Q, Shao J F, Wang Y J, et al. Analysis of banana transcriptome and global gene expression profiles in banana roots in response to infection by Race 1 and Tropical Race 4 of Fusarium oxysporum f. sp. Cubense[J]. BMC genomics, 2013, 14: 851.
[20] Garcia-Sanz J A, Mikulits W, Livingstone A, et al. Translational control: a genera mechanism for gene regulation during T cell activation[J]. FASEB J, 1998, 12(3): 299-306.
[21] Day D A, Tuite M F. Post-transcriptiona regulatory mechanisms in eukaryotes: an overview[J]. J Endocrinol, 1998, 157(3): 361-371.
[22] Steven P. Gygi, Yvan Rochon, B. Robert Franza, et al. Correlation between protein and mRNA abundance in yeast[J]. Mol Cell Biol, 1999, 19: 1 720-1 730.
[23] Smith J L, Zaluzac E J, Wery JP, et al. structure of the allosteric regulatory enzyme of purine biosynthesis[J]. science, 1994, 264(5 164): 1 427-1 433.
[24] Ruppen M E, Switzer R L. Degradation of Bacillus subtilis glutamine phosphoribosylpyrophosphate amidotransferase in vivo[J]. J Biol Chem, 1983, 258(5): 2 843-2 851.
[25] He S P, Zhao J, Song S S, et al. Viral Pseudo-Enzymes Activate RIG-I via Deamidation to Evade Cytokine Production[J]. Molecular Cell, 2015, 58: 1-13.
[26] Kredich N M, Martin D V Jr. Role of S-adenosylhomocysteine in adenosinemediated toxicity in cultured mouse T lymphoma cells[J]. Cell, 1977, 12: 931-938.
[27] 唐永勤. 野油菜黃单胞菌腺苷激酶基因的鉴定及功能分析[D], 南宁: 广西大学硕士论文, 2009.
[28] Hohmann S. Osmotic stress signaling and osmoadapatation in yeasts[J]. Microbiol Mol Biol Rev, 2002, 66(2): 300-372.
[29] Sweigard J A, Carrolla M, Farrall, et al. Magnaporthe grisea pathogenicity genes obtained through hinsertional mutagenesis[J]. Mol Plant-Microbe Interact, 1998, 11: 404-412.
[30] Hudson A L, Sotirchos I M, Davey M W. The activity and hydrogen peroxide sensitivity of the peroxiredoxins from the parasitic nematode Haemonchus contortus[J]. Mol Biochem Parasitol, 2011, 176: 17-24.
[31] Budanov A V, Sablina A A, Feinstein E, et al. Regeneration of peroxiredoxins by p53-regulated sestrins, homologs of bacterial AhpD[J]. Science, 2004, 304(5 670): 596-600
[32] Leake J R, Read D J. Chitin as a nitrogen source form ycorrhizal fungi[J]. Mycological Res, 1990, 94: 993-995
[33] Takaya N, Yamazaki D, Horiuchi H, et al. lntracellular chitinase gene from Rhizopus oligosporus: molecular cloning and characterization[J]. Microbiology, 1998, 144: 2 647-2 654.
[34] Egide Ishimwe, Jeffrey J, Hodgson, A. Lorena Passarelli. Expression of the Cydia pomonella granulovirus matrix metalloprotease enhances Autographa californica multiple nucleopolyhedrovirus virulence and can partially substitute for viral cathepsin[J]. Virology, 2015, 481: 166-178.
[35] Valina A L, Mazumder-Shivakumar D, Bruice T C. Probing the Ser-Ser-Lys catalytic triad mechanism of peptide amidase: computational studies of the ground state, transition state, and intermediate[J]. Biochemistry, 2004, 43 (50): 15 657-15 672.
[36] Michel Monod, Sabrina Capoccia, Barbara Le chenne, et al. Secreted proteases from pathogenic fungi[J]. Int J Med Microbiol, 2002, 292: 405-419.
[37] Iurov D S, Varfolomeev A F, Tukhvatulin A I, et al. Contribution of L, D-carboxypeptidases in virulence of facultative intracellular pathogenic bacteria Listeria monocytogenes[J]. Zh Mikrobiol Epidemiol Immunobiol, 2012, (4): 15-20.
[38] Nakadai T, Nasuno S, Iguchi N. Purification and properties of acid carboxypeptidase IV from Aspergillus oryzae[J]. Agric Biol Chem, 1973, 37: 1 237-1 251.
