拟南芥转录终止因子PDE191的生物信息学分析
2015-10-09熊伟等
熊伟等


摘要:为了预测和分析拟南芥(Arabidopsis thaliana)转录终止因子PDE191蛋白质的结构与功能,采用生物信息学的方法对PDE191蛋白质进行了系统研究,包括PDE191蛋白质的理化性质、跨膜区和信号肽、亚细胞定位、二级结构、功能域、蛋白质的功能分类预测、多重序列比对与系统发育树构建、三级结构建模。结果表明,拟南芥PDE191蛋白质属于植物mTERF蛋白质家族的成员,其蛋白质相对分子质量为37.89 ku,等电点为9.12,不具有信号肽和跨膜区。该蛋白质定位于细胞叶绿体,N端1-30位氨基酸为前导肽序列。其二级结构主要为α螺旋和无规则卷曲,包含7个mTERF基序,三级结构显示结果与二级结构预测结果相符。蛋白质多重序列比对和聚类分析显示,在玉米、蓖麻、杨树、大豆、葡萄、水稻和高粱等高等植物中存在与拟南芥PDE191蛋白质高度同源性的蛋白质,尤其是与玉米PDE191蛋白质相似性高达99%。
关键词:拟南芥(Arabidopsis thaliana);转录终止因子;色素缺失突变191;生物信息学
中图分类号:Q811.4 文献标识码:A 文章编号:0439-8114(2015)17-4332-06
DOI:10.14088/j.cnki.issn0439-8114.2015.17.059
拟南芥(Arabidopsis thaliana)属于十字花科植物,是一种模式植物,其基因组测序已于2000年全部完成,因为具有同类植物无法比拟的条件,在植物学、细胞生物学和分子生物学等研究领域被广泛应用。首先,拟南芥个体小,其各种特征比较简单;其次,拟南芥基因组比较小,并且为单倍体植物,只有5对染色体,其核基因组只有约125 Mbp;再次,拟南芥生长周期短,一个生活史只需要8周左右;最后,拟南芥每代平均能够收获多达数千粒种子。拟南芥具有高等植物的一般特点,所以拟南芥的研究成果很容易借鉴到其他农作物、经济作物等的应用中去,可以产生客观的经济和社会价值。
线粒体转录终止因子(Mitochondrial transcription termination factor, mTERF) 蛋白质家族是一类具有多功能的蛋白质家族,包含4个亚家族,分别命名为mTERF1、mTERF2、mTERF3和mTERF4。通过PSI-BLAST发现该蛋白质家族成员广泛存在于后生动物和植物中,但目前还没有在真菌中发现同源蛋白质[1-3]。张晓雷[4]于2011年首次报道了一个由于T-DNA插入导致的拟南芥色素缺失突变体pde191,表型为白化及幼苗致死,电镜观察超微结构显示叶绿体发育不正常,但是在加入蔗糖的培养基上培养表现出逐渐变绿的表型,通过等位试验和遗传互补试验,充分证明是由于PDE191(Pigment defective 191) 基因的沉默导致了植物出现白化表型。后续的研究表明,拟南芥PDE191蛋白质含有mTERF基序重复序列,且定位于叶绿体中,PDE191基因突变导致一系列的叶绿体基因无法正常转录终止,特别是rpoA基因及其下游的间隔区序列在突变体中的表达比野生型高20多倍[5]。在叶绿体发育过程中,PEP(质体编码的聚合酶)是一类重要的负责质体基因转录的RNA聚合酶,并发挥重要的质体基因表达调控作用,转录的效率主要由PEP的活性所决定[5]。PEP活性下降是pde191突变体出现白化现象的一个主要因素。研究表明拟南芥PDE191基因通过作用叶绿体rpoA基因的正常转录终止,进而影响叶绿体PEP酶活性和基因表达,并最终影响叶绿体的发育[5]。
目前已经确定拟南芥PDE191基因位于第4号染色体,含有4个外显子和3个内含子,全长cDNA序列为1 402 bp,编码的蛋白质由333个氨基酸组成。在本研究中,利用生物信息学方法和工具对拟南芥PDE191蛋白质序列进行系统的预测和研究,通过美国NCBI数据库中检索到的拟南芥PDE191蛋白质数据和其他植物同源蛋白质数据,分析拟南芥PDE191蛋白质的氨基酸组成、理化性质、二级结构、功能结构域和三级结构等信息,同时对不同植物的PDE191同源蛋白质序列进行多重序列比对并构建系统发育树,以期为今后进一步研究该蛋白质的功能提供生物信息学参考。
1 材料与方法
1.1 材料
用于生物信息学分析的数据资料来源于国际互联网上NCBI核酸和蛋白质数据库中已经注册的不同植物与拟南芥PDE191基因同源的mRNA及其蛋白质序列(表1)。
1.2 方法
拟南芥PDE191蛋白质的理化性质采用Protparam tool软件预测;亲水性/疏水性采用ProtScale tool软件进行预测;氨基酸分值参数选用HpHob./Kyte and Doolittle;跨膜区域使用TMHMM Server 2.0软件进行预测;信号肽采用SignalP 4.1 Server软件预测;蛋白质亚细胞定位分别采用PSORT WWW Server中的iPSORT prediction工具、WoLFPSORT软件和TargetP软件进行分析;蛋白质功能分类采用ProtFun 2.2 Server软件进行预测;二级结构采用PSIPRED Server 3.3软件进行分析;结构功能域采用SMART软件预测;三级结构预测采用SWISS-MODELR软件进行同源建模;多重序列比对采用Clustal W软件进行;系统发育树构建采用MEGA6.05软件进行。各在线分析软件的网址见表2。
2 结果与分析
2.1 拟南芥PDE191蛋白质的理化性质
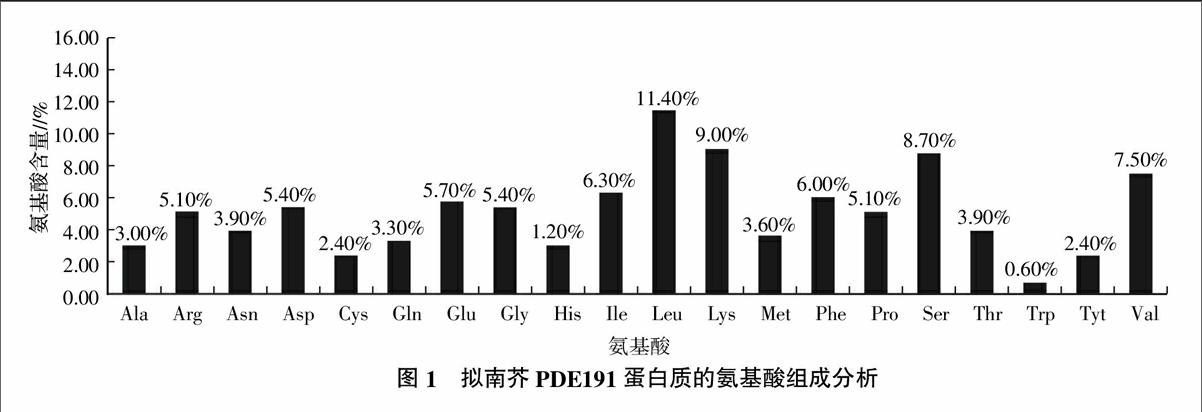
在GenBank注册的拟南芥PDE191基因全长cDNA包括1 002 bp的开放阅读框(Open reading frame,ORF),编码1个由333个氨基酸组成的蛋白质。采用Protparam tool软件预测PDE191蛋白质的理化性质,推测该蛋白质的相对分子质量为37.89 ku,分子式为C1707H2736N448O482S20,等电点为9.12,不稳点参数33.85,根据不稳定参数的数值在40以下是稳定蛋白质的标准,可推定PDE191为稳定蛋白质[6]。软件预测该蛋白质在体外环境下的半衰期为30 h。通过分析该基因编码的氨基酸发现,PDE191蛋白质由20种不同氨基酸组成,Leu、Lys和Ser的含量较多,其中Leu的含量高达11.40%,Trp的相对含量较少,只占0.60%;带正电荷的氨基酸残基(Arg+Lys)总数为47个,带负电荷的氨基酸残基(Asp+Glu)为37个(图1)。疏水性平均系数(Grand average of hydropathicity,GRAVY)为-0.114,预测该蛋白质为亲水性蛋白质。endprint
2.2 拟南芥PDE191蛋白质亲水性/疏水性预测和分析
亲水性/疏水性预测和分析对于进一步预测蛋白质的二级结构和结构功能域具有重要的生物学意义,采用ProtScale tool软件进行亲水性/疏水性预测,结果表明,PDE191蛋白质第154位氨基酸分值最大,为2.622;蛋白质第188位氨基酸分值最小,为-2.122(图2)。整体来看,亲水性氨基酸数量多于疏水性氨基酸,且均匀分布在整个肽链中[7],可推测PDE191是亲水性蛋白质, 与Protparam tool软件预测结果一致。
2.3 拟南芥PDE191蛋白质的跨膜区域与信号肽分析
蛋白质的跨膜区域主要是膜内在蛋白质和细胞膜的膜脂相结合的部位。利用TMHMM Server v2.0在线软件对PDE191蛋白质的跨膜区域进行分析,结果(图3)显示,该蛋白质的跨膜螺旋数量(Number of predicted TMHs)为0,说明PDE191不是跨膜蛋白质。
SignalP是一个信号肽及其剪切位点的预测工具,它采用一个神经网络来区分信号肽和非信号肽,另一个神经网络来识别剪切位点。C值是信号肽酶切位点分值,S值是信号肽分值,Y值是由C值和S值综合得出的剪切位点分值,用于更精确地确定信号肽酶切位点[7]。使用SignalP 4.1 Server在线软件预测平均S值(mean S score)为0.109,依据mean S score>0.5才能判断为分泌蛋白质的标准,推测PDE191蛋白质不具有信号肽,说明它是一种在细胞内发挥生理作用的蛋白质(图4)。
2.4 拟南芥PDE191蛋白质二级结构预测与分析
蛋白质二级结构主要指蛋白质分子中主链骨架原子依赖氢键排列在一维方向上具有周期性的构象,对其进行预测与分析将有助于认识蛋白质的高级结构。使用PSIPRED v3.3软件预测拟南芥PDE191蛋白质的二级结构,结果表明,拟南芥PDE191蛋白质由53.76%的?琢-螺旋(Alpha helix)、1.20%的延伸链(Extended strand)、45.04%的无规则卷曲(Random coil)构成(图5)。可见?琢-螺旋和无规则卷曲是该蛋白质二级结构的主要构成元件,延伸链只出现在2个局部肽链,且没有 ?茁-转角(Beta-turn)出现。
2.5 拟南芥PDE191蛋白质结构功能域的预测
结构功能域是指生物大分子中具有特异结构与独立功能的区域。用SMART在线软件预测PDE191蛋白质的结构功能域,结果表明,该蛋白质4-125、148-272位氨基酸区域为2个内部重复序列(Internal repeat),8-123、112-322位氨基酸区域为2个mTERF蛋白结构域(Pfam),60-91、96-127、132-163、169-201、206-237、275-306、342-374位氨基酸区域为7个mTERF基序重复序列,每个基序由32或33个保守的氨基酸残基组成(图6A)。对这7个基序的序列分析发现,每个基序的第8个氨基酸均为脯氨酸(P),第10、11、15、19、26位氨基酸是亮氨酸(L)或其他疏水性氨基酸,如异亮氨酸(I)、缬氨酸(V)、苯丙氨酸(F),这些结构特征使得PDE191蛋白质可能具有与mTERF同样的结合DNA的性质。
2.6 拟南芥PDE191蛋白质的亚细胞定位和功能分类
分别使用PSORT WWW Server中的WoLFPSORT工具和iPSORT Prediction工具对拟南芥PDE191蛋白质进行细胞定位, 两个不同软件的分析结果均表明PDE191蛋白质定位于拟南芥的叶绿体和线粒体中,蛋白质N端的1-30位氨基酸可能是其前导肽序列。此外,TargetP蛋白质定位分析软件预测拟南芥PDE191蛋白质可能定位于细胞核、线粒体、叶绿体和过氧化物酶体中,但因为该基因突变体为白化突变体,所以推测定位部位在叶绿体中。
采用ProtFun软件对拟南芥PDE191蛋白质进行功能分类,由表3可知,蛋白质功能分类(Functional category)显示该蛋白质可能是转运和结合蛋白质(Transport and binding), 基因本体分类(Gene Ontology category)进一步提示该蛋白质是一种转录调控因子(Transcription factor)。此外,预测结果还显示拟南芥PDE191蛋白质不具有酶活性。
2.7 多重序列比对与系统发育树构建
根据PDE191蛋白质的氨基酸序列在NCBI上进行BLASTP,结果显示在许多物种中存在与拟南芥PDE191同源的蛋白质,进化系统分析显示PDE191蛋白质在双子叶植物中处于一个独立的分支上,表明其在进化中比较保守(图7)。蛋白质多重序列比对结果显示,拟南芥PDE191蛋白质与玉米(Zea mays)的PDE191蛋白质(EU952184.1)相似性为99%,与杨树(Populus trichocarpa)中的一个预测蛋白质(XM_002328250.1)相似性为71%,与蓖麻(Ricinus communis)中的蛋白质(EQ973785.1)相似性为71%,与大豆(glycine max)中的蛋白质(BT095136.1)相似性为65%,与葡萄(Vitis vinifera)中的蛋白质(XM_002280046.1)相似性为66%,与水稻(Oryza sativa)中的蛋白质(NM_001068770.1)相似性为56%,与高粱(Sorghum bicolor)中的蛋白质(XM_002444711.1)相似性为56%(图8)。由此可见,该基因编码的蛋白质序列具有很高的保守性,在各种植物之间都有很高的相似性,尤其和玉米的相似度更是达到了99%。
2.8 拟南芥PDE191蛋白质三级结构预测与分析endprint
采用SWISS-MODEL同源建模的方式得到拟南芥PDE191蛋白质的三维预测模型(图9),经RasMol软件分析显示该蛋白质外形呈椭球状,主要由?琢-螺旋和无规则卷曲构成,与二级结构预测结果基本一致。
3 讨论
随着计算机技术和生物技术的飞速发展,通过计算机模拟的方式对蛋白质进行理化性质、序列结构和功能等方面进行预测的确信度越来越高[8]。在植物中,除了有线粒体之外,还有叶绿体。因为等位突变体的缺乏而引起的转录终止,关于其编码的蛋白质是否是mTERF的研究并不多,且其在叶绿体中是否也存在转录终止功能仍有待研究。拟南芥PDE191基因编码1个线粒体转录终止因子mTERF蛋白质,该家族蛋白质一般有2个独立的DNA结合区和3个亮氨酸拉链结构,以单体的形式作用于DNA上。
通过生物信息学分析发现拟南芥PDE191蛋白质是一个相对分子质量为37.89 ku的亲水性蛋白质,且不具有分泌信号肽的功能。亚细胞定位发现拟南芥PDE191蛋白质定位于叶绿体中,其N端的1~30个氨基酸为前导肽序列,所以它很有可能是作为一个细胞核与细胞质之间相互作用的一个重要蛋白质。通过预测蛋白质的二级结构发现PDE191蛋白质中56.90%的结构是由?琢螺旋和?茁-折叠构成;通过SMART软件预测该蛋白质含有7个mTERF基序,每个基序由大约32或33个保守的氨基酸残基构成。对其蛋白质功能的预测结果显示,PDE191蛋白质不具有酶活性,但在调控叶绿体基因转录过程中发挥一定的生物学功能。
通过对拟南芥PDE191蛋白质与玉米、杨树、蓖麻、大豆、葡萄、水稻、高粱等其他7个不同物种的系统发育树聚类构建分析发现,该蛋白质属于直系同源蛋白质(Orthologous protein),说明在不同物种之间PDE191蛋白质是来源于共同祖先的蛋白质,能够很好地保留其主要序列以及结构,并且具有共同或者相似的生物学功能[9]。虽然该蛋白质在不同物种中具有很好的氨基酸序列保守性,但其在不同物种中的进化关系与物种本身之间的进化关系并不是很一致,推测该蛋白质可能并不是随着生物的进化而进化的,而是在生物中具有某种固有的作用,只是在植物的进化过程中由于意外的原因导致该基因发生突变,从而打乱了其物种之间的进化关系[10]。本研究为今后更进一步研究植物PDE191蛋白质家族的生物学功能及其他物种的直系同源PDE191蛋白质之间的关系奠定了基础。
参考文献:
[1] ROBERTI M, POLOSA P L, BRUNI F, et al. The MTERF family proteins: Mitochondrial transcription regulators and beyond [J]. Biochim Biophys Acta, 2009, 1787(5):303-311.
[2] LINDER T, PARK C B, ASIN-CAYUELA J, et al. A family of putative transcription termination factors shared amongst metazoans and plants [J]. Curr Genet,2005,48(4):265-269.
[3] ROBERTI M, POLOSA P L, BRUNI F, et al. MTERF factors: A multifunction protein family[J]. BioMol Concepts, 2010, 1(2):215-224.
[4] 张晓雷.拟南芥mTERF蛋白PDE191功能的初步研究[D].上海:上海师范大学,2011.
[5] 张 翼.转录终止因子PDE191调控拟南芥rpoA基因的转录终止[D].上海:上海师范大学,2013.
[6] 范 晶,胥成浩,张西玉,等.番茄LeNHX3基因的生物信息学分析[J].湖北农业科学,2009,48(12):2917-2921.
[7] 陶 隽,贾 青,魏星灿,等.猪ACACA基因及其编码蛋白质的生物信息学分析[J].江苏农业科学,2014,42(5):42-45.
[8] 李显航,刘红美.家蝇小热休克蛋白(sHsp20.6)的生物信息学分析[J].生物信息学,2013,11(1):65-71.
[9] 钱叶雄,徐士杰,张亚男,等.玉米精氨酸甲基转移酶蛋白家族生物信息学分析[J].生物技术进展,2014,4(1):22-29.
[10] KLEINE T. Arabidopsis thaliana mTERF proteins: Evolution and functional classification[J]. Front Plant Sci,2012,3:1-16.endprint
