抗生素耐药性的来源与控制对策*
2015-09-26朱永官欧阳纬莹吴楠苏建强乔敏
文/朱永官 欧阳纬莹 吴楠 苏建强 乔敏
1中国科学院城市环境研究所,中国科学院城市环境与健康重点实验室厦门361021
2中国科学院生态环境研究中心,区域与城市生态国家重点实验室北京100085
3天津农学院 天津300384
抗生素耐药性的来源与控制对策*
文/朱永官1,2欧阳纬莹1吴楠3苏建强1乔敏2
1中国科学院城市环境研究所,中国科学院城市环境与健康重点实验室厦门361021
2中国科学院生态环境研究中心,区域与城市生态国家重点实验室北京100085
3天津农学院 天津300384

抗生素除了大量用于人类疾病的治疗外,还作为饲料添加剂被广泛应用于动物养殖业。微生物的抗生素耐药性就是指微生物能够在抗生素存在的情况下生长和繁殖。抗生素耐药性是环境微生物固有的,即所谓的内在抗性,但是人类大量使用抗生素带来的抗生素抗性基因的扩散和传播普遍存在,且已开始威胁到全球人群的健康。微生物对抗生素的抗性主要有3个机制:(1)抗生素的外排;(2)抗生素的降解或修饰;(3)抗生素作用位点的保护。大量研究表明,抗生素的使用和抗生素抗性的蔓延呈现良好的相关性,而且环境微生物的抗性可以通过基因横向转移向人类致病菌扩散,最终可能导致超级细菌的爆发,直接影响人类健康。为了应对全球性的抗生素抗性问题,必须加强:(1)全球抗生素使用和环境排放的监管政策和管理体系;(2)建立快速和透明的抗生素耐药性监测体系,使其涵盖医院、养殖业、污水处理厂等;(3)建立抗生素药物创新基金,通过政府和企业的联合,加快新型药物的研制;同时加强知识产权保护,使新药创制走上可持续之路;(4)加强抗生素耐药性相关的基础与应用研究,包括耐药性发生和传播的生态学机制,消除和缓解耐药性发生和传播的环境技术及其系统解决方案等,包括改进污水处理厂的处理工艺,削减出水中抗性基因和抗性菌的比例;(5)加强抗生素耐药性的科普宣传,提高全社会对耐药性的认知能力,从而在源头上有效控制抗生素在农业和医疗方面的滥用及其环境污染。
城市化,抗生素,耐药性,超级细菌,环境健康
抗生素的发现和大规模生产与使用是人类医学史上的巨大进步,挽救了数以亿计的病人。自1928年弗莱明发现青霉素以来,历史上曾有3次诺贝尔医学或生理学奖颁给了发现抗生素的科学家(图1)。除临床使用外,1950年美国食品与药品管理局(FDA)还首次批准抗生素可作为饲料添加剂,抗生素因此被全面推广应用于动物养殖业,在预防和治疗动物传染性疾病,促进动物生长及提高饲料转化率等方面发挥了重要作用。而几乎在20世纪40年代第一代青霉素开始使用之时,就出现了细菌对其的耐药性,科学家也意识到抗生素的耐药性问题。

图1 人类社会抗生素抗性发展史
事实上,抗生素耐药性是微生物的一种自然进化过程,但是在迄今的70年间,由于抗生素在医疗及养殖领域的大量使用,甚至滥用,这一进化过程被大大加快,导致抗生素耐药性的不断发展,在人类致病菌、动物致病菌、动物肠道传染病原体及人与动物共生菌中都出现了抗生素耐药性,并且由单一耐药性发展到多重耐药性。
近年来,耐药性病原菌特别是多重耐药菌的增多与人类研发新型抗生素进展缓慢间的矛盾日益凸显,有学者惊呼,人类即将进入无药可用的“后抗生素时代”或“耐药时代”。根据英国首相专门任命的一个独立研究委员会的报告指出:如果抗生素耐药性得不到有效控制,至2050年全球每年耐药感染的死亡人数可达1 000万,远远超出癌症所导致的死亡数。从经济角度讲,至2050年抗生素耐药性将造成的全球GDP损失累计达100万亿美元。由于耐药性的蔓延及其健康危害,抗生素已成为全球紧缺的不可再生资源。
我国作为抗生素的生产和消费大国,由于长期不合理的使用及严格监管的缺乏,所导致的抗生素耐药性问题十分严峻,耐药感染性疾病已成为当前临床感染性疾病死亡的主要原因,亟需从国家战略的高度加以重视和采取积极有效的应对策略,一方面避免新的耐药性产生,另一方面阻止已经存在的耐药性传播,从而维护人体健康和生态系统安全。
1 抗生素耐药性
抗生素耐药性是指一些微生物亚群体能够在暴露于一种或多种抗生素的条件下得以生存的现象,其主要机制包括:(1)抗生素失活。通过直接对抗生素的降解或取代活性基团,破坏抗生素的结构,从而使抗生素丧失原本的功能;(2)细胞外排泵。通过特异或通用的抗生素外排泵将抗生素排出细胞外,降低胞内抗生素浓度而表现出抗性;(3)药物靶位点修饰。通过对抗生素靶位点的修饰,使抗生素无法与之结合而表现出抗性。
微生物对抗生素的耐性是自然界固有的,因为抗生素实际上是微生物的次生代谢产物,因此能够合成抗生素的微生物首先应该具有抗性,否则这些微生物就不能持续生长。这种固有的抗生素耐药性,也称作内在抗性(intrinsic resistance),是指存在于环境微生物基因组上的抗性基因的原型、准抗性基因或未表达的抗性基因[1]。这些耐药基因起源于环境微生物,并且在近百万年的时间里进化出不同的功能,如控制产生低浓度的抗生素来抑制竞争者的生长,以及控制微生物的解毒机制,微生物之间的信号传递,新陈代谢等,从而帮助微生物更好地适应环境[2,3]。因此,抗生素耐药性的问题其实是自然和古老的。科学家在北极的冻土中提取到3万年前的古DNA,从中发现了较高多样性的抗生素抗性基因,而且部分抗性蛋白的结构与现代的变体相似,也证实了抗生素耐药性问题是古老的[4]。
虽然一些抗生素抗性微生物和抗性基因很早就存在于自然界,但是抗生素大规模的生产和使用加速了固有抗性微生物和抗性基因的扩散,极大地增加了抗生素耐药性的发生频率[5]。抗生素耐药基因的存在往往与抗生素的使用之间存在良好的相关性[6]。由外源进入并残留在环境中的抗生素对环境微生物的耐药性产生选择压力,携带耐药基因的具有抗性的微生物能存活下来并逐渐成为优势微生物,并不断地将其耐药基因传播给其他微生物。众多研究证实抗生素耐药基因具有较高的移动性,主要是通过基因水平转移(Horizontal gene transfer,HGT)机制,又称基因横向转移(Lateral gene transfer)。即借助基因组中一些可移动遗传因子,如质粒(plasmids)、整合子(integrons)、转座子(transposons)和插入序列(insertion sequences)等,将耐药基因在不同的微生物之间,甚至致病菌和非致病菌之间相互传播(图2)。环境中拥有基因横向转移等内在机制的微生物组成一个巨大的抗性基因储存库[2],并可能将抗生素耐药性转移到人类共生微生物和病原体中[7,8]。医学专家很早就指出,抗生素的广泛使用导致了内源性感染和细菌耐药性的增加。而通过宏基因组学的研究方法,科学家在人类肠道微生物群中也发现了高丰度、高多样性的抗生素耐药基因,也印证了这一观点[9]。
2 人类活动与耐药性

图2 抗生素抗性的分子机制
已有文献和相关统计资料显示,我国是抗生素的生产和消费大国,2007年的一项调查显示,我国抗生素原料生产量约为21万吨,其中有9.7万吨(占年总产量的46.1%)的抗生素用于畜牧养殖业,2009—2010年畜用抗生素的年消耗量均接近10万吨,远超其他国家。全球范围来看,至少50%的抗生素都是用于养殖业,美国年畜用抗生素的消耗量从2002年的9 300吨增至2006年的11 200吨。欧盟实施“限抗令”后,畜用抗生素年消耗量从2002年的5 000吨降至2006年的3 800吨。据预测,我国养殖业抗生素占全球消费总量的比重将从2010年的23%升至2030年的30%。在美国,兽用抗生素甚至是人用的4倍,世界卫生组织的调查表明,当前增加人和动物感染风险的抗生素基本属于同一类。
由于抗生素在医疗以及养殖业中的大量使用,导致环境中出现了大量抗性污染热点区,抗性基因可以通过多种直接或间接的传播途径在其间扩散并最终进入水体和土壤(图3)。其中,城市污水处理厂和集约化养殖场是最为关键和主要的传播途径。主要污染源有3种。
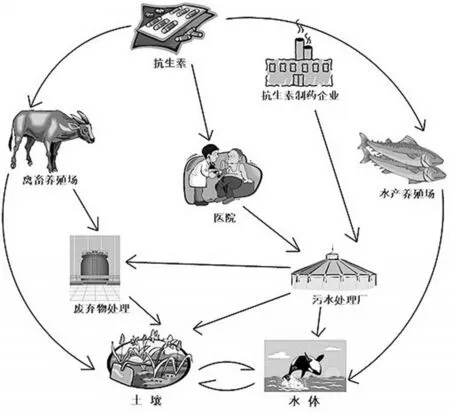
图3 环境中抗性基因传播途径示意图
(1)人类使用抗生素导致医疗废水和生活污水中富含大量耐药菌及其抗性基因,尤其是医疗废水被认为是丰富的整合子基因库[10]。因此城市污水处理厂的集中处理就成为抗生素耐药菌和抗性基因传播的重要源头。研究表明,污水处理厂的进水、出水和污泥中均存在高丰度和极其多样的抗性基因[11],且污水处理厂的出水会引起受纳水体环境中抗性水平的显著升高[12]。此外,城市污水处理厂的中水回用(农田灌溉和城市景观用水等)和污泥施肥亦会导致土壤中抗性基因的富集,从而危及公共健康[13]。
(2)集约化养殖业(包括水产养殖)系统中有机废弃物和污水的排放会直接向环境中释放大量抗性因子。更为严重的是养殖业的环境管理相对粗放,废弃物处置和循环利用技术的相对低下进一步加剧了污染。研究表明,由于集约化养殖业中抗生素和重金属添加剂的滥用可使猪粪中抗性基因(导致微生物产生抗生素耐药性的基因)比背景值富集高达1万倍[14]。Shah等人研究表明,从三文鱼养殖场及其附近海域分离的200株菌中,耐药菌比例高达81%,并从中检测到四环素、磺胺、β-内酰胺类等多种抗性基因[15]。
(3)抗生素制药企业的废水和废渣排放。抗生素制药企业的废弃物中含有高浓度的抗生素残留,长期的选择压力可以导致其成为丰富的抗性基因储库。Liu等人研究了土霉素生产厂的废水与废渣中四环素抗性基因的分布,结果显示,废水中的四环素抗性基因比发酵的废渣中高出2个数量级,且两者均显著高于普通城市污水处理中抗性基因的丰度[16]。
由于耐药菌和抗生素抗性基因污染的广泛性和严重性,如何有效抗击全球范围抗生素耐药性的问题已经得到各国政府和国际机构的高度重视,也被认为是与全球变暖同等重要的全球性挑战。2013年在英国召开的G8峰会把解决全球抗生素耐药性作为首要议题来讨论。英国2014年启动的巨额“经度奖”所要解决的6大难题之一就是抗生素的耐药性。正如世界卫生组织(WHO)指出的“如果今天不采取行动,明天将无药可用”,人类将进入抗生素发明之前的“黑暗时代”。
3 抗击耐药性的研究进展
随着近年来抗生素耐药性严重危害人类健康,各国科学家们开始致力于发展多种策略来抗击耐药性。这些策略主要包括大力挖掘和筛选新型抗生素及抗菌药物、研究新的作用靶点、研发抗生素佐剂等。
3.1 新型抗菌药物及作用靶位
抗菌药物可分为天然结构的抗生素和人工合成的抗菌药物。20世纪40—60年代是微生物学家发现抗生素的“黄金时代”,经过多年的密集筛选,天然结构抗生素的发现进入瓶颈。近年来随着微生物培养技术、宏基因组学、代谢组学以及高通量筛选方法的发展,使得人们再次将目光聚焦于从天然产物中发现新型抗生素[17]。土壤中有约99%的微生物尚未能培养,这使得人们难以获得其产生的活性物质,而通过采用新兴的ichip培养技术,美国与德国科学家从土壤中未培养微生物中筛选出一种新型抗生素Teixobactin,该抗生素可通过与肽聚糖前体Lipid II和磷壁酸的前体Lipid III结合抑制细胞壁的合成,从而杀死多种病原菌,并且细菌很难对该抗生素产生耐药性[18]。宏基因组学技术是人们获得未可培养微生物资源的重要手段之一,采用该技术,Brady小组从type-II polyketide合成基因簇超表达产物中分离纯化到一种新型抗生素Tetarimycin A,对耐甲氧西林金黄色葡萄球菌(MRSA)具有抗菌活性[19]。除了传统的抗生素外,Timothy小组采用CRISPR-Cas技术开发出一类以RNA引导的核酸酶(RNA-guided nucleases,RGNs),RGNs可在DNA水平以特异的DNA序列如抗性基因或细菌毒力因子为目标,通过噬菌体或质粒进入病原菌体内使特异的目标基因失活[20]。
除了开发新型抗菌药物外,科学家们还致力于寻找新的作用靶位蛋白用于开发新型抗菌药物。采用晶体学方法,已鉴定出多种细菌膜蛋白的晶体结构和功能机制,这些膜蛋白包括病原菌福氏志贺氏菌的脂多糖转运(Lpt)蛋白[21]、广泛存在于革兰氏阳性病原菌的能量转运蛋白[22]以及革兰氏阴性菌的分泌独立因子的关键蛋白[23]。这些蛋白晶体结构的解析为针对这类蛋白筛选或设计新的抗菌药提供了理论基础。
3.2 抗生素佐剂
抗生素佐剂是指一类本身并不具有抗菌功能,但可与抗生素协同作用,促进抗生素对于细菌尤其是抗性细菌的杀菌活性的化合物。抗生素佐剂的研制和使用可以大大延长现有抗生素的使用寿命,这类化合物可以分为针对细菌抗性基因和细菌毒力因子的药物[24]。
Wright小组从1 065种现有的非抗生素药物中筛选出69种可与二甲胺四环素协同作用的药物,这些药物可显著降低二甲胺四环素的最小抑制浓度,并在体内和体外实验中均表现出对多重耐药菌株的抗菌活性[25];该小组还筛选出多种抗生素抗性激酶抑制剂,其中黄酮醇槲皮素表现出最强的广谱活性,可抑制由蛋白激酶引起的抗生素耐药性[26]。最近他们还从一株真菌Aspergillus versicolor的代谢产物中筛选出一个可抑制金属β-内酰胺酶(MBLs)活性的化合物Aspergillomarasmine A。该化合物可抑制包括超级细菌的抗性基因NDM-1的耐药活性,从而恢复碳青霉烯抗生素的杀菌活性[27]。此外,人们还发现多种可抑制细菌外排泵的化合物,可降低细菌外排泵的活性、增加抗生素在细菌体内的浓度从而杀死细菌[24]。
与传统抗菌药物不同的是,抗细菌独立因子的药物直接使病原菌特异的毒力因子失活,使其丧失致病能力,病原菌在这种状态下将更容易被抗生素杀死,而且人体的免疫系统和有益微生物将更容易杀死这类病原菌。Curtis等人采用高通量筛选从15万种小分子化合物中筛选出一种化合物LED209,该化合物可与多种重要病原菌毒力因子表达的信号受体QseC结合,从而使病原菌不能表达毒力因子[28]。脂多糖是许多病原菌内毒素的成分,LpxC是其合成的关键酶,针对LpxC的抑制剂可抑制毒性脂多糖的合成,从而降低鲍氏不动杆菌的致病性[29]。
除了上述两方面的研究外,目前关于抗击抗生素抗性的研究还包括[30]:(1)捕食性微生物的研究;(2)抗菌肽的开发;(3)噬菌体;(4)通过基因编码技术发展新的酶;(5)金属离子,如铜和银制剂的开发等。
4 对策与措施
为有效应对全球抗生素耐药性的蔓延及其对人类健康的影响,建议采取如下对策与措施:
(1)分别在全球和国家层面建立跨部门的抗生素耐药性控制委员会(“耐药委”),协调和管理抗生素的生产,人兽使用,废弃物处置及最终向自然环境排放的整个生命周期,该机构的职能包括制定相关政策、规范和监督等。
(2)在“耐药委”的框架下建立快速和透明的抗生素耐药性监测体系,涵盖医院、养殖业、污水处理厂等。
(3)建立抗生素药物创新基金,通过政府和企业的联合,加快新型药物的研制;同时加强知识产权保护,使新药创制走上可持续之路。
(4)加强抗生素耐药性相关的基础与应用研究,包括耐药性发生和传播的生态学机制,消除和缓解耐药性发生和传播的环境技术及其系统解决方案等,包括改进污水处理厂的处理工艺,削减出水中抗性基因和抗性菌的比例。
(5)加强抗生素耐药性的科普宣传,提高全社会对耐药性的认知能力,从而在源头上有效控制抗生素在农业和医疗方面的滥用及其环境污染。
1 Davies J,Davies D.Origins and evolution of antibiotic resistance. Microbiology and Molecular Biology Reviews,2010,74(3):417-433.
2 Martinez J L.Antibiotics and antibiotic resistance genes in natural environments.Science,2008,321(5887):365-367.
3 Baquero F,Alvarez-Ortega C,Martinez J L.Ecology and evolution of antibiotic resistance.Environmental Microbiology Reports,2009,1(6):469-476.
4 D'Costa V M,King C E,Kalan L,et al.Antibiotic resistance is ancient.Nature,2011,477(7365):457-461.
5 Knapp C W,Dolfing J,Ehlert PAI,et al.Evidence of increasing antibiotic resistance gene abundances in archived soils since 1940. Environmental Science&Technology,2010,44(2):580-587.
6 Peak N,Knapp C W,Yang R K,et al.Abundance of six tetracycline resistance genes in wastewater lagoons at cattle feedlots with different antibiotic use strategies.Environmental Microbiology, 2007,9(1):143-151.
7 Smillie C S,Smith M B,Friedman J,et al.Ecology drives a global network of gene exchange connecting the human microbiome.Nature,2011,480(7376):241-244.
8 Forsberg K J,ReyesA,Wang B,et al.The shared antibiotic resistome of soil bacteria and human pathogens.Science,2012, 337(6098):1107-1111.
9 Hu Y,Yang X,Qin J,et al.Metagenome-wide analysis of antibiotic resistance genes in a large cohort of human gut microbiota.Nature Communication,2013,4:2151.DOI:10.1038/ ncomms3151.
10 Stalder T,Barraud O,Jove T,et al.Quantitative and qualitative impact of hospital effluent on dissemination of the integron pool. Isme Journal,2014,8(4):768-777.
11 Yang Y,Li B,Zou S C,et al.Fate of antibiotic resistance genes in sewage treatment plant revealed by metagenomic approach. Water Research,2014,62:97-106.
12 Czekalski N,Diez E G,Burgmann H.Wastewater as a point source of antibiotic-resistance genes in the sediment of a freshwater lake.Isme Journal,2014,8(7):1381-1390.
13 Wang T,Fu G,Pan X,et al.Structure of a bacterial energycoupling factor transporter.Nature,2013,497(7448):272-276.
14 Zhu Y G,Johnson TA,Su J Q,et al.Diverse and abundant antibiotic resistance genes in Chinese swine farms.Proceedings of the NationalAcademy of Sciences of the United States of America,2013,110(9):3435-3440.
15 Shah S QA,Cabello F C,L’Abee-Lund T M,et al.Antimicrobial resistance and antimicrobial resistance genes in marine bacteria from salmon aquaculture and non-aquaculture sites. Environmental Microbiology,2014,16(5):1310-1320.
16 Liu M M,Zhang Y,Yang M,et al.Abundance and distribution of tetracycline resistance genes and mobile elements in an oxytetracycline production wastewater treatment system. Environmental Science&Technology,2012,46(14):7551-7557.
17 HarveyAL,Edrada-Ebel R,Quinn R J.The re-emergence of natural products for drug discovery in the genomics era.Nat Rev Drug Discov,2015,14(2):111-129.
18 Ling L L,Schneider T,PeoplesAJ,et al.Anew antibiotic kills pathogens without detectable resistance.Nature,2015,517(7535):455-459.
19 Taylor P L,Rossi L,Pascale G De,et al.Aforward chemicalscreen identifies antibiotic adjuvants in Escherichia coli. ACS Chem Biol,2012,7(9):1547-1555.
20 Citorik R J,Mimee M,Lu T K.Sequence-specific antimicrobials using efficiently delivered RNA-guided nucleases.Nat Biotech,2014,32(11):1141-1145.
21 Qiao S,Luo Q,Zhao Y,et al.Structural basis for lipopolysaccharide insertion in the bacterial outer membrane.Nature,2014,511(7507):108-111.
22 Wang F H,Qiao M,Su J Q,et al.High throughput profiling of antibiotic resistance genes in urban park soils with reclaimed water irrigation.Environmental Science&Technology,2014,48(16):9079-9085.
23 Gu S,Rehman S,Wang X,et al.Structural and functional insights into the pilotin-secretin complex of the type II secretion system.PLos Pathog,2012,8(2):e1002531.
24 Gill E E,Franco O L,Hancock R E.Antibiotic adjuvants:diverse strategies for controlling drugresistant pathogens.Chem Biol Drug Des,2015,85(1):56-78.
25 Ejim L,Farha MA,Falconer S B,et al.Combinations of antibiotics and nonantibiotic drugs enhance antimicrobial efficacy.Nat Chem Biol,2011,7(6):348-350.
26 Shakya T,Stogios P J,Waglechner N,et al.Asmall molecule discrimination map of the antibiotic resistance kinome.Chem Biol,2011,18(12):1591-1601.
27 KingAM,Reid-Yu SA,Wang W,et al.Aspergillomarasmine A overcomes metallo-[bgr]-lactamase antibiotic resistance.Nature,2014,510(7506):503-506.
28 Curtis M M,Russell R,Moreira C G,et al.QseC inhibitors as an antivirulence approach for gramnegative pathogens.MBio,2014,5(6):e02165.DOI: 10.1128/mBio.02165-14.
29 Lin L,Tan B,Pantapalangkoor P,et al.Inhibition of LpxC protects mice from resistantAcinetobacter baumannii by Modulating Inflammation and Enhancing Phagocytosis.MBio,2012,3(5):e00312-12.DOI: 10.1128/mBio.00312-12
30 Cheng V C C,Wong S C Y,Ho P L,et al.Strategic measures for the control of surging antimicrobial resistance in Hong Kong and mainland of China. Emerging Microbes&Infections,2015,4(2):e8.DOI: 10.1038/emi.2015.8.
朱永官 中科院城市环境所所长、研究员,长期从事环境土壤学和生物地球化学研究,曾获国家自然科学奖二等奖(排名第一)和发展中国家科学院(TWAS)科学奖。2002年获得国家杰出青年基金。先后主持国家自然科学重大项目、中科院知识创新工程重大项目和战略性先导专项B等项目。曾任联合国原子能机构科学顾问,现任国际科学理事会“城市环境与健康”国际计划科学委员会委员,以及多个国内外学术期刊的主编、副主编和编委。研究成果先后得到Science、Nature等刊物的专文评述和报道。E-mail:ygzhu@iue.ac.cn
Zhu Yongguan,Professor of Soil Environmental Sciences and Biogeochemistry,is the director general of the Institute of Urban Environment,CAS.Professor Zhu is a leader in taking multi-scale and multi-disciplinary approaches to soil and environmental problems,for example his systematic contribution to the understanding of the dynamics of arsenic in soil-plant systems and human health impacts.He is a scientific committee member for the ICSU program on Human Health and Wellbeing in Changing Urban Environment,and served for nine years as a member of Standing Advisory Group for Nuclear Application, International Atomic Energy Agency(2004-2012).Professor Zhu is the recipient of many international and Chinese merit awards,including TWAS ScienceAward 2013,National Natural ScienceAward 2009.E-mail:ygzhu@iue.ac.cn
Antibiotic Resistance:Sources and Mitigation
Zhu Yongguan1,2Ouyang Weiying1Wu Nan3Su Jianqiang1Qiao Min2
(1 Institute of Urban Environment,Key Lab of Urban Environment and Health,ChineseAcademy of Sciences,Xiamen 361021,China;2 Research Center for Eco-environmental Sciences,ChineseAcademy of Sciences,Beijing 100085,China;3 TianjinAgricultural College,Tianjin 300384,China)
Alexander Fleming’s discovery of a fungus producing penicillin more than 80 years ago has been regarded as a major breakthrough in medical science in human history.Nobel Prize for medicine or physiology was awarded three times to those who discovered novel antibiotics.Antibiotics have been widely used not only in human beings but also in animals for growth promotion and infectious disease control.Antibiotic resistance is defined as the microbial ability to sustain and multiply in the presence of antibiotics.Antibiotic resistance is ancient and ubiquitous in environmental microbes,particularly soils where many antibiotics have been discovered so far,and this original resistance is viewed as intrinsic resistance.Nevertheless,the intensive use of anti-biotics in humans and animals have undoubtedly increased the emergence and abundance of antibiotic resistance in the environment,and therefore threatening global human health.Overall,there are three mechanisms that enable the microbes to resist antibiotics:(1)efflux pumps;(2)deactivation or degradation of antibiotics;(3)protection of targets in bacterial cells.Numerous studies have demonstrated that the use of antibiotics and residual discharge into the environment is well correlated with the abundance of antibiotic resistance.Antibiotic resistance can spread via not only vertical gene transfer but also horizontal gene transfer(HGT),and eventually to human pathogens,and even the emergence of superbugs.In 2006 Pruden et al.explicitly proposed antibiotic resistance as emerging contaminants,and pointed out that conventional environmental treatment systems were not designed to remove these emerging contaminants.In order to combat global antibiotic resistance,we must take the following actions and measures:(1)to establish global management system to regulate the use and discharge of antibiotics;(2)to develop rapid and transparent global surveillance system on antibiotic resistance,covering clinical environments,animals farms and urban wastewater treatment facilities;(3)to set up global innovation fund for new drug development and intellectual property protection system so that drug develop is sustainable;(4)to strengthen fundamental and applied research on environmental antibiotic resistance,including the emergence and spread of resistance,mitigation technologies and solutions,such as advanced water treatment technologies to reduce resistant bacteria in effluent;(5)and to improve public awareness of antibiotic resistance,therefore control the misuse of antibiotics in agricultural and medical industries and environmental pollution from the root cause.
urbanization,antibiotics,antibiotic resistance,superbug,environmental health
DOI 10.16418/j.issn.1000-3045.2015.04.010
*修改稿收到日期:2015年7月8日
