葡萄糖醛酸木聚糖生物合成研究进展
2015-07-12吴蔼民赵先海解巧丽解新明
吴蔼民,赵先海,解巧丽,解新明
(华南农业大学 林学与风景园林学院,广东 广州510642)
陆生植物千姿百态,但其构成基本单元——细胞壁的基本成分却相对固定,为植物生长提供了支撑,使一些木本植物有可能长成参天大树.植物细胞壁不是静止的,它也参与植物的代谢活动,比如物质交换和信号传导,并且植物初生细胞壁具有可延展性,可随着植物的生长而扩张.在植物细胞停止生长后,次生细胞壁开始形成,这部分结构赋予植物更强的支撑力.而植物通过光合作用产生的核苷糖类及其他代谢产物,最后在特定的细胞壁合成酶(如纤维素合成酶CESA)作用下,主要沉积在次生细胞壁.
植物次生细胞壁主要包括纤维素、半纤维素和木质素,这3 大组分是地球上最丰富的可再生资源[1].纤维素是细胞壁的第1 大组分,由均一的D-葡萄糖以β-(1→4)糖苷键连接而成[2].木质素主要由4-羟基苯丙素类(4-hydroxyphenylpropanoids)氧化生成,是一种不均一的大分子聚合物[3],在高等植物中广泛存在.半纤维素是一类复合聚糖的总称(图1),主要由几种不同类型的五碳糖[β-D-木糖(Xylose,Xyl)、α-L-阿拉伯糖(Arabinose,Ara)]、六碳糖(β-D-葡 萄 糖(Glucose,Glu)、β-D-甘 露 糖(Mannose,Man)、α-D-半乳糖(Galactose,Gal))和糖醛酸(α-D-葡萄糖醛酸、α-D-4-o-甲基-葡萄糖醛酸和α-D-半乳糖醛酸)单体组成,其他糖类如α-L-鼠李糖(Rhamnose,Rha)和α-L-岩藻糖(Fucose,Fuc)等也少量存在于半纤维素中.植物半纤维素结构上主要有木聚糖(Xylan)、木葡聚糖(Xyloglucan)、甘露聚糖(Mannan)和β-(1→3,1→4)-葡聚糖(Glucan),其中β-(1→3,1→4)-葡聚糖为β-(1→3,1→4)-糖苷键主链,多数只存在于草类单子叶植物,其他类型的半纤维素主要为β-(1→4)糖苷键主链,存在于所有陆生植物中[4].木聚糖是双子叶植物和大多数单子叶植物次生壁的主要半纤维素成分,含量与木质素相当,约占20%~30%.木聚糖通过氢键与纤维素交联缠绕在一起,又通过共价键与木质素交联,三者形成植物细胞壁的网络结构.完全丧失木聚糖的植物将基本失去次生生长,植物矮小,几乎不抽薹[5-6].而适当下调木聚糖合成中关键的木糖转移酶活性,不但植物生长正常,生物量不受影响,而且植物的纤维素含量相对提高,木聚糖和木质素含量相对下降[7-10],这种材料对于造纸工业和生物质燃料生产都是非常有利的.为了更加充分地利用木聚糖,必须探明木聚糖的合成机理,以便通过关键基因调控的方式来人为修饰木聚糖的结构和它在木质纤维素中的比例.
木聚糖的研究起步较早,但以前的研究主要集中于对不同植物测定木糖转移酶活性和木聚糖的成分分析上[11],而在分子水平上揭示木聚糖的合成机理则主要发生于最近这10年.其中,报道于2005年[12]的Fragile fiber 8(FRA8)基因第1 次从分子水平上揭示了木聚糖的生物合成.随着生物质能源的兴起,木聚糖的研究更是取得了突破性进展,成为植物细胞壁研究的一个热点.本文综述了近10年来葡萄糖醛酸木聚糖合成分子机理上的研究进展.

图1 半纤维素结构示意图Fig.1 Schematic illustrations of hemicelluloses
1 木聚糖的结构
木聚糖主链由木糖以β-(1→4)糖苷键连接而成,木糖残基o-2 或o-3 位置上常有乙酰基,在双子叶植物和裸子植物的木聚糖主链末端还有1 个还原四糖结构:β-D-木糖-(1→3)-α-L-鼠李糖-(1→2)-α-D-半乳糖醛酸-(1→4)-D-木糖[13].根据侧链的不同,木聚糖可以分为葡萄糖醛酸木聚糖(Glucuronoxylan,GX)、4-o-甲基葡萄糖醛酸木聚糖(4-o-methylglucuronoxylan,MGX)、阿拉伯糖木聚糖(Arabinoxylan,AX)和葡萄糖醛酸阿拉伯糖木聚糖(Glucuronoarabinoxylan,GAX)(图1).(4-o-甲基)葡萄糖醛酸通过α-(1→2)糖苷键与主链相连,形成GX 或MGX,是双子叶植物次生壁最主要的半纤维素成分,可占20%~30%[4],主链上55%的木糖残基可在o-2(25%)或o-3(30%)位置上被乙酰化[14],为木聚糖最常见的修饰形式.通常阔叶木中(4-o-甲基)葡萄糖醛酸基团与木糖基团的物质的量比为1 ∶4~1 ∶16,平均值为1∶10[15-16],在拟南芥Arabidopsis thaliana 中,每8 个木糖基团便会连有1 个(4-o-甲基)葡萄糖醛酸[17].GX 和MGX 在双子叶植物初生壁中也存在(约占5%)[18].Reis 等[19]认为酸性侧链基团有助于纤维素微纤丝的螺旋状排列.草本单子叶植物木聚糖主链也以β-(1→4)-D-木糖为主链,侧链由α-(1→3)或/和α-(1→2)呋喃阿拉伯糖基(Arabinofuranosyl)和α-(1→4)键连接的(4-o-甲基)葡萄糖醛酸组成,称之为(4-o-甲基)葡萄糖醛酸阿拉伯糖基木聚糖(Glucuronoarabinoxylan,GAX).另外,草本植物木聚糖没有还原末端.AX 在草本单子叶植物的胚乳细胞壁中大量存在[20],GAX 在草本单子叶植物的初生壁中可占20%~40%,在次生壁中可占40%~50%[4].本文将主要集中在目前研究最透彻的GX 上介绍木聚糖的合成.
2 葡萄糖醛酸木聚糖合成相关基因
葡萄糖醛酸木聚糖在高尔基体合成,然后经膜泡运输转移到细胞壁上.利用正向遗传学和反向遗传学,在模式植物拟南芥中已发现许多木聚糖合成基因.双子叶植物木聚糖合成涉及多种糖基转移酶(Glycosyltransferase,GT)基因,其存在于GT43 家族、GT47 家族和GT8 家族等[21],这些GTs 可能以蛋白复合体的形式催化木聚糖的合成[22].根据这些基因在木聚糖合成时的功能,可以分为3 组.
2.1 葡萄糖醛酸木聚糖主链合成相关基因
此类基因主要功能涉及木聚糖主链延伸,催化木聚糖主链β-(1→4)糖苷键的形成.从杨树和拟南芥里提取的微囊体具有高β-(1→4)木糖转移酶活性,可将最多7 个木糖添加到外源的寡聚木糖上[23-24].在另外一些研究中,如小麦幼苗和大麦的胚乳,微囊体的β-(1→4)木糖转移酶可将5 个木糖添加到外源的寡聚木糖上[11,25].虽然有报道类纤维素合成酶(Cellulose synthase-like,Csl)基因参与木葡聚糖[26]、葡甘 露聚糖[27-28]和β-(1 →3,1 →4)-葡聚糖[29]的合成,但是到目前为止,还未发现类纤维素合成酶基因成员与木聚糖主链合成有关[20].
与木聚糖主链合成有关的基因包括IRX9、IRX9L、IRX14、IRX14L(GT43 家族)、IRX10 和IRX10L(GT47 家族)[5,13,24,30-32].它们被命名为IRX(Irregular xylem)是由于它们的突变体表现为不正常的木质部,葡萄糖醛酸木聚糖含量和葡萄糖醛酸木聚糖主链长度都受到了影响,irx9、irx14 突变体和irx10 irx10L 双突变体拟南芥微囊体的β-(1→4)木糖转移酶活性减小[17,33].这6 个基因可再分为3 组:IRX9 和IRX9L,IRX10 和IRX10L,IRX14 和IRX14L.每组内的2 个基因为同源基因,且前者为主效基因,后者为次效基因[5-6,34].在每组基因中,除IRX9 外,其他单基因突变体表型不明显,但双突变体则表现为异常矮小、次生生长几乎完全丧失、木糖含量减少、缺失木聚糖等.主效基因的纯合突变体和次效基因的杂合体[如irx10 irx10L(+ /-)]表现为介于双突变体和单突变体之间的中间状态表型,有明显的不规则木质部,维管束导管坍塌,花不能正常授粉,基本不能收到种子.而次效基因的纯合突变体和主效基因的杂合体[如irx10L irx10(+ /-)]则没有表型变化,植物生长正常[5-6,33].暗示了木聚糖的合成存在着剂量效应(Dosage dependent).irx9、irx10 和irx14 突变体只能被相应的同源基因IRX9L、IRX10L和IRX14L 所互补,表明这3 类基因间在功能上是非冗余的[5].目前还不清楚是否FRA8 也参与了木聚糖主链的合成,因为FRA8/F8H 也是成对出现,fra8 fra8H 双突变体生长矮小,木聚糖长度变短,但是FRA8 基因本身可能参与木聚糖还原末端合成.因而,FRA8 基因的功能还有待进一步研究确定[5].这些基因相似的突变体表型和功能的合理解释是这些基因之间可能像纤维素合成酶一样形成复合体.然而,irx14 irx14L 和irx10 irx10L 双突变体表现出比irx9 irx9L 和fra8 fra8h 更严重的生长缺陷,暗示可能有更复杂的合成机理,如涉及木聚糖合成的起始.
IRX9、IRX9L、IRX14 和IRX14L 同属于GT43 家族,在杨树中此家族有7 个基因,其中PtrGT43A、PtrGT43B、PtrGT43E、PtrGT43F 和PtrGT43G 为IRX9的同源基因,PtrGT43C 和PtrGT43D 为IRX14 的同源基因.Ratke 等[35]对杨树中的这些基因进行表达分析发现,它们具有明显的差异表达,因此提出在杨树中可能有2 种木聚糖合成酶复合体形式,次生壁的木聚糖合成酶复合体活性要高于初生壁木聚糖合成酶复合体活性.
Jensen 等[36]通过在毕赤酵母中单独异源表达拟南芥、小立碗藓Physcomitrella patens 和洋车前草Plantago ovata 的IRX10 蛋白,在体外检测到了木糖转移酶活性,并且来自小立碗藓的IRX10 酶活性最高,其次是洋车前草,拟南芥的IRX10 蛋白检测到了相对较弱的活性,表明IRX10 作为木糖转移酶的功能具有保守性,在拟南芥中IRX10 的功能可能还需要与其他蛋白形成复合体,这可能是体外表达IRX10 活性比较低的原因.与此同时,Urbanowicz等[37]通过在人胚胎肾细胞系293 中表达拟南拟的IRX10L 蛋白和ESK1/TBL29 蛋白,分别证明了这2个蛋白具有木糖转移酶和乙酰基转移酶活性,但是IRX10L 的活性比在Jensen 等[36]试验中高很多,这可能是2 个同源蛋白本身之间的差异(虽然二者之间具有86.65%的相似性),也可能是由于利用了不同的异源表达系统.
木聚糖的合成可能需要多个蛋白形成复合体发挥作用,这也可能是这些糖基转移酶在异源表达时没有活性的原因[4].拟南芥的IRX9 和IRX14 基因或杨树的PtGT43B 和PtGT43C 基因在烟草BY2 细胞系中共表达可以检测到XylT 活性,但是单个基因的表达却不能检测到XylT 活性[23,38].这种现象表示IRX9 和IRX14 可能形成蛋白复合体共同行使功能.Zeng 等[39]从小麦中通过免疫共沉淀分离得到了包含GT43、GT47 和GT75 成员的蛋白复合体,这个复合体可合成葡萄糖醛酸阿拉伯糖木聚糖,这是木聚糖合成需要蛋白复合体的直接证据.
2.2 葡萄糖醛酸木聚糖还原四糖末端合成相关基因
在双子叶植物和裸子植物的木聚糖主链末端有1 个还原四糖结构:β-D-木糖-(1 →3)-α-L-鼠李糖-(1→2)-α-D-半乳糖醛酸-(1→4)-D-木糖,在拟南芥中,FRA8、F8H(GT 47 家族)、IRX8 和PARVUS(GT8家族)被认为与这个四糖结构的合成有关.这些基因的突变体多数生长矮小,木质部不规则,含有较少的木聚糖,缺乏还原四糖末端,但木聚糖主链延伸能力没有受到影响,到目前为止,对四糖末端的分析还停留于突变体的生化分析,具体的酶活性还没有检测到[12-13,17,40-41].在杨树中已发现FRA8 和IRX8 基因的同源基因,分别为PoGT47C 和PoGT8D,PARVUS 的同源基因为PoGT8E、PoGT8F、PdGATL1.1 和PdGATL1.2[42-45].
IRX8 和PARVUS 同属于GT8 家族,可利用α-GalA 形 成α-GalA-(1-4)-Xyl.与其他糖基转移酶FRA8、IRX8、IRX9、IRX10 和IRX14 定位于高尔基体不同,AtPARVUS 和它在杨树中的同源基因PoGT8E/F 表达产物主要定位于内质网[45-46],因此,Lee 等[45]认为,木聚糖四糖末端的合成起始于内质网,PARVUS 和PoGT8E/F 可催化木糖连接到一个未知的受体上,然后IRX8 和FRA8 在高尔基体上继续合成四糖末端.IRX8(也称为GAUT12)是半乳糖醛酸转移酶GAUT1 的同源基因,GT8 家族可特异地利用α-型单糖形成α-型糖苷键,因此认为IRX8 可能合成GalAXyl[47].irx8 突变体生长矮小,木聚糖和均质聚半乳糖醛酸含量减少[48].
GT47 家族具有一种反转机制,可以α-型单糖/β-型单糖为底物,形成β-糖苷键/α-糖苷键产物[12],fra8 突变体最明显的表现为还原四糖缺失,因此,Pena 等[13]认为,Fra8 的功能主要是在还原四糖末端形成时,利用UDP-α-Xyl 形成β-糖苷键连接的β-Xyl-(1-3)-α-Rha 或利用UDP-β-Rha 形成α-糖苷键连接的α-Rha-α-GlaA.但是这些分析均是建立在fra8 单突变体的基础上的,因为irx8、parvus 和fra8 单突变体植物生长受影响程度相似,共同参与还原四糖末端合成,这就很难解释fra8/fra8H 双突变体的异常矮小[5].
2.3 葡萄糖醛酸木聚糖侧链合成相关基因
拟南芥木聚糖主链上12%的木糖残基有(4-o-甲基)葡萄糖醛酸侧链,即每8 个木糖残基便会连接有1 个(4-o-甲基)葡萄糖醛酸侧链[17],在拟南芥中有5 个葡萄糖醛酸转移酶(Glucuronic acid substitution of xylan,GUX,GT8 家族)相关基因,包括GUX1、GUX2、GUX3 和GUX4 和GUX5,可催化葡萄糖醛酸连接到木聚糖主链上去[10,49-51].gux1 和gux2 单突变体的葡萄糖醛酸取代度分别降低了大约70%和20%,但是双突变体植株的木聚糖基本检测不到葡萄糖醛酸侧链,并且gux1/gux2 双突变体没有葡萄糖醛酸转移酶活性,gux1/gux2 双突变体的木聚糖主链没有受到影响,表示木聚糖主链的合成与侧链基团的合成不是同步进行的,虽然双突变体植株主茎的应力略微变弱,但是木质部导管没有变形,并且植株大小也很正常,葡萄糖醛酸侧链消失对双突变体最大的影响是木聚糖容易抽提了,并且易于分解[10].Rennie 等[51]通过在烟草瞬时表达拟南芥GUX 蛋白,在烟草微囊体中测定到葡萄糖醛酸转移酶活性,结果表明只有GUX1、GUX2 和GUX4 具有葡萄糖醛酸转移酶活性,而GUX3 和GUX5 是否与木聚糖合成有关还不能确定.对这2 个蛋白的结构进行预测发现,与其他3 个蛋白相比,它们的活性位点和表面结构都不同,但考虑到这5 个蛋白在进化上的保守性,GUX3 和GUX5 可能作为GUX 的非催化亚基起作用[51],这也可以解释为何GUX3 在烟草BY2 细胞中进行表达时,GlcAT 活性增加了[50].但是,在拟南芥培养细胞的高尔基体蛋白组分析表明存在GUX3 蛋白[52-53],由于未分化的培养细胞初生壁占主要成分,因此,GUX3 也可能参与初生壁某些成分的合成.Lee等[50]对5 个GUX 基因的表达模式进行了分析,发现GUX1 和GUX2 主要在茎部表达,GUX3 在根、茎、叶和花部都有表达,并且茎部只在木质部有表达,GUX4 主要在根部表达,GUX5 主要在叶和花部表达.在烟草BY2 细胞中对在茎部有表达的GUX1、GUX2和GUX3 也进行了表达,分别转3 个基因的细胞系都表达出GlcAT 活性,并且同时表达GUX1 和GUX2的细胞系没有表现出更高活性.GUX1、GUX2 和GUX3 这3 个基因同时缺失突变体,完全检测不到GlcA,并且植株表现出矮小,木质部细胞变形,细胞壁变薄,细胞壁密度增大,但是木聚糖含量没有受到影响.Bromley 等[49]认为GUX1 修饰木聚糖时会均匀地间隔6、8、10 或更多偶数个木糖残基,这种修饰为主要模式,而GUX2 修饰木聚糖时则有更小的间隔,会在7 个或更少的木糖残基间隔下行使修饰作用,这种修饰所占比例要少一些,是次要模式,这2 种修饰方式共存于同一木聚糖分子,可能影响到与纤维素或木质素的交联.
葡萄糖醛酸甲基转移酶(Glucuronoxylan methyltransferase,GXM)含有1 个Domain of unknown function 579(DUF579)结构域,是1 种阳离子依赖型的甲基转移酶,特异地将甲基从S-腺苷甲硫氨酸转移到木糖侧链的葡萄糖醛酸o-4 位置[54-55].gxm 突变体葡萄糖醛酸的甲基化程度减小,但植株的生长没有受到影响,gxmt1 突变体的木聚糖变得更容易酶解糖化,这说明由于木聚糖甲基化程度的减小,gxmt1 突变体细胞壁中木聚糖与其他成分间的交联改变了[56].GXM1、GXM2 和GXM3 在大肠埃希菌Escherichia coli 异源表达时具有甲基转移酶活性,但是得到的重组蛋白只能甲基化在木聚糖上的葡萄糖醛酸,而不能甲基化葡萄糖醛酸单糖,这说明在木聚糖合成时,葡萄糖醛酸的甲基化是在连接到木聚糖以后发生的,或者说GXM 基因可以特异性的甲基化木聚糖上的葡萄糖醛酸[54-55].
IRX15 和IRX15L 也含有DUF579 结构域,irx15 irx15L 双突变体木质部导管变形或崩塌,并且木聚糖聚合度也减小[56-57],但是IRX15 和IRX15L 的具体生化功能还不得而知,虽然它们也含有DUF579 结构域,但irx15 irx15L 双突变体木聚糖的甲基化程度升高了,因此,对木聚糖来说,IRX15 和IRX15L 不具有甲基转移酶的功能,它们可能参与到木聚糖合成酶复合体中,或者催化其他成分的甲基化,比如果胶.
RWA1~RWA4(Reduced wall acetyltion)为新型隐球菌Cryptococcus neoformans CAS1 基因在拟南芥中的同源基因,单基因突变体没有明显的表型改变,只有rwa2 表现出乙酰化明显减少(减少约20%),但乙酰化的减少不限于木聚糖,木葡聚糖和果胶等多糖也出现了乙酰化减少,对RWA2 的亚细胞定位表明,RWA2 定位于内质网,因此,RWA2 可能是乙酰基转运蛋白[58].在Lee 等[59]的研究中,rwa1~rwa4的双突变体和三突变体也没有明显表型改变,但是四突变体茎的抗拉力程度变小,细胞壁变薄,导管变形,乙酰化减少.然而在Manabe 等[60]的研究中,三突变体都表现出明显的植株矮小和乙酰化减少,四突变体更为严重,甚至没有次生细胞壁的分化.现在还发现表皮毛双折射状(Trichome birefringence-like,TBL)家族的成员TBL29/ESK1 直接参与催化木聚糖的乙酰化,esk1 突变体次生壁变薄,主茎变弱,乙酰化程度明显减少[61-62],并且Urbanowicz 等[37]通过异源表达ESK1 蛋白检测到了乙酰转移酶活性(图2,表1).
3 葡萄糖醛酸木聚糖主链合成机制
生物体内复杂的碳水化合物有多样的合成机制,聚糖可直接由核苷糖组装,或者单糖首先转移到脂类中间体上形成寡糖,再转移到延伸的聚糖上.聚糖的延伸可经由还原末端或非还原末端,当由还原末端进行延伸时,伸长的糖链为供体,活化的单糖为受体,而当由非还原末端进行延伸时,二者的角色相反,活化的单糖为供体,伸长的糖链为受体[65].葡萄糖醛酸木聚糖具有还原四糖末端结构,但是此结构在木聚糖的合成中的作用还不清楚.根据木聚糖的延伸方向和还原四糖结构在木聚糖合成中的功能,木聚糖的合成可能有2 种机制[13,65].
第1 种机制认为,还原四糖结构在木聚糖的合成中起到起始物的功能,木糖不断添加到非还原末端使之延长.来源于动物的葡糖氨基葡聚糖的合成也是这种形式,葡糖氨基葡聚糖的还原末端有1 个连接寡糖,它经由1 个木糖的还原末端连接到蛋白质上,通过不断在非还原末端添加单糖,葡糖氨基葡聚糖得以延伸[66],然而在植物中,对这种机制的支持证据还比较缺乏.

图2 葡萄糖醛酸木聚糖在高尔基体中的合成(参考Rennie 等[63]改进)Fig.2 Glucuronoxylan biosyntheses in Golgi(referenced by Rennie et al.[63])

表1 葡萄糖醛酸木聚糖合成相关基因Tab.1 Genes involved in the glucuronoxylan biosynthesis
第2 种机制认为,还原四糖结构在木聚糖的合成中起到终止物的功能,通过添加到木聚糖链的还原末端,终止木聚糖链的延伸,这样会产生长度比较均一的木聚糖.在这种机制中木糖添加到糖链的还原末端使之延伸.还原四糖结构如果作为终止物,则可以解释fra8 和irx8 突变体缺失了还原四糖结构后为何会产生非均一长度的木聚糖[13].这种机制与透明质酸[67]和淀粉[68]的合成类似,此二者也是通过添加单糖到还原末端使单糖延伸.
另一方面,在草类单子叶植物的葡萄糖醛酸阿拉伯糖木聚糖中,并没有还原四糖结构存在,支持了还原四糖末端可能作为终止物的假说.但是,是否草本单子叶植物的木聚糖合成不需要起始物或终止物,还是在草本单子叶植物中有另一条木聚糖合成途径现在还不得而知[20].另外,在苔藓和草本单子叶植物中有FRA8、IRX8 和PARVUS 的同源基因,这些基因在苔藓和草本单子叶植物中的功能还有待进一步研究[63](图3).
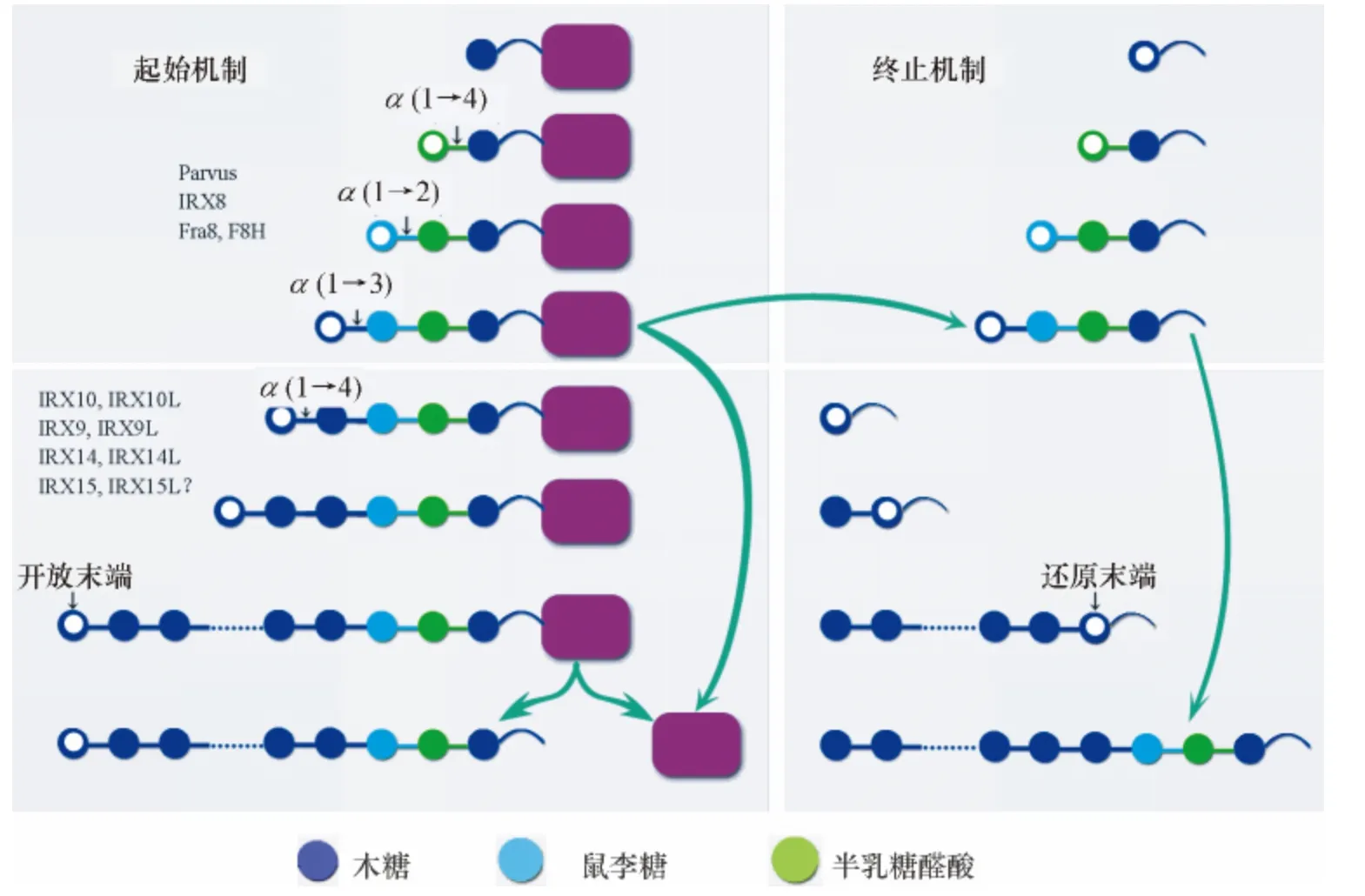
图3 葡萄糖醛酸木聚糖主链合成的2 种机制(参考York 等[65]改进)Fig.3 Two mechanisms of the glucuronoxylan backbone biosynthesis(Referenced by York et al.[65])
4 总结
半纤维素具有重要的商业价值,一些植物的种子中含有大量的半乳甘露聚糖(如瓜尔豆Cyamopsis tetragonoloba、刺槐Robinia pseudoacacia)、葡甘露聚糖(如魔芋Amorphophallus rivieri)、木葡聚糖(如罗望子Tamarindus indica),这些成分可用于食品工业,木聚糖水解得到的木糖可用于生产木糖醇,低聚木糖有利于人体健康,是膳食纤维的主要成分,并且半纤维素对食品和饲料的品质也有很大的影响[4].改性后的木聚糖可应用于日用化工,制作特殊材料.在传统的工业化组分分离中,碱法制浆过程将半纤维素和木质素都降解成小分子排放进了黑液中,并未得到高值化利用.另一方面,酵母只能利用已糖(如葡萄糖或甘露糖)进行生物乙醇发酵,虽然现在也有发展可酵解戊糖的酵母,但是效果不佳[69].因此,对于生物质能源植物,目前增加生物质利用率的有效方法是增加已糖(如葡萄糖或甘露糖)的含量,降低木糖的含量[1].但是,占有木质纤维素1/3 比率的木聚糖仍然是重要的生物质能源,改变木聚糖结构以减少木质纤维素之间的联结,提高木糖酵解效率将是一个重要的研究领域.
近几年,关于木聚糖合成机制的了解取得了长足进步,木聚糖主链合成基因和侧链合成基因都已有许多发现,通过生化方法也测得了一些基因的体外活性,特别是IRX10 的活性[36-37],直接证明了IRX10 是木聚糖合成时的木糖转移酶,但是其他的几个基因IRX9、IRX14 和IRX15 是否直接催化木聚糖主链的延伸,或木聚糖复合体蛋白的结构亚基?存在于双子叶植物中的四糖末端是否绝对不存在于草本单子叶植物植物,此结构对木聚糖合成的作用是什么?如此多的糖基转移酶是如何协作来合成木聚糖,是否形成不同的蛋白复合体?为何不同植物中具有保守的纤维素合成机制,但却具有不同的半纤维素合成机制?半纤维素是在高尔基体中合成的,最终在细胞壁上与纤维素和木质素交联,在这2个结构间半纤维素的运输机制是什么?如此多的未知问题还有待去解决,然而现在发掘出的木聚糖合成基因也为人为调控木聚糖的合成打开了大门,通过改变木聚糖的含量、木聚糖与纤维素、木素间的交联,可以开发出新的生物质能源作物,提高生物质能源利用效率,相信随着测试技术和分析技术的快速发展,木聚糖的合成机理会得到更加充分的解析.
[1]PAULY M,KEEGSTRA K.Cell-wall carbohydrates and their modification as a resource for biofuels[J].Plant J:Cell Mol Biol,2008,54(4):559-568.
[2]PAULY M,KEEGSTRA K.Plant cell wall polymers as precursors for biofuels[J].Curr Opin Plant Biol,2010,13(3):305-312.
[3]SIMMONS B A,LOGUE D,RALPH J.Advances in modifying lignin for enhanced biofuel production[J].Curr Opin Plant Biol,2010,13(3):313-320.
[4]SCHELLER H V,ULVSKOV P.Hemicelluloses[J].Annu Rev Plant Biol,2010,61:263-289.
[5]WU A M,HORNBLAD E,VOXEUR A,et al.Analysis of the arabidopsis IRX9/IRX9-l and IRX14/IRX14-l pairs of glycosyltransferase genes reveals critical contributions to biosynthesis of the hemicellulose glucuronoxylan[J].Plant Physiol,2010,153(2):542-554.
[6]WU A M,RIHOUEY C,SEVENO M,et al.The Arabidopsis irx10 and irx10-like glycosyltransferases are critical for glucuronoxylan biosynthesis during secondary cell wall formation[J].Plant J:Cell Mol Biol,2009,57(4):718-731.
[7]YANG F,MITRA P,ZHANG L,et al.Engineering secondary cell wall deposition in plants[J].Plant Biotechnol J,2013,11(3):325-335.
[8]PETERSEN P D,LAU J,EBERT B,et al.Engineering of plants with improved properties as biofuels feedstocks by vessel-specific complementation of xylan biosynthesis mutants[J].Biotechnol Biofuels,2012,5(1):84.
[9]EUDES A,LIANG Y,MITRA P,et al.Lignin bioengineering[J].Curr Opin Biotechnol,2014,26:189-198.
[10]MORTIMER J C,MILES G P,BROWN D M,et al.Absence of branches from xylan in Arabidopsis gux mutants reveals potential for simplification of lignocellulosic biomass[J].Proc Natl Acad Sci USA,2010,107(40):17409-17414.
[11]KUROYAMA H,TSUMURAYA Y.A xylosyltransferase that synthesizes beta-(1→4)-xylans in wheat(Triticum aestivum L.)seedlings[J].Planta,2001,213(2):231-240.
[12]ZHONG R,PENA M J,ZHOU G K,et al.Arabidopsis fragile fiber8,which encodes a putative glucuronyltransferase,is essential for normal secondary wall synthesis[J].Plant Cell,2005,17(12):3390-3408.
[13]PENA M J,ZHONG R Q,ZHOU G K,et al.Arabidopsis irregular xylem8 and irregular xylem9:Implications for the complexity of glucuronoxylan biosynthesis[J].Plant Cell,2007,19(2):549-563.
[14]GILLE S,PAULY M.O-acetylation of plant cell wall polysaccharides[J].Front Plant Sci,2012,3:12.
[15]VAZQUEZ M J,GARROTE G,ALONSO J L,et al.Refining of autohydrolysis liquors for manufacturing xylooligosaccharides:Evaluation of operational strategies[J].Bioresour Technol,2005,96(8):889-896.
[16]TELEMAN A,LUNDQVIST J,TJERNELD F,et al.Characterization of acetylated 4-o-methylglucuronoxylan isolated from aspen employing1H and13C NMR spectroscopy[J].Carbohydr Res,2000,329(4):807-815.
[17]BROWN D M,GOUBET F,WONG V W,et al.Comparison of five xylan synthesis mutants reveals new insight into the mechanisms of xylan synthesis[J].Plant J:Cell Mol Biol,2007,52(6):1154-1168.
[18]ZABLACKIS E,HUANG J,MULLER B,et al.Characterization of the cell-wall polysaccharides of Arabidopsis thaliana leaves[J].Plant Physiol,1995,107(4):1129-1138.
[19]REIS D,VIAN B.Helicoidal pattern in secondary cell walls and possible role of xylans in their construction[J].Comptes Rendus Biologies,2004,327(9/10):785-790.
[20]PAULY M,GILLE S,LIU L,et al.Hemicellulose biosynthesis[J].Planta,2013,238(4):627-642.
[21]DOERING A,LATHE R,PERSSON S.An update on xylan synthesis[J].Mol Plant,2012,5(4):769-771.
[22]FAIK A.Xylan biosynthesis:News from the grass[J].Plant Physiol,2010,153(2):396-402.
[23]LEE C,ZHONG R,YE Z H.Biochemical characterization of xylan xylosyltransferases involved in wood formation in poplar[J].Plant Signaling Behavior,2012,7(3):332-337.
[24]LEE C,O’NEILL M A,TSUMURAYA Y,et al.The irregular xylem9 mutant is deficient in xylan xylosyltransferase activity[J].Plant Cell Physiol,2007,48(11):1624-1634.
[25]URAHARA T,TSUCHIYA K,KOTAKE T,et al.A β-(1→4)-xylosyltransferase involved in the synthesis of arabinoxylans in developing barley endosperms[J].Physiologia Plantarum,2004,122(2):169-180.
[26]COCURON J C,LEROUXEL O,DRAKAKAKI G,et al.A gene from the cellulose synthase-like c family encodes a beta-1,4 glucan synthase[J].Proc Natl Acad Sci USA,2007,104(20):8550-8555.
[27]DHUGGA K S,BARREIRO R,WHITTEN B,et al.Guar seed beta-mannan synthase is a member of the cellulose synthase super gene family[J].Science,2004,303(5656):363-366.
[28]LIEPMAN A H,WILKERSON C G,KEEGSTRA K.Expression of cellulose synthase-like(csl)genes in insect cells reveals that csla family members encode mannan synthases[J].Proc Natl Acad Sci USA,2005,102(6):2221-2226.
[29]BURTON R A,WILSON S M,HRMOVA M,et al.Cellulose synthase-like cslf genes mediate the synthesis of cell wall(1,3;1,4)-beta-d-glucans[J].Science,2006,311(5769):1940-1942.
[30]CHINIQUY D,VARANASI P,OH T,et al.Three novel rice genes closely related to the Arabidopsis irx9,irx91,and irx14 genes and their roles in xylan biosynthesis[J].Front Plant Sci,2013,4:83.
[31]KEPPLER B D,SHOWALTER A M.IRX14 and IRX14-LIKE,two glycosyl transferases involved in glucuronoxylan biosynthesis and drought tolerance in Arabidopsis[J].Mol Plant,2010,3(5):834-841.
[32]HORNBLAD E,ULFSTEDT M,RONNE H,et al.Partial functional conservation of IRX10 homologs in Physcomitrella patens and Arabidopsis thaliana indicates an evolutionary step contributing to vascular formation in land plants[J].BMC Plant Biol,2013,13:3.
[33]BROWN D M,ZHANG Z,STEPHENS E,et al.Characterization of irx10 and irx10-like reveals an essential role in glucuronoxylan biosynthesis in Arabidopsis[J].Plant J:Cell Mol Biol,2009,57(4):732-746.
[34]LEE C,TENG Q,HUANG W,et al.The Arabidopsis family GT43 glycosyltransferases form two functionally nonredundant groups essential for the elongation of glucuronoxylan backbone[J].Plant Physiol,2010,153(2):526-541.
[35]RATKE C,PAWAR P M,BALASUBRAMANIAN V K,et al.Populus GT43 family members group into distinct sets required for primary and secondary wall xylan biosynthesis and include useful promoters for wood modification[J].Plant Biotechnol J,2015,13(1):26-37.
[36]JENSEN J K,JOHNSON N R,WILKERSON C G.Arabidopsis thaliana irx10 and two related proteins from psyllium and physcomitrella patens are xylan xylosyltransferases[J].Plant J:Cell Mol Bio,2014,80(2):207-215.
[37]URBANOWICZ B R,PENA M J,MONIZ H A,et al.Two Arabidopsis proteins synthesize acetylated xylan in vitro[J].Plant J:Cell Mol Biol,2014,80(2):197-206.
[38]LEE C,ZHONG R,YE Z H.Arabidopsis family GT43 members are xylan xylosyltransferases required for the elongation of the xylan backbone[J].Plant Cell Physiol,2012,53(1):135-143.
[39]ZENG W,JIANG N,NADELLA R,et al.A glucurono(arabino)xylan synthase complex from wheat contains members of the GT43,GT47,and GT75 families and functions cooperatively[J].Plant Physiol,2010,154(1):78-97.
[40]LEE C,TENG Q,HUANG W,et al.The F8H glycosyltransferase is a functional paralog of FRA8 involved in glucuronoxylan biosynthesis in Arabidopsis[J].Plant Cell Physiol,2009,50(4):812-827.
[41]LAO N T,LONG D,KIANG S,et al.Mutation of a family 8 glycosyltransferase gene alters cell wall carbohydrate composition and causes a humidity-sensitive semi-sterile dwarf phenotype in Arabidopsis[J].Plant Mol Biol,2003,53(5):647-661.
[42]ZHOU G K,ZHONG R,RICHARDSON E A,et al.The poplar glycosyltransferase GT47C is functionally conserved with Arabidopsis fragile fiber8[J].Plant Cell Physiol,2006,47(9):1229-1240.
[43]ZHOU G K,ZHONG R,HIMMELSBACH D S,et al.Molecular characterization of PoGT8D and PoGT43B,two secondary wall-associated glycosyltransferases in poplar[J].Plant Cell Physiol,2007,48(5):689-699.
[44]KONG Y,ZHOU G,AVCI U,et al.Two poplar glycosyltransferase genes,PdGATL1.1 and PdGATL1.2,are functional orthologs to PARVUS/AtGATL1 in Arabidopsis[J].Mol Plant,2009,2(5):1040-1050.
[45]LEE C,TENG Q,HUANG W,et al.The poplar GT8E and GT8F glycosyltransferases are functional orthologs of Arabidopsis parvus involved in glucuronoxylan biosynthesis[J].Plant Cell Physiol,2009,50(11):1982-1987.
[46]LEE C,ZHONG R,RICHARDSON E A,et al.The PARVUS gene is expressed in cells undergoing secondary wall thickening and is essential for glucuronoxylan biosynthesis[J].Plant Cell Physiol,2007,48(12):1659-1672.
[47]STERLING J D,ATMODJO M A,INWOOD S E,et al.Functional identification of an Arabidopsis pectin biosynthetic homogalacturonan galacturonosyltransferase[J].Proc Natl Acad Sci USA,2006,103(13):5236-5241.
[48]PERSSON S,CAFFALL K H,FRESHOUR G,et al.The Arabidopsis irregular xylem8 mutant is deficient in glucuronoxylan and homogalacturonan,which are essential for secondary cell wall integrity[J].Plant Cell,2007,19(1):237-255.
[49]BROMLEY J R,BUSSE-WICHER M,TRYFONA T,et al.GUX1 and GUX2 glucuronyltransferases decorate distinct domains of glucuronoxylan with different substitution patterns[J].Plant J:Cell Mol Biol,2013,74(3):423-434.
[50]LEE C,TENG Q,ZHONG R,et al.Arabidopsis GUX proteins are glucuronyltransferases responsible for the addition of glucuronic acid side chains onto xylan[J].Plant Cell Physiol,2012,53(7):1204-1216.
[51]RENNIE E A,HANSEN S F,BAIDOO E E K,et al.Three members of the Arabidopsis glycosyltransferase family 8 are xylan glucuronosyltransferases[J].Plant Physiol,2012,159(4):1408-1417.
[52]DUNKLEY T P J,HESTER S,SHADFORTH I P,et al.Mapping the Arabidopsis organelle proteome[J].Proc Natl Acad Sci USA,2006,103(17):6518-6523.
[53]PARSONS H T,CHRISTIANSEN K,KNIERIM B,et al.Isolation and proteomic characterization of the arabidopsis golgi defines functional and novel components involved in plant cell wall biosynthesis[J].Plant Physiol,2012,159(1):12-26.
[54]LEE C,TENG Q,ZHONG R,et al.Three Arabidopsis DUF579 domain-containing GXM proteins are methyltransferases catalyzing 4-o-methylation of glucuronic acid on xylan[J].Plant Cell Physiol,2012,53(11):1934-1949.
[55]URBANOWICZ B R,PENA M J,RATNAPARKHE S,et al.4-o-methylation of glucuronic acid in Arabidopsis glucuronoxylan is catalyzed by a domain of unknown function family 579 protein[J].Proc Natl Acad Sci USA,2012,109(35):14253-14258.
[56]JENSEN J K,KIM H,COCURON J C,et al.The DUF579 domain containing proteins IRX15 and IRX15-l affect xylan synthesis in Arabidopsis[J].Plant J:Cell Mol Biol,2011,66(3):387-400.
[57]BROWN D,WIGHTMAN R,ZHANG Z,et al.Arabidopsis genes IRREGULAR XYLEM(IRX15)and IRX15l encode DUF579-containing proteins that are essential for normal xylan deposition in the secondary cell wall[J].Plant J:Cell Mol Biol,2011,66(3):401-413.
[58]MANABE Y,NAFISI M,VERHERTBRUGGEN Y,et al.Loss-of-function mutation of REDUCED WALL ACETYLATION2 in Arabidopsis leads to reduced cell wall acetylation and increased resistance to Botrytis cinerea[J].Plant Physiol,2011,155(3):1068-1078.
[59]LEE C,TENG Q,ZHONG R,et al.The four Arabidopsis REDUCED WALL ACETYLATION genes are expressed in secondary wall-containing cells and required for the Acetylation of xylan[J].Plant Cell Physiol,2011,52(8):1289-1301.
[60]MANABE Y,VERHERTBRUGGEN Y,GILLE S,et al.Reduced wall acetylation proteins play vital and distinct roles in cell wall o-acetylation in Arabidopsis[J].Plant Physiol,2013,163(3):1107-1117.
[61]YUAN Y,TENG Q,ZHONG R,et al.The Arabidopsis DUF231 domain-containing protein ESK1 mediates 2-oand 3-o-acetylation of xylosyl residues in xylan[J].Plant Cell Physiol,2013,54(7):1186-1199.
[62]XIONG G,CHENG K,PAULY M.Xylan o-acetylation impacts xylem development and enzymatic recalcitrance as indicated by the Arabidopsis mutant tbl29[J].Molecular Plant,2013,6(4):1373-1375.
[63]RENNIE E A,SCHELLER H V.Xylan biosynthesis[J].Curr Opin Biotechnol,2014,26:100-107.
[64]BROWN D M,ZEEF L A H,ELLIS J,et al.Identification of novel genes in Arabidopsis involved in secondary cell wall formation using expression profiling and reverse genetics[J].Plant Cell,2005,17(8):2281-2295.
[65]YORK W S,O’NEILL M A.Biochemical control of xylan biosynthesis:Which end is up?[J].Curr Opin Plant Biol,2008,11(3):258-265.
[66]PRYDZ K,DALEN K T.Synthesis and sorting of proteoglycans[J].J Cell Sci,2000,113:193-205.
[67]BODEVIN-AUTHELET S,KUSCHE-GULLBERG M,PUMMILL P E,et al.Biosynthesis of hyaluronan:Direction of chain elongation[J].J Biol Chem,2005,280(10):8813-8818.
[68]MUKERJEA R,ROBYT J F.Starch biosynthesis:The primer nonreducing-end mechanism versus the nonprimer reducing-end two-site insertion mechanism[J].Carbohydr Res,2005,340(2):245-255.
[69]VAN VLEET J H,JEFFRIES T W.Yeast metabolic engineering for hemicellulosic ethanol production[J].Curr Opin Biotechnol,2009,20(3):300-306.
