肿瘤细胞膜蛋白体细胞突变分析
2015-07-07吴姗姗周展陈枢青
吴姗姗,周展,陈枢青
(浙江大学 药学院/浙江省抗肿瘤药物临床前研究重点实验室,浙江 杭州 310058)
肿瘤细胞膜蛋白体细胞突变分析
吴姗姗,周展Δ,陈枢青Δ
(浙江大学 药学院/浙江省抗肿瘤药物临床前研究重点实验室,浙江 杭州 310058)
目的 筛选肿瘤细胞特异性膜蛋白胞外突变(特异性膜表面蛋白突变),以期寻找癌症精准医疗的作用靶点。方法 收集7042个肿瘤样本的体细胞突变信息,通过对体细胞突变位点进行基因定位分析,筛选出全部特异性膜蛋白胞外突变,并统计分析这些突变的整体分布情况,鉴定特异性膜蛋白胞外突变频率较高的基因、突变位点以及肿瘤类型。结果 对7042个肿瘤样本的4938362个体细胞突变,共筛选出97193个特异性膜蛋白胞外突变,统计分析结果显示这些突变涉及4347个基因,包括65532个基因位点。进一步分析鉴定出发生特异性膜蛋白胞外突变频率较高的5个基因(MUC16、LRP1B、CSMD3、RYR2、USH2A),1个突变位点(17:37868208)以及6个肿瘤类型(结直肠癌、黑素瘤、子宫癌、脑胶质瘤、肺癌和胃癌)。结论 建立了特异性膜蛋白胞外突变信息库,并得到了该类突变的整体分布情况,为寻找癌症精准医疗的作用靶点提供了信息参考。
癌症精准医疗;作用靶点;体细胞突变;特异性膜蛋白胞外突变
科学界普遍认为体细胞突变是癌症发生发展的重要因素[1],编码区的体细胞突变会造成蛋白质异常,从而形成肿瘤特异性抗原。造成蛋白质异常的体细胞突变大部分是能造成单个氨基酸置换的非同义点突变[2]。膜蛋白是细胞的关键分子,也是重要的药物作用靶点,许多膜蛋白都暴露于细胞表面,且几乎60%的药物都以膜蛋白为受体作药物作用靶点[3-6]。
虽然化疗已经成为癌症治疗的一个必要手段,然而,细胞毒性试剂(化学治疗剂)无法特异性杀伤肿瘤细胞,且存在较大副作用。常规的其他治疗方法如放疗和手术同样存在缺乏特异性的问题。随着测序技术、分子生物学和生物信息学的快速发展,癌症精准医疗方法如靶向治疗和免疫治疗逐渐成为研究热点。靶向治疗如抗体类药物能够特异性的靶向肿瘤细胞,具有特异性抗肿瘤作用,并且毒性明显减少。其中抗体偶联药物(antibody-drug conjugates,ADCs)的肿瘤特异性高,副作用低,且其治疗效果优于一般单克隆抗体[7-9]。研究表明,奥法木单抗偶联有丝分裂抑制剂MMAE(OFA-vcMMAE)能特异性识别慢性淋巴细胞白血病肿瘤细胞表面的CD20并造成有效杀伤[10]。肿瘤坏死因子相关凋亡诱导配体(TNF-related apoptosis-inducing ligand,TRAIL)偶联MMAE(TRAIL-vcMMAE)具有高效靶向杀伤肿瘤细胞的效果,此处TRAIL可作为“靶向弹头”代替抗体[11]。而嵌合抗原受体T细胞免疫疗法(chimeric antigen receptor T-Cell immunotherapy,Car-T)作为免疫治疗的一种也是癌症治疗的研究热点之一。然而,寻找准确的药物作用靶点是靶向治疗和免疫治疗发挥作用的关键,目前还没有较全面的寻找肿瘤特异性抗原的方法。本研究旨在通过生物信息学方法对7042个肿瘤样本(来自30个肿瘤类型)的4938362个体细胞突变进行细胞膜表面蛋白突变分析,通过对突变位点的基因定位分析筛选出全部特异性膜蛋白胞外突变并对其进行统计分析,建立全面筛选靶向治疗和免疫治疗潜在药物作用靶点的参考信息库。
1 材料与方法
1.1 数据来源与数据库 7042个肿瘤样本的体细胞突变数据源于公共数据库COSMIC(Catalogue of somatic mutations in cancer)[12]和TCGA(The cancer genome atlas)[13]及已发表的相关数据[1];细胞膜蛋白预测数据源于The human protein atlas[14](http://www.proteinatlas.org/);Gene Cards人类基因数据库[15]和David基因功能注释数据库[16]提供基因功能注释信息;Ensembl数据库[17]提供基因和编码蛋白的序列信息。
1.2 方法
1.2.1 细胞膜蛋白筛选及其胞外区域氨基酸位点定位:从Ensembl数据库下载人类基因组所有编码蛋白质的序列,利用TMHMM(TransMembrane prediction using Hidden Markov Models)在线预测服务器预测所有蛋白质跨膜区的信息(包括跨膜区数量、跨膜区序列、胞内序列和胞外序列),从中筛选出跨膜区≥1的膜蛋白,并对膜蛋白处于胞外区域的氨基酸位点进行定位,结合人类蛋白质组计划鉴定出的膜蛋白[14]进行基因信息匹配,最终筛选出细胞膜蛋白及其胞外区域。
1.2.2 筛选膜蛋白胞外氨基酸突变:从7042个样本的所有体细胞突变数据中筛选出全部的氨基酸突变,根据细胞膜蛋白及其胞外区域数据信息筛选出膜蛋白胞外氨基酸突变。
1.2.3 分类筛选:样本中包含插入缺失和点突变,由于插入缺失容易造成移码突变,产生与正常蛋白完全不同的多肽段,改变膜蛋白跨膜性质。因此,本研究分析点突变形成的潜在肿瘤特异性抗原,利用生物信息学方法对所有体细胞突变位点进行基因定位分析,筛选造成膜蛋白胞外氨基酸发生置换的体细胞突变,并对突变前后氨基酸性质的变化进行分析,从中筛选出氨基酸性质发生变化的特异性膜蛋白胞外氨基酸突变。
1.3 统计学方法 对同一个基因位点突变所造成的特异性膜蛋白胞外氨基酸突变进行信息整合,并进行样本来源信息匹配,附上样本所对应的肿瘤类型信息。结合生物信息及统计分析软件对筛选得到的特异性膜蛋白胞外突变进行统计分析,主要分析3项内容:每个样本所发生的特异性膜蛋白胞外突变的数量;7042个样本的特异性膜蛋白胞外突变所涉及的基因及其出现次数;7042个样本的特异性膜蛋白胞外突变所涉及的基因突变位点及其出现的次数。
2 结果
2.1 特异性膜蛋白胞外突变筛选情况 特异性膜蛋白胞外突变的筛选流程及结果见图1。共筛选出97193个特异性膜蛋白胞外区突变。研究表明,造成蛋白质异常的突变大部分是能造成单个氨基酸置换的编码区域非同义点突变[2]。这97193个特异性膜蛋白胞外突变是致使膜蛋白胞外区氨基酸发生置换的点突变,本研究对这些突变进行深入考察。

图1 特异性膜蛋白胞外区突变筛选流程图Fig.1 Flow chart of the selection of specific mutations in membrane surface proteins
2.2 特异性膜蛋白胞外突变数在肿瘤样本中的分布 统计结果显示,7042个样本中有5993个样本发生特异性膜蛋白胞外突变,单个样本特异性膜蛋白胞外突变数由1~811共有201类。突变数在100以上的样本主要来自6个肿瘤类型,分别为结直肠癌(colorectal cancer)、黑素瘤(melanoma)、子宫癌(uterine cancer)、脑胶质瘤(brain lower grade glioma)、肺腺癌(lung adenocarcinoma)和胃癌(stomach adenocarcinoma),这说明这些肿瘤被找到潜在靶点的可能性较大。而其他样本的突变数主要在1~20之间密集,10以下尤其密集(4207个样本,占比70.2%)。由于数据量庞大,仅取具有代表性的突变数即10以下和500以上的统计结果作展示,见图2。样本的特异性膜蛋白胞外突变数越大,该样本找到潜在靶点的可能性越大。由统计结果可知,30个肿瘤类型均有可能在肿瘤细胞膜表面蛋白上找到潜在靶点。

图2 单个样本特异性膜蛋白胞外突变数在肿瘤类型中的分布Fig. 2 The distribution of the number of specific mutations in membrane surface proteins in a single sample across human cancer types
2.3 涉及特异性膜蛋白胞外突变的基因 统计分析结果表明97193个特异性膜蛋白胞外突变共涉及到4347个基因。“基因出现次数”指所有样本在该基因上发生特异性膜蛋白胞外突变的数量总和,基因出现的次数越大,该基因发生特异性膜蛋白胞外突变的可能性越大。有28个基因含有超过200次的基因出现次数,见图3。
其中5个基因的出现次数大于600次,分别为ENSG00000181143 (MUC16)、ENSG00000168702(LRP1B)、ENSG00000164796(CSMD3)、ENSG00000198626 (RYR2)和ENSG00000042781(USH2A),见表1。除表1所示的基因功能信息外,有研究表明,胆囊癌和急性淋巴细胞白血病的体细胞突变中分别有CSMD3突变[18]和USH2A突变[19]。
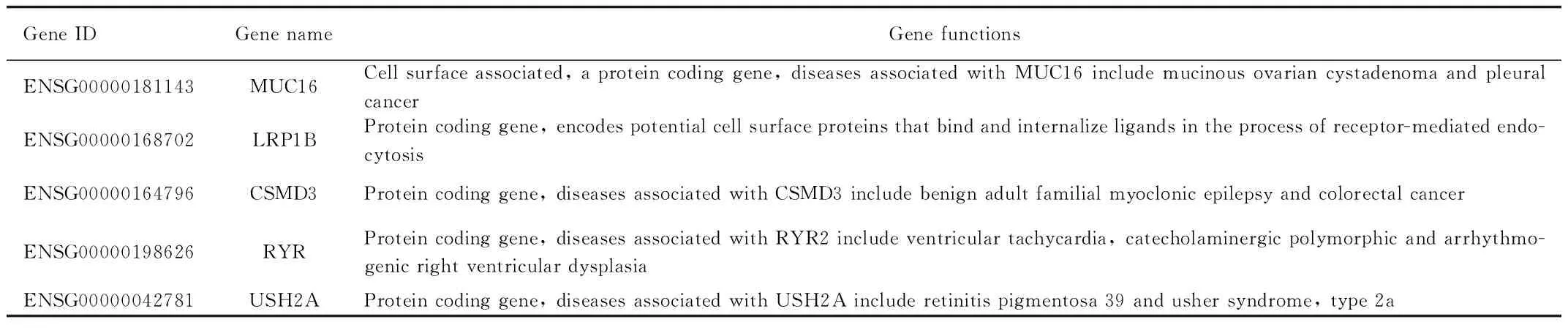
表1 基因功能信息表
2.4 涉及特异性膜蛋白胞外突变的基因位点 由统计分析结果可知,97193个特异性膜蛋白胞外突变是65532个基因位点上的突变。“基因突变位点出现次数”表示在该基因位点上发生特异性膜蛋白胞外突变的样本数,基因突变位点出现次数在各肿瘤类型的样本中的分布代表了该基因突变位点在30个肿瘤类型中的普及性。统计结果表明,大部分基因突变位点只出现1次,仅8个基因突变位点出现次数≥8,见表2。
对这8个基因突变位点进一步分析可得它们在30个肿瘤类型各个样本中的分布情况,见图4。统计结果表示,出现次数最高的基因突变位点是17:37868208(17号染色体的37868208位点),位于ERBB2蛋白质编码基因,且其涉及到的肿瘤类型最多,分别为膀胱癌、乳腺癌、宫颈癌、肺腺癌、卵巢癌和胃癌。发生特异性膜蛋白胞外突变的基因突变位点所涉及的肿瘤类型越多,越有助于拓宽靶向该突变位点药物的肿瘤治疗范围。

表2 基因突变位点出现次数

图4 高频次出现的基因突变位点在肿瘤类型中的分布基因突变位点“A:B”表示“染色体位置:该染色体上的位点”Fig.4 The distribution of gene mutation sites with high frequency of occurrence across human cancer types Gene mutation site “A:B” represents “chromosome:gene site”
3 讨论
本研究筛选出的氨基酸突变数比体细胞突变数多,这种现象与RNA的可变剪切(alternative splicing)有关,同一个基因的剪接位点及拼接位点可以不一致,这就导致了同一个基因能够编码多个转录产物并表达出多个不同生理功能的相关蛋白。RNA可变剪切可以增加真核生物基因表达的复杂程度和蛋白质的功能多样性。因此,同一个基因突变位点在不同转录水平上翻译得到的在氨基酸序列上的氨基酸突变位点可能不一致。
特异性膜蛋白胞外突变的“特异性”源于氨基酸突变后其性质发生了变化。氨基酸的性质主要包括酸碱性、亲水性和疏水性。氨基酸性质的变化可能会引起氨基酸带电荷情况的变化,从而引起相应膜蛋白的分子间作用力的变化。另外,氨基酸性质的变化也可能引起氢键及疏水作用力等的变化,进而影响相应膜蛋白与抗体之间的结合能力。所以,如果膜蛋白胞外氨基酸突变后性质发生了变化,则其与抗体的结合能力就可能区别于正常膜蛋白,从而形成特异性抗原作为药物作用靶点[20]。
传统的癌症治疗方法如手术、放疗、化疗缺乏肿瘤杀伤特异性,存在较大副作用。2015年初,美国总统奥巴马提出精准医疗计划,其中癌症精准医疗是最为重要的短期目标。精准医疗也逐渐成为癌症研究热点,癌症免疫治疗和靶向治疗可以通过肿瘤特异性抗原选择性地对肿瘤细胞造成特异性杀伤,从而降低副作用。因此,寻找肿瘤细胞区别于正常细胞的特异性抗原成为癌症精准医疗的关键。本研究进行的肿瘤细胞膜蛋白体细胞突变分析是对样本的所有体细胞突变进行分析,从中筛选出所有可能形成肿瘤特异性抗原的特异性膜蛋白胞外突变,即造成膜表面蛋白的氨基酸突变并使其性质发生变化的全部突变,构建了一个全面寻找肿瘤特异性膜表面潜在靶点的方法,建立了一个特异性膜蛋白胞外突变的信息库,并发现MUC16、LRP1B、CSMD3、RYR2、USH2A这5个基因及17号染色体的37868208位点(ERBB2基因位点)在肿瘤样本中发生特异性膜蛋白胞外突变的频率较高,为寻找癌症精准医疗,尤其是靶向治疗及免疫治疗的药物作用靶点提供信息参考。然而并不是所有特异性膜蛋白胞外突变都能形成药物作用靶点,这些突变有待后续抗原表位分析及实验验证。同时,本研究发现每个膜蛋白胞外突变位点在所有肿瘤样本中出现的频率较低(最高仅为12/7042),体现了肿瘤细胞极强的异质性,因而癌症精准医疗必将是个性化的。
[1] Alexandrov LB,Nik-Zainal S,Wedge DC,et al.Signatures of mutational processes in human cancer[J].Nature,2013,500(7463):415-421.
[2] Blankenstein T,Leisegang M,Uckert W,et al.Targeting cancer-specific mutations by T cell receptor gene therapy[J].Curr Opin Immunol,2015(33):112-119.
[3] Kampen KR.Membrane Proteins:The Key Players of a Cancer Cell[J].J Membrane Biol,2011,242(2):69-74.
[4] Fagerberg L,Jonasson K,von Heijne G,et al.Prediction of the human membrane proteome[J].Proteomics,2010,10 (6):1141-1149.
[5] Almén MS,Nordström KJV,Fredriksson R,et al.Mapping the human membrane proteome:a majority of the human membrane proteins can be classified according to function and evolutionary origin[J].BMC Biol,2009(7):50.
[6] Kim MS,Yi GS.HMPAS:Human Membrane Protein Analysis System[J].Proteome Science,2013,11(Suppl 1):S7.
[7] Beerli RR,Hell T,Merkel AS,et al.Sortase Enzyme-Mediated Generation of Site-Specifically Conjugated Antibody Drug Conjugates with High In Vitro and In Vivo Potency[J].PLoS ONE,2015,10(7):e0131177.
[8] Sliwkowski MX,Mellman I.Antibody Therapeutics in Cancer[J].Science,2013,341(6151):1192 -1198.
[9] Vlachakis D,Kossida S.Antibody Drug Conjugate Bioinformatics:Drug Delivery through the Letterbox[J].Comput Math Methods Med,2013(2013):282398.
[10] Li ZH,Zhang Q,Wang HB,et al.Preclinical studies of targeted therapies for CD20-positive B lymphoid malignancies by Ofatumumab conjugated with auristatin[J].Invest New Drugs,2014,32(1):75-86.
[11] Pan LQ,Wang HB,Xie ZM,et al.Novel Conjugation of Tumor-Necrosis-Factor-Related Apoptosis-Inducing Ligand (TRAIL) with Monomethyl Auristatin E for Efficient Antitumor Drug Delivery[J].Adv Mater,2013,25(34):4718-4722.
[12] Forbes SA,Bindal N,Bamford S,et al.COSMIC:mining complete cancer genomes in the Catalogue of Somatic Mutations in Cancer[J].Nucleic Acids Res,2011,39(suppl 1):D945-950.
[13] Ding L,Wendl MC,McMichael JF,et al.Expanding the computational toolbox for mining cancer genomes[J].Nat Rev Genet,2014,15(8):556-570.
[14] Uhlén M,Fagerberg L,Hallström BM,et al.Tissue-based map of the human proteome[J].Science,2015,347(6220):1260419.
[15] Safran M,Dalah I,Alexander J,et al.GeneCards Version 3:the human gene integrator[J].Database(Oxford),2010,2010:baq020.
[16] Huang DW,Sherman BT,Lempicki RA.Systematic and integrative analysis of large gene lists using DAVID bioinformatics resources[J].Nat Protoc,2009,4(1):44-57.
[17] Hubbard T,Barker D,Birney E,et al.The Ensembl genome database project[J].Nucleic Acid Res,2002,30 (1):38-41.
[18] Li M,Zhang Z,Li X,et al.Whole-exome and targeted gene sequencing of gallbladder carcinoma identifies recurrent mutations in the ErbB pathway[J].Nat Genet,2014,46(8):872-876.
[19] Ma X,Edmonson M,Yergeau D,et al.Rise and fall of subclones from diagnosis to relapse in pediatric B-acute lymphoblastic leukaemia[J].Nat Commun,2015(6):6604.
[20] Yu J,Kane S,Wu J,et al.Mutation-Specific Antibodies for the Detection of EGFR Mutations in Non-Small-Cell Lung Cancer[J].Clin Cancer Res,2009,15(9):3023-3028.
(编校:吴茜)
Analysis of somatic mutations in membrane proteins of tumor cells
WU Shan-shan, ZHOU ZhanΔ, CHEN Shu-qingΔ
(Zhejiang Province Key Laboratory of Anti-Cancer Drug Research, College of Pharmaceutical Sciences, Zhejiang University, Hangzhou 310058, China)
ObjectiveTo screen specific mutations on extracellular regions of membrane proteins (extracellular membrane protein mutations) in tumor cells and provide the reference information for target searching in cancer precision medicine.MethodsSomatic mutations on extracellular regions of membrane proteins of 7042 tumor samples were collected to screen all specific extracellular membrane protein mutations, and the overall distribution of these mutations were obtained by statistical analysis. Genes, gene site and cancer types occured high frequency of extracellular membrane protein mutations were identified.Results97193 specific extracellular membrane protein mutations were obtained from 4938362 somatic mutations in 7042 tumor samples (30 cancer types), the statistical analysis showed that 4347 genes and 65532 sites were involved in these specific mutations. The study further analyzed five genes (MUC16、LRP1B、CSMD3、RYR2、USH2A), one site (17:37868208) and six cancer types (including colorectal cancer, melanoma, uterine cancer, brain lower grade glioma, lung adenocarcinoma and stomach adenocarcinoma) which occured high frequency of extracellular membrane protein mutations.ConclusionAn information library of specific mutations on extracellular regions of membrane proteins was established and the distribution of these specific mutations was obtained which can provide reference information for target detection in targeted cancer therapy and immunological therapy.
cancer precision medicine; biological target; somatic mutations; specific extracellular membrane protein mutations
国家自然科学基金重点项目(81430081);浙江省自然科学基金面上项目(LY15C06001)
吴姗姗,女,硕士在读,研究方向:微生物与生化药学,E-mail:swodylm@zju.edu.cn;陈枢青,通信作者,男,博士,教授、博士生导师,研究方向:生化药学,E-mail:chenshuqing@zju.edu.cn;周展,共同通信作者,男,博士,讲师、硕士生导师,研究方向:癌症基因组学,E-mail:zhouzhan@zju.edu.cn。
R730.1
A
1005-1678(2015)11-0001-05
