剑豆SRAP-PCR反应体系的建立及优化
2015-06-23刘明骞陈丽君欧阳昆唏丁美美陈晓阳
刘明骞,陈丽君,欧阳昆唏,丁美美,陈晓阳
(1华南农业大学林学院/广东省森林植物种质创新与利用重点实验室,广东广州510642; 2华南农业大学教学科研基地管理中心,广东广州510642)
剑豆SRAP-PCR反应体系的建立及优化
刘明骞1,陈丽君2,欧阳昆唏1,丁美美1,陈晓阳1
(1华南农业大学林学院/广东省森林植物种质创新与利用重点实验室,广东广州510642; 2华南农业大学教学科研基地管理中心,广东广州510642)
【目的】建立和优化剑豆Canavalia ensiformis的SRAP-PCR体系,为分析剑豆遗传多样性和建立遗传图谱打下基础.【方法】首先用单因素试验法对SRAP-PCR反应体系的5个主要影响因素(Mg2+、dNTPs、引物、Taq DNA聚合酶、模板DNA)各设8个浓度梯度进行试验,找到5个因素的适宜浓度范围,再采用均匀设计法进行5因素4水平和5因素3水平的2轮优化.【结果和结论】建立了剑豆SRAP-PCR最佳反应体系(25μL):Mg2+1.75 mmol/L,dNTPs 200μmol/L,引物0.36μmol/L,Taq DNA聚合酶0.06 U/μL,模板DNA 40 ng.所建立的体系稳定可靠,适用于剑豆后续的SRAP分析.
剑豆;SRAP-PCR;均匀设计;优化
剑豆Canavalia ensiformis为豆科刀豆属植物,原产于墨西哥,现主要分布在亚洲、美洲、大洋洲等的热带和亚热带地区[1].剑豆在我国分布广泛,北至黄河流域,南至海南省都有剑豆的分布.剑豆适应性很强,生长期温度在14~27℃的热带、亚热带、温带地区均可生长.由于其根系发达,根瘤发达,耐盐、耐干旱、耐涝,能在蓄水差、养分缺乏、盐碱地等多种土壤中生存,其pH适应范围为4.3~6.8[1],可作为重要的改良土壤和覆草植物[2-3].
由于剑豆富含蛋白质、不饱和脂肪酸、必需氨基酸、维生素和矿物质等营养物质[4-6],被美国科学研究院认为是发展中国家和热带地区潜在的食物来源.剑豆种子中的刀豆蛋白A、脲酶等物质是应用十分广泛的工业和化工原料[7-9].目前对剑豆的研究主要集中在其营养物质的含量和刀豆蛋白A的生理生化作用方面,分子标记相关研究鲜见报道.
相关序列扩增多态性(Sequence-related amplified polymorphism,SRAP)是一种基于PCR的标记系统,由美国加州大学Li等[10]于2001年在研究芸薹属植物时开发.该标记具有简便、高效、产率高、高共显性、重复性好、易测序和便于克隆目标片段的特点,目前己被广泛应用于植物遗传多样性分析、品种鉴定、QTL定位以及遗传图谱的构建等方面的研究[11-14].本文以剑豆的叶片为试验材料,结合单因素试验设计和均匀试验设计,旨在建立剑豆的SRAP反应体系,为剑豆种质资源遗传多样性评价、品种鉴定、遗传图谱构建和重要性状分子标记辅助选择育种等奠定基础.
1 材料与方法
以原产地分别为中国、澳大利亚、巴西、以色列、巴拉圭和尼日利亚的6个剑豆种源为材料,采用改良的CTAB法提取其叶片DNA,利用8~10 g/L的琼脂糖凝胶电泳检测基因组DNA提取效果,并用超微量紫外分光光度计(Thermo nanodrop 2000)检测DNA浓度和纯度,然后将DNA质量浓度稀释为50 ng/μL,-20℃条件下贮存备用.
试验中所用Taq DNA聚合酶、MgCl2、dNTPs和DNA标准相对分子质量(Marker)DL2000均为TaKaRa公司产品,引物由上海生工生物工程有限公司合成,无水乙醇、Tris饱和酚、氯仿等试剂为国产分析纯.
以SRAP-PCR反应产物电泳得到的条带清晰、数目较多为筛选原则,对反应体系中起主要作用的5个因素进行浓度梯度筛选试验.得到各个因素的大概浓度范围,根据均匀设计的原理及特点,以上述单因素试验各因素的适宜浓度范围为基础,采用U16(45)均匀设计表(表1)对SRAP-PCR反应体系进行优化[15];在第1轮优化的基础上采用U12(35)均匀设计表(表2)进行第2轮均匀设计[16].SRAP-PCR反应程序为:94℃预变性5 min;94℃变性1 min,35℃复性1 min,72℃延伸1 min,5个循环;94℃变性1 min,50℃复性1 min,72℃延伸1 min,30个循环;72℃延伸10 min.
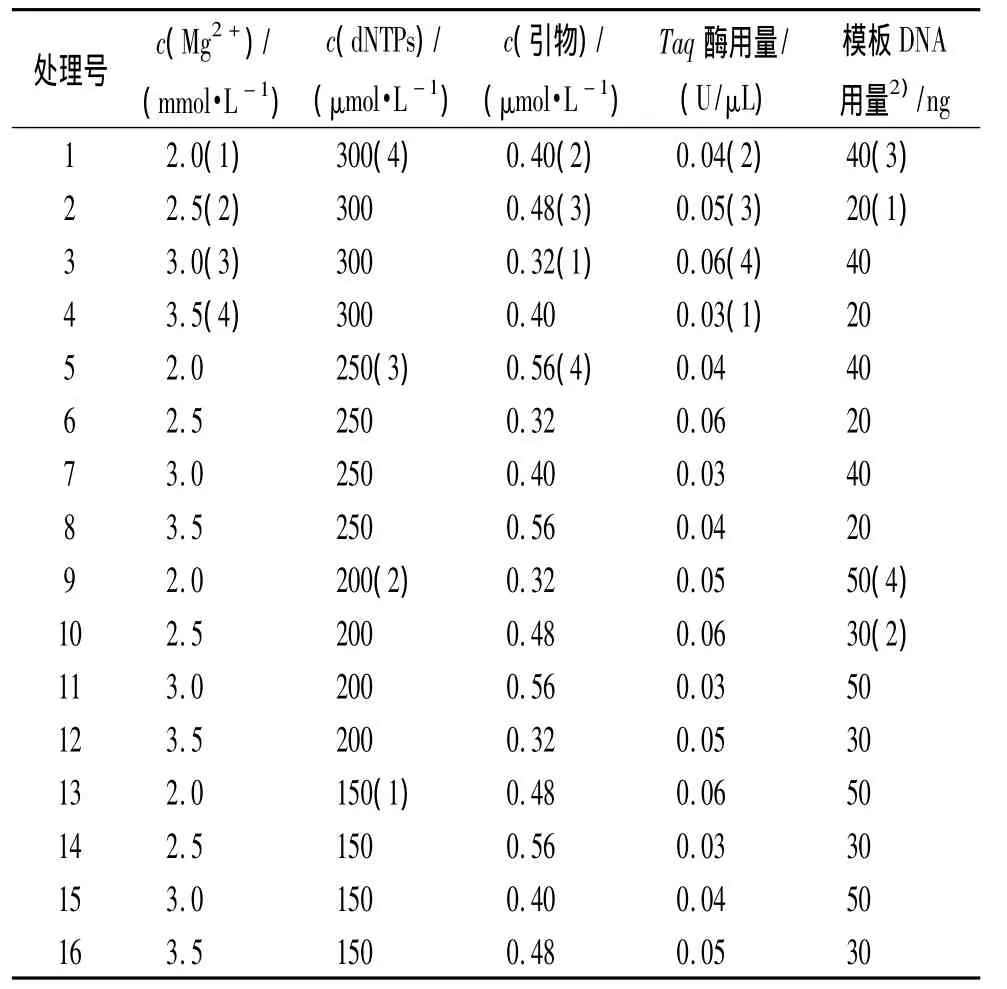
表1 SRAP反应第1轮均匀设计1)Tab.1 The first uniform design for Canavalia ensiform is SRAP reaction system

表2 SRAP反应第2轮均匀设计1)Tab.2 The second uniform design for Canavalia ensiform is SRAP reaction system
2 结果与分析
2.1 单因素试验分析
采用改良CTAB法提取剑豆基因组DNA,经琼脂糖凝胶电泳检测,主带清晰明显.紫外分光光度计检测结果显示:D260nm/D280nm为 1.8 ~2.0,D260nm/D230nm>2.0,说明DNA质量较好,可以用于后续的SRAP-PCR反应.
以SRAP-PCR反应产物电泳得到的条带清晰、数目较多为筛选原则,对反应体系中起主要作用的5个因素进行单因素浓度梯度筛选试验,每个因素设置8个浓度梯度.试验结果显示:当25μL反应体系中DNA用量为20~50 ng、Mg2+浓度范围为2.0~3.5 mmol/L、引物浓度为0.32~0.56μmol/L、Taq DNA聚合酶用量为0.03~0.06 U/μL、dNTPs浓度选取150~300μmol/L时扩增效果好,条带较多且清晰,故将其作为后续均匀设计的适宜浓度范围.
2.2 均匀设计优化
以上述单因素试验各因素的适宜浓度范围为基础,第1轮均匀设计优化结果如图1所示.各泳道均有较清晰的条带,说明单因素筛选试验结果比较可靠.泳道2、6、9、10的条带数目多且较清晰,其余泳道的条带较少或是部分条带较模糊.综合来看,泳道9(处理9)不仅效果好,且其dNTPs、Taq DNA聚合酶和引物的用量最少,最经济实惠.
以处理9为基础,第2轮均匀设计优化结果如图2所示.泳道3(处理3)效果最好,不仅条带最多,且非常清晰.故可以确定25μL的SRAP-PCR反应系中,含10×Buffer(Mg2+free)2.5μL,Mg2+1.75 mmol/L,dNTPs 200μmol/L,引物浓度为0.36μmol/L,Taq DNA聚合酶0.06 U/μL,DNA模板40 ng.
为了验证体系的准确性,以来自中国、澳大利亚、巴西、以色列、巴拉圭和尼日利亚的6个剑豆种源DNA为模板,选取引物M2E3、M3E2、M5E2进行SRAP-PCR验证,其结果如图3所示,每个种源对每个引物均有清晰的条带,且不同种源间条带有差异,由此可见,本试验建立的SRAP-PCR体系稳定可靠,适用于剑豆后续的SRAP分析.

图1 第1轮均匀设计优化SRAP扩增结果Fig.1 The result of the first uniform design for Canavalia ensiformis SRAP reaction system

图2 第2轮均匀设计优化SRAP扩增结果Fig.2 The result of the second uniform design for Canavalia ensiformis SRAP reaction system

图3 SRAP-PCR反应体系的验证Fig.3 The verification of Canavalia ensiformis SRAP-PCR system
3 讨论与结论
目前常使用的SRAP程序大多都是基于美国加州大学Li等[10]于2001年开发建立的程序,或者有微小的改动.SRAP引物是基于外显子富含GC而启动子富含AT的特点而设计的,其正向引物一般长17 bp,反向引物长18 bp,其退火温度大多为50℃左右,本试验采用Li等[10]的程序得到了较好的扩增效果.
本研究利用单因素设计对影响PCR反应的5个因素研究时发现,SRAP反应对其要求并不是非常严格,在一定的范围内均可得到清晰条带,利用均匀设计时也可以得到较为清晰的条带,这和孙佩光等[11]、郭大勇等[17]的试验结果一致.单因素试验结果显示: 25μL SRAP反应体系中模板DNA用量适宜范围为20~50 ng,Mg2+浓度适宜范围为2.0~3.5 mmol/L,引物浓度适宜范围为0.32~0.56μmol/L,Taq DNA聚合酶用量适宜范围为0.03~0.06 U/μL,dNTPs浓度范围为150~300μmol/L.
单因素设计能够快速找到各因子的适宜用量范围,而均匀设计不但能利用最少的试验次数来揭示出各因素对指标的影响程度和规律,而且还能从多个因素中找出影响试验结果的各主要因素和优化结果,另外,它还能通过建立的回归方程对最优试验条件进行有效性预测[15].因此笔者认为在进行体系优化时,先用单因素设计找出各个因子大概的适宜范围,再进一步使用均匀设计对体系进行优化,可以减少试验设计的盲目性,使试验的针对性更强,更容易得到理想的结果.本试验通过2轮均匀设计,得到稳定可靠的25μL SRAP-PCR反应体系为:Mg2+1.75mmol/L,dNTPs200μmol/L,引物0.36μmol/L,Taq DNA聚合酶0.06 U/μL,模板DNA 40 ng.可用于后续剑豆遗传多样性分析、种质鉴定和遗传连锁图谱的构建等.
[1] MORRIS JB.Swordbean(Canavalia ensiformis(L.) DC.)genetic resources regenerated for potentialmedical,nutraceutical and agricultural traits[J].Genet Resour Crop Evol,2007,54(3):585-592.
[2] POHLAN H A J,JANSSENSM JJ,EVERSBUSCH BG.Impact of canavalia cover crop management in Coffea arabica L.on plant-invertebrate associations[J].TOpen Agric J,2008,2:84-89.
[3] BALIGAR V C,FAGERIA N K.Agronomy and physiology of tropical cover crops[J].JPlant Nutr,2007,30(8): 1287-1339.
[4] SRIDHAR K R,SEENA S.Nutritional and antinutritional significance of four unconventional legumes of the genus Canavalia:A comparative study[J].Food Chem,2006,99(2):267-288.
[5] VADIVEL V,JANARDHANAN K.Diversity in nutritional composition of wild jack bean(Canavalia ensiformis L.DC)seeds collected from south India[J].Food Chem,2001,74(5):507-511.
[6] SIDDHURAJU P,BECKER K.Species/variety differences in biochemical composition and nutritional value of Indian tribal legumes of the genus Canavalia[J].Mol Nutr Food Res,2001,45(4):224-233.
[7] BELMAR R,NAVA-MONTER R,SANDOVAL-CASTROL C,et al.Jack bean(Canavalia ensifomis L.DC) in poultry diets:Antinutritional factors and detoxification studies[J].World Poultry Sci J,1999,55:37-59.
[8] AKPAPUNAM M A,SEFA-DEDEH S.Jack bean (Canavalia ensiformis):Nutrition related aspects and needed nutrition research[J].Plant Food Hum Nutr,1997,50 (2):93-99.
[9] MULINARI F,STANISCUASKI F,BERTHOLDO-VARGAS L R,et al.Jaburetox-2Ec:An insecticidal peptide derived from an isoform of urease from the plant Canavalia ensiformis[J].Peptides,2007,28(10):2042-2050.
[10]LIG,QUIROSC F.Sequence-related amplified polymorphism(SRAP),a new marker system based on a simple PCR reaction:Its application tomapping and gene tagging in Brassica[J].Theor Appl Genet,2001,103(2/3):455-461.
[11]孙佩光,奚如春,钮世辉,等.油茶SRAP-PCR反应体系的建立和优化[J].基因组学与应用生物学,2010,29 (6):1192-1199.
[12]LIAO L,GUO Q S,WANG Z Y,et al.Genetic diversity analysis of Prunella vulgaris in China using ISSR and SRAPmarkers[J].Biochem Syst Ecol,2012,45:209-217.
[13]MISHRA M K,SURESH N,BHAT A M,et al.Genetic molecular analysis of Coffea arabica(Rubiaceae)hybrids using SRAPmarkers[J].Rev Biol Trop,2011,59(2): 607-617.
[14]YOUSSEFM,JAMESA C,RIVERA-MADRID R,et al.Musa genetic diversity revealed by SRAP and AFLP[J].Mol Biotechnol,2011,47(3):189-199.
[15]郭丽琴,卫尊征,张金凤,等.均匀设计优化杨属的SRAP-PCR反应体系[J].北京林业大学学报,2010,32 (2):34-38.
[16]王卫,陈义挺,陈婷,等.均匀设计优化猕猴桃SRAP体系[J].福建农林大学学报:自然科学版,2013,42(1): 23-28.
[17]郭大勇,罗正荣,徐育海,等.湖北海棠SRAP-PCR反应体系的优化[J].湖北农业科学,2009,48(3):530-533.
【责任编辑 李晓卉】
Establishment and optim ization of SRAP-PCR system in Canavalia ensiformis
LIU Mingqian1,CHEN Lijun2,OU-YANG Kunxi1,DING Meimei1,CHEN Xiaoyang1
(1 College of Forestry,South China Agricultural University/Guangdong Key Laboratory for Innovative Development and Utilization of Forest Plant Germplasm,Guangzhou 510642;2 Center for Teaching&Research Base,South China Agricultural University,Guangzhou 510642,China)
【Objective】The purpose of this study is to establish the SRAP-PCR reaction system for Canavalia ensiformis.【Method】An optimization experiment with single factor design was conducted,comprising five factors of Taq DNA polymerase,Mg2+,primer,dNTPs and DNA template,each with eight concentration levels,aiming to screen their suitable concentration range.After that,uniform design U16(45)and U12(35)were operated in order to improve the accuracy.【Result and conclusion】The results showed that the optimum SRAP-PCR system was established,including Mg2+1.75mmol/L,dNTPs 200μmol/L,primers0.36μmol/L,Taq DNA polymerase 0.06 U/μL and DNA template 40 ng in the 25 μL reaction system.The reaction system is steady and dependable,which can be applied to the analysis of Canavalia ensiformis by SRAP.
Canavalia ensiformis;SRAP-PCR;uniform design;optimization
S643.2
A
1001-411X(2015)01-0075-04
刘明骞,陈丽君,欧阳昆唏,等.剑豆SRAP-PCR反应体系的建立及优化[J].华南农业大学学报,2015,36(1):75-78.
2014-02-21 优先出版时间:2014-12-02
优先出版网址:http://www.cnki.net/kcms/doi/10.7671/j.issn.1001-411X.2015.01.014.html
刘明骞(1987—),男,博士研究生,E-mail:liumq0123@163.com;陈晓阳(1958—),男,教授,博士,E-mail:xychen @scau.edu.cn.
广州市科技计划项目(2010Z1-E241);农业部948项目(2011-Z50)
