基于蛋白质组学的尿液中肾脏疾病相关蛋白的筛选
2015-06-07郑春霞李芙蓉刘志红
王 玉 郑春霞 李芙蓉 王 霞 左 科 刘志红
基于蛋白质组学的尿液中肾脏疾病相关蛋白的筛选
王 玉 郑春霞 李芙蓉 王 霞 左 科 刘志红
目的:通过对正常人血浆、尿液、肾组织及肾脏疾病患者尿液蛋白质组学获取的数据进行比较分析,寻找尿液中与肾脏疾病及肾脏损伤相关的生物标志物。 方法:肾脏疾病患者尿液蛋白质组数据来自表现为肾病综合征的膜性肾病(MN)和局灶节段性肾小球硬化(FSGS)患者尿液蛋白质组谱图,从PeptideAtlas 2013数据库获得正常人血浆、尿液和肾组织蛋白质组数据。寻找肾脏疾病患者尿液蛋白质组相对正常人尿液蛋白质组富集蛋白,将筛选到的蛋白定义为肾脏疾病相关蛋白。寻找正常人血浆和尿液蛋白质组中不出现,同时在肾脏疾病患者尿液和肾组织蛋白质组中出现,且相对正常人尿液蛋白质组富集的蛋白,将筛选到的蛋白定义为肾损伤相关蛋白。 结果:筛选到40 种肾脏疾病相关蛋白。发现4种与肾损伤相关的蛋白,分别为泛素-60s核糖体蛋白L40、S相激酶相关蛋白1、转化生长因子βⅡ型受体、谷胱甘肽S转移酶A3。 结论:通过对肾脏疾病患者尿液蛋白质组学数据与正常人血浆、尿液和肾组织的蛋白质组学数据进行比较分析,从患者尿液中获得了40 种与肾脏疾病相关的蛋白和4种肾脏组织损伤相关的蛋白。上述蛋白成分在肾脏疾病诊断及病情判断中的意义有待进一步研究阐明。
肾脏疾病 尿液 蛋白质组学 生物标志物
人体从尿液中排泄出蛋白的数量及其成分是反映肾功能和疾病状态的重要指标,当发生肾损伤时,不仅尿蛋白量增加,同时尿液中蛋白成分的变化可能携带反映肾损伤的一些信息。因此,尿液可能是较血液更好的寻找肾脏疾病标志物的来源[1]。
受来自血液中高丰度蛋白的干扰,对表现为蛋白尿尤其是大量蛋白尿的肾脏疾病患者的尿蛋白质组学探索较为困难。长期以来只能检出蛋白质组中一些高丰度的蛋白,探索范围较为狭窄[2-4]。我们前期利用白蛋白/IgG抗体清除联合二维液相色谱分离的方法,有效地减轻了伴大量蛋白尿的患者尿液蛋白质组的高丰度抑制,获得了伴大量蛋白尿的肾小球疾病患者尿液蛋白质组谱图[5]。
Peptide Atlas是人类蛋白质组计划的一个重要组成部分,致力于收集和再处理已发表的各类蛋白质组数据。2013年Peptide Atlas将收集的数据分类处理为正常人血浆、尿液和肾组织三个蛋白质组数据库。并对蛋白质谱图数进行标准化处理,为进行跨蛋白质组水平的比较研究提供了有效的工具[6]。
本研究对肾脏疾病患者尿液、正常人血浆、正常人尿液、正常人肾组织这四个密切联系的蛋白质组进行比较分析,以期寻找疾病相关的生物标志物。
方 法
肾脏疾病患者尿液蛋白质组数据从30例患者尿液中获取,其中膜性肾病(MN)15例,局灶节段性肾小球硬化(FSGS) 15例。蛋白质组学研究策略见前期工作[5],简要介绍如下:来自每例患者的新鲜尿液丙酮沉淀获得富集蛋白,取等量蛋白混合,清除混合样品白蛋白/IgG,酶切获取多肽,行二维反相色谱分离联合串联质谱分析。质谱结果通过Mascot和Scafford软件分析获取鉴定蛋白和谱图数。
正常人血浆、正常人尿液、正常人肾组织蛋白质组数据来自于PeptideAtlas 2013非冗余swiss-prot蛋白质数据库[6]。由于本研究肾脏疾病患者尿液蛋白质组每个蛋白特异性多肽≥2,为使各组数据具有可比性,限定进入本研究的其他各组蛋白满足特异性多肽≥2。
根据简化APEX(Absolute Protein Expression Estimates)公式计算蛋白质组内蛋白的标准化谱图数(normalized spectral count,NSC)[7],简化APEX公式如下:

结 果
正常人尿液中肾脏来源蛋白成分的筛选 经分析共获取符合条件的正常人尿液蛋白质组蛋白1 940个,正常人血浆蛋白质组蛋白2 568个,正常人肾组织蛋白质组蛋白3 375个。其中, 71%(1 379/1 940)的正常人尿液蛋白在正常人血浆蛋白质组中出现,63%(1 229/1 940)的正常人尿液蛋白在正常人肾组织蛋白质组中出现。将同时在正常人尿液和肾组织中检出,但在血浆中未能检出的这组蛋白称为正常人尿液中肾脏来源蛋白(图1),共获得276个蛋白。

图1 正常人尿液中肾脏来源蛋白成分的筛选
肾脏病患者尿液中肾脏来源蛋白成分的筛选 从肾脏疾病患者尿液中获取到809个蛋白。其中,84%(677/809)的肾脏疾病患者尿液蛋白在正常人血浆蛋白质组中出现, 55%(446/809)的肾脏疾病患者尿液蛋白在正常人肾组织蛋白质组中出现。将同时在肾脏疾病患者尿液和正常人肾组织检出,但在正常人血浆中未能检出的这组蛋白称为肾脏疾病患者尿液中肾脏来源蛋白(图2),共获得34个蛋白。
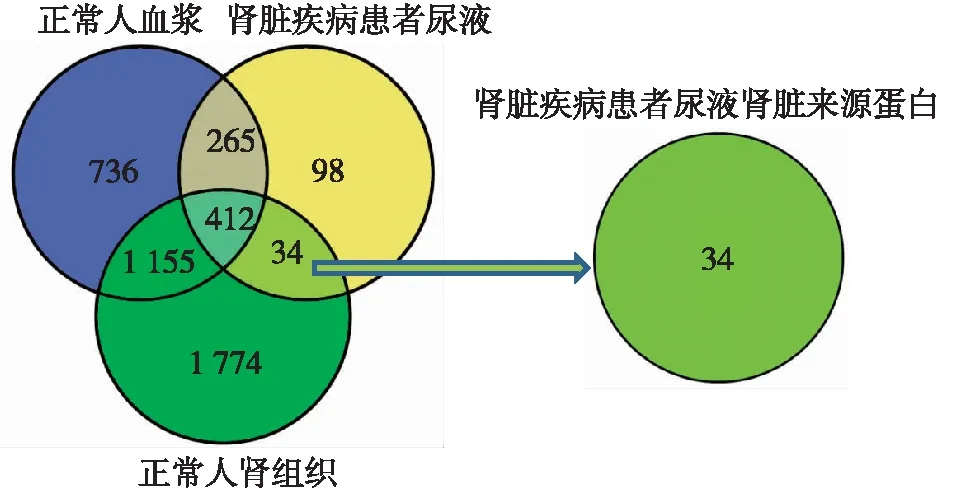
图2 肾脏疾病患者尿液中肾脏来源蛋白成分的筛选
肾脏疾病相关蛋白的筛选 对肾脏疾病患者尿液、正常人尿液蛋白谱图数进行标准化处理,参考Terry等提出的蛋白质丰度富集计算方法[6]:将蛋白质组A中某蛋白的NSC除以蛋白质组B中对应蛋白的NSC(没有在蛋白质组A或蛋白质组B中出现的蛋白考虑为极低浓度蛋白。为进行蛋白质丰度富集分析,假定未出现的蛋白质NSC为该蛋白质组最小NSC的1/2),对获得的比值进行log转换,σ为log转换后比值的标准差。如果蛋白质组A中某蛋白的logNSC是蛋白质组B中该蛋白logNSC的2σ
倍以上,则认为该蛋白在蛋白质组A中相对于白质组B富集。根据以上方法计算肾脏疾病患者尿液蛋白质组相对于正常人尿液蛋白质组富集蛋白,获得大于2σ倍以上的蛋白共40 个,我们将其称为“肾脏疾病相关蛋白”(图3,表1)。肾脏疾病相关蛋白在肾脏病患者尿液蛋白质组中排序远远大于在正常人尿液蛋白质组中排序,提示其可能是与疾病状态有关的异常蛋白。
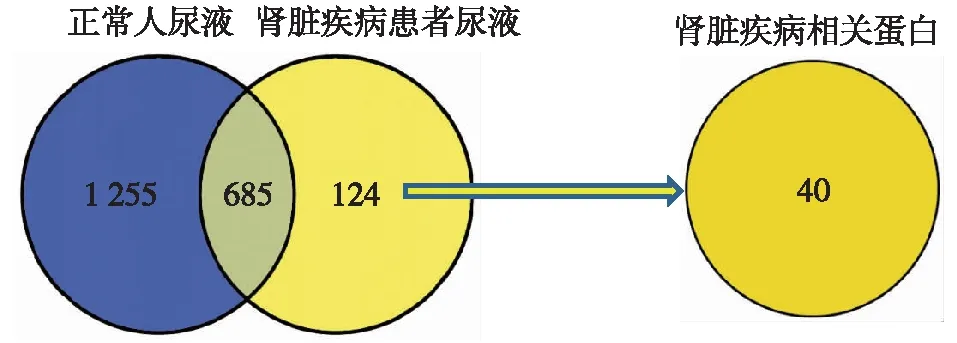
图3 肾脏疾病相关蛋白的筛选示意图

表1 肾脏疾病相关蛋白
肾损伤相关蛋白的筛选 将肾脏疾病患者尿液中肾脏来源蛋白、正常人尿液中肾脏来源蛋白进行比较,挑选出肾脏疾病患者尿液中独有的肾脏来源蛋白。这部分蛋白更可能来源于受损的肾脏组织,从而提供肾脏损伤的相关信息。由于原尿在肾脏形成后,还要流经泌尿道才形成我们可分析的尿液,因此,我们获得的肾脏疾病患者尿液中肾脏来源蛋白有可能受到泌尿道蛋白的污染。为进一步减少这种可能性及增加筛选蛋白作为肾损伤蛋白的可信性,我们将获得的肾脏疾病患者尿液独有肾脏来源蛋白与肾脏病相关蛋白进行比较,挑选出二者重合的蛋白,我们将其称为“肾损伤相关蛋白”(图4,表2)。

图4 肾脏损伤相关蛋白的筛选流程图

表2 肾损伤相关蛋白
讨 论
利用蛋白质组学在肾脏疾病患者尿液中筛选到能够帮助疾病诊断、判断病情和预后的标志物,一直是人们努力的方向。但大量蛋白尿肾脏疾病患者尿液中血液来源的高丰度蛋白阻碍了研究的深入。我们前期研究通过一系列措施减轻高丰度抑制,获得了一份较全面的伴大量蛋白尿肾小球疾病患者尿液蛋白质组学图谱。此外,不同体液、组织蛋白质组数据库的建立提供了从蛋白质组水平进行比较、寻找疾病标志物的机会。本研究通过对肾脏疾病患者尿液蛋白质组和正常人尿液蛋白质组进行比较分析,筛选出40种肾脏疾病相关蛋白。结合文献,以下列3个蛋白为例,介绍这些蛋白可能的意义。
甘露糖结合凝集素(MBL)是一种急性时相反应蛋白,参与补体甘露糖结合凝集素途径的活化。IgA肾病患者尿液中MBL较正常对照明显增多,且与预后相关[8]。补体甘露糖结合凝集素途径的活化在MN发病中发挥着重要作用,对MN肾活检组织进行免疫荧光检测后发现MBL沉积[9]。
蛋白Z依赖性蛋白酶抑制剂(PZI)是一种维生素K依赖的丝氨酸蛋白酶,在Protein Z存在的情况下显著抑制凝血因子Xa,还能直接抑制凝血因子XIa[10]。PZI基因敲除的小鼠在外界刺激下血栓形成明显增强[11]。静脉血栓形成是肾病综合征常见的并发症,PZI在尿液中的过量丢失是否参与肾病综合征血栓的形成有待研究证实。
羧肽酶,又称凝血酶激活的纤溶抑制物(TAFI),是一种分泌性蛋白质。TAFI由肝脏分泌,在外周循环被凝血酶激活,通过清除纤维蛋白溶酶原的C末端残基下调纤溶过程[12]。血浆中TAFI浓度升高被认为是血栓形成的危险因素[13]。疾病状态下尿液中异常排泄的TAFI是否代表了机体对血液中异常升高TAFI的一种代偿调节机制,TAFI能否成为肾病综合征血栓形成的标志物有待进一步探索。
通过对肾脏疾病患者尿液蛋白质组和正常人尿液、血浆和肾组织蛋白质组进行比较,发现4种可能与肾脏损伤相关的蛋白,分别为泛素-60s核糖体蛋白L40、S相激酶相关蛋白1、转化生长因子βⅡ型受体、谷胱甘肽S转移酶A3。这组蛋白的功能及其在肾组织中的分布特点,提示它们有可能参与疾病状态下肾组织损伤的发生。
泛素-60s核糖体蛋白L40(Ubiquitin-60S ribosomal protein L40,UBA52)是由泛素和60s核糖体蛋白L40组成的融合蛋白。泛素是一种存在于大多数真核细胞中的蛋白,主要功能是标记需要分解掉的蛋白质,使其被水解;60s核糖体蛋白L40是核糖体60s亚基的组成部分。UBA52蛋白在肾小球和肾小管均有丰富表达[14],在链霉菌素诱导的糖尿病新生小鼠肾组织内,UBA52 mRNA水平较正常对照明显升高[15]。
S相激酶相关蛋白1(SKP1)是SCF(SKP1-CUL1-F-box protein)泛素连接酶复合体的基本成分,SCF复合体通过介导蛋白的泛素化参与细胞周期调控,信号传导及转录。SKP1主要表达于肾小管,少量表达于肾小球[14]。
转化生长因子βⅡ型受体(TGFBR2)主要由Ⅰ 型/Ⅱ 型受体(GFBR1/TGFBR2 )组成,为丝氨酸/苏氨酸激酶。配体与TGFBR2结合后,TGFBR2自身磷酸化,紧接着TGFBR1 相互聚积成复合体或与TGFBR2结合呈嵌合态,然后TGFBR1的蛋白激酶磷酸化再将信号传递到下游物质,完成信号传递[16]。TGFBR2在肾纤维化和炎症中发挥着多种作用。Meng等[17]将单侧输尿管梗阻模型的TGFBR2基因敲除,发现肾脏纤维化得到抑制,同时伴随着TGF-β/Smad3信号通路的抑制、核因子κB信号通路增强及前炎症因子表达上调。
谷胱甘肽S转移酶A3 (GSTA3)是谷胱甘肽S转移酶家族的一员,GST是具有解毒功能的三大酶系之一。除了解毒作用,GST也参与胞内物质转运、激素的合成、细胞氧化应激损伤的保护。在肾脏,GSTA3主要见于肾小管,肾小球也可见少量表达[14]。在链霉菌素诱导的糖尿病小鼠肾组织中,Gong等[18]运用iTRAQ标记的蛋白质组学技术发现GSTA3表达较正常对照下调。Shin等[19]使用顺铂诱导小鼠发生急性肾损伤,发现尿液GSTA3较正常对照明显升高。
本研究的不足之处是没有这组患者血浆蛋白质组数据,只能使用正常人血浆蛋白质组数据。另外,无法排除肾病综合征患者血浆蛋白质组某蛋白较正常人血浆发生明显异常,导致其在正常人血浆和尿液中没有,但在蛋白尿中异常富集。
从尿液中寻找无创性的肾脏疾病诊断、判断病情和预后的标志物具有重要的临床实际应用价值。由于蛋白质组学技术的进步和生物信息学的发展,各种组织、体液蛋白质组学数据库正在被构建和健全,从而为组学水平上进行蛋白质组数据的比较研究提供了可能。本研究利用肾脏疾病患者尿液蛋白质组数据,结合已有的正常人血浆、尿液和肾组织蛋白质组数据库,筛选出40个肾脏疾病相关蛋白和4个候选肾损伤标志物。上述蛋白成分在肾脏疾病诊断及病情判断中的意义有待进一步研究阐明。
1 Gao Y.Urine is a better biomarker source than blood especially for kidney diseases.Adv Exp Med Biol,2015,845:3-12.
2 Candiano G,Musante L,Bruschi M,et al.Repetitive fragmentation products of albumin and alpha1-antitrypsin in glomerular diseases associated with nephrotic syndrome.J Am Soc Nephrol,2006,17(11):3139-3148.
3 Varghese SA,Powell TB,Budisavljevic MN,et al.Urine biomarkers predict the cause of glomerular disease.J Am Soc Nephrol,2007,18(3):913-922.
4 Bhensdadia NM,Hunt KJ,Lopes-Virella MF,et al.Urine haptoglobin levels predict early renal functional decline in patients with type 2 diabetes.Kidney Int,2013,83(6):1136-1143.
5 王玉,孙伟,左科,等.肾小球疾病患者尿液比较蛋白质组学研究方法的建立.肾脏病与透析肾移植杂志,2014,23(2):141-143.
6 Farrah T,Deutsch EW,Omenn GS,et al.State of the human proteome in 2013 as viewed through peptideatlas:comparing the kidney,urine,and plasma proteomes for the biology- and disease-driven human proteome project.J Proteome Res,2014,13(1):60-75.
7 Lu P,Vogel C,Wang R,et al.Absolute protein expression profiling estimates the relative contributions of transcriptional and translational regulation.Nat Biotechnol,2007,25(1):117-124.
8 Liu LL,Jiang Y,Wang LN,et al.Urinary mannose-binding lectin is a biomarker for predicting the progression of immunoglobulin (Ig)A nephropathy.Clin Exp Immunol,2012,169(2):148-155.
9 Segawa Y,Hisano S,Matsushita M,et al.IgG subclasses and complement pathway in segmental and global membranous nephropathy.Pediatr Nephrol,2010,25(6):1091-1099.
10 Girard TJ,Lasky NM,Tuley EA,et al.Protein Z,protein Z-dependent protease inhibitor (serpinA10),and the acute-phase response.J Thromb Haemost,2013,11(2):375-378.
11 Zhang J,Tu Y,Lu L.et al.Protein Z-dependent protease inhibitor deficiency produces a more severe murine phenotype than protein Z deficiency.Blood,2008,15,111(10):4973-4978.
12 Fidan E,Kavgaci H,Orem A, et al.Thrombin activatable fibrinolysis inhibitor and thrombinantithrombin-III-complex levels in patients with gastric cancer.Tumour Biol,2012,33(5):1519-1525.
13 Li SJ,Guo JZ,Zuo K,et al.Thromboembolic complications in membranous nephropathy patients with nephrotic syndrome-a prospective study.Thromb Res,2012,130(3):501-505.
14 Uhlen M,Oksvold P,Fagerberg L,et al.Towards a knowledge-based Human Protein Atlas.Nat Biotechnol,2010,28(12):1248-1250.
15 Wada J,Sun L,Kanwar YS.Discovery of genes related to diabetic nephropathy in various animal models by current techniques.Contrib Nephrol,2011,169:161-174.
16 Kawabata M,Imamura T,Miyazono K,et al.Interaction of the transforming growth factor-beta type I receptor with farnesyl-protein transferase-alpha.J Biol Chem,1995,270(50):29628-29631.
17 Meng XM,Huang XR,Xiao J,et al.Diverse roles of TGF-β receptor II in renal fibrosis and inflammation in vivo and in vitro.J Pathol,2012,227(2):175-188.
18 Gong D,Chen X,Middleditch M,et al.Quantitative proteomic profiling identifies new renal targets of copper(II)-selective chelation in the reversal of diabetic nephropathy in rats.Proteomics,2009,9(18):4309-4320.
19 Shin YJ,Kim TH,Won AJ,et al.Age-related differences in kidney injury biomarkers induced by cisplatin.Environ Toxicol Pharmacol,2014,37(3):1028-1039.
(本文编辑 青 松)
Screening of urinary disease-related proteins in patients with kidney disease based on proteomics data analysis
WANGYu,ZHENGChunxia,LIFuyong,WANGXia,ZUOKe,LIUZhihong
NationalClinicalResearchCenterofKidneyDisease,JinlingHospital,NanjingUniversitySchoolofMedicine,Nanjing210016,China
LIUZhihong(E-mail:liuzhihong@nju.edu.cn)
Objective:To explore kidney disease-related proteins and kidney damage-related proteins in urine of patients with kidney disease. Methodology:Urine proteomic data of patients with kidney disease came from urine proteomic profiles of FSGS and MN with nephrotic syndrome.Plasma, urine and kidney proteomic data of healthy subjects were obtained from Peptide Atlas 2013.The data from urine proteomic of patients with kidney disease were compared to the data from plasma, urine and kidney proteomic of healthy subjects.The proteins which enriched in urine proteomic of patients with kidney disease to urine proteomic of healthy subjects were named by kidney disease-related protein.Coincidence analysis for above four proteomics was made to search kidney damage-related proteins which are not in plasma proteomic of healthy subjects, shared in urine proteomic of patients with kidney disease and kidney proteomic of healthy subjects, and enriched in urine proteomic of patients with kidney disease to urine proteomic of healthy subjects. Results:A total of 40 kidney disease-related proteins and 4 kidney damage-related proteins, Ubiquitin-60S ribosomal protein L40, S-phase kinase-associated protein 1, TGF-beta receptor type-2 and Glutathione S-transferase A3 was found. Conclusion:By comparing data from above four proteomic, 40 kidney disease-related proteins and 4 kidney damage-related proteins were achieved from urine of patients with kidney disease.These proteins need to be further determined.
kidney diseases urine proteomic biomarker
国家重点基础研究发展计划(973计划)[2012CB517600(2012CB517606)],江苏省自然科学基金项目(BK2012054)
南京大学医学院附属金陵医院(南京军区南京总医院)肾脏科 博士研究生(王 玉),国家肾脏疾病临床医学研究中心 全军肾脏病研究所(南京,210016)。
刘志红(E-mail:liuzhihong@nju.edu.cn)
ⓒ 2015年版权归《肾脏病与透析肾移植杂志》编辑部所有
2015-01-29
