割手密过氧化物酶基因SsPOD—1cDNA克隆与功能分析
2015-05-30胡小文姚艳丽邢淑莲徐磊刘洋
胡小文 姚艳丽 邢淑莲 徐磊 刘洋

摘 要 割手密(Saccharum spontaneum)属于甘蔗属细茎野生种,具有较强的抗逆性,是甘蔗育种的优良材料。过氧化物酶(POD)是活性氧系统(ROS)中的一个关键酶,是植物应对干旱胁迫的关键调控酶之一。利用同源克隆的方法,以割手密为材料,成功克隆2个POD等位基因序列(SsPOD-1a和SsPOD-1b,GenBank登录号为KJ002565和KJ002566)。经测序鉴定,这2个基因长度均为1 083 bp,其中包括27 bp的5′非编码区、60 bp的3′非编码区及一个996 bp的开放阅读框,共编码331个氨基酸,这2个等位基因的序列相似性为98.8%。蛋白质保守结构域分析结果表明,此2个基因均具有POD基因典型保守结构域,属于依赖于亚铁血红素Ⅲ型POD亚类。原核表达结果表明,这2个基因编码区能正常表达出目的蛋白,说明得到的这2个基因为功能基因,而不是假基因。POD基因分子系统分生分析结果表明,割手密POD基因与高粱、玉米、水稻等禾本科植物亲缘关系较近,与一些双子叶植物及裸子植物也有一定的亲缘关系,这也证实了POD基因起源较为古老且存在高度保守区。
关键词 割手密;POD;基因克隆;抗旱
中图分类号 S566.1 文献标识码 A
Abstract Saccharum spontaneum L. is a wild variety of sugarcane with tolerance to drought-stress and can be used in sugarcane breeding programs. Gene encoding peroxidases respond to stress-related signals and are believed to play key roles in these processes. In this study, two novel peroxidase(POD) allele genes(SsPOD-1a and SsPOD-1b) were cloned by using the homologous cloning strategy. Sequencing data showed both of the two cloned genes had 1 083 base pair, including 5′ untranslated regions 27 bp, 3′ untranslated regions 60 bp, and a 996 bp ORF, which coding 331 amino acid, and the similarity of the two allele gene is 98.8%. The structural domain of proteins analysis showed the two genic protein had typical well-conserved amino-acid residues about peroxidases, and were classified to heme containing class Ⅲ peroxidases. The protein expression experiment revealed that both of gene coding areas could express aim protein, indicating SsPOD-1a and SsPOD-1b were not pseudogene, but function genes. The phylogeny analysis indicated that peroxidase genes of S. spontaneum L. were similar to the grass family, including Sorghum bicolor, Zea maize and Oryza sativa, also had relationship to the dicotyledon plants, even to some gymnosperms, vertifying the origin of peroxidase genes are old and exist well-conserved region.
Key words Saccharum spontaneum L.;Peroxidases;Gene clone;Drought resistance
doi 10.3969/j.issn.1000-2561.2015.07.017
割手密(Saccharum spontaneum)属于复杂多倍体甘蔗属细茎野生种,现大多数甘蔗栽培种是割手密与热带甘蔗栽培种的杂交后代。割手密分布范围广、类型多、分蘖多、宿根性好、抗逆性强等优良性状,是甘蔗改良育种的优良材料[1]。植物在受干旱胁迫时,细胞内会产生大量的活性氧,而活性氧的大量积累会严重威胁植物的生存[2]。过氧化物酶(POD)以H2O2为电子受体催化底物氧化,是植物体内有效清除活性氧的关键酶之一,对植物应对逆境胁迫有非常重要的作用[3]。植物中POD的结构和反应机理已研究的较为清楚[4-5],拟南芥、水稻、小麦、棉花等植物POD基因的研究也均有报道[6-9],但抗旱能力非常强的割手密其POD基因相关研究还未曾报道,割手密POD基因的研究有助于阐明高抗旱植物水分代谢调控机理。因此,本研究以与割手密亲缘关系较近的高粱POD基因序列为探针,利用同源克隆技术克隆获得割手密POD基因,通过对其基因结构及遗传关系的比较分析,旨在基因水平上对POD基因在植物抗旱性方面有一些新的认识,从而为进一步通过生物技术改良甘蔗品种的抗逆性提供理论依据。
1 材料与方法
1.1 材料
1.1.1 植物材料采集 本试验所采用的割手密来源于云南国家甘蔗种质资源圃割手密82-114,在广东湛江进行盆栽试种,出苗2个月后,取顶端第2片叶于液氮中速冻,提取总RNA。
1.1.2 主要试剂 RNA提取采用北京全式金生物技术有限公司EasyPure Plant RNA Kit试剂盒,反转录采用Thermo Scientific公司的 Thermo Scientific RevertAid Reverse Transcriptase试剂盒,DNA聚合酶采用大连宝生物公司的Master Mix Taq(已包含Loading Bufer和dNTPs),DNA纯化采用天根生化科技有限公司的“普通DNA产物纯化试剂盒”,基因克隆转化采用北京全式金公司的pEASY-T5 Zero Cloning Kit。基因蛋白的原核表达采用北京全式金公司的pEASY-E1 Expression Kit试剂盒。
1.2 方法
1.2.1 RNA的提取 按照EasyPure PlantRNA kit试剂盒提供的方法提取割手密叶片的RNA。
1.2.2 cDNA的合成 利用Thermo Scientific RevertAid Reverse Transcriptase试剂盒对获得的RNA进行反转录,获得到割手密的cDNA。
1.2.3 割手密POD基因cDNA参考序列的推导 以与割手密亲缘关系较近的高粱POD基因cDNA序列为探针,在NCBI中检索甘蔗属(Saccharum officinarum complex)EST数据库,获得覆盖编码区的相似性最高的EST序列,再将这些EST序列进行拼接,去除重复区,获得假定的割手密POD基因的cDNA序列。
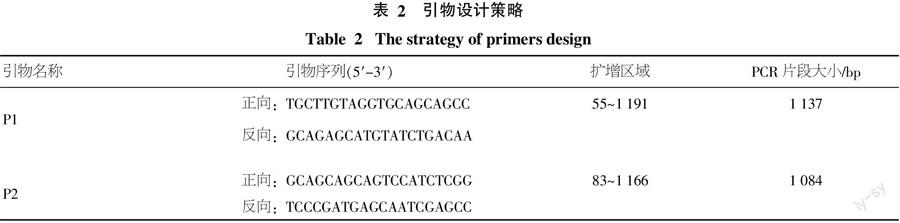
1.2.4 引物设计与PCR扩增 为提高PCR扩增的产量和特异性,采用巢式PCR,以假定的割手密POD基因的cDNA序列为模板,设计2对引物对目标区域进行2轮PCR扩增。2轮PCR程序均采用:94 ℃预热5 min;然后94 ℃预热30 s,60 ℃退火30 s,72 ℃延伸90 s ,30个循环;最后72 ℃延伸10 min。采用琼脂糖凝胶电泳检测PCR产物,将目的PCR产物利用“普通DNA产物纯化试剂盒”进行纯化。
1.2.5 基因克隆与检测 将纯化的目的PCR产物按照“pEASY-T5 Zero Cloning Kit”试剂盒的方法转入克隆载体,利用感受态细胞对克隆进行转化,PCR扩增鉴定阳性重组子,挑选2个阳性克隆送到英骏(广州)生物技术公司测序部进行测序,获得割手密POD基因的cDNA序列。最后对获得的序列进行基因结构功能分析与预测。
1.2.6 SsPOD-1a和SsPOD-1b基因的原核表达 将克隆得到的SsPOD-1a和SsPOD-1b基因利用PCR得到其蛋白表达区(ORF),按照pEASY-E1 Expression Kit试剂盒的方法步骤构建原核表达载体,将得到的表达载体转入BL21(DE3)pLysS感受态细胞,接入新鲜LB液体培养基37 ℃,200 r/min培养到OD600为0.5后加入IPTG(终浓度为1 mmol/L)诱导目的基因表达。到达一定时间后,取1 mL菌液,12 000 r/min离心10 min,弃上清,加入100 μL的ddH2O重悬,并加入等体积的2X SDS凝胶上样缓冲液(100 mmol/L Tris-Cl pH6.8,20%甘油,0.2%溴酚蓝,4%SDS,2%DDT),沸水加热10 min,4 000 r/min离心5 min,取上清液进行SDS-PAGE。电泳缓冲液为TGE,分离胶浓度为12%,浓缩胶浓度为5%,电泳电压为120 V。电泳完成后用考马斯这蓝R-250(质量浓度为0.1%)染色1 h。染色完成后采用脱色液进行脱色(冰乙酸 ∶ 甲醇 ∶ H2O=1 ∶ 3 ∶ 6)。
2 结果与分析
2.1 获得割手密POD基因cDNA参考序列
以高粱POD基因cDNA序列(XM_002437414.1)为探针,采用1.2.3介绍的方法,提取3条与XM_002437414.1相似性较高且覆盖编码区的EST序列(表1和图1),对这3条EST序列进行拼接去除重复区后,获得1 409 bp的割手密POD基因的cDNA参考序列。
2.2 引物设计与PCR扩增
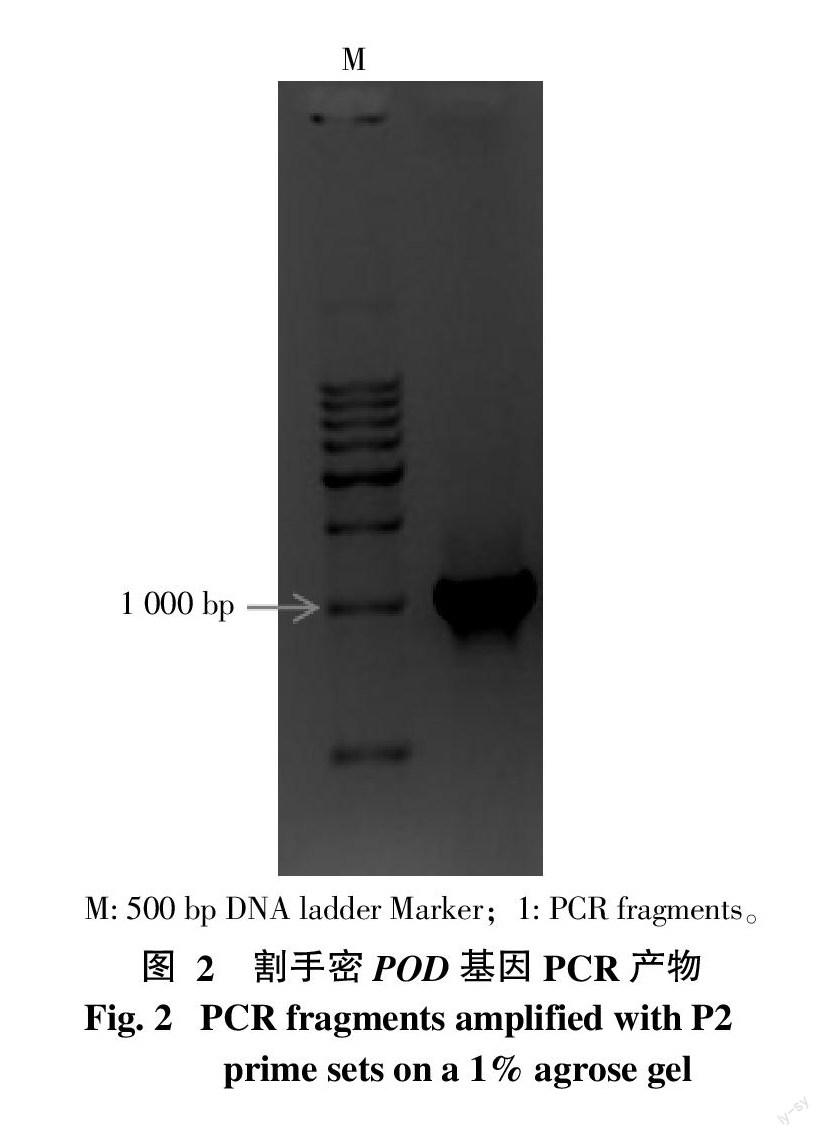
按照上述引物设计的方法,设计的2对引物情况见表2(其中扩增区域大小与位置以拼接的割手密POD基因的cDNA参考序列计算),预计最终PCR产物长度为1 084 bp。将割手密POD基因的cDNA以此2对引物经2轮PCR后,进行琼脂糖凝胶电泳检测,结果见图2。从图2中可以看出,PCR扩增产物大小约为1 000 bp,与预测的结果一致,可认定为目的条带。
2.3 目的序列的确定和结构分析
测序获得的2个序列长度均为1 083 bp,比预测的目的片段少1个碱基,经过比对结果发现,缺少的这个碱基出现在3′非编码区(第1 024个碱基),未影响其开放阅读框,且这2个序列编码区均能翻译成氨基酸序列,可认为此2个克隆均为目的基因克隆。将此2条序列命名为SsPOD-1a和SsPOD-1b,并将序列提交GenBank数据库,获得登录号KJ002565、KJ002566。
测序获得的序列与参考序列比较,共有27个差异位点(图3),涉及到13个氨基酸差异,其中SsPOD-1a和SsPOD-1b之间有7个差异位点(表3),有4个氨基酸差异位点。
2.4 蛋白功能预测
SsPOD-1a和SsPOD-1b基因的序列全长为1 083 bp,其中开放阅读框长度为996 bp,5′非翻译区27 bp,3′非翻译区60 bp。这2个基因均编码331个氨基酸,序列相似性为98.8%。利用Expasy中的protparam对SsPOD-1a和SsPOD-1b的蛋白质理化性质进行预测,蛋白分子量大小分别为35.84、35.82 ku,等电点pI均为8.59,属于碱性蛋白,GRAVY值(Grand average of hydropathicity)均为0.044,为疏水蛋白,不稳定(稳定系数分别为40.08、39.70)。利用NCBI(http://www.ncbi.nlm.nih.gov/ Structure/cdd/docs/cdd_search.html)进行蛋白质保守结构域分析,结果表明,此蛋白属于依赖于亚铁血红素Ⅲ型POD亚类,此类POD主要出现在细胞间质和液泡中,主要与过氧化氢分解、生长素代谢、木质素合成和抗逆反应有关[10],包含4个保守的二硫键和2个保守的钙结合位点(图4)。
2.5 SsPOD-1a和SsPOD-1b在BL21感受态细胞中的表达
按照上述方法将SsPOD-1a和SsPOD-1b转入BL21感受态细胞,分别诱导表达0、1、2、4 h后的表达情况见图5。由图5可以看出,在刚开始诱导时(第2和第6泳道),目的蛋白(35.8 ku)几乎没有表达,但在诱导1、2、4 h后(第3,4,5泳道和第7,8,9泳道),目的蛋白均有表达。说明克隆得到的SsPOD-1a和SsPOD-1b的表达区域能正常表达出蛋白。
2.6 POD基因蛋白进化分析
将SsPOD-1a和SsPOD-1b的氨基酸序列在NCBI做blastp,找到其它物种的POD氨基酸序列,并利用MEGA6.06软件,采用Neighbor-joining法做系统发育分析(图6),结果发现克隆得到的割手密POD基因与高粱的POD基因的亲缘关系最近,其次为禾本科植物的玉米和水稻,与一些双子叶植物及裸子植物也有一定的亲缘关系,且亲缘关系的远近与植物分化的程度相关性很大,说明POD基因起源较为古老且较为保守,可为植物分类作参考。
3 讨论与结论
现代基因克隆技术主要有同源克隆、功能克隆和图位克隆等,各种基因克隆方法均有各自的优势和不足[11],结合本试验的实际情况,采取同源克隆方法比较准确和方便。传统同源克隆一般是找到亲缘关系较近的物种基因结构和功能比较清楚的同源基因序列,利用基因保守性,在同源区域设计引物,克隆得到基因。但此种方法有一定的局限性,如果在进行引物设计时,引物区与实际序列存在差异,就很难扩增到目的片段,为尽量避免这种情况的出现,本研究做了一些改进,在得到割手密亲缘关系较近的高粱POD基因序列(XM_002437414.1)后,将此序列和割手密关系更近的甘蔗EST库进行blast比对,找到相似度更高的EST拼接成一条假设的割手密POD基因的cDNA序列,根据此cDNA序列设计引物,扩增POD基因,为进一步提高扩增效率和特异性,采用巢式PCR,设计2对引物,成功获得目的基因。但巢式PCR也有一定的不足,即第二轮扩增丢失了一段5′UTR和3′URT区域,但对重点研究表达区域的基因来说,并不影响其表达序列的分析。
包含亚铁血红素的POD根据来源可分为2个亚族,来源于哺乳动物的为一类,来源于细菌、真菌和植物的为另一类,而第二种POD又可分为3类,一种是与细菌POD类似的Ⅰ型,一种是存在于真菌细胞外的Ⅱ型,还有一种是与植物分泌途径相关的Ⅲ型,这也是研究得最为广泛的一种类型[12],根据蛋白质保守区域结构域分析结果表明,本试验克隆得到的割手密POD基因也属于此类。在植物POD亚族中,构成POD基因的原始序列在个体中是高度可变的,整个氨基酸序列在不同植物种类中相似性有时会少于35%,然而90%参与催化的残基在不同植物中均存在[6],这表明对于不同植物个体,虽然POD基因编码序列存在较大差异,但功能区相对保守,而序列的差异也导致了POD在应对环境刺激时的差异。Hiraga等[7]利用基因特异探针研究了21个水稻POD基因在环境胁迫时基因的表达情况,结果表明它们在面对相同或不同环境胁迫时均存在显著差异。郭媖等[13]从陆地棉中克隆得到2个相似性达99%的POD基因,通过RT-PCR研究结果表明,这2个基因在植株不同部位、不同生长阶段的表达情况均有所不同。这表明POD在植物体内存在很多同工酶,且这些酶在植物不同部位、不同时间或不同环境的分布均有所不同,很难确定单一的POD在面对环境刺激所起的作用。从本研究克隆得到的割手密POD基因的氨基酸序列系统发育分析结果表明,不同物种POD基因的氨基酸组成之间存在较大差异,且差异随着亲缘关系的远近而变化,依此推断,同属于甘蔗属的割手密与栽培甘蔗的POD基因差异应较小。POD是应对干旱胁迫的关键调控酶之一[3]。割手密和栽培甘蔗的POD基因的氨基酸组成差异应不是很大,而割手密耐旱性却远高于栽培甘蔗,这说明植物应对干旱胁迫是一个复杂的过程,有多个基因参与应对环境胁迫,单一的POD基因并不能决定植物的耐旱性,所以需要研究更多的割手密和栽培甘蔗的POD基因才能找到响应干旱胁迫的关键基因,才能进一步阐明POD基因在应对干旱胁迫时所起的作用,为研究植物应对干旱逆境时基因调控机制奠定基础,以解释各植物耐旱性差异的根本原因。
致谢 感谢中国热带农业科学院热带生物技术研究所给予的经费支持,感谢中国热带农业科学院湛江实验站提供本实验所需的科研条件,感谢在本研究中给予帮助和关心的各位老师和工作人员!
参考文献
[1] 陈 辉,范源洪,史宪伟,等. 甘蔗细茎野生种(Saccarun spontaneum L.)的遗传多样性和系统演化研究[J]. 作物学报,2001, 27(5): 645-652.
[2] 张木清, 陈如凯. 作物抗旱分子生理与遗传改良[M]. 北京: 科学出版社, 2005.
[3] 梁艳荣, 胡晓红, 张颍力,等. 植物过氧化物酶生理功能研究进展[J]. 内蒙古农业大学学报, 2003, 24(2): 110-113.
[4] Schuller D J, Ban N, Huystee R B, et al. The crystal structure of peanut peroxidase[J]. Structure, 1996, 4(2): 311-321.
[5] Berglund G I, Carlsson G H, Smith A T, et al. The catalytic pathway of horseradish peroxidase at high resolution[J]. Nature,2002, 417(3): 463-468.
[6] Kjaersgard I V, Jespersen H M, Rasmussen S K, et al. Sequence and RT-PCR expression analysis of two peroxidases from Arabidopsis thaliana belonging to a novel evolutionary branch of plant peroxidases[J]. Plant Molecular Biology, 1997, 33(4): 699-708.
[7] Hiraga S, Yamamoto K, Ito H, et al. Diverse expression profiles of 21 rice peroxidase genes[J]. FEBS Lett, 2000, 471: 245-250.
[8] 单丽伟, 罗海霞, 范三红, 等. 小麦种子过氧化物酶基因wp1的克隆及在大肠杆菌中的表达[J]. 西北农林科技大学学报(自然科学版), 2009, 37(3): 213-218.
[9] Delannoy E, Marmey P, Jalloul A. Molecular analysis of class III peroxidases from cotton[J]. J Cotton Sci, 2006, 10(1): 53-60.
[10] Hiraga S, Sasaki K, Ito H, et al. A large family of class Ⅲ plant peroxidases[J]. Plant Cell Physiol, 2001, 42(3): 462-468.
[11] 陈儒钢, 巩振辉, 逮明辉,等. 植物基因克隆技术的发展与展望[J]. 长江蔬菜, 2009, 23(20): 13-18.
[12] Duroux L, Welinder K G. The peroxidase gene family in plants: a phylogenetic overview[J]. J Mol Evol, 2003, 57(2):397-407.
[13] 郭 媖, 郭旺珍, 张天真. 两个陆地棉过氧化物酶cDNA的克隆和鉴定[J]. 作物学报, 2007, 33(6): 891-897.
