应用噬菌体展示肽库技术淘选大鼠CCR5膜外第一、二胞外环特异性结合的活性拮抗肽与初步鉴定*
2015-05-16刘思雪叶小研黄花荣钟英强中山大学孙逸仙纪念医院消化内科儿科广东广州500广东药学院附属第一医院消化内科广东广州50080
刘思雪,胡 梅,叶小研,黄花荣,钟英强(中山大学孙逸仙纪念医院消化内科,儿科,广东广州500;广东药学院附属第一医院消化内科,广东广州50080)
应用噬菌体展示肽库技术淘选大鼠CCR5膜外第一、二胞外环特异性结合的活性拮抗肽与初步鉴定*
刘思雪1▲,胡梅1▲,叶小研1,3,黄花荣2△,钟英强1△
(中山大学孙逸仙纪念医院1消化内科,2儿科,广东广州510120;3广东药学院附属第一医院消化内科,广东广州510080)
[摘要]目的:利用噬菌体展示肽库技术淘选与大鼠CC趋化因子受体5( CCR5)膜外第一、二胞外环特异性结合的短肽,并鉴定其与CCR5的结合能力。方法:在蛋白质数据库中查得大鼠CCR5第一、二胞外环的氨基酸序列,合成相应的线性短肽作为淘选的靶分子,利用噬菌体展示7肽文库进行3~4轮淘选,用ELISA法鉴定所选肽与靶分子的结合,并测定其与浓度的关系。结果:与CCR5第一、二胞外环特异性结合的噬菌体展示的短肽序列分别为GHWKVWL和HYIDFRW,ELISA鉴定呈阳性反应,且短肽与靶分子的结合具有浓度依赖性和可饱和性。结论:利用噬菌体展示技术成功获得了2条CCR5特异性结合的短肽,并在体外证明其可与CCR5第一、二胞外环具有结合能力。
[关键词]CC趋化因子受体5;拮抗肽;噬菌体展示
▲并列第1作者
CC趋化因子受体5( CC chemokine receptor 5,CCR5)是趋化因子受体超家族的重要成员之一,属于G蛋白偶联受体,具有典型的7次跨膜区段( transmembrane region)和3个胞外环( extracellular loop,ECL)及3个胞内环( intracellular loop,ICL),其胞外部分可与配体结合而发挥相应的生物学效应[1]。CCR5主要分布于炎症细胞表面,如淋巴细胞、单核细胞、嗜酸性粒细胞等,参与炎症细胞在体内的趋化性迁移,在炎症性肠病( inflammatory bowel disease,IBD)的免疫炎症反应中起重要作用,其可能为IBD治疗的新靶点[2],而针对CCR5的各类拮抗剂可用于人类多种自身免疫性疾病、器官移植后排斥反应和艾滋病等的治疗,具有广阔的应用前景。我们拟应用噬菌体展示技术淘选能与大鼠CCR5第一、二胞外环特异性结合的活性拮抗短肽,并在体外对其结合能力作了初步鉴定。
材料和方法
1主要材料和试剂
噬菌体表面展示肽库试剂盒Ph.D.TM-7(随机7肽库)购自New England BioLabs,库容1×1016pfu/L,宿主菌E.coli ER2738,-96 gⅢ测序引物5’-OHCCC TCA TAG TTA GCG TAA CG-3’。辣根过氧化物酶( horseradish peroxidase,HRP)标记的抗M13单克隆抗体( HRP/anti-M13)购自GE Healthcare。HRP底物2,2-联氮-二( 3-乙基苯并噻唑-6-磺酸)二铵盐[2,2-azino-bis( 3-ethylbenzthiazoline-6-sulfonic acid) diammonium salt,ABTS]购自Sigma。大鼠CCR5第一、二胞外环( ECL1、ECL2)的氨基酸序列在OWL蛋白质数据库中查得,ECL1为AANEW VFGNI MCK ( 13 aa),ECL2为MRSQK EGSHY TCSPH FLHIQ YRFWK HFQTL KM ( 32 aa),交由中国上海吉尔生化有限公司合成,纯度为95%。异丙基-β-D-硫代半乳糖苷( isopropyl-β-D-thiogalactoside,IPTG) 和5-溴-4-氯-3-吲哚-β-D-半乳糖苷( 5-bromo-4-chloro-3-indolyl-β-D-galactopyranoside,X-Gal)即用型溶液购自AMRESCO。
2方法
2.1亲和淘选分别将ECL1和ECL2冻干粉溶于0.1 mmol/L pH 8.6的NaHCO3溶液,ECL1用1%二甲基亚砜( dimethylsulfoxide,DMSO)助溶,制备100 μg/L的靶分子溶液包被6孔板,每孔0.5 mL,在增湿容器中4℃轻微振荡,孵育过夜。第2天倒掉包被液,加满封闭液( 1 mol/L pH 8.6 NaHCO3+5 g/L BSA),4℃作用1 h。去除封闭液,用TBST缓冲液快速洗板6次。用350 μL的TBST缓冲液稀释2 ×1011pfu的噬菌体,加到已包被好的孔中,室温温和摇动60 min。TBST缓冲液洗板10次,加入非特异性缓冲液( 0.2 mol/L pH 2.2的glycine-HCl +1 g/L BSA) 350 μL,室温作用15 min,将洗脱液吸入另一干净微量离心管中,用50 μL 1 mol/L Tris-HCl( pH 9.1)中和上述洗脱液。取1 μL测定洗脱物的滴度。剩余洗脱物加入20 mL ER2738培养物中(菌体应处于对数前期,A600=0. 01~0. 05),37℃剧烈摇动培养4. 5 h。将扩增培养物经聚乙二醇( polyethylene glycol,PEG) /NaCl两次浓缩纯化后得到噬菌体扩增液,测定其滴度后可进行下一轮淘选。如此共进行3轮淘选,若第3轮淘选后没有明显的共有序列发现,按说明书要求继续扩增进行第4轮淘选。其后的淘选中噬菌体投入量均为2×1011pfu,TBST洗涤浓度增至0.5%。
2.2噬菌体滴度测定接种ER2738单菌落于10 mL LB培养基中,摇床培养5 h至对数中期( A600≈0. 5)。用LB系列稀释噬菌体,淘选洗脱物稀释度为101~104;扩增后为108~1011。取10 μL稀释的噬菌体与200 μL菌液混匀,37℃放置5 min,再将上述混合液与3 mL 45℃预热的顶层琼脂( LB培养基+7 g/L琼脂粉+1g/L MgCl2·6H2O)混匀,立即平铺于37℃预温的LB/IPTG/X-Gal平板( LB培养基+15 g/L琼脂粉,高压灭菌,冷却至70℃时加入1 mL IPTG/X-Gal即用型溶液)上,冷却后倒置于37℃细菌培养箱中过夜。第2天检查平板,计数有约100个蓝色噬菌斑的平板上的斑数,乘以稀释倍数即为每10 μL噬菌体的滴度。
2.3噬菌斑扩增测序挑一ER2738单克隆于LBTet培养基中培养过夜,将过夜培养物按1∶100稀释接种于LB培养基,分1 mL到每一个培养管中。在第3轮淘选洗脱物滴度测定的平板中,在总量不到100个噬菌斑的平板上随机挑取15个克隆,分别接种到培养管中。37℃摇床培养4.5 h。14 000 r/min离心30 s,上清液转入新鲜离心管中,再离心,取80%的上清到另一离心管,即为扩增的噬菌体贮液。取200 μL由中国北京金唯智生物科技有限公司完成测序,测序成功的标准:读取序列长度大于800 bp,背景峰高度不高过主峰的1/3。
2.4ELISA鉴定阳性噬菌体克隆将过夜培养的ER2738按1∶100稀释于20 mL LB培养基,于每管加入5 μL测序剩余的噬菌体上清,37℃通气培养4.5 h,PEG/NaCl两次浓缩纯化后得到噬菌体扩增液。测定噬菌体滴度。制备100 mg/L的ECL1、ECL2靶分子溶液(方法同淘选步骤)包被96孔酶标板,150 μL/孔,每个待鉴定克隆1个包被孔及1个空白对照孔,4℃孵育过夜。第2天去除残液,加满封闭液4℃作用1.5 h。甩出封闭液,TBST洗板6次。包被孔和空白对照孔各加入5 μL噬菌体扩增液,0.5% TBST稀释至100 μL,室温振荡作用1.5 h。0.5% TBST洗板6次,每孔加入200 μL HRP/anti-M13抗体( 1∶5 000稀释),室温振荡作用1 h。0.5% TBST洗板6次,每孔加200 μL显色底物ABTS-H2O2溶液,室温作用30 min,酶标仪读取405 nm处的吸光度。每项实验重复3次,阳性克隆定义为反应孔吸光值高于阴性对照3倍[3-4]。
2.5ELISA检测噬菌体浓度与其结合能力的关系
将含有共有序列的噬菌体克隆进行扩增并测定滴度,在一个已封闭的酶标板中稀释噬菌体,第1孔加入约含5×1011个病毒子的噬菌体悬液,TBS稀释至200 μL,吸取100 μL至第2孔,按此方法依次进行2倍系列稀释。将稀释好的噬菌体全部转移至包被有靶分子的酶标板中,进行定量ELISA,方法同2.4,分别读取不同浓度的噬菌体与靶分子结合后的吸光度。
3统计学处理
定量数据以均值±标准差( mean±SD)表示,两组均数比较采用独立样本t检验。应用SPSS 13.0统计学软件分析处理,以P<0.05为差异有统计学意义。
结果
1 亲和淘选与CCR5第一、二胞外环特异性结合的噬菌体
以人工合成的CCR5第一、二胞外环氨基酸序列作为靶分子,对噬菌体7肽库进行淘选,随着筛选轮次的增加,从固相平板洗脱的噬菌体数逐渐增加,回收率(回收/投入噬菌体量)逐轮升高,ECL1和ECL2特异性结合的噬菌体得到了选择性富集,见表1。将第3轮淘选洗脱物测序后发现,ECL1已得到共有序列( 10/15),而ECL2的一致率较低( 3/15),故ECL2增加了第4轮淘选,共有序列即明显出现( 11/15)。
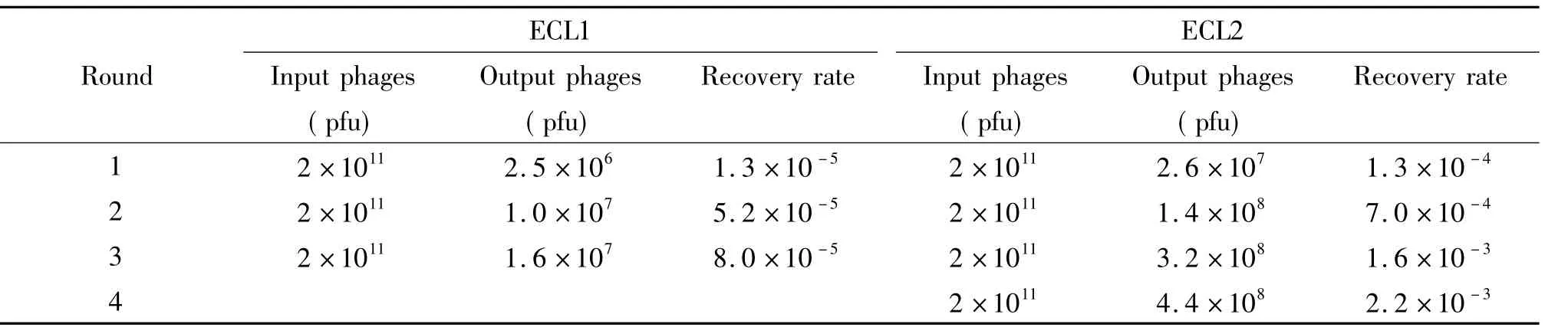
表1 不同淘选轮次噬菌体淘选的回收率Table 1.Recovery rate of phages for each round of panning
2 淘选所得噬菌体克隆的氨基酸序列分析
最后一轮淘选物滴度测定时,从总量不到100个蓝色噬菌斑的平板上随机挑取15个克隆扩增后测序,根据测序结果推断噬菌体展示的氨基酸序列。ECL1和ECL2分别有10和11个噬菌体序列一致,见表2。其共有序列分别为GHWKVWL和HYIDFRW,基因测序图见图1、2。
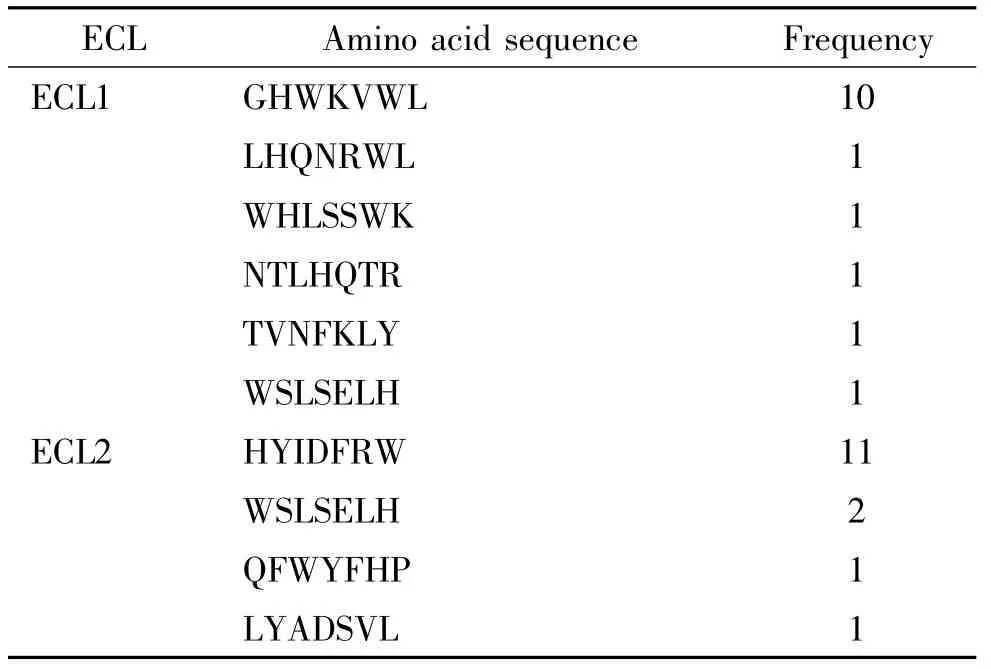
表2 淘选所得噬菌体克隆的氨基酸序列Table 2.Exogenous amino acid sequences of selected phage clones
3 ELISA法鉴定噬菌体克隆与ECL1、ECL2的特异性结合

Figure 1.Gene sequencing of consensus peptide displayed on ECL1-specifically bound phage.图1 ECL1特异性结合噬菌体克隆所展示的共有结合肽的基因测序图
Figure 2.Gene sequencing of consensus peptide displayed on ECL2-specifically bound phage.图2 ECL2特异性结合噬菌体克隆所展示的共有结合肽的基因测序图
将噬菌体上清液扩增后做ELISA检测,除空白对照外,每个克隆均设置阴性对照(不包被靶分子、仅用牛血清白蛋白溶液进行封阻)。ECL1结合的15个噬菌体克隆中,有4个为阴性克隆( P>0. 05,或噬菌体A值<阴性对照A值×3) ; 11个为阳性克隆( P<0. 05,并且噬菌体A值>阴性对照A值×3),其中10个氨基酸序列为GHWKVWL,1个WHLSSWK,见表3。ECL2结合的15个噬菌体克隆全部为阳性克隆,其中11个氨基酸序列为HYIDFRW,2个 WSLSELH,1个QFWYFHP,1个LYADSVL,见表4。

表3 ELISA法鉴定ECL1淘选所得噬菌体克隆的结合能力Table 3.Identification of binding efficiency of selected phage clones targeting ECL1 by ELISA ( Mean±SD.n =3)
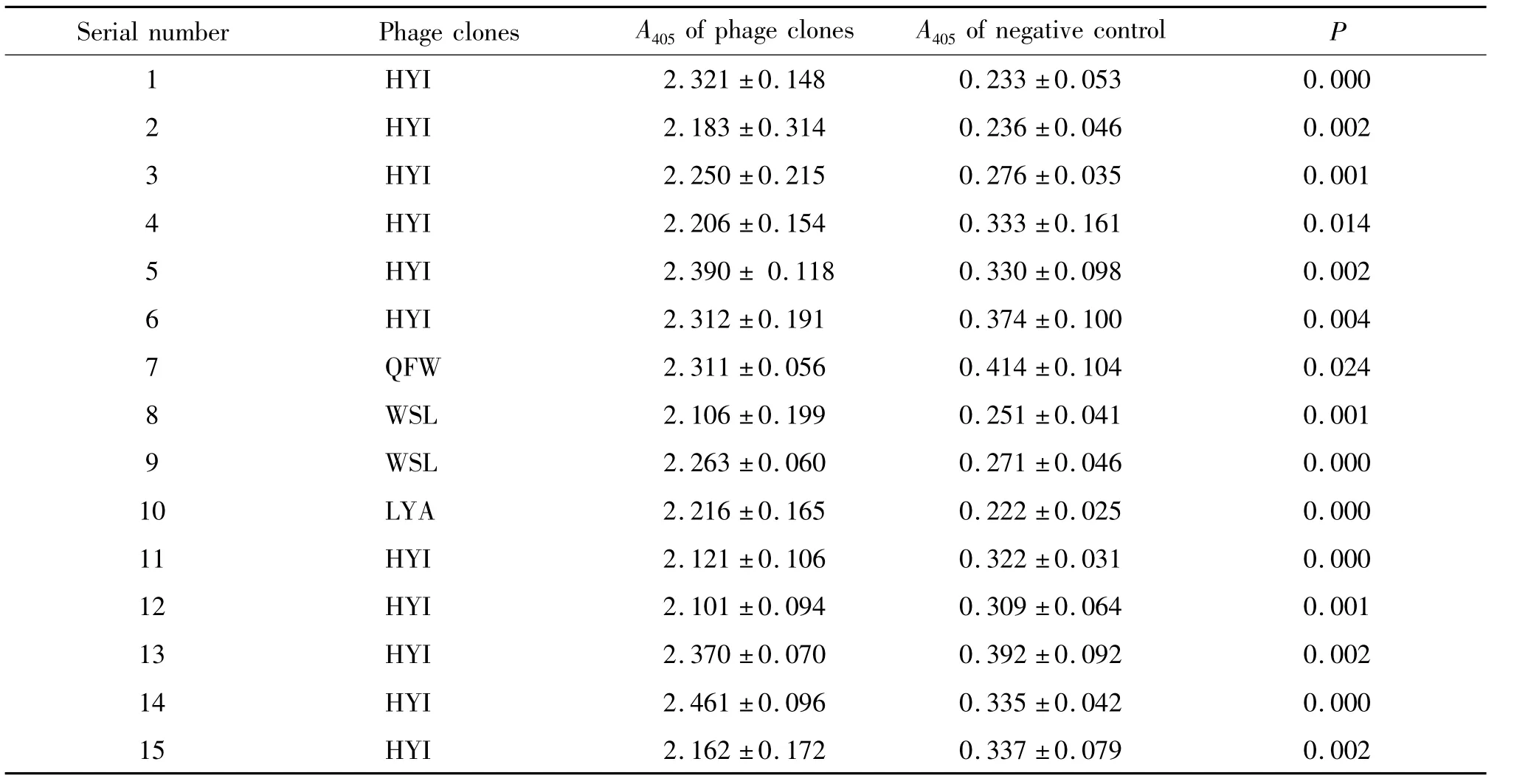
表4 ELISA法鉴定ECL2淘选所得噬菌体克隆的结合能力Table 4.Identification of binding efficiency of selected phage clones targeting ECL2 by ELISA ( Mean±SD.n =3)
4 共有序列噬菌体克隆与靶分子的结合能力与浓度的关系
与ECL1和ECL2特异性结合的共有序列为GHWKVWL和HYIDFRW,将其对应的噬菌体克隆进行扩增,进行定量ELISA检测。结果表明,在噬菌体浓度较低时,其与靶分子的结合能力随浓度的增加呈线性上升趋势;当浓度达到3.125×1014pfu/L(第3孔)及以上时,其与靶分子的结合达到饱和,见图3。
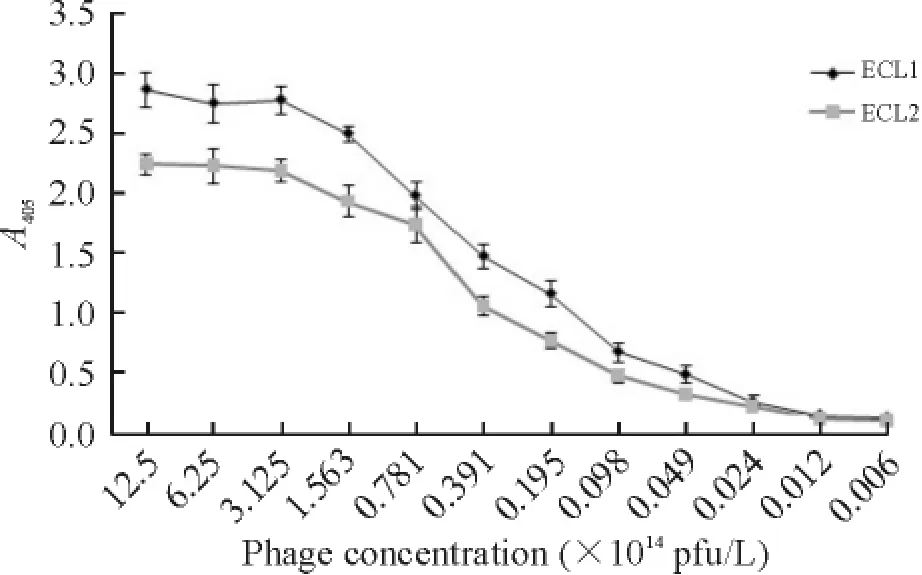
Figure 3.Relationship of concentration and binding efficiency of phage clones.Mean±SD.n =3.图3 噬菌体浓度与结合能力的关系
讨论
噬菌体展示技术是一种选择技术,它利用基因重组的方法将大量随机多肽与噬菌体的一种衣壳蛋白融合表达,将其展示在病毒颗粒的表面,在体外通过亲和淘选的方法,可将与靶分子特异性结合的噬菌体从肽库中淘选出来,经扩增后进行序列测定可确定表达的肽的氨基酸序列。该技术是蛋白质组学研究中的一种常用方法,可广泛用于确定抗原表位、确定蛋白质相互作用位点、鉴定酶的底物或抑制物、鉴定受体激活物或抑制剂、开发多肽药物、研制肿瘤疫苗等。
在本实验中,我们首先进行了常规的3轮淘选,在洗脱物培养平板上,随机选取15个独立克隆进行测序,发现ECL1有10个克隆所展示的外源肽序列完全一致,说明特异性短肽已得到选择性富集,淘选成功。而ECL2的15个测序克隆中仅有3个序列一致,其它12个各不相同,增加了第4轮淘选后发现,相同的序列一致率从3/15上升到11/15,认为淘选成功,这可能与ECL2氨基酸序列长、与其亲和力高的短肽多而需要更强的淘选有关。
淘选得到的短肽应用ELISA法进行验证,发现除了共有序列外,其它非特异性的序列也可表现出阳性反应,说明与CCR5第一、二胞外环有亲和力的7肽并不唯一,特别是具有32个氨基酸长度的ECL2,能与其随机结合的七肽种类很多,而淘选之所以能使一种序列呈明显优势,是因为这种短肽与靶分子的结合作用最强,能在数轮的洗脱中“幸存”下来。另外,我们将这6种阳性的多肽序列通过Gen-Bank的BLAST序列比对工具,在RefSeq protein数据库中进行同源性搜索,未发现其与大鼠趋化因子具有同源性,说明它们与CCR5第一、二胞外环的结合可能是通过模拟趋化因子的构象表位而发挥作用的。噬菌体浓度定量的ELISA实验中发现,噬菌体在体外与CCR5的亲合力具有浓度依赖性和可饱和性,这跟受体与配体结合具有可饱和性的理论一致,推测饱和的主要原因是靶分子包被的数目有限。
在噬菌体展示肽库的选择上,美国New England Biolabs公司有7肽库( Ph.D.TM-7)、12肽库( Ph.D.TM-12)和环7肽库( Ph.D.TM-C7C) 3种可供选择。当配体序列要求在一个短的区域内有几个结合位点时,Ph.D.TM-7肽文库更好一些;当配体序列不能以足够强的亲和力与靶分子结合而被筛选时,Ph.D.TM-C7C文库会更好一些,因为此文库中所有展示肽结构均被局限于1个7残基二硫环内,其结合构象比线性形式表达的肽文库更有效、结合更紧密;而Ph.D.TM-12肽文库提供了更大的随机区域,会有利于选择到有多个弱结合位点的序列,而不是只有少数几个强结合位点的序列。因此,由于我们的实验是以相对较短的线性氨基酸( 13 aa和32 aa)作为靶分子,淘选是为得到高亲和力、高特异性的短肽,故选择使用了结合位点密集、需要强淘选的7肽库。Wang等[5]以细胞表达的人CCR5作为靶分子,用12肽库也淘选到与其特异性结合的亲和短肽,并在细胞水平上初步证明了该十二肽对其天然配体RANTES及CCR5单克隆抗体有竞争性结合作用,这项实验以CCR5整体结构作为靶点,12肽库的选择则可能更有利于特异性的提高。
研究显示CCR5主要表达于炎症细胞表面,其天然配体包括趋化因子RANTES、MIP-1α和MIP-1β,CCR5的N端及第二胞外环是其与配体结合的主要位点[6-7],结合后可诱导炎症细胞向特定组织和器官趋化迁移,从而参与机体免疫反应,并在变态反应性疾病中发挥重要作用,如IBD[8]、多发性硬化[9]、类风湿性关节炎[10]等。另外,CCR5还是HIV-1感染靶细胞的辅助受体,已成为抗艾滋病药物研发的理想靶点之一[11]。在CCR5拮抗剂的研发方面,小分子药物马拉维诺是目前FDA唯一批准用于临床的CCR5拮抗剂,主要用于艾滋病的治疗。其它类型的CCR5拮抗剂包括趋化因子衍生物、单克隆抗体及肽类化合物等,尚在实验室研究阶段,有许多已在实验室证明有效或进入I期临床试验[12],本实验淘选的亲和短肽即属于肽类拮抗剂一类,这类多肽具有高亲和力和特异性结合的特点,生产成本低、稳定性好、副反应发生率低,是蛋白制剂的一种可靠替代品,具有很大的研发空间。
在IBD的发病机制中,CC趋化因子与相应的受体结合后可触发多种炎症反应,包括白细胞的激活、趋化、胞吐作用,产生基质金属蛋白酶降解基质、上调炎症的级联反应等。研究证明IBD患者病变黏膜中CCR5+细胞浸润显著,与UC疾病活动度呈正相关[13],并与CD患者非干酪性肉芽肿形成有关[14]。Ajuebor等[8]及Kucuk等[15]用RANTES受体拮抗剂Met-RANTES分别治疗三硝基苯磺酸诱导的小鼠、大鼠结肠炎,发现病变黏膜的炎症反应可明显减轻、肠内细菌异位明显下降。Katchar等[16]用MIP-3α单克隆抗体治疗小鼠结肠炎后,也可发现结肠损伤明显减轻。由此推断,趋化因子受体拮抗剂可能是IBD治疗的有效方法之一。本实验淘选得到针对大鼠的2个CCR5拮抗短肽,可进一步在构建结肠炎的大鼠模型中来验证其生理作用。
[参考文献]
[1]Paterlini MG.Structure modeling of the chemokine receptor CCR5: implications for ligand binding and selectivity [J].Biophys J,2002,83( 6) : 3012-3031.
[2]叶小研,钟英强.CC趋化因子受体与炎症性肠病[J].胃肠病学,2014,19( 1) : 50-53.
[3]丁宁,肖慧,高巨,等.应用噬菌体展示技术筛选HMGB1启动子结合蛋白[J].中国病理生理杂志,2010,26( 1) : 28-31.
[4]Zhang ZF,Shan X,Wang YX,et al.Screening and selection of peptides specific for esophageal cancer cells from a phage display peptide library[J].J Cardiothorac Surg,2014,9: 76.
[5]Wang FY,Zhang TY,Luo JX,et al.Selection of CC chemokine receptor 5-binding peptide from a phage display peptide library[J].Biosci Biotechnol Biochem,2006,70 ( 9) : 2035-2041.
[6]Schnur E,Kessler N,Zherdev Y,et al.NMR mapping of RANTES surfaces interacting with CCR5 using linked extracellular domains[J].FEBS J,2013,280 ( 9) : 2068-2084.
[7]Lee B,Sharron M,Blanpain C,et al.Epitope mapping of CCR5 reveals multiple conformational states and distinct but overlapping structures involved in chemokine and coreceptor function[J].J Biol Chem,1999,274( 14) : 9617-9626.
[8]Ajuebor MN,Hogaboam CM,Kunkel SL,et al.The chemokine RANTES is a crucial mediator of the progression from acute to chronic colitis in the rat[J].J Immunol,2001,166( 1) : 552-558.
[9]Eltayeb S,Sunnemark D,Berg AL,et al.Effector stage CC chemokine receptor-1 selective antagonism reduces multiple sclerosis-like rat disease[J].J Neuroimmunol,2003,142( 1-2) : 75-85.
[10]Cumming JG,Tucker H,Oldfield J,et al.Balancing hERGaffinity and absorption in the discovery of AZD5672,an orally active CCR5 antagonist for the treatment of rheumatoid arthritis[J].Bioorg Med Chem Lett,2012,22( 4) : 1655-1659.
[11]Dragic T,Litwin V,Allaway GP,et al.HIV-1 entry into CD4+cells is mediated by the chemokine receptor CCCKR-5[J].Nature,1996,381( 6584) : 667-673.
[12]吴英萍,吴文言.CCR5及其拮抗剂的研究进展[J].中国生物工程杂志,2008,28( 11) : 89-96.
[13]Matsuzaki K,Hokari R,Kato S,et al.Differential expression of CCR5 and CRTH2 on infiltrated cells in colonic mucosa of patients with ulcerative colitis[J].J Gastroenterol Hepatol,2003,18( 9) : 1081-1088.
[14]Oki M,Ohtani H,Kinouchi Y,et al.Accumulation of CCR5+T cells around RANTES + granulomas in Crohn’s disease: a pivotal site of Th1-shifted immune response? [J].Lab Invest,2005,85( 1) : 137-145.
[15]Kucuk C,Sozuer E,Gursoy S,et al.Treatment with Met-RANTES decreases bacterial translocation in experimental colitis[J].Am J Surg,2006,191( 1) : 77-83.
[16]Katchar K,Kelly CP,Keates S,et al.MIP-3α neutralizing monoclonal antibody protects against TNBS-induced colonic injury and inflammation in mice[J].Am J Physiol Gastrointest Liver Physiol,2007,292( 5) : G1263-G1271.
Panning and identification of antagonistic active peptides specifically binding to the first and second extracellular membrane loops of rat CCR5 by technique of phage display peptide library
LIU Si-xue1,HU Mei1,YE Xiao-yan1,3,HUANG Hua-rong2,ZHONG Ying-qiang1
(1Department of Gastroenterology,2Department of Pediatric,Sun Yat-sen Memorial Hospital,Sun Yat-sen University,Guangzhou 510120,China;3The First Affiliated Hospital of Guangdong Pharmaceutical University,Guangzhou 510080,China.E-mail: zhongyingqiang@21cn.com; hhrvivi@21cn.com)
[ABSTRACT]AIM: To pan the active peptides which specifically bound to the first and second extracellular membrane loops of rat CC chemokine receptor 5 ( CCR5).METHODS: The technique of phage display peptide library was used and binding ability of the peptides was identified.The amino acid sequences of the first and second extracellular loops of rat CCR5 were searched in the protein database and chemically synthesized corresponding linear peptides were used as targets in the biopanning.After 3 to 4 rounds of screening with Ph.D.(TM)-7 Phage Display Peptide Library were performed,the specific phages were collected and primarily identified by ELISA.RESULTS: The sequences of the peptides displayed on the selected phages were GHWKVWL and HYIDFRW,both of them exhibited positive in phage binding ELISA and the binding to phages and targets were concentration dependent and saturable.CONCLUSION: Two antagonistic active peptides specifically binding to CCR5 were successfully obtained by the technique of phage display peptide library,and the binding ability to the first and second extracellular membrane loops of rat CCR5 were proved in vitro.
[KEY WORDS]CC chemokine receptor 5; Antagonistic peptide; Phage display
通讯作者△钟英强Tel: 020-81332598; E-mail: zhongyingqiang@21cn.com;黄花荣020-81332446; E-mail: hhrvivi@21cn.com
*[基金项目]国家自然科学资金基助项目( No.81370499) ;广东省自然科学基金资助项目( No.2014-A030313020)
[收稿日期]2015-03-03[修回日期]2015-04-11
[文章编号]1000-4718( 2015)07-1225-06
[中图分类号]R363
[文献标志码]A
doi:10.3969/j.issn.1000-4718.2015.07.013
