HMGCR基因多态性与肉牛屠宰性状相关性分析*
2015-04-18姚玉妮宋恩亮刘晓牧成海建刘桂芬
姚玉妮,宋恩亮,2△,刘晓牧,2,成海建,2,游 伟,2 ,韩 红,2,刘桂芬,2*
(1.山东省畜禽疫病防治与繁育重点实验室,山东 济南 250100;2.山东省农业科学院畜牧兽医研究所,山东 济南 250100)
科学研究
HMGCR基因多态性与肉牛屠宰性状相关性分析*
姚玉妮1,宋恩亮1,2△,刘晓牧1,2,成海建1,2,游 伟1,2,韩 红1,2,刘桂芬1,2*
(1.山东省畜禽疫病防治与繁育重点实验室,山东 济南 250100;2.山东省农业科学院畜牧兽医研究所,山东 济南 250100)
MHGCR是胆固醇生物合成过程中的限速酶,在内源胆固醇生物合成过程中起着重要的作用。研究以牛HMGCR基因为候选基因,采用PCR-SSCP和测序技术在利杂牛、利西杂牛、秦川牛、西杂牛和夏西杂牛五个群体共123个个体中检测遗传变异情况。结果表明,检测到AA和AB共2种基因型,没有发现BB基因型,测序分析发现一处SNP多态位点(GenBank:BC153262.1 11137T>C)。不同基因型与屠宰性状的相关性分析显示,在利西杂群体中AA基因型个体与AB基因型个体相比,AA基因型个体的屠宰率和牛柳指标达到了极显著水平(P<0.01),而AA基因型的上脑重显著高于AB基因型(P<0.05),其余各项指标基因型间差异不显著(P>0.05)。因此,HMGCR基因单核苷酸多态性对利西杂牛脂肪沉积有一定的影响,可作为影响屠宰性状的候选基因,指导肉牛的育种工作。
肉牛;HMGCR;PCR-SSCP;基因多态性;屠宰性状
胆固醇生物合成途径,也称为甲羟戊酸(Mevalonic acid,MVA)途径,参与多种细胞功能必不可少的细胞途径。3-羟基-3-甲基戊二酸单酰辅酶A(3-hydroxy-3-methylglutaryl-CoA,HMG-CoA)还原酶(Three-hydroxy-3-methylglutaryl-CoA Reductase,HMGCR)是胆固醇生物合成过程中的限速酶[1-3],在内源胆固醇生物合成过程中起着重要的作用[4],且是体内催化HMG-CoA生成MVA这一代谢途径的关键酶[5-7]。HMGCR的表达受转录和转录后水平的严格调控,HMGCR通过转录抑制和酶修饰负反馈机制维持胆固醇的动态平衡[8]。HMGCR基因在胆固醇代谢过程中的关键作用决定了其在动物肉质及其它生长性能中的重要作用[9]。猪体内胆固醇来自日粮中的吸收和机体自身合成,但以机体自身合成为主[10]。肝脏及其他器官合成胆固醇的原料是乙酰CoA,经过多步酶促反应生成一系列的中间产物,如HMG-CoA、鲨烯、羊毛固醇,最后转变为胆固醇,其中HMG-CoA还原酶是限速酶,决定反应的快慢,也是外界调控体内胆固醇合成的中介。Cánovas等研究表明:不同基因型,猪HMGCR基因在肌肉中的表达水平、脂肪沉淀和总胆固醇水平有所不同。HMGCR基因的表达与血清高密度脂蛋白水平成负相关,而与胴体瘦肉率和硬脂酸含量和亚油酸含量成正相关。刘桂芬等对不同猪种HMGCR基因在13个组织的表达量差异研究表明,HMGCR在不同组织的表达量有明显差异,在哺乳动物上HMGCR很可能也是一个结构表达基因[11]。本研究利用PCR-SSCP技术分析牛HMGCR基因的遗传变异,寻找与屠宰性能相关的多态性位点,为探讨HMGCR在动物脂肪沉积中的作用机制,进而通过营养因子调控技术改善牛肉肉质比率、改善牛肉品质提供一定的理论依据。
1 材料与方法
1.1 试验材料
随机抽取18月龄利杂牛(利木赞牛×地方黄牛,LZ)、利西杂牛(利木赞×鲁西黄牛,LXZ)、秦川牛(QC)、夏西杂牛(夏洛莱牛×鲁西黄牛,XXZ)、西杂牛(鲁西黄牛×地方黄牛,XZ)五个牛群体共123头(除秦川牛外其它牛群均为杂交后代),颈静脉采血,放入加有ACD抗凝剂的离心管中,然后摇匀迅速放入冰盒中带回实验室,-20 ℃保存待用。牛的屠宰指标的数据来自于东营凯银清真肉类有限公司牛场的生产和屠宰记录,且与血样均来自相同的牛。
1.2 方 法
1.2.1 引物设计 本试验所用HMGCR引物根据GenBank中牛HMGCR基因序列(GenBank:BC153262.),扩增出牛HMGCR基因的第三内含子、第四外显子、第五外显子、第六外显子、第七外显子、第八外显子、5'调控区。七对引物由上海博尚生物工程有限公司合成,分别命名为P1、P2、P3、P4、P5、P6、P7。引物序列、PCR产物大小、位置以及引物的最佳退火温度见表1。

表1 HMGCR基因引物序列、产物大小、位置及退火温度
1.2.2 PCR扩增体系(20 μL) DNA模板30 ng;引物(上下游等体积混合10 pmol/L)1.6 μL;2×EasyTaqPCR SuperMix(北京全式金生物技术有限公司)10.0 μL加水至20 μL。
1.2.3 PCR扩增条件 95 ℃ 预变性5 min;94 ℃ 变性30 s;50.2 ℃ 复性35 s;72 ℃ 延伸40 s;35个循环;72 ℃ 延伸10 min;4 ℃保存。扩增产物用1.5%琼脂糖凝胶电泳检测。
1.2.4 PCR-SSCP检测与扩增产物的测序 3 μL PCR产物和8 μL 上样缓冲液(98%甲酰胺、0.025%溴酚蓝、0.025%二甲苯氰、10 mmol/L EDTA(pH8.0))混匀,98 ℃ 变性10 min,然后迅速冰浴10 min 后上样。将样品加到10%非变性聚丙烯酰胺凝胶中过夜电泳。电泳结束后银染显带,biorad凝胶成像系统照相分析,判型。对不同基因型个体的PCR扩增产物送交山东省农科院测序中心直接在ABI3730自动测序仪上测序。
1.2.5 数据分析 利用PopGen32软件计算遗传多态性指标;用SPSS 13.0软件的一般线性模型(GLM)分析个体基因型对牛屠宰指标的影响。Y=u+marker+e,式中Y为性状表型值,u为群体平均值,marker为基因型效应,e为环境效应值。
2 结果与分析
2.1HMGCR基因PCR-SSCP和序列分析
检测5个牛群体HMGCR基因第三内含子、第四外显子、第五外显子、第六外显子、第七外显子、第八外显子、5'调控区的多态性,对于引物P5扩增片段,5个牛群体中均检测到AA和AB 2种基因型(图1),测序对比分析发现,AA型与基因号为GenBank:BC153262.的序列对应位置完全一致,AB型与GenBank:BC153262.序列比对发现在第11 137 bp处有1个T→C的杂合突变。

图1 样点分布图 图2HMGCR基因P5基因座的测序结果
Fig.1 Distribution map of samples Fig. 2 The sequencing result of the SIRT1 gene P5 locus
2.2HMGCR基因在不同牛群体中的遗传多态性
5个牛群体中HMGCR基因P5基因座的等位基因频率和基因型频率见表2。由表2可见:HMGCR基因座的等位基因频率(PA/PB)在利杂牛、利西杂牛、秦川牛、西杂牛和夏西杂5个群体中的比率分别为0.85/0.15、0.67/0.33、1/0、0.95/0.05、0.44/0.56,秦川牛未检测到AB基因型,在五个牛群体中均未检测到BB基因型,优势等位基因均为A。检验结果表明,在 P5位点处各群体均处于 Hardy- Weinberg 不平衡状态。
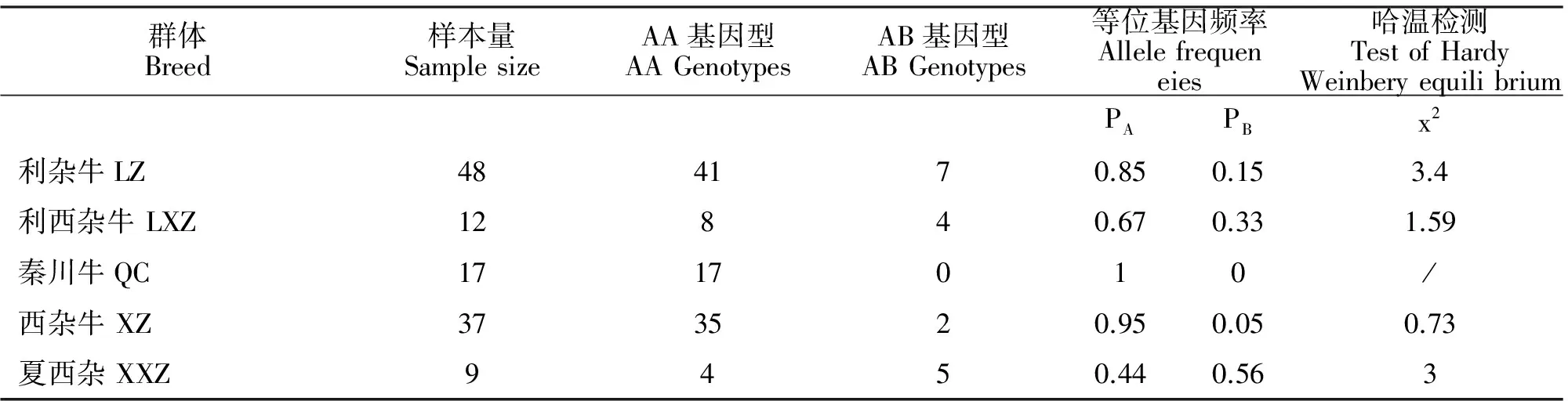
表2 5个牛群体HMGCR基因的突变率分析


2.3 遗传多态性分析
该突变位点在利西杂牛和夏西杂牛群体中的多态信息含量(PIC)值分别为0.3444和0.3714,属于中度多态(0.25 表3 五个牛群体中HMGCR基因P5基因座的遗传参数 表4 不同牛群体间HMGCR多态基因座基因型分布的卡方独立性检验 注:括号外表示不同群体基因型频率分布的P值,括号内表示不同群体基因频率分布的x2值;标有*和**的数值显著水平分别为P<0.05和P<0.01。 Note:The number out parentheses showed P value of genotype distribution,the number in parentheses showed x2of genotype.Value with*and** differ significantly atP<0.05 andP<0.01,respectively. 不同牛群体间多态基因座基因型分布的卡方独立性检验结果显示:利西杂牛与秦川牛、利西杂牛与西杂牛、秦川牛与夏西杂牛及西杂牛与夏西杂的差异极显著(P<0.01)。其余群体间差异不显著(P>0.05)。 表5列出了利西杂牛、利杂牛、西杂牛及夏西杂牛HMGCR基因不同基因型与屠宰率、胴体重、牛柳、西冷、眼肉、上脑、背膘厚和高档肉块占活重的比例的最小二乘均值差异显著性检验结果。结果显示,在利西杂群体中AA基因型个体与AB基因型个体相比,AA基因型个体的屠宰率和牛柳指标达到了极显著水平(P<0.01),而AA基因型的上脑重显著高于AB基因型(P<0.05),其余各项指标基因型间差异不显著。而在利杂牛、西杂牛及夏西杂牛HMGCR基因不同基因型与屠宰率、胴体重、牛柳、西冷、眼肉、上脑、背膘厚和高档肉块占活重的比例的最小二乘均值差异显著性检验结果显示,所有各项指标基因型间差异不显著。 表5 不同牛群体HMGCR基因不同基因型之间屠宰性状最小二乘均值差异显著性检验 注:同行数据肩标为不同小写字母表示差异显著(P<0.05),不同大写字母表示差异极显著(P<0.01)。 Notes:Estimates are given as ±SE.Data with a different letter (a,b) within the same line differ significantly at P<0.05,resepectiveZly. 3.1 牛HMGCR基因多态性及其分布规律 现阶段对影响牛屠宰性状的基因的多态性研究较多,但对牛的HMGCR基因多态性与屠宰性状的相关性还未见报道。安光全在鸡HMGCR基因的部分外显子和部分内含子序列,检测到3个多态位点,分别为内含子l处的SNP1(789bp,G/A)和SNP2(937bp,C/G)、内含子4处的SNP3(2316bp,A/C)[12]。本研究在牛的HMGCR基因发现了一个新的SNP多态位点(GeneID:407159.11137T>C),这个SNP处于内含子区域,所以没有引起氨基酸的变化。在五个群体中未检测到突变纯合子BB基因型,秦川牛中只检测到AA基因型,这是由于突变基因频率在群体中很低,导致有时检测不到相应的突变纯合体,也有可能本研究中的样本数不多导致,或者抽样存在误差未检测到。通常群体中频率最高的等位基因是该物种中最原始最保守的基因,其余的等位基因是进化过程中由该等位基因突变造成的。利杂牛、利西杂牛、西杂牛和夏西杂牛均为杂合群体。本研究所发现的SNPs位点遗传多态性比较丰富,在利西杂牛和夏西杂牛群体中属于高度多态(0.25 在利西杂牛、利杂牛、西杂牛及秦川牛中,AA基因型占明显优势,而在夏西杂牛中,AB基因型所占比例较高。在五个牛群体中,等位基因A的频率明显高于等位基因B的频率。说明在利西杂牛、利杂牛、西杂牛及秦川牛中A等位基因为最原始、最保守的基因。值得一提的是,该位点秦川牛全部表现为A,表现为群体内单态,有可能该位点为秦川牛群体的独特位点,能否用来区分与其他牛种的不同,需要增加样本量进行进一步的研究。不同牛群体间HMGCR基因的基因型分布的卡方独立性检验结果显示,利西杂牛与秦川牛和西杂牛、夏西杂牛与秦川和西杂牛的差异极显著(P<0.01)。其余群体间差异不显著(P>0.05)。这说明夏西杂牛和利西杂牛可能由于选择压力和长期的肉用性状的选育造成该基因座的基因型分布发生改变,而肉用牛群体之间基因型分布也存在显著差异,说明各个肉牛群体的选育程度不同。而其他牛间不存在差异,也从另外一方面证明选育造成了基因型分布的改变。提示今后在育种过程中要加大选择力度,加强选育。 3.2HMGCR基因多态性对牛屠宰性状的影响 哺乳动物HMGCR是一种附着于内质网上的N-甘露糖糖蛋白,且是体内催化HMG-CoA生成甲羟戊酸(MVA)这一代谢途径的关键酶。它主要参与胆固醇的生物代谢,其生物活性的改变能够影响血脂水平[13-14],HMGCR基因对低密度脂蛋白受体的活性及mRNA表达有所影响[15-17]。对高脂血症大鼠肝脏mRNA的表达显著下调(P<0.05),对低密度脂蛋白受体mRNA的表达水平显着上调[18];HMGCR还参与其它化合物的代谢途径,如Ras蛋白的合成过程。即HMGCR基因,能够降低动物脂肪含量,改变动物胴体品质,因此HMGCR对研究新的胴体品质改良剂,提高动物胴体品质有重要意义。魏杨等[19]证明,HMGCR基因的变异可能使鸡蛋品质和胆固醇含量及总蛋白和低密度脂蛋白有显著影响。Cánovas等[20]通过表达和结构分析的方法,研究了HMGCR基因在猪脂肪代谢中的作用。对HMGCR基因编码区两个同义单核苷酸多态性与脂质沉积和胆固醇含量进行了了相关性分析,结果显示,c.807A等位基因与血清低密度脂蛋白(LDL)、胆固醇和甘油三酯以及肌内脂肪(IMF)含量成正相关。同时还评估了在肌肉和肝脏中HMGCR基因的表达水平,结果显示,HMGCR基因与血清高密度脂蛋白(HDL)的含量,胴体瘦肉率和硬脂酸含量呈负相关,在肌肉中的HMGCR的表达与胴体瘦肉率,硬脂酸和亚油酸含量成负相关,在肝脏中HMGCR表达与血脂胆固醇(高密度脂蛋白,低密度脂蛋白和总胆固醇)成正相关。结合我们的研究结果,认为HMGCR基因为牛体脂肪的调节基因,该位点两种基因型对屠宰性状产生了不同的影响。HMGCR基因多态性与利西杂牛、利杂牛、西杂牛及夏西杂牛屠宰性状的相关分析表明:在利西杂群体中AA基因型个体的屠宰率和牛柳极显著高于AB基因型个体(P<0.01);同时AA基因型个体与AB基因型个体相比,上脑达到了显著水平(P<0.05),这也证明了HMGCR基因可以作为脂肪代谢相关性状有用的分子标记。突变AB基因型的屠宰率、牛柳、上脑三个性状都及其显著低于纯合子AA基因型,可能是突变降低了氨基酸的合成速度,而且新的肽链结构也可能发生变化,从而降低了转录活性和细胞定位,导致生产性能的降低。 综上所述,HMGCR基因多态性对肉牛的胴体和肉质性状有一定的影响,尤其在脂肪沉积方面有一定的作用。本试验通过对HMGCR基因多态性研究为其功能研究奠定了良好的基础。并可以该多态性位点对肉牛的胴体、肉质性状进行分子标记辅助选择,指导肉牛的育种工作。 [1] Stormo C,Kringen M K,Grimholt R M,et al.A novel 3-hydroxy-3-methylglutaryl-coenzyme A reductase (HMGCR) splice variant with an alternative exon 1 potentially encoding an extended N-terminus[J].BMC Molecular Biology,2012,13(1):29-30. [2] Olof B,Quinci R,Pär-Ola B,et al.Targeting HMG-CoA reductase with statins in a window-of-opportunity breast cancer trial[J].Breast Cancer Res Treat,2013,138(2):499-508. [3] Yegneh B,Wiechec E,Andes S R,et al.Targeting the mevalonate cascade as a new therapeutic approach in heart disease,cancer and pulmonary disease[J].Pharmacology & therapeutics,2014,143(3):87-110. [4] Karthik M V K,Satya Deepak M V K N,Pratyoosh Shukla.Explication of interactions between HMGCR isoform 2 and various statins through In silico modeling and docking[J].Computers in Biology and Medicine,2012,42(2):156-163. [5] Kubota T,Fujisaki K,Itoh Y,et al.Apoptotic injury in cultured human hepatocytes induced by HMG-CoA reductase inhibitors[J].Biochemical pharmacology,2004,67(12):2 175-2 186. [6] Shahram E B,Gary H,Sandra N,et al.The 3-hydroxy-3-methylglutaryl-CoA reductase (HMGCR) pathway regulates developmental cerebral-vascular stability via prenylation-dependent signalling pathway[J]. Developmental Biology,2013,237(1):258-266. [7] Bengtsson E,Nerjovaj P,Wangefjord S,et al.HMG-CoA reductase expression in primary colorectal cancer correlates with favourable clinicopathological characteristics and an improved clinical outcome[J].Diagnostic Pathology,2014,9(1):78-79. [8] Debose-Boyd R A.Feedback regulation of cholesterol synthesis:sterol-accelerated ubiquitination and degradation of HMG CoA reductase[J].Cell Research,2008,18(6):609-621. [9] Sharpe L J,Brown A J.Controlling cholesterol synthesis beyond 3-hydroxy-3-methylglutaryl-CoA reductase (HMGCR)[J].Journal of Biological Chemistry,2013,288(26):18707-18715. [10] Canovas A,Quintanilla R,Gallardo D,et al.Functional and association studies on the pig HMGCR gene,a cholesterol-synthesis limiting enzyme[J].Animal,2010,4(2): 224-233. [11] 刘桂芬,王爱国,傅金恋.不同猪种HMGR基因的组织表达特性研究[J].家畜生态学报,2008,29(3):15-19. [12] 安光全.鸡HMGCR基因多态性与血清VLDL浓度和繁殖性状的关联研究[D].四川雅安:四川农业大学,2010. [13] Simko V,Ginter E.Understanding cholesterol:high is bad but too low may also be risky-is low cholesterol associated with cancer[J].Bratisl Lek Listy,2014,115(2):59-65. [14] Li Z,Meina H,Zengnan M,et al.A genome wide association study identifies common variants associated with lipid levels in the Chinese population[J].Plos One,2013,8(12):1-9. [15] Inmaculada N G,Horacio P S,Gala M P,et al.The inhibitory effects of bioactive compounds of tomato juice binding to hepatic HMGCR: In vivo study and molecular modeling[J].Plos One,2014,9(1):1-9. [16] Yu C Y,Theuhch E,Lo K,et al.HNRNPA1 regulates HMGCR alternative splicing and modulates cellular cholesterol metabolism[J].Human Molecular Genetics,2014,23(2):319-332. [17] Akadam T B,Kumaz O,Coskunpinar E,et al.The effects of age and gender on the relationship between HMGCR promoter-911 SNP (rs33761740) and serum lipids in patients with coronary heart disease[J].Gene,2013,28(2):93-98. [18] Shi Y,Guo R,Wang X,et al.The Regulation of Alfalfa Saponin Extract on Key Genes Involved in Hepatic Cholesterol Metabolism in Hyperlipidemic Rats[J].PloS one,2014,9(2):e88282. [19] 魏 杨.鸡HMGCR基因多态性及其与经济性状相关性研究[D].河南郑州:河南农业大学,2012. [20] Canovas A,Quintanilla R,Gallardo D,et al.Functional and association studies on the pig HMGCR gene,a cholesterol-synthesis limiting enzyme[J].Animal,2010,4(2):224-233. Three-Hydroxy-3-Methylglutaryl-CoA Reductase Gene and Its Correlation with Carcass Traits in Beef Cattle YAO Yu-ni1,SONG En-liang1,2△,LIU Xiao-mu1,2,CHENG Hai-jian1,2, YOU Wei1,2,HAN Hong1,2,LIU Gui-fen1,2* (1.ShandongProvincalKeyLaboratoryofAnimalDiseaseControlandBreeding, 2.ItituteofAimalSienceandVterinaryMicineShandongAademyofAriculturalSiences,Jinan250100,China) The 3-hydroxy-3-methylglutaryl-coenzyme A (HMG-CoA) reductase (HMGCR) is the rate-limiting enzyme in the cholesterol biosynthesis pathway,which plays an important role in the process of endogenouscholesterol biosynthesis.(Method)In this paper,PCR-SSCP technique and Sequencing Techniques were used to analyze the relationship between the polymorphisms ofHMGCRgene which was studied as a candidate gene.LZ cattle,LXZ cattle,QC cattle,XZ cattle and XXZ cattle,totally 123 beef cattle,were used as research populations.(Result)The fragments showing different SSCP (single strand conformation polymorphism) patterns were sequenced,and only one SNP (GenBank:BC153262.1 11137T>C) were found. The results showed that there were polymorphisms in this locus,but only AA and AB genotypes.Correlations betweenHMGCRgene and beef performances were analyzed,In LXZ cattle,the slaughter rate and tenderloin index of AA genotype compared with AB genotype reached a significant level(P<0.01), high rib of AA genotype was significantly higher than that of AB genotype (P<0.05),the difference of other index between genotypes was not significant (P>0.05). (Conclusion)The results indicated that the SNPs ofHMGCRgene had effect on the slaughter traits of LXZ cattle,it could be a candidate gene for beef modify and directed to the beef cattle breeding. beef;three-hydroxy-3-methylglutaryl-CoA reductase gene;PCR-SSCP 2014-04-28, 2014-07-14 [基金项目] 国家自然科学基金(31100890);2010年山东省优秀中青年科学家科研奖励基金(BS2010SW026);2011年山东省优秀中青年科学家科研奖励基金(BS2011SW043);“十二五”农村领域国家科技计划(2011BAD28B04-02) 姚玉妮(1981-),女,山东莱阳人,硕士,主要从事动物遗传育种研究。E-mail:yuniyao2008@163.com △与第一作者同等贡献。 *[通讯作者] 刘桂芬(1980-),女,山东东明人,博士,助理研究员,主要从事动物分子遗传研究。E-mail:liuguifen126@126.com S811.6 A 1005-5228(2015)03-0018-07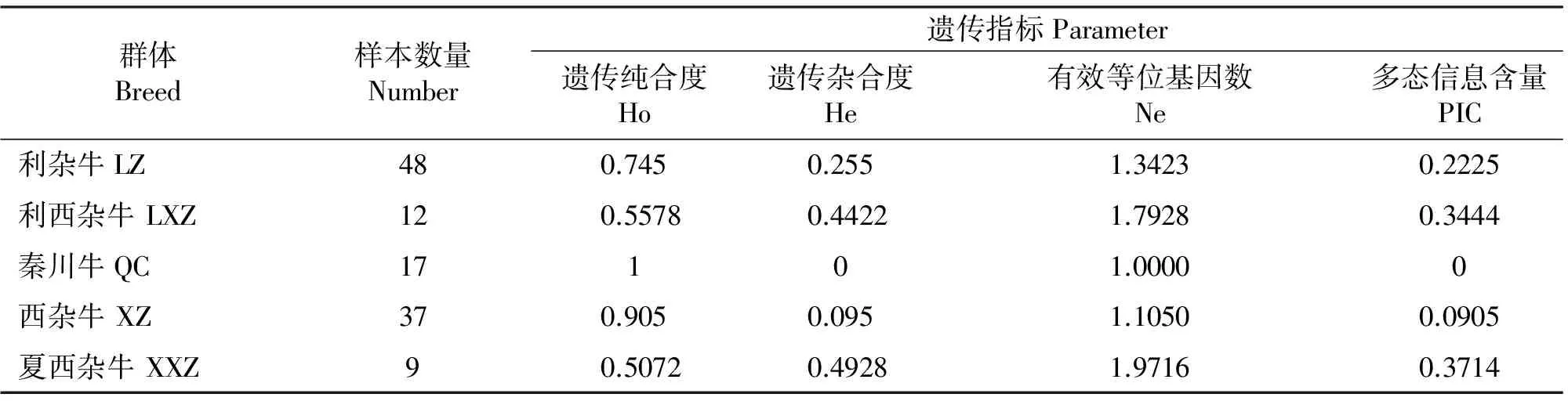


3 讨 论
4 结 论
