梨CDPK 基因家族全基因组序列鉴定分析
2015-04-02许园园李晓刚常有宏
许园园, 李晓刚, 李 慧, 蔺 经, 常有宏
(江苏省农业科学院园艺研究所,江苏省高效园艺作物遗传改良重点实验室,江苏 南京210014)
梨是重要的温带果树之一,属于蔷薇科苹果亚科,大多数的栽培品种都是二倍体(2n =34)。作为国际市场上受欢迎的水果之一,梨在6 个大洲都有广泛的种植,梨的主产国家有中国、美国、意大利、阿根廷、西班牙、韩国、土耳其、南非、日本和比利时,其中,中国为梨世界第一大生产国,年产量超过世界总产量的60%[1-3]。
钙依赖性蛋白激酶(CDPK)是植物和一些原生生物所特有的一类丝氨酸/苏氨酸蛋白激酶,可不需钙调素而被钙信号直接激活,在植物中,CDPK基因以翻译后形成的单肽链形式存在,典型的CDPK 蛋白分子由1 条多肽链组成,从N 端到C端存在4 个功能域[4-6]。在植物的器官水平上,CDPK基因广泛存在于根、茎、叶、果实和种子等器官,在植物钙信号转导中具有重要作用,在细胞水平上,分生细胞、木质部细胞、花粉细胞、保卫细胞和胚细胞中也均发现CDPK 蛋白组分的存在,且越来越多的研究结果表明,在植物碳氮代谢、离子和水分跨膜运输、气孔运动、细胞骨架与生长发育调节中均有CDPK 基因的参与,CDPK 在植物耐非生物胁迫的伤害应答和抗真菌的防卫应答中也发挥重要作用[7]。CDPK 基因在植物中数量众多,在模式植物拟南芥的基因组中,迄今已鉴定了34 个CDPK 基因,分布位于所有5 条染色体上[8];在水稻中,也已鉴定出29 ~31 个CDPK 家族成员[9];据推测,小麦基因组中也至少存在20 个CDPK 基因[10];另外,在大豆[11]、玉米[12]、番茄[13]、烟草[14]和棉花[7]中也鉴定了一些CDPK 基因;尽管对CDPK 基因的研究有了重大进展,但大多局限于模式植物中,而且仍有大部分的CDPK 基因未得到分离与功能鉴定。梨基因组序列的公布为利用生物信息学手段研究该重要果树作物成为可能,但是目前在全基因组水平上对梨CDPK 基因家族成员进行分离鉴定的工作尚未见报道。
本研究利用生物信息学分析方法,基于已公布的梨基因组全序列信息,分离鉴定出全部CDPK 基因家族成员,从基因组水平上分析了梨CDPK 基因的数目,基因结构与进化,为在基因组范围内研究CDPK 基因家族的生物学功能奠定基础。
1 材料与方法
1.1 材料
梨全基因组序列,基因及其注释信息均下载于梨基因组数据库(http://peargenome. njau. edu. cn:8004/)。拟南芥CDPK 基因(34 个)序列和蛋白序列下载于TAIR(http://www. arabidopsis. org/)。水稻CDPK 基因根据Asano 等[15]的研究结果,下载自http://rgp. dna. affrc. go. jp/E/IRGSP/rap-db1. html数据库,共得到31 条CDPK 蛋白序列。
试验于2014 年在江苏省农业科学院园艺研究所高效园艺作物遗传改良重点实验室进行。
1.2 CDPK 基因家族成员鉴定
利用现有梨注释基因的蛋白序列,与拟南芥、水稻CDPK 蛋白序列进行本地blastp 比对,E-value值为le-5,输出最优比对结果,根据比对长度(>400 aa)和相似度(>40%)筛选梨候选CDPK基因。同时结合Pfam 数据库,采用hmmscan 鉴定候选CDPK 基因蛋白结构域,筛选含有CDPK 蛋白典型结构域丝氨酸/苏氨酸蛋白激酶区以及EF-手型结构区(PF07714,PF00036)的蛋白序列[16]。综合上述结果,去除重复基因,得到26 条梨CDPK基因,根据其与拟南芥CDPK基因的相似性来命名。利用ExPASy Proteomics Sever (http://expasy.org/)对所有的CDPK 蛋白氨基酸序列进行分子量、等电点预测[17]。氨基酸修饰,同样采用在线工具进 行,Myristoylator 工具(http://prosite. expasy.org/scanprosite/)用于进行N-myristoylation 预测(http://prosite. expasy. org/scanprosite/)[18],palmitoylation 预测工具为CSS-Plam program[19],在线工具http://www. ebi. ac. uk/Tools/pfa/ps_scan/用于进行EF-手型结构预测[20]。
在梨26 个CDPK 基因中进行重复基因对筛选,筛选标准为:(1)比对长度大于两个基因中最长基因序列长度的80%;(2)比对相似性>80%;(3)只有一个重复事件才被认为是紧密关联的基因对。重复CDPK 基因对的Ka、Ks 值计算采用DnaSP5.0 软件,最后根据Ka/Ks 值来判断每对基因的选择压力[21]。
1.3 系统发育树的构建与蛋白保守域序列比对分析
通过ClustalW 模块对拟南芥、水稻、梨CDPK 蛋白进行多序列联配比对分析,多序列比对结果使用MEGA6.0(http://megasoftware. net)程序[22]采用邻接法(Neighbor-Joining,NJ)生成CDPK 基因的无根系统进化树,校验参数Bootstrap 重复1 000 次。CDPK基因家族保守性分析采用ClustalX 生物学软件进行多序列比对,并参照Schneider 等[23]的方法进行蛋白序列保守性分析。
1.4 CDPK 基因结构分析
根据预测结果,利用perl 程序从梨基因组注释信息(pear. gene. gff)中提取梨CDPK 基因的基因组注释信息(gff),得到梨CDPK 基因的基因组(Scaffold)位置信息。基于CDPK 的基因和CDS序列,选取GSDS(http://gsds. cbi. pku. edu. cn/)工具进行基因结构分析,绘制外显子-内含子结构图。
1.5 梨CDPK 基因与水稻、拟南芥的无根系统进化树构建
采用MEGA6.0 程序中的ClustalW 模块对梨、拟南芥和水稻的CDPK 蛋白序列进行多序列比对,采用邻接法(Neighbor-Joining,NJ)生成无根进化树。
2 结果
2.1 梨PbCDPKs 基因家族成员鉴定
利用生物信息学方法,从梨全基因组中鉴定获得26 个候选CDPK 基因家族成员,根据与拟南芥CDPK 基因的同源性分别命名为PbCDPK1 ~PbCDPK26(表1)。通过ExPASy 工具,对梨CDPK 基因进行了蛋白质长度、分子量及等电点等生化属性分析。基因组序列分析结果表明梨CDPK 基因非常保守,最长的梨CDPK 蛋白(PbCDPK6,Pbr010446.1)编码811 个氨基酸,最短的CDPK 蛋白(PbCDPK24,Pbr027545.1)仅编码499 个氨基酸。蛋白质生化属性分析发现,其等电点范围从4. 79(PbCDPK22,Pbr028710.1)到8. 77(PbCDPK16,Pbr007825. 1)(表1)。
2.2 梨PbCDPK 家族成员氨基酸序列保守性分析
利用PFAM 及NCBI-CDD 工具对CDPK 家族成员进行蛋白质结构分析发现,在与Ca2+结合的调控区,CDPK 成员均含有一段结构和功能类似于CaM 的氨基酸序列,这段序列中除PbCDPK16 含有3 个与Ca2+结合的EF-手型结构,其余均包含4 个EF 手型结构(图1)。此外,部分CDPK 成员N 端还发现了含有与蛋白质定位(膜定位)相关的豆蔻酰化和十六烷酰化所需的保守序列MGXXC(S/Q)XXT 位点(表1)。
2.3 梨PbCDPKs 家族基因结构与系统进化分析
为了解梨CDPK 蛋白系统进化关系,利用梨CDPK 蛋白全长序列构建了系统进化树,结果显示,26 个CDPK 基因可以分为4 类,参考拟南芥的研究结果,将4 类亚家族命名为Class Ⅰ、Class Ⅱ、Class Ⅲ和Class Ⅳ,分别含10 个、4 个、10 个和2 个CDPK 基因。此外,26 个CDPK 基因形成11 个旁系同源基因对,其中只有1 对基因步长值低于90,为PbCDPK17/PbCDPK15(图2)。对家族成员的基因结构分析显示,梨CDPK 基因结构相对复杂,有18 个CDPK 基因内含子数目为6 ~7 个,仅有1 个成员含有5 个内含子(PbCDPK3),1 个 成 员 含 有 10 个 内 含 子(PbCDPK16),3 个 成 员 含 有 8 个 内 含 子(PbCDPK6、PbCDPK14),2 个成员含有9 个内含子(PbCDPK1、PbCDPK22),另有2 个CDPK 基因不含内含子(PbCDPK7、PbCDPK24)。对CDPK成员聚类结果的进一步分析发现,位于同一旁系同源基因对中的基因其结构同样类似(图2)。另外,对PbCDPK 家族成员编码序列与基因组序列进行比较分析发现,在9 个PbCDPK 家族成员基因序列中存在UTR 非翻译区域(图2)。
为了深入分析梨与其他物种的同源进化关系,构建了梨与拟南芥、水稻的CDPK 基因系统进化树(图3),根据进化树聚类,可将所有的CDPK蛋白分为四类,分别为CDPK I,CDPK II,CDPK III,CDPK IV。CDPK I 包含了10 个AtCDPK,8 个OsCDPK 和10 个PbCDPK;CDPK II 中分别包含13个AtCDPK,5 个OsCDPK 和4 个PbCDPK;CDPK III 中分别包含8 个AtCDPK,8 个OsCDPK 和10 个PbCDPK;CDPK IV 中在四类分组中含有基因数目最少,分别包含3 个AtCDPK,2 个OsCDPK 和2 个PbCDPK。
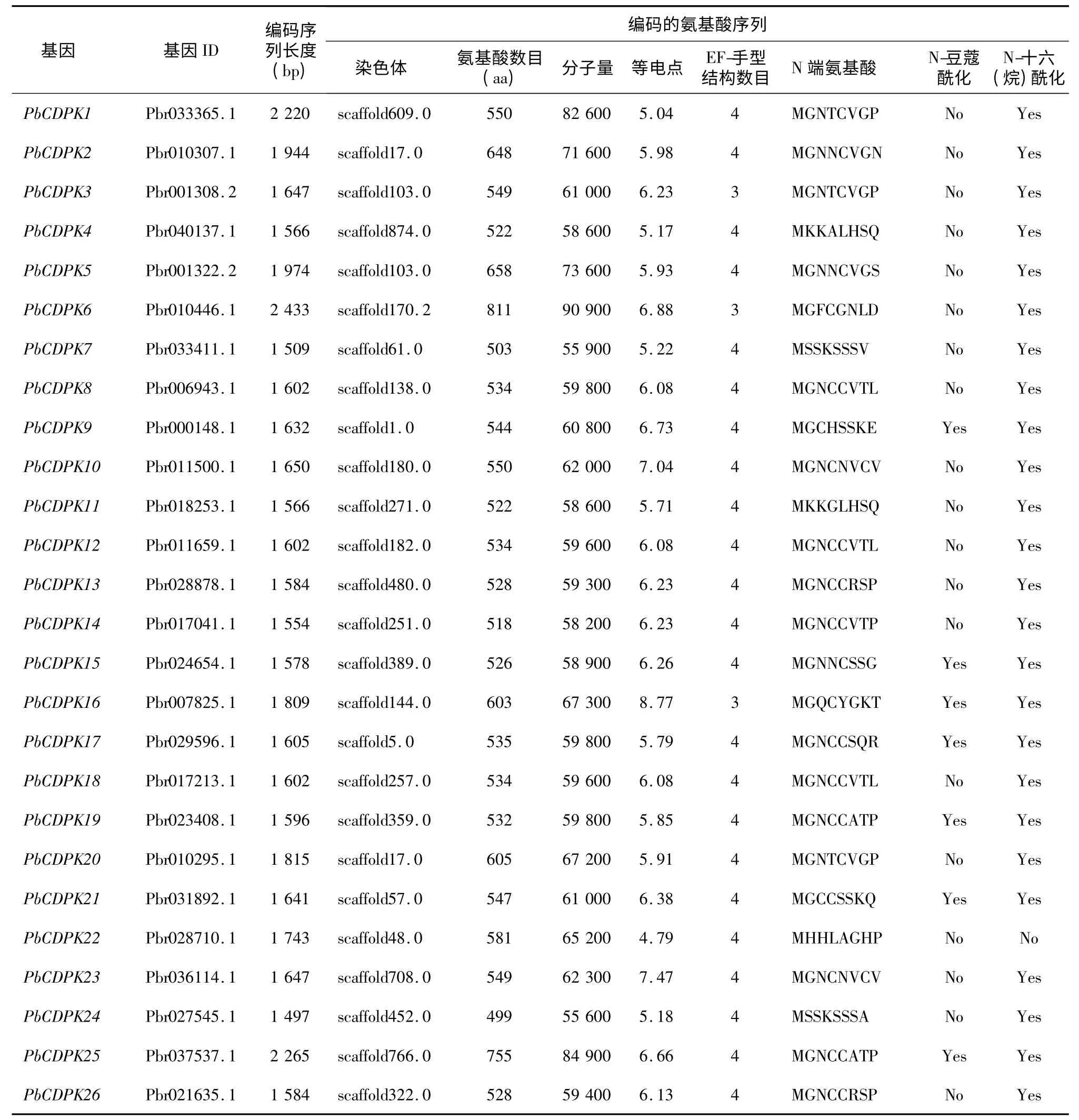
表1 梨CDPK 基因家族成员信息Table 1 The information of CDPK gene family in pear
3 讨论
目前,基因家族分析已在多种作物中有类似报道,如苹果SDH 和IPT 基因家族[24-25],大豆LEA 与ARF 基因家族[26-27],番茄LBD 基因家族等[28],为各个基因家族的功能分析提供了理论基础。CDPK 是植物体内含有Ser/Thr 激酶活性的一类蛋白激酶,研究结果表明,CDPK 是介导植物生长发育与逆境信号的关键信号传递体[7,29-30]。在植物体内,CDPK基因以家族基因的形式存在,在对模式植物拟南芥基因组的搜索中发现CDPK 含有34 个成员,杨树基因组中被鉴定含有30 个成员[31],在其他植物如马铃薯、豌豆等作物中也发现了部分CDPK 基因家族成员。梨作为一种重要的果树作物,基因组测序完成后,有关梨CDPK 家族基因的分析研究尚未见报道。本研究通过对梨进行全基因组扫描分析,从梨基因组中成功鉴定出26 个CDPK 家族成员,通过对其进行基因结构、系统进化等生物信息学分析,以期为深入揭示该基因家族在梨生长发育与信号转导中的功能和作用机制奠定基础。

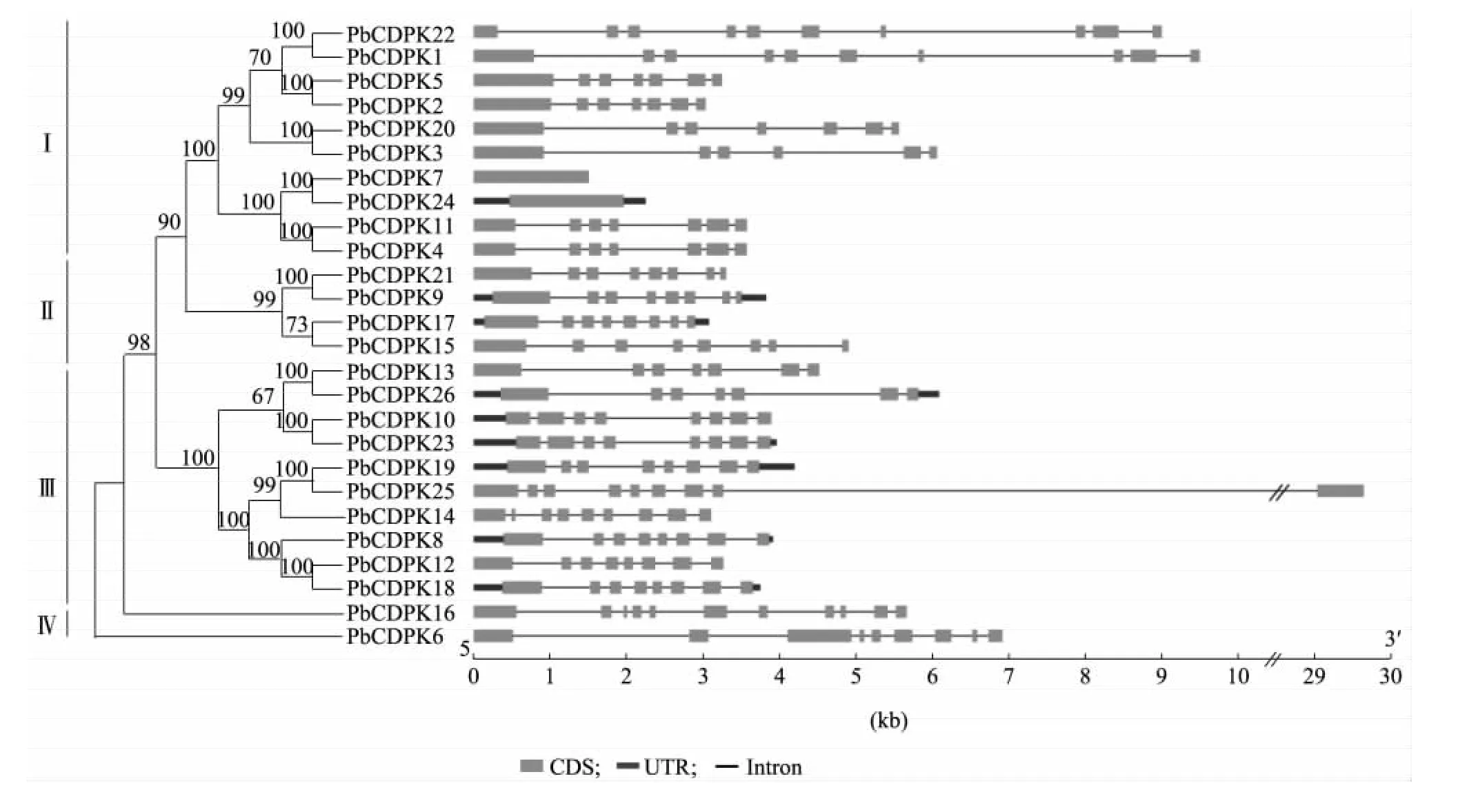
图2 梨CDPK 基因内含子外显子结构与家族进化树分析Fig.2 The intron-exon structures and the phylogenetic tree of CDPK genes in pear
总体来说,梨CDPK 基因结构较为复杂,外显子数目较多(6 ~11 个),这种复杂的基因结构可能会导致基因结构不稳定,且复制时容易产生可变剪切。研究报道,大多数的CDPK 基因在调控区含有4 个保守的与Ca2+结合的EF-手型结构,少数CDPK 含有3 个EF-手型结构,本研究中,还发现了2 个CDPK成员含有6 个EF-手型结构。通过这些手型结构可使CDPK 在不依赖于CaM 的条件下与Ca2+高度亲和。另外,部分CDPK 成员N 端存在与蛋白质定位(膜定位)相关的豆蔻酰化和十六烷酰化所需的保守序列MGXXC(S/Q)XXT 位点,推测这些结构可能在参与蛋白质与质膜的可逆结合或蛋白间相互作用过程中发挥重要作用。
通过系统进化树的构建,可以分析基因之间的起源关系,预测基因的功能。同一亚家族或小的分枝往往具备相似的功能,依据聚类分析,梨26 个CDPK 蛋白成员划分为4 组(CDPK I、CDPK II、CDPK III 和CDPK IV),表明梨CDPK 蛋白来自于不同的祖先,另外研究结果显示,CDPK I、CDPK III 和CDPK IV 在3 个物种(梨、拟南芥和水稻)中分别含有大致相同数量的CDPK,而较另外两个物种(梨和水稻),CDPK II 则仅在拟南芥中含有数量较多的CDPK 成员,该聚类结果与棉花CDPK 蛋白分类相一致[7],由于植物种属的CDPK 可能来自于蛋白激酶和CaM 基因的融合,因此,CDPK 各成员的结构与序列组成上的较大差异,可能与蛋白激酶或CaM 在序列上存在的差异有关。3 个物种的亲缘关系聚类分析结果表明,所有的CDPK 蛋白在3 个物种中均被分成不同的4 个类别,暗示这些分组在单子叶植物与双子叶植物分化前就已存在,该结论同样支持了在藻类和陆生植物分化前CDPK 家族基因就已被分为4 类的假设[32]。
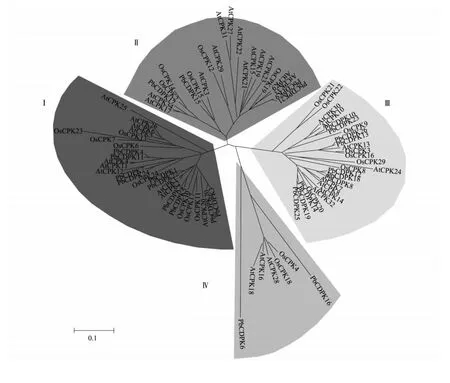
图3 梨CDPK 蛋白与拟南芥、水稻CDPK 蛋白的亲缘关系Fig.3 Phylogenetic relationships of CDPK proteins in Arabidopsis,rice and pear
随着研究深入,可能会发现更多的梨CDPK同源基因,这些CDPK 家族基因如何响应激素信号调控梨生长发育,如何参与对逆境反应的信号传导等都将成为今后研究的重点。
[1] 李刚波,李 慧,丛 郁,等. 非生物胁迫下杜梨PbCBL4 基因的表达[J]. 江苏农业学报,2014,30(5):1132-1138.
[2] 蔺 经,李晓刚,李 慧,等.沙梨新品种苏翠1 号组培快繁体系研究[J].江苏农业科学,2014,42(11):66-67.
[3] 王程亮,徐丽君,倪 萌,等.无锡地区桃园梨小食心虫发生规律[J].江苏农业科学,2014,42(9):117-119.
[4] HARMON A C,GRIBSKOV M,GUBRIUM E,et al. The CDPK superfamily of protein kinases[J]. New Phytologist,2001,151:175-183.
[5] HARPER J F,SUSSMAN M R,SCHALLER G E,et al. A calcium-dependent protein kinase with a regulatory domain similar to calmodulin[J]. Science,1991,252:951-954.
[6] HRABAK E M,CHAN C W,GRIBSKOV M,et al. The arabidopsis CDPK-SnRK superfamily of protein kinases [J]. Plant Physiology,2003,132:666-680.
[7] LIU W,LI W,HE Q,et al. Genome-wide survey and expression analysis of calcium-dependent protein kinase in Gossypium raimondii[J]. PLoS One,2014,9(6):e98189.
[8] CHENG S H,WILLMANN M R,CHEN H C,et al. Calcium signaling through protein kinases the Arabidopsis calcium-dependent protein kinase gene family[J]. Plant Physiology,2002,129(2):469-485.
[9] RAY S,AGARWAL P,ARORA R,et al. Expression analysis of calcium-dependent protein kinase gene family during reproductive development and abiotic stress conditions in rice (Oryza sativa L.ssp. indica)[J]. Molecular Genetics and Genomics,2007,278:493-505.
[10] LI A L,ZHU Y F,TAN X M,et al. Evolutionary and functional study of the CDPK gene family in wheat (Triticum aestivum L.)[J]. Plant Molecular Biology,2008,66:429-443.
[11] LEE J Y,YOO B C,HARMON A C. Kinetic and calcium-binding properties of three calcium-dependent protein kinase isoenzymes from soybean [J]. Biochemistry,1998,37:6801-6809.
[12] KONG X,LV W,JIANG S,et al. Genome-wide identification and expression analysis of calcium-dependent protein kinase in maize[J]. BMC Genomics,2013,14:433.
[13] CHICO J M,RAICES M,TERESA M T,et al. A calcium-dependent protein kinase is systemically induced upon wounding in tomato plants[J]. Plant Physiology,2002,128:256-270.
[14] 太帅帅,刘贯山,孙玉合,等. 普通烟草CDPK 基因家族的克隆及表达分析[J].中国农业科学,2009,42(10):3600-3608.
[15] ASANO T,TANAKA N,YANG G,et al. Genome-wide identification of the rice calcium-dependent protein kinase and its closely related gene families:comprehensive analysis of the CDPKs gene family in rice[J]. Plant Cell Physiology,2005,46(2):356-366.
[16] XU Q,DUNBRACK R L. Assignment of protein sequences to existing domain and family classification systems:Pfam and the PDB[J]. Bioinformatics,2012,28(21):2763-2772.
[17] ARTIMO P,JONNALAGEDDA M,ARNOLD K,et al. ExPASy:SIB bioinformatics resource portal[J]. Nucleic Acids Research,2012,40(Web Server issue):597-603.
[18] BOLOGNA G,YVON C,DUVAUD S,et al. N-Terminal myristoylation predictions by ensembles of neural networks[J]. Proteomics,2004,4:1626-1632.
[19] REN J,WEN L,GAO X,et al. CSS-Palm 2.0:an updated software for palmitoylation sites prediction[J]. Protein Engineering Design Selection,2008,21:639-644.
[20] DE CASTRO E,SIQRIST C J,GATTIKER A,et al. ScanProsite:detection of PROSITE signature matches and ProRule-associated functional and structural residues in proteins[J]. Nucleic Acids Research,2006,34 (Web Server issue):362-365.
[21] LIBRADO P,ROZAS J. DnaSP v5:a software for comprehensive analysis of DNA polymorphism data[J]. Bioinformatics,2009,25:1451-1452.
[22] TAMURA K,STECHER G,PETERSON D,et al. MEGA6:Molecular evolutionary genetics analysis version 6.0[J]. Molecular Biology Evolution,2013,30(12):2725-2729.
[23] SCHNEIDER T D,STEPHENS R M. Sequence logos:a new way to display consensus sequences [J]. Nucleic Acids Research,1990,18:6097-6100.
[24] 梁 东,吴 钐,王素芳,等. 苹果山梨醇脱氢酶基因家族的克隆及表达分析[J]. 中国农业科学,2012,45(1):102-110.
[25] 李 皓,张 文,赵旭勉,等. 苹果异戊烯基转移酶基因家族(MdIPTs)的克隆与MdIPT5a 功能分析[J]. 中国农业科学,2011,44(19):4029-4036.
[26] 李 乐,许红亮,杨兴露,等. 大豆LEA 基因家族全基因组鉴定、分类和表达[J]. 中国农业科学,2011,44(19):3945-3954.
[27] HA C V,LE D T,NISHIYAMA R,et al. The auxin response factor transcription factor family in soybean:genome-wide identification and expression analyses during development and water stress[J]. DNA Research,2013,20(5):511-524.
[28] 王小非,刘 鑫,苏 玲,等. 番茄LBD 基因家族的全基因组序列鉴定及其进化和表达分析[J]. 中国农业科学,2013,46(12):2501-2513.
[29] DUBROVINA A S,KISELEV K V,KHRISTENKO V S. Expression of calcium-dependent protein kinase (CDPK)genes under abiotic stress conditions in wild-growing grapevine Vitis amurensis[J]. Journal Plant Physiology,2013,170:1491-1500.
[30] WITTE C P,KEINATH N,DUBIELLA U,et al. Tobacco calcium-dependent protein kinases are differentially phosphorylated in vivo as part of a kinase cascade that regulates stress response[J].Journal Biological Chemistry,2010,285:9740-9748.
[31] ZUO R,HU R,CHAI G,et al. Genome-wide identification,classification,and expression analysis of CDPK and its closely related gene families in poplar (Populus trichocarpa)[J]. Molecular Biology Reporter,2013,40:2645-2662.
[32] CHEN F,FASOLI M,TORNIELLI G B,et al. The evolutionary history and diverse physiological roles of the grapevine calcium-dependent protein kinase gene family [J]. PLoS One,2013,8:80818.
