南极冰鱼和伯氏肩孔南极鱼EPO基因预测及生物信息学分析*
2015-03-22姜华鹏王丛丛许强华
张 驰 姜华鹏 王丛丛, 许强华,,
(1. 上海海洋大学海洋科学学院 上海 201306; 2. 大洋渔业资源可持续开发省部共建教育部重点实验室 上海 201306;3. 农业部大洋渔业资源环境科学观测试验站 上海 201306; 4. 国家远洋渔业工程技术研究中心 上海 201306;5. 远洋渔业协同创新中心 上海 201306)
南极位于地球最南端, 又称“第七大陆”, 为全球气候变化敏感区。南极水域低温高氧, 生存着许多鲜为人知的极地水生生物。南极所具有的独特地理及气候特征, 造就了极地生物特殊的生理特征, 使其能够适应南极低温高氧极地环境, 在生物进化中极具研究价值。南极圈内生活的南极鱼已成为研究鱼类适应性进化和耐寒机制的优良模型。
作者在本文中以独角雪冰鱼(Chionodraco hamatus,CH)和伯氏肩孔南极鱼(Trematomus bernacchii, TB)为主要研究对象。独角雪冰鱼属于鲈形目(Perciformes)、南极鱼亚目(Notothenioidei)、鳄冰鱼科(Channichthyidae),生活在南极水深200—800 m的水域中, 体表无鳞片,体内不含血红蛋白, 可能通过鳃和极薄的皮肤吸收海水中的氧(Bargelloniet al, 1994; Eastmanet al,2004)。伯氏肩孔南极鱼为鲈形目(Perciformes)、南极鱼亚目(Notothenioidei)、南极鱼科(Notothenioidei),生活在南极水深700m以上的水域中, 其生存环境与独角雪冰鱼相似且与独角雪冰鱼有较近的亲缘关系,但体内表达有正常的血红蛋白。本文以探究独角雪冰鱼的进化机制为大背景, 从对血红蛋白生成有促进作用关键因子之一 EPO入手, 来探究血红蛋白丢失的潜在机制以及EPO在其中起的作用。
促红细胞生成素(erythropoietin, EPO) 是哺乳动物调节红细胞生成的主要调控因子, 主要由肾脏和胚胎肝脏分泌。研究表明EPO为氧依赖型基因, 缺氧可以诱导 EPO基因的转录, 从而达到调控效果(葛全兴等, 2009)。尤其是低氧诱导因子-1(hypoxia inducing factor-1, HIF-1), 在缺氧条件下能够进入核内形成异二聚体HIF-1复合物, 并结合在EPO的3¢端约120 bp的区域内的顺式调控元件上(桂长云等, 1999)。该顺势调控元件为(5¢-RCGTG-3¢)保守序列构成, 是典型的低氧反应元件(hypoxia response element, HRE)(桂长云等, 1999)。EPO作为一种糖蛋白激素, 能够刺激骨髓造血功能, 及时有效地增加红细胞的数量, 从而提高血液的携氧能力, 增加血红蛋白含量与红细胞比积(血液中红细胞百分比)(Katakuraet al, 2013)。高海拔哺乳动物低氧促进红细胞生成是通过提高血浆EPO水平来实现的, 以改善缺氧引起的生理反应; 红细胞增多又可反馈性抑制 EPO的生成, 以使红细胞量精确地适应机体的需要。在非哺乳动物中, EPO的保守性以及刺激肾脏中红色祖先细胞分裂和分化作用同哺乳动物是一致的(Stachuraet al, 2009, 2011)。在非洲爪蟾体外实验中也同样证明 EPO系统在正常状态下刺激幼红血细胞的增殖, 并使其在急性严重贫血情况中恢复过来(Nogawa-Kosakaet al, 2010)。这表明 EPO系统在红系器官发育中具有关键作用。近年来, 体外和动物实验报道都暗示了 EPO的作用不仅仅局限于红系细胞生成。在非造血系统中, EPO在低氧状态下具有神经营养和神经保护作用(Kulkarniet al, 2010; Ostrowskiet al, 2011)。
值得一提的是, 鱼类 EPO功能的研究尚处于起步阶段。人们先后在河豚、斑马鱼中发现了 EPO基因的结构、HRE(低氧反应元件)所在位点, 对EPO在鱼类缺氧应答机制中所起的作用有了初步认识(Chouet al, 2004; Chuet al, 2007; Paffett-Lugassyet al,2007)。对鱼类EPO结构和功能的研究仍将继续。本研究首次获得了独角雪冰鱼、伯氏肩孔南极鱼的EPO基因序列, 对其分子进化水平进行了初步探索, 以期在 EPO对鱼类的功能研究方面做出贡献, 为极端环境下的生物长期适应机制提供帮助。本研究为揭示EPO基因在南极冰鱼内的遗传特性提供理论依据,并为进一步探讨 EPO基因在极端低温高氧环境中的鱼类适应性进化研究奠定了基础。
1 材料与方法
1.1 材料
1.1.1 实验动物 独角雪冰鱼和伯氏肩孔南极鱼样本分别采集于南极埃默里冰架和南极熊猫码头。利用冰凿打冰洞, 再进行渔钓的方式, 获得活体南极鱼样本数十条。活体迅速冻存于–80°C超低温冰箱。由雪龙号带回的冻存样本用锡箔纸包裹后液氮带回上海海洋大学海洋科学学院保护遗传学实验室, –80°C保存备用。
1.1.2 主要试剂 Taq DNA Polymerase (艾德莱);Agarose N、氯仿、异丙醇、5×TAE 缓冲液、琼脂糖、氯化钠、酵母提取物(上海生工); 6×loading buffer、0.1%DEPC 及 75%乙醇(天根); X-gal、IPTG (Amersco);DNA胶回收试剂盒(Axygen Biosciences); TRIzol®试剂 (Invitrogen Corp); 胰蛋白胨 (OXOID); pMD19-T载体、DH5α感受态细胞及DL5000 DNA标记(TaKaRa);Trans 2K Plus DNA标记(TransGen)。
1.2 方法
1.2.1 南极鱼基因组 DNA(gDNA)提取及 PCR扩增取独角雪冰鱼和伯氏肩孔南极鱼的肌肉组织, 参照Trizol法(Invitrogen Corp, USA)分别提取其基因组DNA。用NanoDrop®2000 (Thermo)与1.5%琼脂糖凝胶电泳检测提取质量。根据GenBank数据库中EPO相应的同源序列(表 1)(Katakuraet al, 2013), 利用Primer Premier 5软件设计特异性引物, 引物由上海生工合成(表2)。

表1 EPO相应的同源序列信息Tab.1 The information of the respective counterparts of EPO homologous sequences

表2 EPO基因PCR引物信息Tab.2 The information of PCR primers of EPO gene
PCR 总反应体系为 25 μL, 含 0.5 μL 10 μmol/L正向和反向引物、2.5 μL 10×Buffer (含 MgCl2)、2 μL dNTP Mixture (各 2.5 mmol/L)、0.5 μL Taq DNA 聚合酶(5U/μL)。反应程序为 94°C 预变性 5 min; 94°C 变性30 s, 58—64°C退火30 s (退火温度依引物而异),72°C延伸1—2 min (延伸温度依扩增片段长短而异),32个循环; 72°C终延伸10 min。PCR产物用1%的琼脂糖凝胶电泳检测。
1.2.2 PCR扩增产物的克隆、转化与测序 琼脂糖凝胶回收试剂盒回收目的片段, 与 pMD19-T克隆载体于 16°C下连接过夜; 以连接产物转化大肠杆菌DH5α菌株感受态细胞, 蓝白斑筛选阳性克隆。菌落PCR (以阳性菌落为模板)鉴定重组转化菌(莎姆布鲁克, 2002; 陈书霞等, 2006)。经菌落PCR鉴定为阳性克隆的菌落重新挑入500 μL含Amp的LB培养基中37°C 210 r/min培养14 h后, 送上海生工进行双向测序, 测序引物为载体通用引物。
1.2.3 生物信息学分析 使用Seqman、Editseq等软件进行测序序列片段拼接; 通过 NCBI blast two sequences完成mRNA序列与基因组DNA比对, 利用相关软件处理结果, 并仔细核对序列信息; EPO基因开放阅读框(ORF)利用NCBI的ORF Finder程序分析(罗轶, 2008); 使用 Smart在线分析网站(http://smart.embl-heidelberg.de/)以及 NCBI中的 CDD 库(Conserved Domain Database)进行结构域的保守性分析(Marchler-Baueret al, 2005); 使用Scanprosite在线软件(http://prosite.expasy.org/cgi-bin/prosite/)分析其功能位点差异; 使用SignalP 4.1 server在线处理网站(http://www.cbs.dtu.dk/services/ SignalP/)分析蛋白潜在信号肽; 利用 ProtParam (http://web.expasy.org/protparam/)在线分析氨基酸序列的理化特性(Wilkinset al, 1999); 利用Protscale网站(http://web.expasy.org/protscale/)分析蛋白质亲疏水性; 使用 PredictProtein网站(http://www.predictprotein.org)和 Psipred分析网站(http://bioinf.cs.ucl.ac.uk/psipred/)预测蛋白质二级结构(李盛杰等, 2013); 根据PSD数据库和SWISS -MODEL在线建模网站(http://swissmodel.expasy.org/)进行同源建模, 构建蛋白质的3D模型(陶士珩, 2007;刘虎岐等, 2010); 利用PyMOL和Spdbv软件修饰并输出其 3D 结构(李盛杰等, 2013)。最后使用DNAMAN、MEGA软件进行同源性分析并构建其系统进化树(季舒涵等, 2010; 张宝等, 2013)。
2 结果与分析
2.1 南极鱼EPO基因结构及序列比较
通过 PCR分段扩增, 分别获得了独角雪冰鱼和伯氏肩孔南极鱼DNA片段(图1)。阳性克隆菌经过测序后获得测序结果, 利用Editseq和Seqman将测序结果进行序列拼接后, 获得了该基因扩增的核心区及侧翼区部分序列。其中, 得到独角雪冰鱼基因全长2727 bp, 基因编码区558 bp, 而伯氏肩孔南极鱼基因全长2820 bp, 基因编码区558 bp。为保证分析序列结果的可靠性, 拼装EPO 序列与红旗东方 鲀、罗非鱼等同源性高的相关物种进行序列特征性比对, 结果证明其拼接序列位于基因组EPO区域。
对 EPO基因的结构分析表明, 独角雪冰鱼和伯氏肩孔南极鱼的EPO皆由5个外显子组成, 中间穿插有4个内含子。其序列差异比对结果表明, 这两个基因gDNA序列相似性为80.1%, 其主要的序列差异位于内含子非编码区, 共有6个Gap, 平均Gap长度为19 bp, 最大一致序列长度为640 bp。独角雪冰鱼同红鳍东方 鲀序列相似性仅48.9%, 表明EPO基因的种属差异性(图2)。
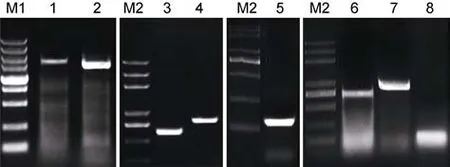
图1 南极鱼扩增产物电泳图Fig.1 Electrophoresis of Antarctic ice fish EPO amplified fragmentsM1: DL5000 DNA marker; M2: Trans 2K Plus DNA marker; 独角雪冰鱼EPO扩增片段1, 2, 3, 4 (1, 3, 4, 5); 伯氏肩孔南极鱼EPO扩增片段1, 2, 3, 4 (2, 6, 7, 8)
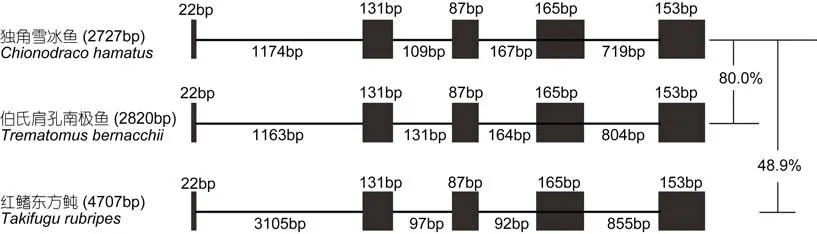
图2 南极鱼基因结构示意图Fig.2 Diagram of EPO genetic structures of Antarctic icefish黑色方框表示为外显子区域, 直线表示为内含子区域。红旗东方 鲀EPO基因为外源参考基因
2.2 南极鱼EPO基因 CDS区核苷酸序列及编码氨基酸序列分析
为了探讨EPO基因的功能域在独角雪冰鱼、伯氏肩孔南极鱼中的差异, 对克隆的基因序列进行比对(图3)。两者之间的ORF序列之间同源性为95.2%,表明南极鱼中的相对保守性。EPO蛋白前体为一种跨膜糖蛋白分子。根据SignalP 4.1 server、SMART软件和NCBI检测得到两者的EPO都包含一个信号肽区和一个EPO_TPO保守结构域(图4)。独角雪冰鱼和伯氏肩孔南极鱼 EPO基因 ORF推导氨基酸序列之间存在16处位点差异, 其中EPO_TPO区域有15个氨基酸的差异。经过Scanprosite的分析, 结果表明伯氏肩孔南极鱼有两个苏氨酸(Thr)和一个丝氨酸(Ser)可以成为蛋白激酶 C磷酸化位点, 有一个苏氨酸和三个丝氨酸可以成为酪蛋白激酶Ⅱ的磷酸化位点; 而相比较而言, 独角雪冰鱼分别增加了一个丝氨酸位点可以被蛋白酶C和酪蛋白激酶Ⅱ磷酸化(图 4)。

图3 南极鱼核苷酸序列间比对Fig.3 Alignment of nucleotide sequences of Antarctic ice fish EPO灰色部分表示物种间氨基酸核苷酸序列相同; 白色表示物种间核苷酸序列不同
2.3 南极鱼EPO蛋白质结构分析
2.3.1 EPO蛋白理化性质分析 用ProtParam软件预测南极鱼 EPO的理化性质, 结果显示独角雪冰鱼和伯氏肩孔南极鱼皆由185个氨基酸组成, 其分子式分别为 C904H1472N262O271S8和 C907H1469N257O267S9, 相应分子量分别为20.60 kDa和20.53 kDa, 理论等电点(pI)分别为6.65和6.28, 蛋白呈酸性。独角雪冰鱼EPO氨基酸残基中Leu (16.8%)、Ser (9.2%)及 Val (8.1%)的频率较高, 疏水性氨基酸占 43.8%, 极性不带电荷氨基酸占 32.4%, 极性带电荷氨基酸占 23.8%, 其中包括 10.3%的酸性氨基酸(D, E)及 13.5%的碱性氨基酸(H, R, K); 伯氏肩孔南极鱼中 Leu (18.4%)、Ser(8.1%)、Ala (7.0%)的频率较高, 疏水性氨基酸占45.4%, 极性不带电荷氨基酸占 30.8%, 极性带电荷氨基酸占 23.8%, 其中包括 10.3%的酸性氨基酸及13.5%的碱性氨基酸(表3)。独角雪冰鱼和伯氏肩孔南极鱼 EPO 蛋白消光系数(M–1cm–1γ=280nm)同样为23740, 其不稳定系数分别为36.00和36.87(通常当不稳定系数小于 40时, 可将蛋白质确定为稳定类蛋白质), 皆属于稳定类蛋白质(Guruprasadet al, 1990),且预计在体外哺乳动物网织红细胞内的半衰期为30 h。独角雪冰鱼疏水指数为106.43, 伯氏肩孔南极鱼疏水指数为110.6, 平均亲水性分别为 –0.071和0.022, 都属可溶性蛋白范围(Kyteet al, 1982)。
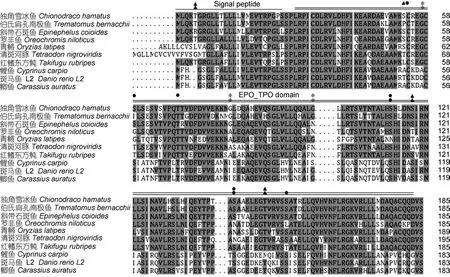
图4 EPO氨基酸序列多重比对及保守结构域和功能位点分析Fig. 4 Alignment of amino acid sequences and conservative domain structure and functional sites of Antarctic icefish EPO
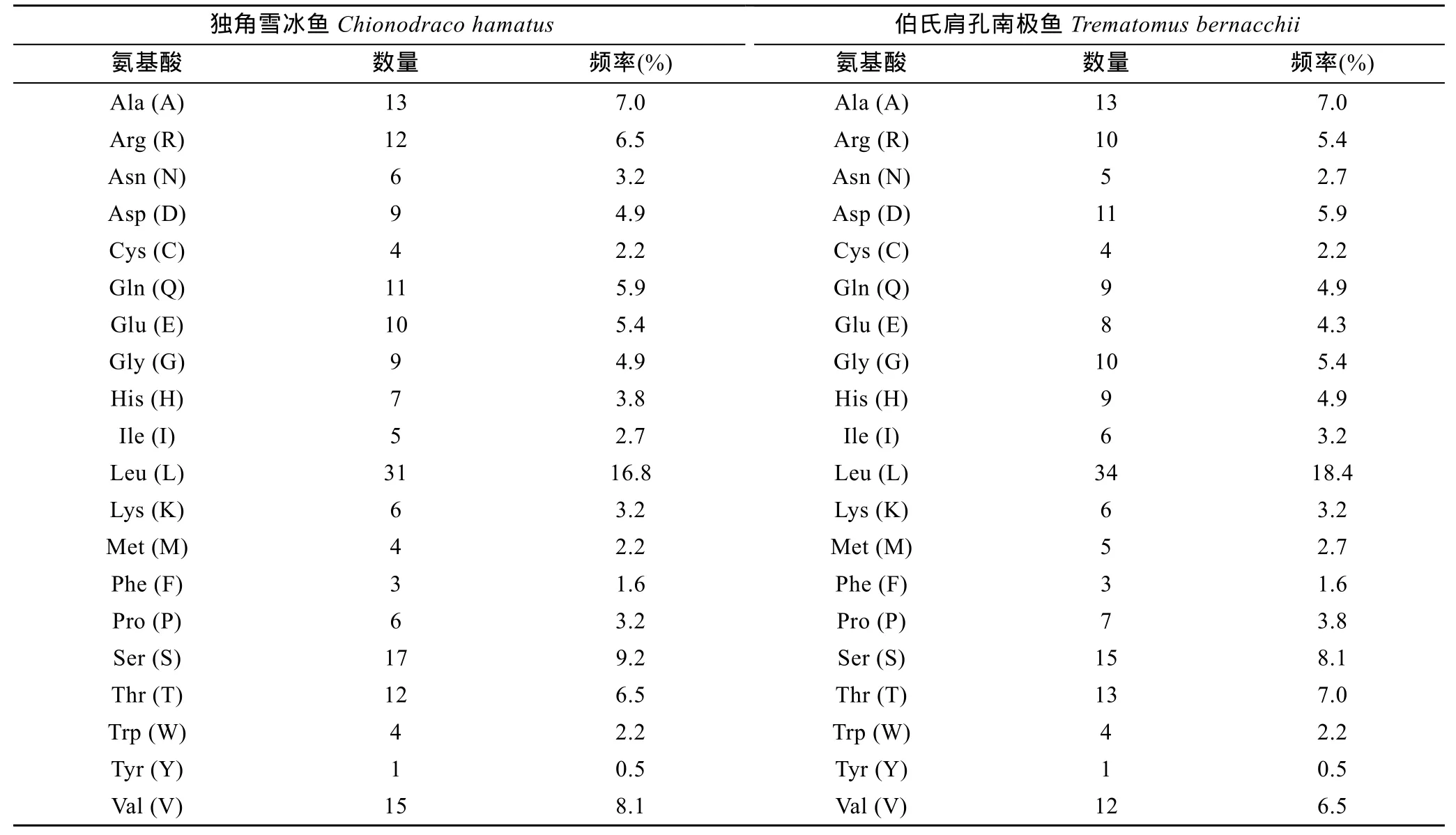
表3 南极鱼EPO的氨基酸组成Tab.3 The composition of amino acids in EPO
2.3.2 南极鱼 EPO二级、三级结构预测 使用PredictProtein和 PSIPRED软件预测南极鱼的二级结构, 结果表明: 独角雪冰鱼和伯氏肩孔南极鱼EPO_TPO域都主要由 4个反相平行的 α螺旋和两长两短的环状结构(Loops)区域构成, 为 EPO蛋白的典型结构(Boisselet al, 1993), 仅伯氏肩孔南极鱼预测出置信度较低的两个小的β折叠区域(图5)。利用 SWISS-MODEL同源建模网站和 PyMol软件对南极冰鱼 EPO建模获得三级结构模型, 该结果表明独角雪冰鱼和伯氏肩孔南极鱼都呈4个反向平行的α螺旋结构, 由α螺旋与无规卷曲构成。即使同源构建模型所用模板不同, 其主要的4个螺旋结构也是保守的, 这与二级结构预测结果基本一致(图 6)。
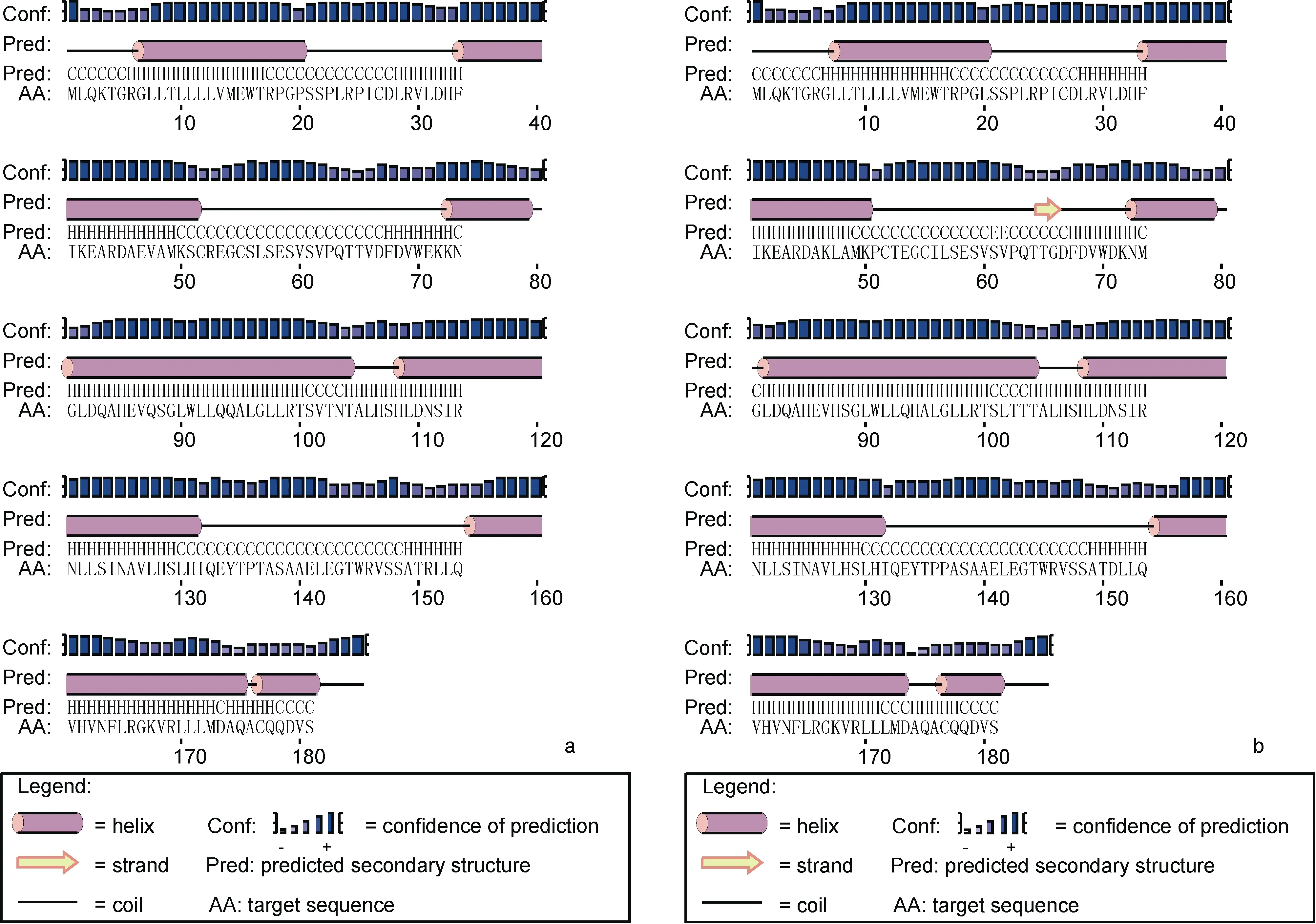
图5 南极鱼EPO的二级结构预测Fig.5 Predictions of the secondary structure of the Antarctic icefish EPOa. 独角雪冰鱼二级结构预测结果; b. 伯氏肩孔南极鱼二级结构预测结果
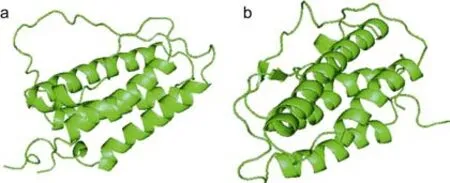
图6 南极鱼EPO的三级结构预测Fig.6 Predictions of the tertiary structure of the Antarctic icefish EPOa. 独角雪冰鱼EPO同源建模结果(以PDB数据库中1eer.1.A为模板); b. 伯氏肩孔南极鱼EPO同源建模结果(以PDB数据库中1cn4.1.C为模板)
2.4 南极鱼EPO同源性及系统发育分析
将独角雪冰鱼和伯氏肩孔南极鱼 EPO基因的氨基酸序列与其它物种的相应序列进行同源性分析(图7)。其中, 独角雪冰鱼EPO与其它鱼类的同源性在 70%—90%之间, 与两栖类的同源性约 85.5%, 与哺乳动物同源性在 53.9%—85.5%之间。数据表明,EPO种内差异较小, 同源性总体较高, 进化比较保守;但EPO基因的种间差异比较明显。用MEGA5软件分析南极冰鱼及其它鱼类、两栖类、哺乳类 EPO基因编码区氨基酸系统发生树, 其中独角雪冰鱼和伯氏肩孔南极鱼与其它鱼类的遗传关系很近, 与两栖类、哺乳类的遗传距离较远(图8)。
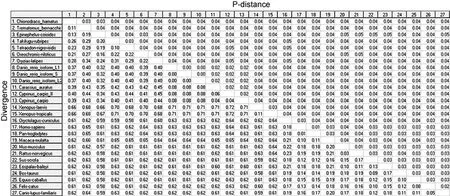
图7 各物种EPO基因推定的氨基酸序列间的遗传距离比较Fig.7 The sequence identity of inferred EPO amino acid sequences
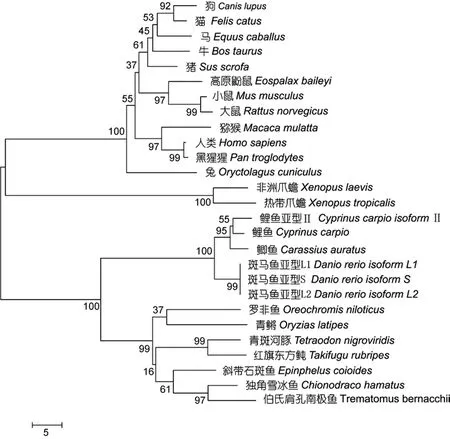
图8 利用MEGA软件中N-J法构建的EPO系统发育树Fig. 8 The Neighbor-Joining tree of EPO constructed with MEGA5 software
3 讨论
南极作为地球上特殊的极端环境之一, 其特殊物种南极冰鱼在长期进化选择过程中所占据的重要位置值得去做深入研究。EPO是与低氧适应、组织保护和血系生成相关的重要基因, 但目前还未获得其在极端环境下尤其是低温下的表达调控的机制和功能。尤其是南极冰鱼的特殊存在以及其基因组研究价值, 为人们提供了一个优秀的耐寒基因库(许强华等,2014)。南极鱼类通过三千万年的快速进化中, 在南大洋逐渐变冷直至冰冻的期间演化成了南大洋最多样化的鱼种。作者希望通过比较基因组学、生态学、生理学以及生物统计学等相关专业知识, 对 EPO基因获得更深层的认识, 推测它在生物适应性进化过程中所起的作用。尤其是在全球变暖的生态大背景下,南极冰鱼如何适应正在慢慢变化的南极低温环境。目前, 针对南极水生动物 EPO的研究成果在国内外鲜有发布, 而对于南极特有的水生动物之一南极冰鱼的研究更是少之甚少。
对独角雪冰鱼和伯氏肩孔南极鱼 EPO基因结构进行分析, 长度分别为2727 bp和2820 bp。南极鱼EPO基因结构在鱼类中具有一致性, 都包含5个外显子和4个内含子, 种属内部同源性较高, 差异性主要体现在非编码区。而针对编码区的研究发现, 南极鱼EPO推导氨基酸数目与其它鱼类基本一致, 但与两栖类、哺乳动物类差异性较大。独角雪冰鱼与伯氏肩孔南极鱼相比, 尽管存在有 26个碱基差异, 其中 10个碱基突变均属同义突变, 所编码的氨基酸序列同源性为91.35%。说明独角雪冰鱼EPO与伯氏肩孔南极鱼EPO一致, 两者之间有生物学功能上的一致性。基于氨基酸序列构建的系统发育树中, 独角雪冰鱼和伯氏肩孔南极鱼EPO聚类, 亲缘关系极近, 与鱼类的亲缘关系也在91.35%—70%之间, 符合进化规律。这说明 EPO在生物长期进化过程中保持着较强的保守性。
在蛋白性质的研究中, 蛋白质的稳定性的探究一直都与其半衰期长短相联系。研究结果表明, 半衰期长则蛋白质稳定性高, 反之半衰期短则表明蛋白质稳定性低(贾浩等, 2011)。在本研究中, 独角雪冰鱼和伯氏肩孔南极鱼的 EPO都具有较长的半衰期, 且属于稳定性蛋白, 将其与 EPO的稳定性分析结果结合来看, 能够确保独角雪冰鱼和伯氏肩孔南极鱼都属于稳定型亲水性蛋白, 这也就表明了 EPO蛋白在体内的生理功能发挥将会维持较稳定的状态。根据以往的研究表明, 成熟 EPO的某些螺旋区域能够影响EPO的生物活性, 其主要原理是能够增加蛋白质三级结构的灵活度(Boisselet al, 1993)。而伯氏肩孔南极鱼在二级结构预测中的两个小 β折叠区域能够在确保不会影响 EPO的功能结构特点的基础上, 潜在增加 EPO的生物活性。两者的三级结构分析结果表明南极冰鱼的 EPO同哺乳动物研究结果一致, 都是一个典型的4个反向平行的α螺旋折叠结构构成的球形分子(桂长云等, 1999; 葛全兴等, 2009)。虽然独角雪冰鱼和伯氏肩孔南极鱼的 EPO的理化性质分析结果以及二、三级结构预测都能够确保 EPO能够正常行使其功能, 但是两者之间由碱基突变造成的功能位点的改变对于该蛋白与受体的结合效率、转运、以及参与能量代谢过程的影响仍是一个有待深入研究的过程。该基因的成功克隆及分析为揭示南极冰鱼EPO基因的遗传特性以及生物在极端寒冷环境中的长期进化适应提供了理论依据。
刘虎岐, 刘应保, 宋云鹏等, 2010. 淡色库蚊氯菊酯抗性相关基因 PR-OP全长 cDNA的克隆及生物信息学分析. 西北农林科技大学学报(自然科学版), 38(9): 109—117
许强华, 吴智超, 陈良标, 2014. 南极鱼类多样性和适应性进化研究进展. 生物多样性, 22(1): 80—87
李盛杰, 杜晓华, 罗玉柱等, 2013. 天祝白牦牛NGB基因的克隆及生物信息学分析. 畜牧兽医学报, 44(3): 395—398
张 宝, 黄克勇, 郭劲松等, 2013. H7N9病毒的来源和重组模式. 南方医科大学学报, 33(7): 1017—1021
陈书霞, 王晓武, 房玉林, 2006. 单菌落PCR法直接快速鉴定重组克隆. 微生物学通报, 33(3): 52—56
罗 轶, 2008. 鸡FATP1基因cDNA的克隆、组织表达及其生物信息学分析. 雅安: 四川农业大学硕士学位论文,22—23
季舒涵, 昝林森, 王洪宝等, 2010. 秦川牛 A-FABP基因的生物信息学分析. 西北农林科技大学学报(自然科学版),38(6): 77—81
桂长云, 钱若兰, 1999. EPO, EPOR及其信号传递研究进展.细胞生物学杂志,21(4): 157—163
贾 浩, 张小白, 宋晓峰, 2011. 人类胞内蛋白半衰期与其亚细胞定位的相关性研究. 计算机与应用化学, 28(4):411—414
陶士珩, 2007. 生物信息学. 北京: 科学出版社, 177—179
葛全兴, 陈 垦, 龙友明等, 2009. EPO/ EPOR及其信号转导机制研究进展. 国际病理科学与临床杂志, 29(5):416—420
莎姆布鲁克 J, 拉塞尔 D W编著, 黄培堂译, 2002. 分子克隆实验指南. 北京: 科学出版社, 103—105
Bargelloni L, Ritchie P A, Patarnello Tet al, 1994. Molecular evolution at subzero temperatures: mitochondrial and nuclear phylogenies of fishes from Antarctica (suborder Notothenioidei), and the evolution of antifreeze glycopeptides. Molecular Biology and Evolution, 11(6):854—863
Boissel J P, Lee W R, Presnell S Ret al, 1993. Erythropoietin structure-function relationships. Mutant proteins that test a model of tertiary structure. Journal of Biological Chemistry,268(21): 15983—15993
Chou C F, Tohari S, Brenner Set al, 2004. Erythropoietin gene from a teleost fish,Fugu rubripes. Blood, 104(5):1498—1503
Chu C Y, Cheng C H, Chen G Det al, 2007. The zebrafish erythropoietin: functional identification and biochemical characterization. FEBS Letters, 581(22): 4265—4271
Eastman J T, Lannoo M J, 2004. Brain and sense organ anatomy and histology in hemoglobinless Antarctic icefishes(Perciformes: Notothenioidei: Channichthyidae). Journal of Morphology, 260(1): 117—140
Guruprasad K, Reddy B V B, Pandit M W, 1990. Correlation between stability of a protein and its dipeptide composition:a novel approach for predicting in vivo stability of a protein from its primary sequence. Protein Engineering, 4(2):155—161
Katakura F, Katzenback B A, Belosevic M, 2013. Molecular and functional characterization of erythropoietin receptor of the goldfish (Carassius auratusL.). Developmental &Comparative Immunology, 45(2): 191—1598
Kulkarni R P, Tohari S, Ho Aet al, 2010. Characterization of a hypoxia-response element in the Epo locus of the pufferfish,Takifugu rubripes. Marine Genomics, 3(2): 63—70
Kyte J, Doolittle R F, 1982. A simple method for displaying the hydropathic character of a protein. Journal of Molecular Biology, 157(1): 105—132
Marchler-Bauer A, Anderson J B, Cherukuri P Fet al, 2005. CDD:A conserved domain database for protein classification.Nucleic Acids Research, 33(suppl1): D192—D196
Nogawa-Kosaka N, Hirose T, Kosaka Net al, 2010. Structural and biological properties of erythropoietin inXenopus laevis.Experimental Hematology, 38(5): 363—372
Ostrowski D, Ehrenreich H, Heinrich R, 2011. Erythropoietin promotes survival and regeneration of insect neuronsin vivoandin vitro. Neuroscience, 188: 95—108
Paffett-Lugassy N, Hsia N, Fraenkel P Get al, 2007. Functional conservation of erythropoietin signaling in zebrafish. Blood,110(7): 2718—2726
Stachura D L, Reyes J R, Bartunek Pet al, 2009. Zebrafish kidney stromal cell lines support multilineage hematopoiesis.Blood, 114(2): 279—289
Stachura D L, Svoboda O, Lau R Pet al, 2011. Clonal analysis of hematopoietic progenitor cells in the zebrafish. Blood,118(5): 1274—1282
Wilkins M R, Gasteiger E, Bairoch Aet al, 1999. Protein identification and analysis tools in the ExPASy Server.Methods in Molecular Biology, 112: 531—552
