盐胁迫下甜菜M14品系BvM14-ATPaseF、BvM14-ATPaseH、BvM14-PsaD基因的表达分析
2015-03-17张咏雪戚诗诗马春泉陈思学李海英
张咏雪,戚诗诗,潘 钰,马春泉,于 冰,陈思学,李海英,*
(黑龙江大学 a. 生命科学学院; b. 黑龙江省普通高等学校分子生物学重点实验室;c. 农业微生物技术教育部工程研究中心,哈尔滨 150080)
盐胁迫下甜菜M14品系BvM14-ATPaseF、BvM14-ATPaseH、BvM14-PsaD基因的表达分析
张咏雪a,b,c,戚诗诗a,b,c,潘 钰a,b,c,马春泉a,b,c,于 冰a,b,c,陈思学a,b,c,李海英a,b,c,*
(黑龙江大学 a. 生命科学学院; b. 黑龙江省普通高等学校分子生物学重点实验室;c. 农业微生物技术教育部工程研究中心,哈尔滨 150080)
甜菜M14品系是由栽培甜菜和野生白花甜菜杂交后获得的带有野生白花甜菜第9号染色体的单体附加系,具有抗逆、无融合等优良特性。在前期工作中,对盐胁迫下叶片膜蛋白进行了定量蛋白质组学分析,与未处理对照相比,200、400 mM盐胁迫下共获得50个差异蛋白质点,选择其中3个蛋白质点为研究对象,与实验室前期相同盐处理下获得的转录组数据库相匹配,获得基因cDNA全长,将此3个基因命名为BvM14-ATPaseF、BvM14-ATPaseH和BvM14-PsaD;进行在线功能预测;设计引物,对盐胁迫(0、200、400 mM)处理下此3个基因进行实时荧光定量分析,了解在盐胁迫下基因转录与蛋白水平上表达量的关系。结果表明,与未处理对照相比,200、400 mM盐浓度处理下BvM14-ATPaseF基因分别上调3.70与4.47倍,BvM14-ATPaseH基因在盐胁迫处理下分别上调0.85与1.36倍,而BvM14-PsaD基因随着盐浓度的增加却下调0.97与2.54倍,表明在盐胁迫下不同基因具有的功能不同,而导致转录水平与蛋白水平的表达量并不完全相同。
甜菜M14品系;盐胁迫;膜蛋白基因;荧光定量PCR
0 引 言
甜菜M14品系是郭德栋教授[1]利用栽培甜菜(B.vulgarisL.)与野生白花甜菜(BetacorollifloraZoss.)进行种间杂交及回交获得的附加有野生白花甜菜第9号染色体的单体附加系。具有耐旱、耐盐、耐寒和无融合生殖等优良特性[2-3],是挖掘甜菜优质基因和蛋白质资源的优质材料。
实验室前期,杨乐[4]等对甜菜M14品系盐胁迫下蛋白质组进行了深入研究。潘钰[5]等利用iTRAQ技术对0、200、400 mM 盐处理下甜菜M14品系叶片膜蛋白进行了定量蛋白质组学分析,共得到50个差异表达蛋白质,对差异蛋白质进一步鉴定,发现与运输、光合作用、代谢、蛋白折叠和降解、蛋白合成、转录、胁迫和防御等相关。由于膜蛋白是生物膜的主要体现者,盐胁迫下膜蛋白的损伤会引起植物一系列的生理变化[6-7],进行膜蛋白的研究是对前期蛋白质组学的进一步补充。
本研究选择3个与膜蛋白功能密切相关的蛋白质点进行分析,并与实验室前期相同盐处理下获得的转录组数据库相匹配,获得基因cDNA全长;将3个基因分别命名为BvM14-ATPaseF、BvM14-ATPaseH和BvM14-PsaD;进行在线功能预测;设计引物,对盐胁迫(0、200、400 mM)处理下基因进行实时荧光定量分析,探讨3个基因在转录水平与蛋白水平表达量的关系。
1 材料与方法
1.1 植物材料
甜菜M14品系(公开号:CN1263695A),植物材料种植于黑龙江大学分子生物学实验室的试验田。
1.2 主要试剂
SYBR PremixEXTaqTMII(Perfect Real Time)试剂盒与RNA PCR Kit (AMV) Ver 3.0 试剂盒均购自TAKARA公司; PCR检测引物由上海生物工程有限公司合成。
1.3 基因功能预测
根据实验室前期获得的3个蛋白质点与相同盐处理下获得的转录组数据库相匹配,获得基因cDNA全长,并利用NCBI与UniProt网站在线进行基因功能预测。
1.4 基因设计引物
利用AlleleID 6.0软件根据实验室前期相同盐处理下获得的转录组数据库的基因cDNA序列设计荧光定量特异引物(s:正向引物;as:反向引物):
Ug16039s:5′-GAATCGTATCTGTGGCTA-CTG-3′
Ug16039as:5′-CGGACTTATTGCTATTG-ATGC-3′
Ug16567s:5′-GTGCTGGTGCTGCTTGTC-3′
Ug16567as:5′-GCTCCGTCTTCCCTCTCC-3′
Cl2797s:5′-AATATAAGAGCCTGACTGA-TTGTG-3′
Cl2797as:5′-CTGAAGCAAGAGCGGTTA-TTAC-3′
根据吴川等[8]设计甜菜M14品系18S rRNA管家基因荧光定量特异引物:
18S-s:5′-TGACGGAGAATTAGGGTTCG-3′
18S-as:5′-CCCCAATGGATCCTCGTTA-3′
1.5 盐胁迫下甜菜M14品系叶片总RNA的提取与浓度、纯度测定
采用TRIzol一步法分别提取0、200、400 mM 盐处理下甜菜M14品系叶片总RNA[9]。利用DNA计算器测定RNA样品在260 nm和280 nm下的吸收值,测定RNA溶液浓度和纯度。利用1%琼脂糖凝胶检测28S、18S条带的清晰度和相对含量,对总RNA的完整性进行评估。
1.6 RNA反转录为cDNA
根据TaKaRa公司RNA PCR Kit试剂盒,分别合成0、200、400 mM 盐处理下甜菜M14品系叶片cDNA 产物。按下列条件进行反转录反应:30 ℃,10 min;42 ℃,30 min;99 ℃,5 min;5 ℃,5 min;1 cycle。
1.7 实时荧光定量PCR反应
荧光定量PCR采用TaKaRa公司SYBR PremixEXTaqTMII(Perfect Real Time)试剂盒进行试验,在Bio-Rad公司的MyiQTM2荧光定量PCR仪反应,以甜菜M14品系的18S rRNA管家基因作为内参,PCR反应体系(35.0 μL)包括:SYBR PremixEXTaqTMII(2×)17.5 μL,cDNA模板3.5 μL,正向、反向引物各1.4 μL,ddH2O补至35.0 μL。反应分为两部分:第一部分扩增反应,95 ℃ 10 min;95 ℃ 15 s;59 ℃ 1 min;72 ℃ 30 s循环40次;第二部分建立溶解曲线,当扩增反应结束后将温度升至95 ℃ 15 s;59 ℃ 1 min;再升至95 ℃反应全部结束。
1.8 数据分析
本研究采用比较Ct值法,即-ΔΔCT值法,对盐胁迫下甜菜M14品系的3个基因进行相对定量分析。计算公式为ΔΔCt=(Ct.差异基因-Ct.18s)Timex-(Ct.差异基因-Ct.18s)Time0。本研究中建立的反应体系,每个反应均设有3次技术重复和3次生物学重复。
2 结果与分析
2.1 甜菜M14品系基因生物信息学分析
实验室前期利用iTRAQ技术对0、200、400 mM 盐处理下甜菜M14品系叶片膜蛋白进行了定量蛋白质组学分析,3个蛋白质点的信息见表1。通过3个蛋白质点的Accession号与相同盐处理下获得的转录组数据库进行匹配,结果见表2。查询匹配获得基因cDNA全长,利用NCBI功能预测,将蛋白质点ATP synthase CF1 alpha subunit命名为BvM14-ATPaseF(gi|126022791, Ug16039_R)基因,其完整的ORF为1 524 bp,共编码507个氨基酸,利用UniProt网站预测该基因编码的蛋白定位于叶绿体类囊体膜上,主要是一类CF1型ATP合成酶,是生物体中能量转化的关键酶,主要参与氧化磷酸化和光合磷酸化过程,具有ATP合成或水解的活性[10-13];同时,通过NCBI功能预测将蛋白质点Chloroplastic photosystem I reaction center subunit II命名为BvM14-PsaD(gi|19855891, Ug16567_R),其完整ORF为861 bp,编码286个氨基酸,利用UniProt网站预测该基因编码的蛋白也定位于叶绿体类囊体膜上,主要是一类膜外周蛋白,为PSI反应中心亚基II类酶,其功能是在光合电子传递链中催化电子从质体色素经过一系列的电子传递到铁硫蛋白的过程,并稳固光系统I(PSI)光合膜上的色素蛋白复合物[14-15]。将蛋白质点ATPase 11命名为BvM14-ATPaseH(gi|12230459, Cl2797),基因完整ORF为2 259 bp,共编码752个氨基酸,利用UniProt网站预测该蛋白定位细胞膜上,该酶是高度保守的多亚基内膜质子泵,以质子驱动力的形式推动各种离子和小分子进行跨膜运输[16],由此产生细胞外部磷酸化和内部的碱化来调节增长反应[17],结果见图1。

表1 盐胁迫下甜菜M14品系iTRAQ质谱鉴定的蛋白质序列信息
表2 盐胁迫下甜菜M14品系转录组数据库基因信息

图1 BvM14-ATPaseF、BvM14-PsaD、BvM14-ATPaseH氨基酸序列保守结构域分析Fig.1 Analysis of amino acid conservative regions of three genes
2.2 甜菜M14品系叶片总RNA的提取鉴定
盐处理(0、200、400 mM)下甜菜M14品系叶片总RNA浓度分别为1.055 μg/μL、2.421 μg/μL、1.516 μg/μL,A260/A280比值分别为1.896、1.904、1.890,结果表明提取的甜菜M14品系叶片总RNA纯度较高。用1%琼脂糖凝胶分离总RNA,结果见图2,28S rRNA条带亮度约为18S rRNA条带的2倍,说明所提取的总RNA完整性较好。

图2 盐胁迫下甜菜M14品系叶片总RNA检测结果Fig.2 Total RNA of leaves in M14 under salt stress
2.3 荧光定量PCR引物特异性检测
荧光染料可以与所有的DNA双链结合(包括引物二聚体)产生荧光,影响定量的准确程度。由此,通过荧光定量PCR产物的溶解曲线分析,可以判断引物是否具有特异性。内参基因18S rRNA与BvM14-ATPaseF、BvM14-PsaD和BvM14-ATPaseH基因在甜菜M14品系不同浓度盐胁迫下叶片实时荧光定量PCR产物的溶解曲线见图3。
图3 基因荧光定量PCR溶解曲线Fig.3 Real time fluorescent quantization PCR melting curve of genes
由图3对内参基因18S rRNA和甜菜M14品系BvM14-ATPaseF、BvM14-PsaD和BvM14-ATPaseH基因荧光定量PCR产物的溶解曲线可见,随温度升高,其荧光染料会与DNA双链分离,未出现杂峰而且峰型单一,可表明试验中未有其它污染发生,无假阳性存在,可用于差异基因的扩增检测。
2.4 基因的表达定量分析
对甜菜M14品系叶片BvM14-ATPaseF、BvM14-PsaD和BvM14-ATPaseH基因进行相对定量分析,该研究采用Ct值法,即-ΔΔCT值法,以未盐胁迫处理的Ct值为对照计算得出数值,结果见图4。
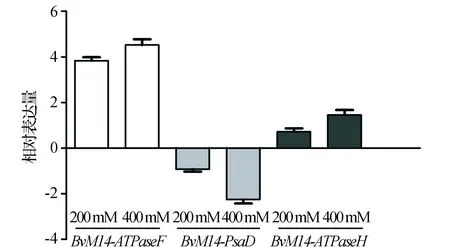
图4 甜菜M14品系中BvM14-ATPaseF、BvM14-PsaD、BvM14-ATPaseH基因的组织特异性表达Fig.4 Detection the expression level of BvM14-ATPaseF、BvM14-PsaD、BvM14-ATPaseH genes in M14 line
根据实时荧光定量PCR结果显示,与未处理对照组相比,200、400 mM盐胁迫下BvM14-ATPaseF基因分别上调3.70与4.47倍,BvM14-ATPaseH基因分别上调0.85与1.36倍,BvM14-PsaD基因分别下调0.97与2.54倍。在400 mM盐浓度处理下BvM14-ATPaseF蛋白上调0.54倍,BvM14-ATPaseH蛋白上调1.39倍,二者转录水平与蛋白水平表达含量相一致;在200 mM盐胁迫处理下,BvM14-PsaD蛋白上调5.15倍,其转录水平与蛋白质水平含量不同,表明在盐胁迫下不同基因具有的功能不同,而导致转录水平与蛋白水平的表达量并不完全相同。
3 结 论
数据显示,在盐处理下转录水平与蛋白水平的表达含量并不完全一致,转录水平盐胁迫下BvM14-PsaD基因下调表达,但在200 mM盐胁迫下,该蛋白却上调5.15倍,可能是因为盐胁迫会导致植物气孔不均匀的关闭,进而可能影响色素与光的结合能力减弱,从而影响其PsaD蛋白参与光合作用[18]。BvM14-PsaD基因下调表达可能由于光合作用的降低导致含量降低。本研究中,盐胁迫处理下BvM14-ATPaseF与BvM14-ATPaseH基因在甜菜M14品系叶片中显著表达,且转录水平与蛋白水平含量一致。虽然目前关于植物在盐胁迫对呼吸作用的影响报道并不一致,但在逆境条件下,植物可能需要提供额外的能量来维持自身的生长发育。盐胁迫后,ATPase酶参与呼吸作用的加强用来满足离子运输以及局部盐分的变化[19-20]。由此可见ATP合成酶对维持细胞内渗透平衡有重要作用。
甜菜M14品系BvM14-ATPaseF、BvM14-PsaD和BvM14-ATPaseH基因与抵御盐胁迫是否存在直接关系,还需要进一步进行基因功能验证。这一结果也对探讨甜菜M14品系与盐胁迫的关系奠定了基础。
[1]郭德栋, 康传红, 刘丽萍. 异源三倍体甜菜( VVC) 无融合生殖的研究[J]. 中国农业科学, 1999, 32(40):1-5.
[2]于 冰, 李海英, 郭德栋, 等. 甜菜无融合生殖系花期差异表达基因cDNA文库的构建[J]. 高技术通讯, 2006, 16( 9):954-957.
[3]郭德栋, 刘丽萍, 康传红, 等. 甜菜无融合生殖单体附加系的繁殖传递特性[J]. 黑龙江大学自然科学学报, 2001, 18(3):104-107.
[4]Yang L, Zhang Y, Zhu N, et al. Proteomic analysis of salt tolerance in sugar beet monosomic addition line M14[J]. J Proteome Res.,2013, 12(11):4 931-4 950.
[5]Li H, Pan Y, Zhang Y. Salt stress response of membrane proteome of Sugar beet monosomic addition line M14[J]. J Proteomics, 2015,127(8):18-23.
[6]Munns R, Tester M. Mechanisms of salinity tolerance[J]. Annual Review of Plant Biology, 2008, 59:651-681.
[7]Matsui H, Takahashi A, Hirochika H. Rice immune regulator, OsPti1a, is specifically phosphorylated at the plasma membrane[J]. Plant Signal Behav., 2015,10(3):e991569.
[8]Wu C, Ma C, Pan Y. Sugar beet M14 glyoxalase I gene can enhance plant tolerance to abiotic stresses[J]. Journal of Plant Research, 2013, 126(3):415-425.
[9]赵晨曦,王 淋,谷 丹,等.利用SSH技术分析盐胁迫下甜菜M14品系的差异基因[J]. 黑龙江大学自然科学学报,2014, 31(2):238-243.
[10]Futal M, Sun W, Wada Y. Proton translocating ATPase: including unique enzymes coupling catalysis and proton translocation through mechanical rotation[C]// Futai M, Wada Y, Kaplan (Eds.) J. Handbook of ATPases: Biochemistry, Cell Biology, Pathology, Wiley-VCH Verlag, Weinheim Germany, 2004:237-260.
[11]Angevine C M, Dmitriev O Y. Mechanics of coupling proton movements to c-ring rotation in ATP synthase[J]. FEBS Lett, 2003, 555(1):29-34.
[12]Maeda M. ATP synthases: bioinformaticts based insights into how their electrochemically driven motor comprised of subunits a and c might serve as a drug target[J]. J Bioenerg Biomembr, 2008, 40(2):117-121.
[13]Kuhnert W L, Hahn K. Genetics of acid adaptation in oral streptococci[J]. Crit Rev Oral Biol Med, 2001, 12(4):301-314.
[14]Ihnatowica A, Pesaresi P, Varotto C, et al. Mutants for photosystem I subunit D of Arabidopsis thaliana: effects on photosynthesis, photosystem I stability and expression of nuclear genes for chloroplast functions[J]. Plant J. 2004,37(6):839-852.
[15]Haldrup A, Lunde C, Scheller H V. Arabidopsis thaliana plants lacking the PSI-D subunit of photosystem I suffer severe photoinhibition, have unstable photosystem I complexes, and altered redox homeostasis in the chloroplast stroma[J]. J Biol Chem., 2003, 278(35):33 276-33 283.
[16]Reiland S, Messerli G, Baerenfaller K. Large-scale Arabidopsis phosphoproteome profiling reveals novel chloroplast kinase substrates and phosphorylation networks [J]. Plant Physiol., 2009, 150(2):889-903.
[17]Krebs M, Beyhl D, Gorlich E. ArabidopsisV-ATPase activity at the tonoplast is required forefficient nutrient storage but not for sodium accumulation[J]. Proc Natl Acad Sci USA, 2010, 107(7):3 251-3 256.
[18]Hussain T, Hazara M, Sultan Z, et al. Recent advances in salt stress biology a review[J]. Biotechnology and Molecular Biology Reviews, 2008,3:8-13.
[19]Maxson M, Grinstein S. The vacuolar-type H+-ATPase at a glance-more than a proton pump[J]. J Cell Sci., 2014, 127(23):4 987-4 993.
[20]赵晨曦,南景东,马春泉,等.甜菜M14品系硫氧还蛋白过氧化物酶(BvM14-Tpx)基因左原核及真核细胞中的抗氧化及抗盐能力分析[J].黑龙江大学工程学报,2015,6(3):62-67.
Specific expression analysis of BvM14-ATPaseF、BvM14-ATPaseH and BvM14-PsaD under salt stress of sugar beet M14 lines
ZHANG Yong-Xuea,b,c,QI Shi-Shia,b,c,PAN Yua,b,c,MA Chun-Quana,b,c,YU Binga,b,c,CHEN Si-Xuea,b,c,LI Hai-Yinga,b,c,*
(Heilongjiang University a. College of Life Sciences; b.Key Laboratory of Molecular Biology, College of Heilongjiang Province; c. Engineering Research Center of Agricultural Microbiology Technology,Ministry of Education, Harbin 150500, China)
Sugar beet M14 line is a unique germplasm that contains genetic materials from Beta vulgaris L and Beta corolliflora Zoss and exhibits tolerance to salt stress. In previous work, it was focusing on analyzing proteomic protein quantitative of the leaf membrane. Under salt stress, 50 proteins exhibited differential protein level changes. We regard 3 of 50 as research object to get full length of genes with prediction function online, and design primers by using the transcriptome data. Using Real-time PCR to analysis the expression of the genes which under the treatment of salt stress (0, 200, 400 mM). The result shows that, theBvM14-ATPaseFgene is up-regulated 3.7 and 4.47 respectively compared with control. And the expression ofBvM14-ATPaseHgene is also up-regulated 0.85 and 1.36. However, different results appear inBvM14-PsaDgene, 0.97 down-regulations in 200 mM and 2.54 in 400 mM salt stress processing. The function of gene is not consistent so that the level of transcription is not entirely consistent with the protein level. Therefore, M14 has been used as an abundant genetic resource for isolating valuable genes in wild species.
sugar beet M14; salt stress; membrane genes; real-time PCR
10.13524/j.2095-008x.2015.04.064
2015-07-21
国家自然科学基金资助项目(31471552、31401441);黑龙江省自然科学基金资助项目(C201202);黑龙江省高校创新团队建设计划项目(2014TD004);黑龙江大学创新团队计划项目(hdtd2010-05);黑龙江省研究生创新科研项目(YJSCX2015-030HLJU)
张咏雪(1990-),女,黑龙江双鸭山人,硕士研究生,研究方向:植物分子生物学,E-mail:xuezylemon@foxmail.com;*通讯作者:李海英(1968-),女,黑龙江肇东人,教授,博士,博士研究生导师,研究方向:植物分子生物学,E-mail:lvzh3000@sina.com。
O436
A
2095-008X(2015)04-0049-06
