Pseudomonas syringae褐藻胶裂解酶基因的克隆及信息学分析
2015-03-11吴丽云肖安风蔡慧农朱艳冰
吴丽云,刘 韩,倪 辉,2,3,4 ,肖安风,2,3,4,蔡慧农,2,3,4,朱艳冰,2,3,4
(1.集美大学食品与生物工程学院,福建 厦门 361021; 2.福建省食品微生物与酶工程重点实验室,
福建 厦门 361021;3.厦门市食品与生物工程技术研究中心,福建 厦门 361021;
4.厦门市南方海洋研究中心经济海藻资源化利用与深加工重点实验室,福建 厦门 361021)
Pseudomonassyringae褐藻胶裂解酶基因的克隆及信息学分析
吴丽云1,刘韩1,倪辉1,2,3,4,肖安风1,2,3,4,蔡慧农1,2,3,4,朱艳冰1,2,3,4
(1.集美大学食品与生物工程学院,福建 厦门 361021; 2.福建省食品微生物与酶工程重点实验室,
福建 厦门 361021;3.厦门市食品与生物工程技术研究中心,福建 厦门 361021;
4.厦门市南方海洋研究中心经济海藻资源化利用与深加工重点实验室,福建 厦门 361021)
[摘要]以Pseudomonas syringae的基因组为模板,使用褐藻胶裂解酶引物进行PCR扩增,将目的基因克隆至pMD18-T载体后进行测序.结果显示,克隆基因的大小为1137 bp,预测编码含有378个氨基酸残基的蛋白质.对该蛋白质进一步进行生物信息学分析,结果表明,该蛋白质序列与其他菌株来源的褐藻胶裂解酶具有高的相似性,预测本研究克隆的基因编码褐藻胶裂解酶.该褐藻胶裂解酶的理论分子质量为42.5 ku,理论等电点为8.15.采用同源建模法建立P.syringae褐藻胶裂解酶的三维结构,富含螺旋结构.
[关键词]假单胞菌;褐藻胶裂解酶;克隆;生物信息学
0引言
褐藻胶为主要来源于褐藻类植物细胞壁的结构多糖,它是一种阴离子酸性直链多糖,由β-D-甘露糖醛酸(M)和α-L-古罗糖醛酸(G)通过共价键随机结合形成的线性无支链的高分子聚合物[1].褐藻胶来源丰富,以其凝胶性、增稠性、稳定性和螯合金属离子等特点被广泛应用于食品、医疗、印染和化工等领域[2-4].褐藻胶的降解产物褐藻胶寡糖因其具有特殊的化学结构而表现出多种生物活性,例如抗氧化、抗菌、抗炎、抗肿瘤、免疫调节、促进植物生长等[5-9]. 研究表明,利用褐藻胶裂解酶催化裂解褐藻胶糖苷键,从而在非还原端形成具有双键的不饱和糖醛酸寡糖,这种不饱和双键对褐藻胶寡糖所具有的生理活性功能有重要的影响[10].根据褐藻胶裂解酶的底物专一性,褐藻胶裂解酶分为多聚β-D-1,4-甘露糖醛酸裂解酶(EC 4.2.2.3)和多聚α-L-1,4-古罗糖醛酸裂解酶(EC 4.2.2.11).已经报道的褐藻胶裂解酶的来源主要包括海洋藻类、海洋软体动物、棘皮动物和微生物(包括海洋细菌、陆生真菌和极少数的病毒),其中细菌类是褐藻胶裂解酶研究最为广泛的来源[11-14].利用基因工程技术将褐藻胶裂解酶基因在工业化生产的宿主细胞中高效表达,以获得高产量、高纯度、高活性、高稳定性的褐藻胶裂解酶制剂,具有高的产业潜力. 本文进行Pseudomonassyringae褐藻胶裂解酶基因的克隆及其生物信息学分析,为该菌株褐藻胶裂解酶的酶学性质、酶的结构与功能研究奠定基础.
1材料与方法
1.1 材料
P.syringae购自德国微生物菌种保藏中心(No.21482),E.coliDH5α菌株由本实验室保存.pMD18-T载体、Taq DNA聚合酶、dNTPs和DNA标准均为TaKaRa公司产品,T4DNA连接酶为Fermentas公司产品,细菌基因组DNA提取试剂盒购自北京百泰克生物技术有限公司,柱式质粒DNA提取试剂盒和柱式DNA胶回收试剂盒均为生工生物工程(上海)股份有限公司产品,寡核苷酸序列的合成及核酸序列的测定均委托生工生物工程(上海)股份有限公司完成,其余试剂均为分析纯产品.
1.2 实验方法
1.2.1褐藻胶裂解酶的活力测定
将P.syringae接种于培养基(0.25% D-葡萄糖,0.3%大豆蛋白胨,1.7%干酪素,0.5% NaCl,0.25% K2HPO4,均为质量分数),25 ℃培养36 h后,取20 mL培养液于4 ℃、10000 r/min离心10 min,收集上清液,即为胞外液.用2 mL 50 mmol/L NaH2PO4-Na2HPO4缓冲液(pH=7.5)重悬菌体,在冰浴条件下超声破菌后,4 ℃、10000 r/min离心10 min,获得上清液,即为胞内液.取0.25 mL含0.5%(质量分数)褐藻胶的50 mmol/L NaH2PO4-Na2HPO4缓冲液(pH=7.5),加入0.25 mL粗酶液,30 ℃反应30 min后,沸水终止反应.加入0.5 mL DNS试剂,混匀后于540 nm波长下测定反应液的吸光度值.通过制作葡萄糖标准曲线确定还原糖含量,根据还原糖的产生计算褐藻胶裂解酶的活力.褐藻胶裂解酶活力定义为:在上述条件下,每分钟水解底物产生1 μg还原糖(以葡萄糖计)所需的酶量为1个酶活力单位(U).
1.2.2褐藻胶裂解酶基因的克隆
根据GenBank中P.syringae的褐藻胶裂解酶基因序列,合成以下引物,P1:5′-ATGCAGACTCCTAAACTGAT-3′,P2:5′-TCACGAACCGTCGTTATCGC-3′.利用细菌基因组DNA提取试剂盒提取P.syringae的基因组DNA,以此为模板进行PCR扩增,反应条件为:95 ℃ 5 min预变性;94 ℃ 45 s,50 ℃ 45 s,72 ℃,90 s,30个循环;72 ℃延伸10 min.PCR产物经1.2%(质量分数)琼脂糖凝胶电泳检测后,凝胶回收目的基因片段.将回收产物与T载体进行连接,转化E.coliDH5α感受态细胞.细胞涂布在含有100 μg/mL氨苄青霉素的LB固体培养平板上(平板上预先涂布10 μL 0.5 mol/L IPTG和50 μL 20 mg/mL X-gal).从平板挑选白色单菌落进行菌落PCR鉴定重组子,并经测序鉴定重组质粒中的插入序列.
1.2.3基因及其编码产物的生物学信息分析
利用NCBI的BLAST程序在GenBank数据库中进行蛋白质序列的同源性搜索,利用MEGA 6.0[15]的邻接法(Neighbor-Joining,NJ)构建系统发育树,利用ExPASy的Protparam工具进行蛋白质的理化性质预测.蛋白质二级结构预测采用SOPMA程序[16]进行,蛋白质的结构域及信号肽分析利用SMART[17]进行.利用SWISS-MODEL[18]进行蛋白质结构的三维模建,采用VMD软件显示蛋白质的空间结构.
2结果
2.1 褐藻胶裂解酶的活力
采用DNS法测定菌株P.syringae所产的细胞内外褐藻胶裂解酶的活性,结果显示,胞内粗酶液的活力达7 U/mL,未检测到胞外样品的酶活力.由此可见,菌株P.syringae以胞内形式产褐藻胶裂解酶.
2.2 褐藻胶裂解酶基因的克隆
以P.syringae的基因组DNA(见图1a)为模板,利用褐藻胶裂解酶引物进行PCR扩增.琼脂糖凝胶电泳显示,扩增产物为大约1100 bp的DNA片段(见图1b).将该基因克隆入T载体后进行测序分析,结果显示目的基因的大小为1137 bp.
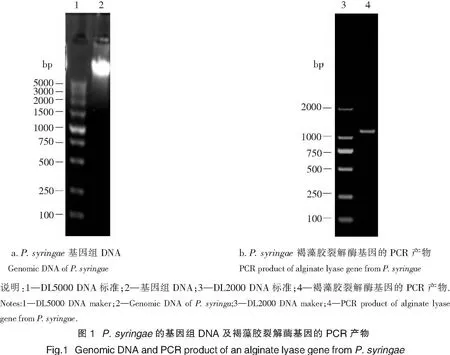
2.3 褐藻胶裂解酶基因及其编码产物的序列分析
P.syringae褐藻胶裂解酶基因(1137 bp)预测编码378个氨基酸.该蛋白质的前28个氨基酸残基预测为信号肽,预测的切割位点位于A28和A29之间.将该蛋白质序列在Genbank Reference proteins数据库中进行同源性搜索,结果显示,目的基因编码的蛋白质序列与来自P.syringae(WP_003365409)、P.amygdale(WP_005752927)、P.savastanoi(WP_002552250)、P.syringae(WP_016567310)、P.syringae(WP_003376044)、P.viridiflava(WP_025994636)和P.syringae(WP_024686395)的多聚β-D-甘露糖醛酸裂解酶或褐藻胶裂解酶的序列分别具有100%、99%、99%、99%、96%、94%和94%的相似性,所以预测该基因编码褐藻胶裂解酶.将本研究的P.syringae褐藻胶裂解酶序列与其他菌株来源的褐藻胶裂解酶序列进行比对,结果如图2所示.N203、H204、R251和Y258构成可能的P.syringae褐藻胶裂解酶催化活性位点.其中H204位于NNHSYW保守结构中.将本研究的P.syringae褐藻胶裂解酶序列与相似性较高的其他菌株来源的酶序列进行系统发育分析,结果(见图3)显示,本研究的褐藻胶裂解酶与来自P.syringae的多聚β-D-甘露糖醛酸裂解酶(WP_003365409)聚在一个分支.


2.4 褐藻胶裂解酶基因编码蛋白质的理化性质
利用ExPASy的ProtParam工具分析目的基因所编码蛋白质的序列,结果如下:氨基酸个数为378,理论分子质量大小为42.5 ku;理论等电点为8.15,负电荷残基总数为50(D+E),正电荷残基总数为52(R+K);分子式为C1883H2942N522O567S18,原子总数为5932;预测在大肠杆菌细胞内半衰期大于10 h,不稳定指数为31.78,该蛋白质分类为稳定蛋白;脂溶指数为68.76,亲水性平均指数(Grand average of hydropathicity,GRAVY)为-0.593.
2.5 褐藻胶裂解酶的二级结构和结构域
预测P.syringae褐藻胶裂解酶的二级结构,α-螺旋、β-折叠、β-转角和不规则卷曲所占比例分别为51.59%、7.94%、7.14%和33.33%.该蛋白质包含褐藻胶裂解酶结构域,位于64~305位氨基酸残基.
2.6 褐藻胶裂解酶的三级结构
将P.syringae褐藻胶裂解酶进行三维模建,模板来自Sphingomonassp.A1的褐藻胶裂解酶(PDB登录号为1QAZ).模建的残基范围为目的序列中的47~363位氨基酸残基,在此范围内,目的序列与模板序列的相似性为28%.P.syringae褐藻胶裂解酶富含螺旋结构,由螺旋构成的栅状结构中有个类似隧道的深裂隙(见图4a).通过与模板序列进行比对,N203、H204、R251和Y258组成可能的P.syringae褐藻胶裂解酶催化活性位点[19](见图4b).一些保守的具有芳香基侧链的残基(W148,W207,Y261和Y258)位于裂隙中,两个保守的精氨酸残基(R251和R354)存在于裂隙入口的两侧(见图4a).

3讨论
本研究中,利用Genbank数据库中已有的P.syringae褐藻胶裂解酶基因序列设计引物,从目的菌株的基因组中成功扩增了1137bp的基因片段.通过NCBI的蛋白质BLAST分析,发现该基因编码的蛋白质与Pseudomonas属中其他菌株来源的褐藻胶裂解酶具有很高的相似性.所以,本研究克隆的基因预测编码褐藻胶裂解酶.
褐藻胶裂解酶是多糖裂解酶家族(PLs,EC4.2.2.X)的成员,根据酶的初级结构可将PLs分为22个家族,褐藻胶裂解酶属于7个家族,分别为PL-5,PL-6,PL-7,PL-14,PL-15,PL-17和PL-18,大多数细菌来源的内切褐藻胶裂解酶属于PL-5和PL-7,而前者多具有降解多聚β-D-甘露糖醛酸底物的特异性,后者多具有降解多聚α-L-古罗糖醛酸底物的特异性[20].研究表明,PL-5和PL-7家族之间的褐藻胶裂解酶序列相似性比较低,然而对催化活性起重要作用的氨基酸,如精氨酸、天冬酰胺(或谷氨酰胺)、组氨酸和酪氨酸都在活性位点的功能区域[21].本研究中,以Sphingomonassp.A1的褐藻胶裂解酶为模板,将本研究的P.syringae褐藻胶裂解酶进行同源模建,它们的三维结构呈现相似的空间结构.P.syringae褐藻胶裂解酶主要结构为由螺旋构成的栅状结构,类似的结构常见于葡萄糖淀粉酶和纤维素酶[21].这两个酶的活性中心包括Asn、His、Arg和Tyr,组氨酸位于NNHSYW保守结构中.与Sphingomonassp.A1褐藻胶裂解酶[19]类似,本研究的P.syringae褐藻胶裂解酶结构中也包含一个很深的隧道缝隙,预测该结构有利于褐藻胶底物分子的渗入,并与酶催化位点相互作用.
P.syringae褐藻胶裂解酶的克隆及生物信息学分析为该酶的进一步研究打下良好的基础.在未来的研究中,可以进行P.syringae褐藻胶裂解酶的表达及酶学性质研究,或进一步利用定向进化技术提高该褐藻胶裂解酶的催化活性或热稳定性,为该褐藻胶裂解酶的工业化生产和应用打下良好的基础.
[参考文献]
[1]VERA J,CASTRO J,GONZALEZ A,et al.Seaweed polysaccharides and derived oligosaccharides stimulate defense responses and protection against pathogens in plants[J].Mar Drugs,2011,9(12):2514-2525.
[2]LEE K Y,MOONEY D J.Alginate:properties and biomedical applications[J].Prog Polym Sci,2012,37(1):106-126.
[3]PAWAR S N,EDGAR K J.Alginate derivatization:a review of chemistry,properties and applications[J].Biomaterials,2012,33(11):3279-3305.
[4]TSENG C K.Algal biotechnology industries and research activities in China[J].J Appl Phycol,2001,13(4):375-380.
[5]孙丽萍,薛长湖,许家超,等.褐藻胶寡糖体外清除自由基活性的研究[J].中国海洋大学学报,2005,35(5):811-814.
[6]LASKY L A.Selection-carbohydrate interactions and the initiation of the inflammatory response[J].Annu Rev Biochem,1995,64:113-139.
[7]HU X K,JIANG X L,WANG H M,et al.Antitumour activities of alginate-derived oligosaccharides and their substitution derivatives[J].Eur J Phycol,2004,39:67-71.
[8]OTTERLEI M,OSTGAARD K,SKJAK BRAEK G,et al.Induction of cytokine production from human monocytes stimulated with alginate[J].J Immunother,1991,10(4):286-291.
[9]YOKOSE T,NISHIKAWA T,YAMAMOTO Y,et al.Growth-promoting effect of alginate oligosaccharIdes on a unicellular marine mieroalga,Nannochloropsisoculata[J].Biosci Biotechnol Biochem,2009,73(2):450- 453.
[10]WONG T Y,PRESTON L A,SCHILLER N L.Alginate lyase:review of major sources and enzyme characteristics,structure-function analysis,biological roles,and applications[J].Annu Rev of Microbiol,2000,54:289-340.
[11]LIN Y T,HASSID W Z.Pathway of alginic acid synthesis in the marine brown alga,Fucus gardneri Silva[J].J Biol Chem,1966,241(22):5284-5297.
[12]WANG L,RAHMAN M M,INOUE A,et al.Heat-stability and primary structure of the major alginate lyase isozyme LbAly35 fromLittorinabrevicula[J].Fish Sci,2012,78(4):889-896.
[13]WANG Y,GUO E W,YU W G,et al.Purification and characterization of a new alginate lyase from a marine bacteriumVibriosp.[J].Biotechnol Lett,2013,35(5):703-708.
[14]CAO L X,XIE L J,XUE X L,et al.Purification and characterization of alginate lyase fromStreptomycesspecies strain A5 isolated from banana rhizosphere[J].J Agric Food Chem,2007,55(13):5113-5117.
[15]TAMURA K,STECHER G,PETERSON D,et al.MEGA6:molecular evolutionary genetics analysis version 6.0[J].Mol Biol Evol,2013,30(12):2725-2729.
[16]GEOURJON C,DELEAGE G.SOPMA:significant improvements in protein secondary structure prediction by consensus prediction from multiple alignments[J].Comput Appl Biosci,1995,11(6):681-684.
[17]LETUNIC I,DOERKS T,BORK P.SMART 7:recent updates to the protein domain annotation resource[J].Nucleic Acids Res,2012,40:302-305.
[18]AMOLD K,BORDOLI L,KOPP J,et al.The SWISS-MODEL workspace:a web-based environment for protein structure homology modelling[J].Bioinformatics,2006,22(2):195-201.
[19]YOON H J,MIKAMI B,HASHIMOTO W,et al.Crystal structure of alginate lyase A1-III fromSphingomonasspecies A1 at 1.78 A resolution[J].J Mol Biol,1999,290(2):505-514.
[20]MIYAKE O,OCHIAI A,HASHIMOTO W,et al.Origin and diversity of alginate lyases of families PL-5 and-7 inSphingomonassp.strain A1[J].J Bacteriol,2004,186(9):2891-2896.
[21]KIM H S,LEE C,LEE E Y.Alginate lyase:structure,property,and application[J].Biotechnol Bioproc E,2011,16:843-851.
(责任编辑马建华英文审校曹敏杰)
Cloning and Bioinformatics Analysis of an Alginate Lyase GenefromPseudomonassyringae
WU Li-yun1,LIU Han1,NI Hui1,2,3,4,XIAO An-feng1,2,3,4,CAI Hui-nong1,2,3,4,ZHU Yan-bing1,2,3,4
(1.College of Food and Biological Engineering,Jimei University,Xiamen 361021,China;
2.Key Laboratory of Food Microbiology and Enzyme Engineering of Fujian Province,Xiamen 361021,China;
3.Research Center of Food Biotechnology of Xiamen City,Xiamen 361021,China;4.Key Laboratory of Systemic Utilization
and In-depth Processing of Economic Seaweed,Xiamen Southern Ocean Technology Center of China,Xiamen 361021,China)
Abstract:The genomic DNA of Pseudomonas syringae was used as the template for amplification of alginate lyase gene by PCR using a pair of specific primers.The target gene was cloned into pMD18-T vector and then sequenced.The results showed that the cloned gene was 1137 bp,encoding 378 amino acid residues.This protein was further analyzed by bioinformatics.The results showed that the target protein sequence shared high identities with the alginate lyase sequences from other bacterial strains.So the cloned gene was predicted to be an alginate lyase.The theoretical molecular weight and pI of the alginate lyase were 42.5 ku and 8.15,respectively.The three-dimensional structure of P.syringae the alginate lyase was constructed by homology modeling and presented α-helix rich structure.
Key words:Pseudomonas sp.;alginate lyase;cloning;bioinformatics
[文献标志码]A
[中图分类号]Q 939.97
[文章编号]1007-7405(2015)03-0179-07
[作者简介]吴丽云(1989―),女,硕士生,从事微生物学方向研究.通信作者:朱艳冰(1976―),女,副教授,从事海洋分子生物学方向研究,E-mail:yanbingzhu@163.com.
[基金项目]国家自然科学基金资助项目(31401632);厦门南方海洋研究中心项目(13GZP004NF10);福建省优良品种同生物与酶工程重点实验室开放基金项目(M20130905)
[收稿日期]2014-10-08[修回日期] 2014-11-15
