乌金猪FTO基因的克隆和表达分析
2014-10-27黄英杨明华毕保良潘洪彬俞政全高士争李永能赵素梅
黄英,杨明华,毕保良,潘洪彬,俞政全,高士争,李永能,赵素梅
云南农业大学 a.云南省动物营养与饲料重点实验室;b.科技管理处;c.招生就业处;云南 昆明 650201
随着经济的快速发展和生活水平的不断提高,肥胖已成为现代社会最常见的营养障碍性疾病之一,肥胖患病率呈现明显上升的趋势。肥胖可导致心血管疾病、高血压、2型糖尿病和某些癌症,从而增加死亡风险[1]。随着对肥胖发生在基因水平上的深入研究,与肥胖相关的肥胖易感基因——脂肪和肥胖相关基因(fat mass and obesity associated gene,FTO基因)受到人们的关注。猪具有与人类相似的器官构成和代谢类型,尤其是在脂肪代谢和沉积方面,是研究人类肥胖的最佳模型。然而,猪FTO基因的相关研究十分欠缺,对乌金猪FTO基因的研究未见报道。乌金猪是云南省地方猪种之一,具有耐粗饲、适应性强、腹部脂肪沉积较多等性状特点,是研究人类肥胖疾病机理的理想动物模型。
我们采用反转录PCR技术,从乌金猪脂肪组织中克隆FTO基因,并对该基因的序列结构和特征进行了生物信息学分析,同时通过软件对该蛋白质的空间结构进行了预测,构建其分子系统进化树,并采用实时PCR方法对7种组织进行了组织表达谱分析,为深入研究乌金猪FTO基因的生物学功能奠定了重要基础。
1 材料与方法
1.1 材料
屠宰成年乌金猪3头,取所需各种组织样本,液氮速冻,-80℃保存。TRIzol-A+试剂购于北京天根生化科技有限公司;EasyScript第一链cDNA合成试剂盒购于北京全式金生物技术有限公司;pMD18-T载体、DL2000 DNA marker等均购于大连宝生物工程有限公司;其他试剂为国产分析纯产品。
1.2 RNA提取
用TRIzol-A+试剂提取1头乌金猪脂肪组织总RNA,用核酸蛋白定量仪测定RNA的浓度及纯度,D260nm/D280nm=1.8~2.0,并经10 g/L琼脂糖凝胶电泳鉴定其完整性后备用。
1.3 PCR引物设计及PCR反应条件优化
根据GenBank中公布的猪的相应基因序列(登录号:NM_001112692.1),用Primer Primer 5.0软件针对该基因设计2对引物,一对用于FTO基因的克隆,另一对用于FTO基因多组织的实时PCR,并以18S rRNA看家基因为内参。引物序列见表1,由生工生物工程(上海)有限公司合成。
FTO基因克隆PCR反应条件:95℃预变性5 min;95℃变性 40 s,58℃退火 40 s,72℃延伸 30 s,35个循环;72℃延伸10 min。用ddH2O代替反转录产物作为空白对照,用以检验是否有外源基因和基因组DNA污染。
1.4 cDNA合成与基因克隆
从1头乌金猪脂肪组织中提取总RNA,根据所测定的浓度,各取2 μg总RNA进行反转录,用Ea⁃syScript第一链cDNA合成试剂盒进行反转录(42℃反转录30 min,85℃加热5 min以使反转录酶失活),反应体系为20 μL。反转录后的cDNA单链可于-20℃保存或立即用于PCR。用生工生物工程(上海)有限公司的胶回收试剂盒回收PCR扩增的目的片段,并将其连接到pMD18-T载体上,转入大肠杆菌,涂布LB培养基平板,长出菌落后经蓝白斑筛选挑取白色单菌落,于LB培养液中振荡培养12 h左右,分别取PCR鉴定过的菌液提取重组质粒,用KpnⅠ/HindⅢ双酶切鉴定,将鉴定过的菌液及质粒送生工生物工程(上海)有限公司进行DNA序列测定。
1.5 生物信息学分析
将反转录PCR所得基因片段测序后,用DNA⁃Star 6.0软件包中的Seqman程序进行序列拼接,用BLAST工具和DNAStar 6.0软件包进行序列同源性比对和cDNA序列分析,用Meg4软件进行系统进化树分析;利用 ExPASy、SignalP、ProtScale、NetPhos、NetNGlyc、TMHMM、SMART、PSIPRED 和 SWISSMODEL网站在线程序对FTO蛋白的等电点、相对分子质量、信号肽、疏水性轮廓、磷酸化位点、糖基化位点、跨膜信息、二级结构及三级结构进行预测。所用相关分析软件的生物信息学网址见表2。
1.6 实时PCR分析组织表达谱
分别提取3头乌金猪心、肝、脾、肺、肾、肌肉和脂肪等7种组织的RNA,反转录为cDNA,以FTO F2/R2为引物进行实时PCR。反应条件:95℃预变性30 s;95℃变性5 s,60℃退火30 s,72℃延伸30 s,40个循环;72℃延伸10 min。18S rRNA和FTO基因荧光定量PCR反应条件只有退火温度不同为55℃。每样品3头猪做3次重复,每个重复做2个平行,同时用ddH2O代替反转录产物和荧光试剂作为空白对照,用以检验是否有外源基因和基因组DNA污染。将纯化的目的基因或内参基因的PCR产物进行1/10梯度稀释后做标准曲线,共8个梯度浓度,每个浓度2个平行重复。相对表达量的分析以18S rRNA基因作为内参基因,通过参照基因表达水平校正待测样本中目标基因的相对表达量,然后将各样品的Ct值与标准曲线进行比较,得出其起始模板拷贝数。

表1 PCR引物

表2 生物信息学软件及网址
2 结果
2.1 乌金猪脂肪组织总RNA的提取
取3 μL提取的总RNA经10 g/L琼脂糖凝胶电泳分离,结果见图1,28S和18S条带清晰,且28S和18S灰度比约为2∶1,无脱尾现象出现,点样孔无残留,表明RNA无降解,质量可靠,可用于后续实验。

图1 乌金猪脂肪组织总RNA的琼脂糖凝胶电泳
2.2 FTO基因的克隆
反转录PCR得到1518 bp的片段,经10 g/L琼脂糖凝胶电泳检测,确认扩增产物与目的片段大小一致。将克隆的片段与pMD18-T载体连接,挑取阳性克隆,提取重组质粒,用KpnⅠ/HindⅢ双酶切鉴定,表明目的基因FTO已成功插入载体。见图2。
2.3 乌金猪FTO基因序列及对应蛋白
通过对PCR产物原液双向测序及克隆后提取的阳性质粒双向测序,确定了1518 bp的编码区(CDS)序列(已提交GenBank,登录号为JQ031263),编码由505个氨基酸残基组成的蛋白(图3)。
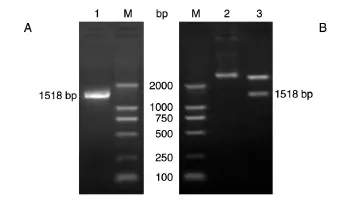
图2 PCR扩增FTO基因(A)及重组质粒的双酶切鉴定(B)

图3 乌金猪FTO基因编码区序列及对应的氨基酸序列
2.4 生物信息学分析
2.4.1 同源性分析 用DNAMAN软件将由FTO CDS推导的氨基酸序列与NCBI网站BLAST后下载的牛(登录号:BC140478.1)、人(登录号:NM_001080432.2)、羊(登录号:EU072419.1)和大鼠(登录号:BC168239.1)等4个物种的FTO CDS编码的氨基酸序列进行比对,同源性分别为91%、89%、90%和83%(图4)。
2.4.2 等电点与相对分子质量预测 利用ExPASy网站在线工具预测的FTO蛋白的理论等电点为5.18,相对分子质量为58.16×103。
2.4.3 进化树分析 用Meg4软件构建乌金猪、牛、人、羊和大鼠FTO蛋白的系统进化树,结果显示乌金猪与牛、羊的亲缘性较近,其次是人、大鼠(图5)。
2.4.4 FTO蛋白的特征分析 用TMHMM程序预测FTO蛋白的跨膜结构,发现该蛋白无跨膜区,表明该蛋白不是跨膜蛋白;用SignalP 3.0 Server软件分析,发现该蛋白无信号肽出现;用NetPhos 2.0 Server对蛋白序列的磷酸化位点进行预测,发现乌金猪FTO蛋白结构中的磷酸化修饰位点共22个,包括10个丝氨酸蛋白激酶磷酸化位点、7个苏氨酸蛋白激酶磷酸化位点、5个酪氨酸蛋白激酶磷酸化位点(图6);用NetNGlyc 1.0 Server对FTO蛋白的糖基化位点进行预测,发现在200、246、302位氨基酸残基处有糖基化位点(图7);用网上在线工具ExPASy-ProtScale预测FTO蛋白的疏水性,发现该蛋白的疏水性最大值为3.056,最小值为-3.222,表明该蛋白是一个亲水性蛋白(图8)。

图4 人(Homo)、大鼠(Rattus)、牛(Bos)、羊(Ovis)和乌金猪FTO CDS编码的氨基酸序列的同源性比较

图5 人、大鼠、牛、羊和乌金猪的FTO进化树
2.4.5 FTO蛋白的二级结构和三级结构 用PSIPRED程序预测该蛋白的二级结构,结果表明共有201个螺旋(helix)、33个伸展链(strand)和271个卷曲结构(coil)(图9)。用网上在线工具SWISSMODEL分析了该蛋白的三级结构(图10)。

图6 FTO蛋白的磷酸化位点预测

图7 FTO基因的糖基化位点预测

图8 FTO蛋白的疏水性预测

图9 FTO蛋白的二级结构预测
2.5 FTO基因的组织表达谱
用FTO F2/R2引物分别扩增了3头乌金猪的7种组织的cDNA,实时PCR结果表明,FTO基因mRNA在肝脏组织中的表达最高,在肾、脂肪、肺和脾组织中也大量表达,在心脏和肌肉组织中少量表达(图11)。
3 讨论
1999年,一篇关于脚趾融合小鼠的文献对FTO基因进行了报道[2],但随后的研究进展缓慢,直至2007年发现人的该基因与肥胖有关,才逐渐成为研究热点。FTO基因是一个与肥胖相关的等位基因[3],属于非血红色加双氧酶基因超家族,编码2-酮戊二酸依赖的核酸脱甲基酶[4-5]。FTO广泛存在于脊椎动物和海洋藻类中,是一个携带核定位信号的球状蛋白。FTO基因mRNA在人、鼠大多数组织中均有表达,尤其在下丘脑中表达丰富[4,6]。本研究表明,FTO基因mRNA在乌金猪7个组织中的表达量有差异,在肝组织中表达量最高,脂肪、肾、脾中也有大量表达,心脏、肌肉中表达量最少。Frayling等用实时PCR发现FTO基因编码蛋白在人体组织中广泛分布,包括脂肪组织、胰岛β细胞、垂体、肾上腺、大脑,尤其是在下丘脑弓状核中高表达[3-4]。郭兵等研究表明,梅山猪FTO基因在海马、脂肪、肝脏和肌肉组织中均有表达,在海马中的表达量最高,在脂肪和肝脏组织中表达量较高,肌肉组织中的表达量较少[7]。

图10 FTO蛋白的三级结构预测
我们克隆了乌金猪FTO基因编码区全长系列,并和人、牛、羊和鼠等4个物种进行了进化树分析,表明乌金猪FTO与牛、羊的亲缘性较近,其次是人、大鼠。

图11 乌金猪FTO基因的组织表达谱
生物信息学是通过数学、统计学和计算机科学等学科对生物数据进行分析的方法和技术,由于它快速、高效和低成本,已被广泛用于预测蛋白质的结构、功能和其他生物学特征[8]。蛋白质分析一般包括信号肽、跨膜结构和磷酸化位点、二级结构等的分析。蛋白质磷酸化是最常见、最重要的一种蛋白质翻译后修饰方式[9],它参与和调控生物体内的许多生命活动,如信号转导、基因表达、细胞周期调控等诸多细胞过程[10]。蛋白质磷酸化位点主要发生在丝氨酸、苏氨酸和酪氨酸残基上[11]。本研究表明乌金猪FTO蛋白结构中的磷酸化修饰位点共22个,这些磷酸化位点将是调控FTO蛋白功能的关键位点。糖基化是真核生物蛋白质翻译后重要的修饰之一,有调节蛋白质功能的作用[12],我们推测的乌金猪FTO氨基酸序列在200、246、302位有糖基化位点。从氨基酸序列到蛋白质二级结构,是理解蛋白质结构和功能的重要一步,对蛋白质二级结构的预测有助于了解其三维构象和分析其结构域[13]。本研究结果表明,推测的乌金猪FTO氨基酸序列共有201个螺旋、33个伸展链和271个卷曲结构。蛋白质的结构与功能关系密切,以上关于FTO推测的氨基酸序列分析将为其蛋白质的生物学功能研究提供理论依据。
[1]Kopelman P.Health risks associated with overweight and obe⁃sity[J].Obesity Rev,2007,8(1):13-17.
[2]Peters T,Ausmeier K,Ruther U.Cloning of Fatso(Fto),a nov⁃elgene deleted by the Fused toes(Ft)mouse mutation[J].Mamm Genome,1999,10(10):983-986.
[3]Frayling T M,Timpson N J,Weedon M N,et al.A common variant in the FTO gene is associated with body mass index and predisposestochildhood and adultobesity[J].Science,2007,316(5826):889-894.
[4]Gerken T,Girard C A,Tung Y C L,et al.The obesity-asso⁃ciated FTO gene encodes a 2-oxoglutarate-dependent nucleic acid demethylase[J].Science,2007,318(5855):1469-1472.
[5]Fernandez A M,Kim J K,Yakar S,et al.Functional inactiva⁃tion of the IGF-1 and insulin receptors in skeletal muscle causes type 2 diabetes[J].Genes Dev,2001,15:1926-1934.
[6]Fredriksson R,Hagglund M,Olszewski P K,et al.The obesi⁃ty gene,FTO,is of ancient origin,up-regulated during food deprivation and expressed in neurons of feeding-related nu⁃clei of the brain[J].Endocrine,2008,149(5):2062-2071.
[7]郭兵.猪FTO的组织特异性表达及其调控[D].南京:南京农业大学,2011.
[8]Romano P,Giugno R,Pulvirenti A.Tools and collaborative environmentsforbioinformatics research[J].BriefBioinform,2011,12(6):549-561.
[9]Kim J H,Lee J,Oh B,et al.Prediction of phosphorylation sites using SVMs[J].Bioinformatics,2004,20(17):3179-3184.
[10]姜铮,王芳,何湘,等.蛋白质磷酸化修饰的研究进展[J].生物技术通讯,2009,20(2):233-237.
[11]Blom N,Gammeltoft S,Brunak S.Sequence and structurebased prediction of eukaryotic protein phosphorylation sites[J].J Mol Biol,1999,294(5):1351-1362.
[12]Gupta R,Brunak S.Prediction of glycosylation across the hu⁃man proteome and the correlation to protein function[J].Pac Symp Biocomput,2002,7:310-322.
[13]张成岗,贺福初.生物信息学方法与实践[M].北京:科学出版社,2002:158-230.
