基于Cytb与D-loop的罗非鱼选育群体遗传结构对比分析
2014-09-28颉晓勇钟金香李思发
颉晓勇,钟金香,李思发
(1. 中国水产科学研究院南海水产研究所,广东省渔业生态环境重点实验室,广东 广州510300;2. 广东省水产技术推广总站,广东 广州 510220;3.上海海洋大学,上海 201306)
线粒体DNA(Mitochondrial DNA,mtDNA)位于细胞核之外,以非孟德尔遗传方式控制和影响线粒体功能。由于动物mtDNA 碱基替换速率相对较快,比核内碱基有更高的变异,其高效的单倍体母系遗传方式可缩减用于检测的有效种群大小,提高对遗传漂变的敏感性[1]。mtDNA 标记已经被广泛应用于鱼类群体遗传结构和系统演化关系的相关研究[2-3]。细胞色素b 基因(Cytb)是线粒体13个蛋白质编码基因中应用得最广泛的基因[4]。控制区(D-loop)序列是整个mtDNA 序列和长度变异最大的区域[5]。Cytb 和D-loop 序列应用于群体遗传结构和系统进化有大量文献报道[5-6]。然而,有关Cytb 和D-loop 序列应用于群体遗传结构研究结果之间区别和联系的研究报道相对较少。吉富罗非鱼由上海水产大学引进中国之后,经历约10 a 的遗传改良,成为生产性状优良的养殖新品系[6]。该研究开展了新吉富罗非鱼基于mtDNA Cytb 和D-loop 基因序列的群体遗传结构对比分析,旨在探索2种分子标记分析结果的差异与关系,进而为针对不同研究材料,选择不同的分析方法,以及扩大研究结果之间横向比较提供技术上的指导,使得研究结论更加客观和真实。
1 材料与方法
1.1 试验材料
中国的吉富品系罗非鱼由菲律宾引进。基础群体(F0)是1994年引进的5 000 尾尼罗罗非鱼,从1996年起按照系统选育方法,人工选育每年产生一代。本试验的材料是从当初引进的基础群体F0、选育群体F6~F9共5 代中随机采样各20 尾,编号后剪取尾鳍以95%乙醇分别保存。
1.2 基因组DNA 提取
参照常规的酚-氯仿抽提方法提取尾鳍样品中的基因组DNA,通过琼脂糖凝胶电泳方法检查所提取DNA 质量并初步定量。
1.3 PCR 扩增
控制区扩增所采用的引物参考Jean 等[7],引物序列为DL1:5’ACC CCT GGC TCC CAA AGC 3’和DH2:5’ATC TTA GCA TCT TCA GTG 3’。细胞色素b 基因所用引物参考彭作刚等[8],引物序列为L14724:5’GAC TTG AAA AAC CAC CGT TG 3’和H15915:5’CTC TCT CCG GAT TAC AAG AC 3’。PCR 反应体系为50 μL,其中包含10×Buffer 溶液5 μL,dNTP 10 mmol,上游引物20 pmol,下游引物20 pmol,模板DNA 200 ng,Taq DNA 聚合酶2.5 U,不足部分用去离子灭菌水补充至反应体系总体积50 μL。所采用的PCR 扩增反应条件:94℃模板预变性4 min;94℃模板DNA 变性30 s,模板与引物退火30 s,模板- 引物结合物于72℃延伸1 min,每次扩增包含35个循环;最后72℃延伸10 min;扩增完成后于4℃保存。其中,控制区退火温度50℃,细胞色素b 基因退火温度56℃。
1.4 DNA 测序
扩增产物用1%的琼脂糖凝胶电泳进行质量检测,选择扩增效果好的PCR 产物采用DNA 纯化试剂盒进行纯化,委托上海生工生物工程技术服务有限公司用ABI 377 DNA 自动测序仪进行扩增片段碱基序列的双向测定。
1.5 数据处理与分析
在GenBank 数据库中,用BLAST 软件[9]搜索尼罗罗非鱼Cytb 和D-loop 基因序列,与本研究委托测序所得到的不同选育群体序列结果进行比较;对测序结果采用Bioedit 软件[10]进行编辑,采用CLUSTL W 软件[11]进行碱基顺序重排和DNA 序列同源性比较,同时进行人工核对校正确保DNA 序列结果的准确性。用DNASP软件[12]进行碱基序列统计相关分析。用Arlequin 软件[13]对整理重排确定的DNA 序列进行分子方差分析,同时采用该软件计算群体遗传分化指数Fst,其中Fst 的显著性采用排列测验法(Permutation test)进行检验(重复次数为1 000)。遗传距离采用MEGA 软件[14]基于Kimura 2-parameter 模型进行计算。按Cytb/D-loop 分别计算由Cytb 和D-loop 2种方法所得到的群体间遗传距离比率和群体间遗传分化指数比率。
2 结果与分析
2.1 基于Cytb 与D-loop 序列分析的群体内遗传距离比率
本研究基于Cytb 与D-loop 序列分析得到的群体内遗传距离比率结果如表1 所示。根据2种方法得到的群体内遗传距离比率范围0.754 67~0.872 81,平均值0.795 15±0.048 57。
2.2 基于Cytb 与D-loop 序列分析的群体间遗传分化指数比率
本研究基于Cytb 与D-loop 序列分析得到群体间遗传分化指数比率结果如表2 所示。根据2种方法得到的群体间遗传分化指数比率范围为1.051 40~2.914 65,平均值为1.506 55±0.564 65。
3 小结与讨论
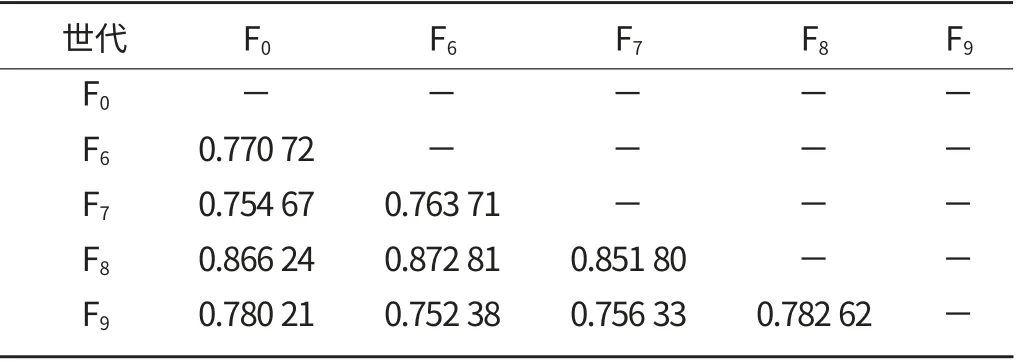
表1 基于Cytb 和D-loop 序列的罗非鱼5个选育群体间遗传距离比率
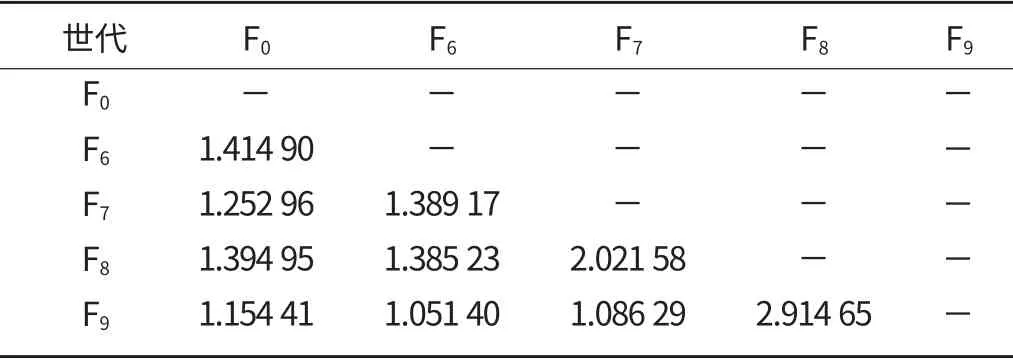
表2 基于Cytb 和D-loop 序列的罗非鱼5个选育群体间遗传分化指数比率
鱼类线粒体DNA 是细胞核外具自主复制、转录和翻译能力的遗传因子,其分子结构包括一条重链、一条轻链,呈共价闭合环状。mtDNA 在遗传方式上,遵从母系遗传,一般不发生重组。但是mtDNA 的遗传并不完全独立,在一定程度上受到核基因的影响和控制,其复制、转录等过程所需要的各种蛋白酶体系都由核基因编码。与核DNA 相比,鱼类mtDNA 具有分子量小、碱基堆叠方式相对简单、进化速度相对较快而且其中不同区段的进化速度存在差异等特点,因而成为细胞内一个相对独立的遗传信息复制单位。细胞中mtDNA 的碱基突变会导致一个细胞中同时存在两种类型mtDNA,即形成异质性细胞,这种类型的细胞在连续分裂过程中会改变突变型和野生型的比例。因此,在对群体进行严谨的分子遗传结构特征分析时,有必要考虑核DNA 与mtDNA的遗传差异,选择合适的mtDNA 区域进行遗传变异分析。有大量采用Cytb 序列、D-loop 序列或其他mtDNA基因区域进行分析的研究报道[5,15-16]。mtDNA 不同区域的进化速率不同,采用mtDNA 不同区域分析得到的群体遗传结构特征必然有所差异,对于mtDNA 不同区域遗传得到的群体遗传结构特征进行比较和量化分析有助于进一步深入理解群体之间的遗传结构特征差异。
当前大多数采用Cytb 或(和)D-loop 分子标记的研究停留于分析不同种类生物群体的遗传结构特征,未对Cytb 和D-loop 研究结果进一步对比分析,在一定程度上限制了研究结果的参考和应用价值。该研究基于Cytb方法所得到的群体间遗传距离均小于基于D-loop 标记所得到的相应遗传距离,表明D-loop 区域序列碱基变异速率大于Cytb 区域,D-loop 标记相对于Cytb 标记具有更高的区分能力,这一结论与其他类似研究结论相一致[17-19]。该研究Cytb 方法所得到的群体间遗传距离仅为基于D-loop 标记所得到相应遗传距离的0.795 15,D-loop 标记所得群体间遗传距离是Cytb 方法所得相应遗传距离的约1.25 倍。群体遗传分化指数是群体遗传结构的另一项重要考量指标[6]。根据杨慧荣等[20]研究赤眼鳟的资料,采用Cytb 与D-loop 方法的群体间Fst 比值与本研究呈现相似的规律,基于Cytb 的遗传分化指数在程度上大于D-loop 的遗传分化指数,剔除其Fst 偏离较大及出现负值的情况,可比的Cytb/D-loop 比率范围在1.172 03~1.283 45 之间。该研究根据2种方法得到的群体间遗传分化指数比率范围1.051 40~2.914 65,平均值1.506 55,即基于Cytb 的遗传分化指数约为D-loop 方法所得遗传分化指数的1.5 倍。本研究遗传距离比率和遗传分化指数比率结果可为其他采用单种Cytb 或D-loop标记方法研究对像遗传结构特征提供参考。这同时也表明,采用DNA 不同区域、不同分子标记方法联合分析结果将更为客观。
[1]廖顺尧,鲁 成.动物线粒体基因组研究进展[J].生物化学与生物物理进展,2000,27(5):508-512.
[2]Oleinik A G,Skurikhina L A,Brykov V A.Divergence of Salvelinus species from northeast Asia based on mitochondrial DNA[J].Ecology of freshwater Fish,2007,16(1):87-98.
[3]TeletcheaF.Molecularidentificationmethodsof fish species:reassessment and possibleapplications[J].Reviews in Fish Biology and Fisheries,2009,19(3):265-293.
[4]Lydeard C,Roe K J.The phylogenetic utility of the mitochondrial cytochrome b gene for inferring relationshipsamong actinoptergian fishes[A].Kocher T D,Stepien S A.Molecular systematics of fishes[C].San Diego:Academic Press,1997.
[5]郭新红,刘少军,刘 巧,等.鱼类线粒体DNA研究新进展[J].遗传学报,2004,31(9):983-1000.
[6]颉晓勇,李思发,蔡完其.基于D-loop序列的罗非鱼选育群体遗传变异分析[J].上海海洋大学学报,2011,20(3):336-341.
[7]JeanFA,BeatriceAG,EddieKA,etal.Geneticdifferentiationamong naturalpopulationsoftheNiletilapiaOreaochronisniloticus(Teleostei Cichlidae)[J].Heredity,1997,79:88-96.
[8]彭作刚,张耀光,何舜平,等.从细胞色素b基因序列变异分析中国鲇形目鱼类的系统发育[J].遗传学报,2005,32(2):145-154.
[9]AltschulSF,ThomasLM,AlejandroA,etal.GappedBLASTand PSI-BLAST:anewgenerationofproteindatabasesearchprograms[J].NucleicAcidsRes.1997,25:3389-3402.
[10]HallTA.BioEdit:user-friendlybiologicalsequencealignmenteditor and analysisprogram forWindows95/98/NT [J].Nucleic Acids SymposiumSeries,1998,41:95-98.
[11]ThompsonJD,HigginsDG,GibsonTJ.CLUSTALW:improvingthe sensitivityofprogressivemultiplesequencealignmentthroughweighting,positioin-specificgappenaltiesandweightmatrixchoice[J].Nucleic AcidsResearch.1994,22:4673-4680.
[12]RozasJ,Sanchez-DelbarrioJC,MesseguerX,etal.DnaSP,DNA polymorphism analysesby the coalescentand othermethods[J].Bioinformatics,2003,19:2496-2497.
[13]SchneiderS,RoessliD,ExcoffierL.ARLEQUIN Version2.000:a softwareforpopulationgeneticsdataanalysis[M].Geneticsandbiometry laboratory,UniversityofGeneva,Switzerland.2000.
[14]KumarS,TamuraK,NeiM.MEGA3:Integratedsoftwareformolecular evolutionarygeneticsanalysisandsequencealignmentbriefings[J].Bioinformatics,2004,5:150-163.
[15]于旭蓉,仇雪梅,柳晓瑜,等.线粒体DNA多态性在海洋动物群体遗传结构研究中的应用[J].生物技术通报,2011,10:49-54.
[16]陈善元,肖 蘅.鱼类分子系统发育研究中的分子标记与推断方法[J].云南大学学报(自然科学版),2003,25(增刊):146-152.
[17]李栋元,李景云,杨具田,等.兰州大尾羊mtDNAD-loop和Cytb区序列分析与多态性研究[J].中兽医医药杂志,2011,5:14-17.
[18]GerlachG,MusolfKF.Fragmentationoflandscapeasacauseforgenetic subdivisioninbankvoles[J].ConservationBiology,2000,14:1066-1074.
[19]赵 亮,谢本贵,刘志瑾,等.太湖新银鱼线粒体D-loop和cytb片段序列结构与进化速率比较[J].动物学杂志,2010,45(2):27-38.
[20]杨慧荣,赵会宏,蒙子宁,等.赤眼鳟线粒体D-loop和Cytb基因序列的对比分析[J].中山大学学报(自然科学版),2012,51(5):100-106,136.
