胞质型磷脂酶A2基因克隆及其表达量随大黄鱼仔稚鱼生长发育的变化
2014-09-20冯硕恒谢奉军蔡佐楠麦康森艾庆辉
冯硕恒 谢奉军 蔡佐楠 麦康森 艾庆辉
(中国海洋大学,农业部水产动物营养与饲料重点实验室,海水养殖教育部重点实验室,青岛 266003)
磷脂是分子中含有磷酸的复合极性脂,在仔稚鱼发育过程中发挥着重要作用。首先,磷脂是细胞膜的重要组成成分[1],对维持细胞膜功能的完善起着重要作用;其次,鱼类利用极性脂的能力要高于中性脂,因此,磷脂在胚胎发育阶段和仔稚鱼早期阶段发挥着重要的供能作用[2-4];此外,磷脂分解代谢还能产生类二十烷酸、二脂酰甘油和肌醇磷酸等生物活性物质,对维持鱼类正常的生理活动具有重要意义[5]。目前,在水产动物上的研究表明,微颗粒饲料中添加磷脂可显著提高仔稚鱼的生长、存活、抗应激能力、脂肪吸收和转运以及饲料的适口性[6-13]。然而,在鱼类上对磷脂代谢的研究却较少。
目前,诸多学者认为鱼类磷脂分解代谢机制与哺乳动物是一致的,即在肠道内由胰腺分泌的磷脂酶A2(PLA2)水解产生游离的脂肪酸和溶血磷脂,再被肠细胞吸收[14]。根据磷脂酶起作用的酯键不同,磷脂酶分为 A1、A2、C和 D型。由于PLA2能特异性水解磷脂sn-2位的脂肪酸,产生溶血磷脂和游离脂肪酸,溶血磷脂和游离的脂肪酸分别在高等动物膜磷脂更新[15]和炎性反应[16]过程中发挥重要作用,因此对PLA2的研究在哺乳动物中已成为焦点。Zambonino等[17]的研究发现,在海水仔稚鱼开口后几天就能检测到PLA2。早期在真鲷肝胰腺中的研究发现PLA2有2种亚型,且其分子质量较低的属于Ca2+依赖型酶[18]。此外,Saele等[19]在大西洋鲑上确定了 PLA2-ⅠB 的序列,并在基因和蛋白质水平上阐述了其随大西洋鲑生长发育的变化。随后,Fujikawa等[20]在真鲷上也发现了PLA2的新亚型PLA2-ⅠB,并对其在消化器官中的组织分布做了详细阐述。在鱼类上,已克隆到斑马鱼(Danio rerio,GenBank登录号:AAA53229.1)、尼罗罗非鱼(Oreochromis niloticus,GenBank 登录号:XP_003445168.2)、青鳉(Oryziaslatipes,GenBank登 录 号:XP_004068057.1)和红鳍东方鲀(Takifugu rubripes,GenBank登录号:XP_003978485.1)等胞质型磷脂酶A2(cPLA2)的cDNA全长或部分序列。但是,cPLA2基因表达与仔稚鱼生长发育之间的关系鲜有报道。
Mai等[21]从组织学的角度系统分析了大黄鱼仔稚鱼到幼鱼早期阶段消化系统的变化情况。近年来,Zhao 等[13]、Xie 等[22]和 Li等[23]的研究均发现消化酶活力的大小在一定程度上能反映大黄鱼仔稚鱼消化系统的完善与否。然而,在转录水平上cPLA2的基因表达与大黄鱼仔稚鱼消化系统发育之间的关系却未见报道。
大黄鱼是我国特有的养殖种类,因其具有较高的营养价值和经济价值,在我国东南沿海地区大黄鱼的人工养殖得到广泛推广[24-26]。因此,本试验旨在克隆大黄鱼磷脂分解代谢关键酶cPLA2基因的cDNA全长,并分析cPLA2基因表达量随大黄鱼仔稚鱼生长发育的变化趋势,同时探究大黄鱼消化系统发育与cPLA2基因表达变化之间的关系。
1 材料与方法
1.1 试验用鱼与饲养管理
试验在福建省宁德水产技术推广站进行。试验用鱼为该推广站2012年3月份人工繁育的大黄鱼鱼苗。试验期间所用海水经室外蓄水池沉淀,2级砂滤池滤过,进育苗池前再经过滤袋过滤。试验期间,水温(23±1)℃,pH 8.0±0.2,盐度(23±2)‰。每桶内设1个气石,桶内海水每天的换水量为150%~300%。白天水表面的光强度为100 lx左右。每天用聚乙烯管将漂浮在水面的污物及时去除,用虹吸管将桶底的残余饲料、粪便和死苗及时吸出。从3~8日龄投喂轮虫(Brachionus plicatilis),6~11日龄投喂卤虫无节幼体,10~16日龄投喂鲜活桡足类,之后到40日龄投喂冷藏桡足类,每天饱食投喂 5 次(06:00、08:30、12:00、14:30和 17:00)。
1.2 取样
鱼苗孵化出来后,从1日龄开始取样,随后在3、7、11、15日龄时直接从育苗池中取样。每个时间点随机取3组,每组50尾鱼。15日龄时随机挑选规格整齐、体质健壮、活力好的初始体重为(4.08±0.11)mg鱼苗9 000尾,随机放养于3个蓝色塑料桶(180 L)内,每桶3 000尾。在19、25、30、35和40日龄分别从3个养殖桶内随机取50尾鱼,样品保存在-80℃,用来分析cPLA2基因的表达。40日龄时,随机取6尾大黄鱼仔稚鱼的肝脏,取后马上置于液氮中,并储存于-80℃中,用于cPLA2基因的克隆。
1.3 总RNA提取、cDNA合成和cPLA2基因的克隆
采用Trizol Reagent提取大黄鱼幼鱼(体重在20 g左右)肝脏总RNA,经1.2%琼脂糖凝胶电泳、GoldView染色显示28S、18S和5S以检测RNA的完整性,并用分光光度计检测总RNA的浓度及纯度。采用TaKaRa反转录用PrimerScript RT Reagent Kit合成cDNA第1链。以GenBank中斑马鱼、大西洋鲑、尼罗罗非鱼、青鳉、红鳍东方鲀、人和鼠等物种的氨基酸序列为模板,利用CODHOP(http://bioinformatics.weizmann.ac.il/blocks/codehop.html)设计简并引物cPLA21F和cPLA21R(表1)。然后进行PCR扩增反应,克隆大黄鱼cPLA2基因的部分片段。cPLA25'端和3'端非编码区的扩增采用Clonetech SMARTTMRACE cDNA扩增试剂盒,按照说明书进行操作,采用的特异性引物分别为cPLA22F和cPLA22R(表1)。将获得的PCR产物经纯化后克隆到pEASY-T1载体上,转化到大肠杆菌感受态细胞上,挑选阳性克隆送往上海博尚生物科技有限公司进行测序。
1.4 序列分析和进化树构建
应用NCBI上的BLAST工具(http://www.ncbi.nlm.nih.gov/BLAST/)将克隆的基因进行同源性比对分析,并推断其开放阅读框(ORF)和编码的氨基酸序列。使用软件TMHMM(http:www.cbs.dtu.dk/services)来预测基因的跨膜域。使用SMART(http://smart.emblheidelberg.de/)和PROSITE软件(http://kr.expasy.org/prosite/)来预测氨基酸序列中的功能位点或氨基酸结构域。使用ProtParam程序(http://www.expasy.ch/tools/protparam.html)来计算氨基酸序列的物理和化学参数。根据选择的相关基因序列使用CLUSTAL X1.83[27]和 MEGA 4.0[28]软 件 来 构 建 物 种 间cPLA2基因的物种系统发生关系。使用邻位相连(neighbor-joining)法构建进化树[29]。
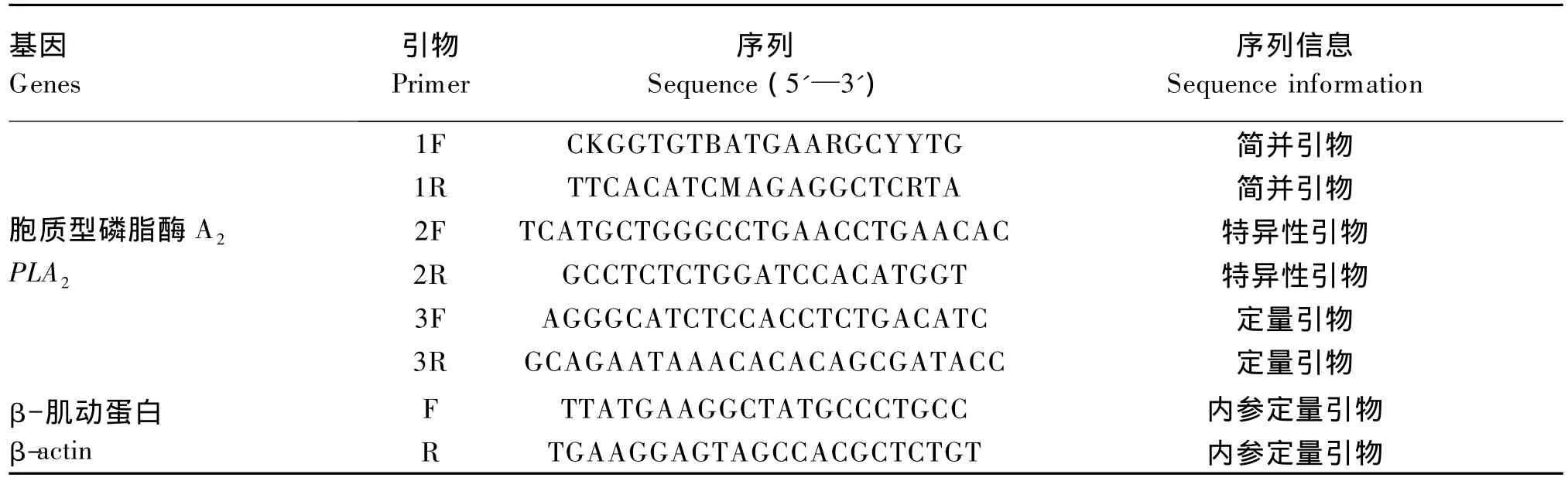
表1 本试验中用到的引物序列Table 1 Sequences of primers used in this experiment
1.5 cPLA2基因的定量表达
采用实时定量PCR法来检测cPLA2基因的表达量随大黄鱼仔稚鱼生长发育的变化。实时定量PCR引物为PLA23F和PLA23R(表1)。
实时定量PCR体系为25 μL,其中上、下游引物(10 μmol/L)各 1 μL,第 1 链 cDNA 1 μL,2×SYBR Premix Ex TaqTMⅡ (TaKaRa,日 本)12.5 μL和9.5 μL的无菌水。定量仪器为实时定量 PCR 仪 (Mastercyclereprealplex,Eppendorf,德国)。实时定量的程序为95℃持续2 min,1个循环;95℃持续10 s,58.7℃持续10 s,72℃持续20 s,共计40个循环。之后,进行熔解曲线以检验每个PCR反应只有1个PCR产物。通过4倍梯度稀释得到5个不同浓度的cDNA模板,以每个浓度cDNA为模板,通过实时定量PCR得出每对引物每个浓度cDNA的循环数(Ct)值,以Ct值为纵坐标,模板拷贝数的对数为横坐标,拟合得到一条随拷贝数变化的Ct值的直线,根据拟合得到的直线斜率和扩增效率的计算公式E=10(-1/Slope)-1(式中:E为扩增效率;Slope为斜率),得出每对引物的扩增效率。在本试验中,目的基因cPLA2的扩增效率为0.909 8,内参基因β-actin的扩增效率为 0.978 3。由于 ΔCt(ΔCt=Ct目的基因-Ct内参基因)的绝对值为0.041,约等于0,表明目的基因和内参基因扩增效率基本一致,采用2-ΔΔCt方法测定目的基因的表达量[30]。
1.6 统计分析
结果采用SPSS 16.0软件进行统计分析,在单因素方差分析(one-way ANOVA)达到显著水平(P<0.05)时,采用Tukey’s检验进行多重比较,数据表示为平均值±标准误。
2 结果与分析
2.1 基因序列特征分析
通过序列分析可知,cPLA2基因cDNA全长共计2 550 bp,其中5'端非编码区176 bp,开放阅读框 2 169 bp,3'端非编码区 205 bp,共编码 723 个氨基酸,预测所编码的蛋白质的分子质量为81.98 ku,理论等电点为5.21,将该序列提交至GenBank,获得登录号为 KF006240.1。软件分析预测该蛋白质可能含有42个磷酸化位点,包括24个丝氨酸位点、9个苏氨酸位点和9个酪氨酸位点(图 1)。
2.2 序列比对和进化树分析
推导的大黄鱼cPLA2基因的氨基酸序列与斑马鱼、红鳍东方鲀、尼罗罗非鱼和青鳉的cPLA2基因的氨基酸序列分别有75.80%、89.35%、87.96%和86.31%的相似性(图2)。系统进化树也显示克隆到的大黄鱼cPLA2基因与上述这几种鱼类的亲缘关系较近,与其他物种的亲缘关系较远(图3)。

图1 大黄鱼cPLA2基因的cDNA全长核苷酸序列及其推导的氨基酸序列Fig.1 cDNA full long nucleotide sequence and deduced amino acid sequence of cPLA2gene of large yellow croaker

图2 大黄鱼与其他9种生物cPLA2基因氨基酸序列比对结果*Fig.2 Alignment results of cPLA2gene amino acid sequence between large yellow croaker and other 9 species
* 因像素较低,排版后图片显示不清,如有需要,可与作者联系索取。
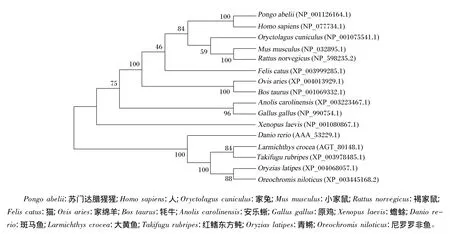
图3 大黄鱼与其他物种cPLA2基因氨基酸序列系统进化树聚类分析Fig.3 Cluster analysis of phylogenetic tree of cPLA2gene amino acid sequence between large yellow croaker and other species
2.3 cPLA2基因表达量随大黄鱼仔稚鱼生长发育的变化
由图4可知,孵化后,大黄鱼仔稚鱼cPLA2基因的表达量随日龄的增加先显著升高(1、3、7日龄vs.15日龄,P<0.05),在15日龄时达到最高值,随后显著下降(15日龄vs.19日龄,P<0.05),之后趋于平稳。
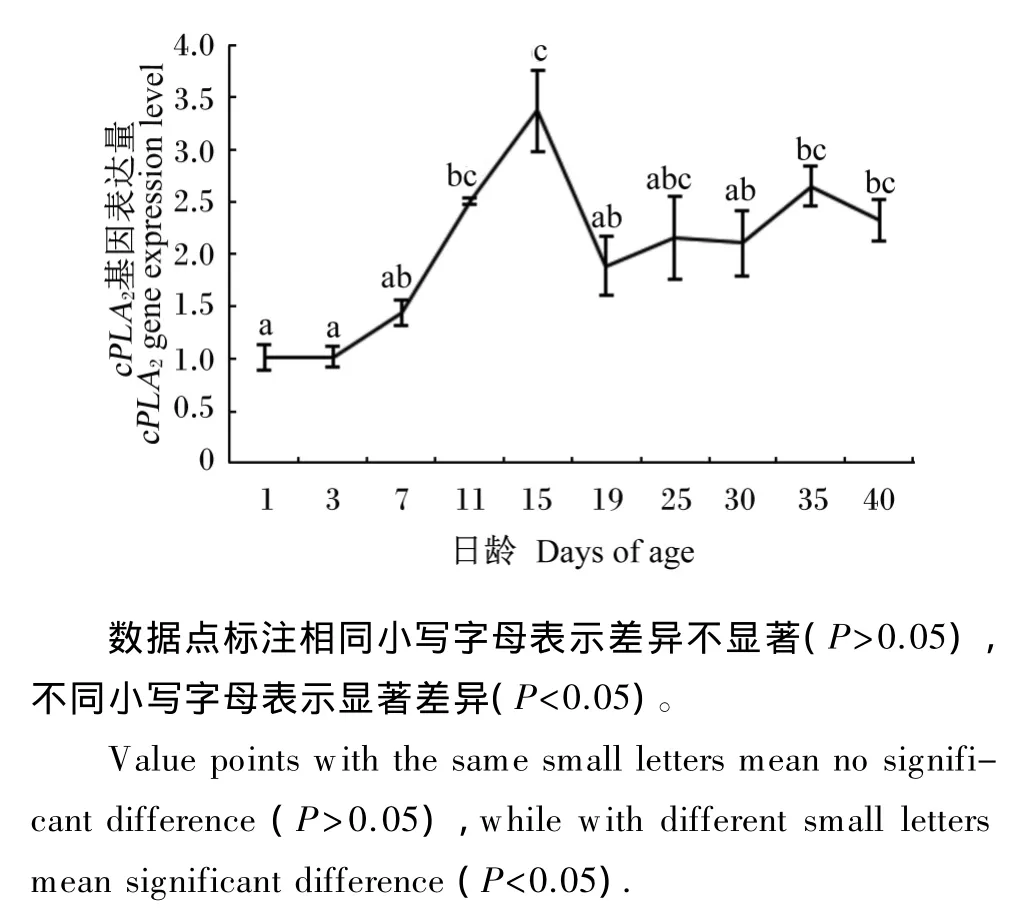
图4 大黄鱼仔稚鱼cPLA2基因表达量随日龄增加的变化Fig.4 Change of expression level of cPLA2gene with days of age increasing of large yellow croaker larvae
3 讨论
采用同源克隆和cDNA末端快速扩增(RACE)技术克隆到大黄鱼cPLA2基因的cDNA全长。首先通过分子质量大小确定本试验中克隆到的PLA2属于胞质型的,多序列比对分析发现克隆到的基因与cPLA2基因相似度较高。序列比对发现大黄鱼cPLA2基因与其他鱼类亲缘关系较近,与哺乳动物亲缘较远,符合进化规律。在鼠上的研究发现,cPLA2基因包括脂质结合的Ca2+依赖的C2结构域和起催化作用的α/β水解酶结构域[31]。这 2 个结构域影响 cPLA2的催化活性[32]。C2结构域与内膜结合是由神经酰胺-1-磷酸的作用决定的,C2结构域包含一段精氨酸、精氨酸和组氨酸高度保守序列(RxRH),而RxRH可以调节神经酰胺-1磷酸的特异性[33]。通过多序列比对发现,只有斑马鱼的cPLA2基因存在RxRH,而其他几种鱼类的保守序列是精氨酸、苏氨酸、赖氨酸和组氨酸(RTKH)。这说明大黄鱼cPLA2与膜结合的调节机制可能与哺乳动物的不太一致,需要进一步的研究。此外,脂肪酶的保守序列甘氨酸、丝氨酸、甘氨酸和丝氨酸(GxSGS)存在大黄鱼的cPLA2基因中,且与其他物种高度保守。GxSGS还是区别cPLA2与其他类型PLA2的标志[34]。
磷脂是鱼类胚胎发育和早期发育阶段重要的供能物质[5]。3日龄以前大黄鱼仔鱼生长主要依靠卵黄囊营养,在这个阶段磷脂被磷脂酶水解,进而发挥其供能作用,因此在刚孵化出(1日龄)的大黄鱼仔鱼可中检测到cPLA2基因的表达。Zambonino等[17]发现海鲈仔稚鱼开口觅食后几天就能检测到PLA2,这与本试验的结果一致。尽管从3日龄起大黄鱼已经开口摄食,开始接受外源性营养(轮虫),但是在7日龄之前卵黄囊内营养成分并未完全消耗掉[21]。从 3~7日龄,PLA2基因的表达量开始升高,但是并没有出现显著性差异,这可能因为生物饵料体内磷脂酶存在的缘故。随着生长发育的继续,7日龄时大黄鱼稚鱼的食道延长,分为明显的黏液分泌部位和起运输功能的部位[21],消化机能逐渐增强,开始投喂不同种类的生物饵料。15日龄(投喂鲜活桡足类)时cPLA2基因的表达量到最大值,这可能是大黄鱼保持机体磷脂动态平衡的一种机制。19日龄时,cPLA2基因的表达量出现显著下降,之后变得平稳。21日龄时大黄鱼稚鱼形成胃腺[21],胃腺的形成减轻了胰腺的负担,进而使cPLA2基因的表达量下降,cPLA2的分泌也下降。
4 结 论
大黄鱼从仔稚鱼到幼鱼早期的变形过程中,磷脂分解代谢的关键酶cPLA2基因的表达量呈现有规律的变化,这可能对机体保持体内磷脂的动态平衡、维护细胞膜的流动性具有重要的意义。大黄鱼仔稚鱼cPLA2基因表达量的变化趋势在一定程度上可以衡量大黄鱼消化系统的发育情况。
[1]FAGONE P,JACKOWSKI S.Phosphatidylcholine and the CDP-choline cycle[J].Biochimica et BiophysicaActa:Molecular and Cell Biology of Lipids,2013,1831(3):523-532.
[2]FRASER A J,GAMBLE J C,SARGENT J R.Changes in lipid content,lipid class composition and fatty acid composition of developing eggs and unfed larvae of cod(Gadus morhua)[J].Marine Biology,1988,99(3):307-313.
[3]RAINUZZO J R,REITAN K I,JØRGENSEN L.Comparative study on the fatty acid and lipid composition of four marine fish larvae[J].Comparative Biochemistry and Physiology Part B:Comparative Biochemistry,1992,103(1):21-26.
[4]TOCHER D R,FRASER A J,SARGENT J R,etal.Fatty acid composition of phospholipids and neutral lipids during embryonic and early larval development in Atlantic herring(Clupea harengus,L.)[J].Lipids,1985,20(2):69-74.
[5]TOCHER D R,BENDIKSEN E Å,CAMPBELL P J,etal.The role of phospholipids in nutrition and metabolism of teleost fish[J].Aquaculture,2008,280(1):21-34.
[6]POSTON H A.Effect of body size on growth,survival,and chemical composition of Atlantic salmon fed soy lecithin and choline[J].The Progressive Fish-Culturist,1990,52(4):226-230.
[7]CAHU C,GUILLAUME J C,STÉPHAN G,etal.Influence of phospholipid and highly unsaturated fatty acids on spawning rate and egg and tissue composition in Penaeus vannamei fed semi-purified diets[J].Aquaculture,1994,126(1/2):159-170.
[8]DAPRÀ F,GEURDEN I,CORRAZE G,etal.Physiological and molecular responses to dietary phospholipids vary between fry and early juvenile stages of rainbow trout(Oncorhynchus mykiss)[J].Aquaculture,2011,319(3/4):377-384.
[9]GEURDEN I,COUTTEAU P,SORGELOOS P.Effect of a dietary phospholipid supplementation on growth and fatty acid composition of European sea bass(Dicentrarchus labrax L.)and turbot(Scophthal musmaximus L.)juveniles from weaning onwards[J].Fish Physiology and Biochemistry,1997,16(4):259-272.
[10]HARADA K.Relationships between structure and feeding attraction activity of certain L-amino acids and lecithin in aquatic animals[J].Nippon Suisan Gakkaishi,1987,53(12):2243-2247.
[11]KANAZAWA A.Effects of docosahexaenoic acid and phospholipids on stress tolerance of fish[J].Aquaculture,1997,155(1/2/3/4):129-134.
[12]SEOKA M,KURATA M,TAMAGAWA R,etal.Dietary supplementation of salmon roe phospholipid enhances the growth and survival of Pacific bluefin tuna Thunnus orientalis larvae and juveniles[J].Aquaculture,2008,275(1/2/3/4):225-234.
[13]ZHAO J Z,AI Q H,MAI K S,etal.Effects of dietary phospholipids on survival,growth,digestive enzymes and stress resistance of large yellow croaker,Larmich-thys crocea larvae[J].Aquaculture,2013,410/411:122-128.
[14]HENDERSON R J,TOCHER D R.The lipid composition and biochemistry of freshwater fish[J].Progress in Lipid Research,1987,26(4):281-347.
[15]SOUPENE E,FYRST H,KUYPERS F A.Mammalianacyl-CoA:lysophosphatidylcholine acyltransferase enzymes[J].Proceedings of the National Academy of Sciencesof the United States of America,2008,105(1):88-93.
[16]BURKE J E,DENNIS E A.Phospholipase A2structure/function,mechanism,and signaling[J].Journal of Lipid Research,2009,50(Suppl.):S237-S242.
[17]ZAMBONINO I J L,CAHU C L.Ontogeny of the gastrointestinal tract of marine fish larvae[J].Comparative Biochemistry and Physiology Part C:Toxicology& Pharmacology,2001,130(4):477-487.
[18]ONO H,IIJIMA N.Purification and characterization of phospholipase A2isoforms from the hepatopancreas of red sea bream,Pagrus major[J].Fish Physiology and Biochemistry,1998,18(2):135-147.
[19]SAELE Ø,NORDGREEN A,OLSVIK P A,etal.Characterisation and expression of secretory phospholipase A2 groupⅠB during ontogeny of Atlantic cod(Gadus morhua)[J].British Journal of Nutrition,2011,105(2):228-237.
[20]FUJIKAWA Y,SHIMOKAWA M,SATOH F,etal.Ontogeny of gene expression of group IB phospholipase A2 isoforms in the red sea bream,Pagrus(Chrysophrys)major[J].Comparative Biochemistry and Physiology Part A:Molecular&Integrative Physiology,2012,161(2):185-192.
[21]MAI K,YU H,MA H,etal.A histological study on the development of the digestive system of Pseudosciaena crocea larvae and juveniles[J].Journal of Fish Biology,2005,67(4):1094-1106.
[22]XIE F J,AI Q H,MAI K S,etal.Dietary lysine requirement of large yellow croaker(Pseudosciaena crocea,Richardson 1846)larvae[J].Aquaculture Research,2012,43(6):917-928.
[23]LI W J,AI Q H,MAI K S,etal.Effects of dietary amino acid patterns on growth and protein metabolism of large yellow croaker(Larimichthys crocea)larvae[J].Aquaculture,2013,406/407:1-8.
[24]XIE F J,AI Q H,MAI K S,etal.The optimal feeding frequency of large yellow croaker(Pseudosciaena crocea,Richardson)larvae[J].Aquaculture,2011,311(1/2/3/4):162-167.
[25]AI Q H,ZHAO J Z,MAI K S,etal.Optimal dietary lipid level for large yellow croaker(Pseudosciaena crocea)larvae[J].Aquaculture Nutrition,2008,14(6):515-522.
[26]ZUO R,AI Q,MAI K,etal.Effects of dietary n-3 highly unsaturated fatty acids on growth,nonspecificimmunity,expression of some immune related genes and disease resistance of large yellow croaker(Larmichthys crocea)following natural infestation of parasites(Cryptocaryon irritans)[J].Fish&Shellfish Immunology,2012,32(2):249-258.
[27]THOMPSON J D,HIGGINS D G,GIBSON T J.CLUSTAL W:improving the sensitivity of progressive multiple sequence alignment through sequence weighting,position-specific gap penalties and weight matrix choice[J].Nucleic Acids Research,1994,22(22):4673-4680.
[28]TAMURA K,DUDLEY J,NEI M,et al.MEGA4:molecular evolutionary genetics analysis(MEGA)software version 4.0[J].Molecular Biology and Evolution,2007,24(8):1596-1599.
[29]KUMAR S,TAMURA K,JAKOBSEN I B,et al.MEGA2:molecularevolutionary geneticsanalysis software[J].Bioinformatics,2001,17(12):1244-1245.
[30]YAO C L,KONG P,WANG Z Y,etal.Cloning and expression analysis of two alternative splicing toll-like receptor 9 isoforms A and B in large yellow croaker,Pseudosciaena crocea[J].Fish &Shellfish Immunology,2008,25(5):648-656.
[31]UOZUMI N,SHIMIZU T.Roles for cytosolic phospholipase A2α as revealed by gene-targeted mice[J].Prostaglandins & Other Lipid Mediators,2002,68:59-69.
[32]HSU Y H,BURKE J E,STEPHENS D L,etal.Calcium binding rigidifies the C2 domain and the intradomain interaction of GIVA phospholipase A2 as revealed by hydrogen/deuterium exchange mass spectrometry[J].Journal of Biological Chemistry,2008,283(15):9820-9827.
[33]LEE L K,BRYANT K J,BOUVERET R,etal.Selectiveinhibition of human groupⅡA-secreted phospholipase A2(hGⅡA)signaling reveals arachidonicacid metabolism is associated with colocalization of hGⅡA to vimentin in rheumatoid synoviocytes[J].Journal of Biological Chemistry,2013,288(21):15269-15279.
[34]MANSFELD J,GEBAUER S,DATHE K,etal.Secretory phospholipase A2from Arabidopsis thaliana:insights into the three-dimensional structure and the amino acids involved in catalysis[J].Biochemistry,2006,45(18):5687-5694.
